研究发现对基因进行修饰改变,可能会直接引发各类疾病,作为基因修饰功能实施者的蛋白质,在基因改变致病的过程中起着重要作用。所以了解蛋白质的功能十分重要[1]。对于生物体内存在的蛋白质,如果知道它在细胞中的位置,就能了解该蛋白质的生物学功能[2]。研究表明,帕金森病[3]和阿尔茨海默病[4]等疾病都与蛋白质的亚高尔基体位置相关,正确识别高尔基体驻留蛋白的类型,对于了解高尔基体蛋白质的功能有着非常重要的作用。亚细胞分离等实验识别亚高尔基体蛋白质位置的方法既费时又费钱,而且实验过程中还会出现很多问题。利用蛋白质序列及结构信息预测其亚细胞位置,成本低而且不耗费时间。
高尔基体是由许多扁平的囊泡构成的以分泌为主要功能的细胞器,是真核细胞中内膜系统的组成之一,它不仅存在于动植物细胞中,而且也存在于原生动物和真菌细胞内。高尔基体由三个扁平的膜囊组成,包括:顺面膜囊、中间膜囊和反面膜囊,结构如图 1所示。

|
图 1 亚高尔基体的结构 Figure 1 Structure of sub-Golgi apparatus |
高尔基体[5]在蛋白质糖基化、膜上的转化作用以及溶酶体的形成和植物细胞壁的形成等方面都发挥着重要作用。对亚高尔基体蛋白质的定位预测有助于理解高尔基体内发生的各种生物过程的机制,并了解亚高尔基体蛋白质的功能,许多疾病发生的致病机理都与亚高尔基体的位置相关,因此,亚高尔基体蛋白质的定位预测也可以为与高尔基体相关的疾病设计药物提供一定的帮助[6]。2008年,van Dijk等[7]对II型跨膜蛋白的单通道跨膜结构域(TMD)序列进行了预测,证明了亚高尔基体蛋白质的位置与聚糖生物合成中不同步骤的顺序之间有明显的关系。2011年Ding Hui等[8]建立了两类亚高尔基体蛋白质的数据集并将离散增量(ID)和信息熵(IH)与改进的马氏判别式相结合对高尔基体蛋白质类型进行预测,预测成功率为74.7%。2013年,Ding Hui等[9]提取了间隔二肽组分(g-gap dipeptide)信息来识别高尔基体蛋白质的类型。2015年,Jiao Yasen等[10]。基于Ding Hui的数据集,对cis-Golgi蛋白和trans-Golgi蛋白进行了区分,预测成功率为86.9%。2016年,Yang Runtao等[11]基于SwissProt数据库建立了新的亚高尔基体蛋白质数据集,他们基于共同空间模式(CSP)的概念,开发了一种新的特征提取技术从蛋白质序列中提取进化信息,预测成功率为86.4%。近几年,一些研究者对亚高尔基体蛋白质进行了深入研究,对亚高尔基体蛋白质的cis-Golgi蛋白和trans-Golgi蛋白进行预测,得到了较高的成功率[12-15]。2019年,Zhao Wei等[16]建立了三类亚高尔基体蛋白质的数据集,将伪氨基酸组分信息与功能域富集得分(FunDES)结合,预测成功率为78.4%。2020年,Cui Qingyu等[17]。在特征提取中没有使用完整的蛋白质序列,提出了529种切割的蛋白质序列,训练集和测试集都根据这529种切割类型进行切割,预测成功率为78.13%。
本文基于最新版本SwissProt数据库建立了包含顺面膜囊(cis-Golgi)和反面膜囊(trans-Golgi)两类亚高尔基体蛋白质的数据集,通过输入多种特征信息进行预测实验,最后选取6种预测结果较好的特征信息,6种特征信息中基因本体(GO)注释信息的预测结果最好。在此基础上对6种特征信息进行融合,利用支持向量机算法对两类亚高尔基体蛋白质定位进行预测,融合特征最高预测成功率为87.43%,特征信息的融合对预测结果有一些提升作用,本文的预测方法对反面膜囊(trans-Golgi)蛋白质的识别效果较好。
1 材料与方法 1.1 数据集的构建在过去的几十年中,通过计算方法对蛋白质进行定位预测成为了生物信息学研究的重点,本文基于Swiss-Prot数据库,建立了两类亚高尔基体蛋白质的数据集,建立该数据集有以下几个步骤:
1) 在Swiss-Prot数据库中搜索关键词“Golgi apparatus”,选择经过实验验证的、亚细胞位置在“cis-Golgi”和“trans-Golgi”的蛋白质。
2) 去除Fragment序列。
3) 去掉具有模糊蛋白质注释的序列,例如含有“By similarity”和“Probably”等注释的序列。
4) 删除重复位置的蛋白质序列。
经过上述步骤得到977条亚高尔基体蛋白质,使用CD-HIT在线工具去除序列相似性,由于两类亚高尔基体蛋白质的数目差距较大,当阈值设为25%时,顺面膜囊(cis-Golgi)蛋白质的数目较少,会导致两类亚高尔基体蛋白质数据集更加不平衡,所以我们将阈值设为40%,得到两类亚高尔基体蛋白质的数据集,见表 1。
| 表 1 亚高尔基体蛋白质数据集中序列数目 Table 1 Number of sequences in sub-Golgi protein dataset |
2016年Yang等[11]基于Swiss-Prot数据库建立的两类亚高尔基体蛋白质数据集,该数据库被研究者广泛应用,这个数据集共304条蛋白质序列,其中包括87条顺面膜囊(cis-Golgi)蛋白质序列和217条反面膜囊(trans-Golgi)蛋白质序列。但是随着测序技术的发展,Swiss-Prot数据库不断更新,顺面膜囊(cis-Golgi)蛋白质序列和反面膜囊(trans-Golgi)蛋白质序列数目不断变化,本文基于最新版本Swiss-Prot数据库建立了两类亚高尔基体蛋白质定位数据集,新建的数据集与Yang等[11]构建的数据集相比亚高尔基体蛋白质序列数目增加54条。
1.2 特征参数的选取 1.2.1 氨基酸组分信息单肽组分信息(AAC)是对每一条蛋白质中20种氨基酸出现的概率进行统计得到的[18]。单肽组分信息可表示如下:
| $\begin{gathered} P=\left[v_1, v_2, v_3, \cdots v_i \cdots, v_{20}\right] \end{gathered} $ | (1) |
| $ v_i=\frac{n_i}{L} $ | (2) |
其中,L表示蛋白质序列的长度,ni表示蛋白质中第i个氨基酸出现的个数。
二肽组分信息[19]。(DC)是通过计算每两个紧邻氨基酸出现的概率得到的。对于给定的蛋白质,二肽组分信息可表示如下:
| $\begin{gathered} F=\left[f_1, f_2, \cdots f_i, \cdots f_{400}\right] \end{gathered} $ | (3) |
| $f_i=\frac{n_i}{L-1} $ | (4) |
其中,L是蛋白质序列的长度,ni表示蛋白质中第i个二肽出现的个数。
1.2.2 两亲性伪氨基酸组分信息两亲性伪氨基酸组分(APAAC)是由Chou提出的[20],可以有效地识别蛋白质,并已广泛应用于不同蛋白质的序列分析。与传统AAC不同的是,APAAC利用蛋白质中氨基酸的疏水性和亲水性,将蛋白质序列顺序与传统AAC结合起来。APAAC可以表示如下:
| $ \begin{gathered} P_{A P A A C}=\left[P_1, P_2, \cdots, P_{20}, P_{20+1}, P_{20+2} \cdots\right. \\ \left.P_{20+\lambda}, \cdots P_{20+2 \lambda}\right] \end{gathered} $ | (5) |
其中,前20个向量代表的是传统的AAC,λ表示相关系数。
1.2.3 联合三联体特征联合三联体特征(CTF)是Shen等人[21]基于预测蛋白质相互作用所提出的特征信息,他们考虑了一个氨基酸及其紧邻氨基酸的性质,20种氨基酸被分为7类,见表 2。CTF具体可表示为:
| $\begin{gathered} P_{C T F}=\left[f_1, f_2, f_3, \cdots f_i \cdots, f_{343}\right] \end{gathered} $ | (6) |
| $ f_i=\frac{k_i}{L-2} $ | (7) |
| 表 2 氨基酸分类 Table 2 Classification of amino acids |
其中ki是每种联合三联体在蛋白质中出现的个数,L是蛋白质序列的长度。
1.2.4 平均化学位移本文通过将蛋白质序列提交到PSIPRED网站获得亚高尔基体蛋白质的二级结构,然后将蛋白质序列和其相对应的二级结构提交到Fan等人构建的平均化学位移[22]服务网站acACS(http://202.207.14.87:8032/bioinformation/acACS/index.asp)中,得到化学位移的结果,表示如下:
| $A C S_i^k(j)=\frac{1}{N} \sum \omega_i^k(j) $ | (8) |
其中i表示4种骨架原子(15N、13C、1H、1HN),k表示蛋白质二级结构的类别(H、E、C),j表示20种氨基酸,N表示蛋白质序列中氨基酸的个数。
对于给定蛋白质P,P可表示如下:
| $ P=\left[C_1^i, C_2^i, \cdots C_L^i\right]\left(i={ }^{15} N, { }^{13} C_\alpha, { }^1 H_\alpha, { }^1 H_N\right) $ | (9) |
该化学位移的自相关协方差可表示如下:
| $\begin{gathered} \psi_\lambda^i=\frac{1}{L-\lambda} \sum\limits_l^{1-\lambda}\left[C_1^i-C_{1+\lambda}^i\right]^2\left(i={ }^{15} N, { }^{13} C_\alpha, \right. \\ \left.{ }^1 H_\alpha, { }^1 H_N ; 0<\lambda<L\right) \end{gathered} $ | (10) |
则PacACS可表示如下:
| $\begin{gathered} P_{a c A C S}=\left[\psi_0^i, \psi_1^i, \psi_2^i, \cdots \psi_\lambda^i\right]\left(i={ }^{15} N, { }^{13} C_\alpha, \right. \\ \left.{ }^1 H_\alpha, { }^1 H_N ; 0<\lambda<L\right) \end{gathered} $ | (11) |
其中λ为相关长度,L为蛋白质序列的长度。对于不同蛋白质,要选择不同的骨架原子组合及合适的λ值,得到预测结果最好的特征参数。
1.2.5 基因本体注释信息基因本体(GO)注释信息包括分子功能(F)、生物过程P)及细胞组分(C)三部分[23],由于细胞组分(C)包含亚高尔基体蛋白质亚细胞位置的注释信息,为了预测结果的客观性,本文使用的GO注释信息去掉了细胞组分(C),本文提取的GO特征信息只包含生物过程(P)和生物功能(F)两个注释信息。GO注释信息可以描述基因和基因产物的功能,许多研究中通过检测蛋白质或者遗传基因的GO号来提取基因本体注释信息,如果蛋白质的GO号未知,则使用同源序列的GO号,同源序列可由BLAST软件检测得到。
本文通过AC号在Swiss-Prot数据库中找到每条蛋白质对应的生物过程(P)和分子功能(F)的注释信息,没有GO注释信息的利用BLAST软件查找该序列的同源序列,同源阈值设为60%,下载所有亚高尔基体蛋白质序列的GO注释信息后,整理、去重复后得到1646个GO注释信息,将对应位置有GO注释信息的设为1,对应位置没有GO注释信息的设为0,得到特征向量,表示如下:
| $P_{G 0}=\left[f_1, f_2 \cdots f_n \cdots f_{1646}\right] $ | (12) |
其中,是指在位置n,该蛋白质序列有无GO注释信息,它的定义如下:
| $f_n= \begin{cases}1 & \text { 在该位置存在 GO} \text { 信息 } \\ 0 & \text { 在该位置不存在 GO} \text { 信息 }\end{cases} $ | (13) |
支持向量机(SVM)算法最早由Vapnik等[24]提出,已成功用于蛋白质结构和功能的预测。该算法的核心理念是将数据从低维向量映射到高维向量,在高维空间中使正集和负集之间的距离最大化。本文使用LibSVM软件包对亚高尔基体蛋白质进行预测。
1.4 评价指标本文采用5-折交叉检验来评估预测性能。选用预测成功率(Accuracy,Acc)、敏感性(Sensitivity,Sn)、特异性(Specificity,Sp)和Matthew相关系数(Matthew’s correlation coefficient,MCC)作为评价指标对算法进行评价[25]。评价指标的定义如下:
| $ \begin{gathered} A c c=\frac{\mathrm{TP}+\mathrm{TN}}{\mathrm{TP}+\mathrm{TN}+\mathrm{FP}+\mathrm{FN}} \end{gathered} $ | (14) |
| $ S n=\frac{\mathrm{TP}}{\mathrm{TP}+\mathrm{FN}} $ | (15) |
| $ S p=\frac{\mathrm{TN}}{\mathrm{TN}+\mathrm{FP}} $ | (16) |
| $ \begin{aligned} & M C C= \\ & \frac{\mathrm{TP} \times \mathrm{TN}-\mathrm{FP} \times \mathrm{FN}}{\sqrt{(\mathrm{TP}+\mathrm{TN}) \times(\mathrm{TN}+\mathrm{FN}) \times(\mathrm{TP}+\mathrm{FN}) \times(\mathrm{TN}+\mathrm{FP})}} \end{aligned} $ | (17) |
其中,TP表示真阳性,TN表示真阴性,FP表示假阳性,FN表示假阴性。
2 结果与讨论 2.1 特征向量参数选取两亲性伪氨基酸组分信息(APAAC)提取时,要选取不同λ值进行预测,当λ=30时,预测结果最好; 平均化学位移(acACS)提取时,要选取不同λ值和不同骨架原子组合进行预测,当λ=30,骨架原子组合为15N+13Cα+1HN时,对亚高尔基体蛋白质的预测结果最好。
2.2 预测结果 2.2.1 单特征预测结果本文采用支持向量机算法,利用提取的6类不同特征信息,对两类亚高尔基体蛋白质进行预测,单特征的预测结果见表 3。
| 表 3 不同特征参数的预测结果 Table 3 Prediction results of different feature parameters |
由表 3可以看出,单个特征信息预测成功率Acc(除CTF外)都在80%以上,所有特征信息的敏感性Sn都较高,最高可达到100%,说明针对本文建立的两类亚高尔基体蛋白质数据集,该预测方法对反面膜囊(Trans-Golgi)蛋白质的预测结果较好,可以有效地识别反面膜囊(Trans-Golgi)蛋白质,而特异性Sp的结果都偏低,说明本文的方法对顺面膜囊(Cis-Golgi)蛋白质的预测结果较差。
为了直观的比较单个参数的预测成功率,以特征参数为横坐标,预测成功率Acc为纵坐标作图,图 2表示不同特征参数的预测成功率Acc,由图 2可知,基因本体GO注释信息的预测成功率Acc最高,为87.15%。
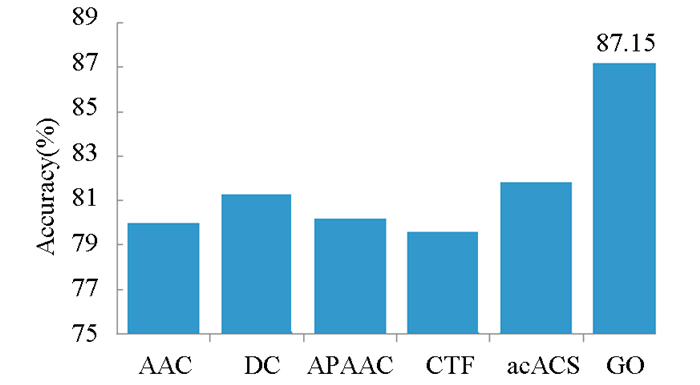
|
图 2 不同特征参数的预测成功率 Figure 2 Prediction accuracy of different feature parameters |
在选取单个特征对亚高尔基体蛋白质定位预测的基础上,进一步对单个特征进行融合,利用融合特征对亚高尔基体蛋白质定位预测,预测结果表明:GO特征与其它特征的融合结果好于没有GO特征的融合。表 4是GO特征与其它一些特征信息融合后的预测结果。
| 表 4 融合特征参数的预测结果 Table 4 Prediction results of fusion feature parameters |
图 3表示融合特征参数的预测准确率Acc,由图 3可知,单肽组分信息和基因本体注释信息融合; 单肽组分信息、两亲性伪氨基酸组分信息和基因本体注释信息融合,这两组融合特征参数的Acc最高。由表 4可以看出,融合特征对区分cis-Golgi蛋白质和trans-Golgi蛋白质有一定的作用。两种融合特征信息中,单肽组分信息和基因本体注释信息融合后预测成功率提高到87.43%,其它特征信息与基因本体注释信息融合以后预测成功率没有提高,还有所下降,出现这种结果的原因可能是:首先,融合特征信息以后,特征信息维数过大,数据冗余,影响预测结果; 其次,特征信息融合以后一些蕴含亚高尔基体位置的关键信息可能被覆盖或者丢失,不能有效识别亚高尔基体蛋白质。三种特征信息融合中,单肽组分信息、两亲性伪氨基酸组分信息和基因本体注释信息融合的预测成功率提高到87.43%,以上结果说明两种和三种特征信息适当的融合对亚高尔基体蛋白质的识别有一定的作用。由表 3和表 4可以看出,选取单个特征和融合特征对亚高尔基体蛋白质的预测,敏感性Sn都较高,这表明本文的预测方法对trans-Golgi蛋白质的识别具有一定的优势,一些特征信息融合以后,特异性Sp和Matthew相关系数MCC比单个特征信息会有一些提高,当基因本体(GO)注释信息分别与氨基酸单肽组分信息(AAC)和平均化学位移(acACS)融合以后Sp和MCC有一定的提升,说明这两类融合特征对cis-Golgi蛋白质识别的准确率有一些提升作用,其它融合特征的特异性Sp和Matthew相关系数MCC并没有提高。
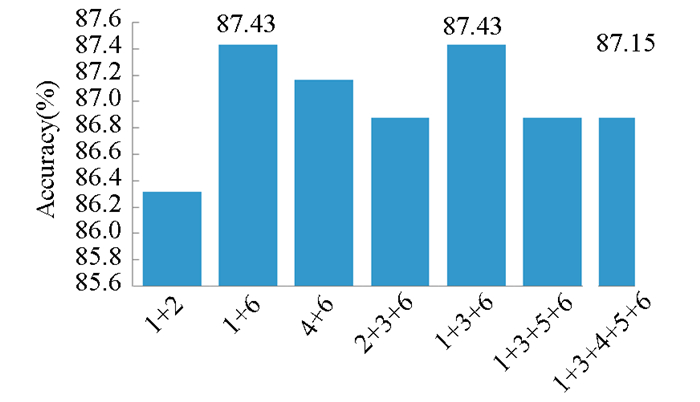
|
图 3 融合特征参数的预测成功率 Figure 3 Prediction accuracy of fusion feature parameters 注:其中1代表AAC,2代表DC,3代表APAAC,4代表CTF,5代表acACS,6代表GO. |
本文对亚高尔基体蛋白质的预测结果相比于前人的预测结果,总体预测成功率略有提高,但是由于本文的两类亚高尔基体数据集是新建立的数据集,新建的数据集蛋白质数量有所增加,本文是针对新建立的数据集进行预测的,所以预测结果与前人的预测结果不具可比性。
3 结论正确识别亚高尔基体蛋白质的类型对治疗由高尔基体蛋白质功能紊乱而引发的疾病有一定的作用。本文建立了两类亚高尔基体蛋白质数据集,选取多种特征信息,利用支持向量机算法,基于5-折交叉检验对该数据集进行预测,预测结果表明:1)选取的6种特征信息中基因本体GO注释信息对亚高尔基体蛋白质定位有较高识别率,本文可以有效识别trans-Golgi蛋白质,但是对于cis-Golgi蛋白质的识别效果较差; 2)基因本体GO注释信息与其它特征信息进行恰当融合后对预测结果有一定的提升作用。
| [1] |
李明俊. 利用多信息融合方法预测蛋白质亚核定位[D]. 呼和浩特: 内蒙古农业大学, 2019. DOI: 10.27229/d.cnki.gnmnu.2019.000115. LI Mingjun. Prediction of protein subnuclear localization based on different features[D]. Hohhot: Inner Mongolia Agricultural University, 2019. DOI: 10.27229/d.cnki.gnmnu.2019.000115. (  0) 0) |
| [2] |
张松, 黄波, 夏学峰, 等. 蛋白质亚细胞定位的生物信息学研究[J]. 生物化学与生物物理进展, 2007, 34(6): 573-579. ZHANG Song, HUANG Bo, XIA Xuefeng, et al. Bioinformatics research in subcellular localization of protein[J]. Progress in Biochemistry and Biophysics, 2007, 34(6): 573-579. DOI:10.3321/j.issn:1000-3282.2007.06.004 (  0) 0) |
| [3] |
FUJITA Y, OHAMA E, TAKATAMA M, et al. Fragmentation of Golgi apparatus of nigral neurons with alpha-synuclein-positive inclusions in patients with Parkinson's disease[J]. Acta Neuropathologica, 2006, 112(3): 261-265. DOI:10.1007/s00401-006-0114-4 (  0) 0) |
| [4] |
GONATAS N K, GONATAS J O, STIEBER A. The involvement of the Golgi apparatus in the pathogenesis of amyotrophic lateral sclerosis, Alzheimer's disease, and ricin intoxication[J]. Histochemistry & Cell Biology, 1998, 109(5/6): 591-600. DOI:10.1007/s004180050257 (  0) 0) |
| [5] |
苗芳芳, 范隆, 王天龙. 高尔基体与神经退行性疾病[J]. 北京医学, 2017, 39(3): 294-296. DOI: CNKI:SUN:BJYX.0.2017-03-024. MIAO Fangfang, FAN Long, WANG Tianlong. Golgi apparatus and neurodegenerative diseases[J]. Beijing Medicine, 2017, 39(3): 294-296. DOI: CNKI:SUN:BJYX.0.2017-03-024. (  0) 0) |
| [6] |
张瑜, 周文胜, 王佳. 高尔基体与神经退行性疾病研究进展[J]. 中风与神经疾病杂志, 2017, 34(5): 474-477. DOI: CNKI:SUN:ZFSJ.0.2017-05-026. ZHANG Yu, ZHOU Wensheng, WANG Jia. Research progress of Golgi body and neurodegenerative diseases[J]. Journal of Stroke and Neurological Diseases, 2017, 34(5): 474-477. DOI: J.0.2017-05-026. (  0) 0) |
| [7] |
DIJK A, BRAAK C. Predicting sub-Golgi localization of type Ⅱ membrane proteins[J]. Bioinformatics, 2008, 24(16): 1779-1786. DOI:10.1093/bioinformatics/btn309 (  0) 0) |
| [8] |
DING H, LIU L, GAO F B, et al. Identify Golgi protein types with modified Mahalanobis discriminant algorithm and pseudo amino acid composition[J]. Protein and Peptide Letters, 2011, 18(1): 58-63. DOI:10.2174/092986611794328708 (  0) 0) |
| [9] |
DING H, GUO S H, DENG E Z, et al. Prediction of Golgi-resident protein types by using feature selection technique[J]. Chemometrics and Intelligent Laboratory Systems, 2013, 124(6): 9-13. DOI:10.1016/j.chemolab.2013.03.005 (  0) 0) |
| [10] |
JIAO Y S, DU P F. Predicting Golgi-resident protein types using pseudo amino acid compositions: Approaches with positional specific physicochemical properties[J]. Journal of Theoretical Biology, 2016, 391(5): 35-42. DOI:10.1016/j.jtbi.2015.11.009 (  0) 0) |
| [11] |
YANG R T, ZHANG C J, GAO R, et al. A novel feature extraction method with feature selection to identify golgi-resident protein types from imbalanced data[J]. IJMS, 2016, 17(2): 218. DOI:10.3390/ijms17020218 (  0) 0) |
| [12] |
AHMAD J, JAVED F, HAYAT M. Intelligent computational model for classification of sub-Golgi protein using oversampling and fisher feature selection methods[J]. Artificial Intelligence in Medicine, 2017, 78(5): 14-22. DOI:10.1016/j.artmed.2017.05.001 (  0) 0) |
| [13] |
RAHMAN M S, RAHMAN M K, KAYKOBAD M, et al. isGPT: An optimized model to identify sub-Golgi protein types using SVM and Random Forest based feature selection[J]. Artificial Intelligence in Medicine, 2018, 84(5): 90-100. DOI:10.1016/j.artmed.2017.11.003 (  0) 0) |
| [14] |
张蕾. 基于序列和结构信息预测蛋白质亚高尔基体定位[D]. 呼和浩特: 内蒙古大学, 2018. DOI: 10.7666/d.D01535807. ZHANG Lei. Prediction of protein sub-Golgi localization based on sequence and structure information[D]. Hohhot: Inner Mongolia University, 2018. DOI: 10.7666/d.D01535807. (  0) 0) |
| [15] |
LV Z B, JIN S S, DING H, et al. A random forest sub-golgi protein classifier optimized via dipeptide and amino acid composition features[J]. Frontiers in Bioengineering and Biotechnology, 2019, 7: 215. DOI:10.3389/fbioe.2019.00215 (  0) 0) |
| [16] |
ZHAO W, LI G P, WANG J, et al. Predicting protein sub-Golgi locations by combining functional domain enrichment scores with pseudo-amino acid compositions[J]. Journal of Theoretical Biology, 2019, 473: 38-43. DOI:10.1016/j.jtbi.2019.04.025 (  0) 0) |
| [17] |
CUI Q Y, CAO Y, BAO W Z, et al. SubRF_Seq: Identification of Sub-Golgi protein types with random forest with partial sequence information[J]. Scientific Programming, 2020, 2020(9): 1-7. DOI:10.1155/2020/8862468 (  0) 0) |
| [18] |
罗林波, 陈绮. 氨基酸序列特征提取方法研究[J]. 计算机技术与发展, 2010, 20(2): 206-208. LUO Linbo, CHEN Qi. Research on feature extraction method of amino acid sequence[J]. Computer Technology and Development, 2010, 20(2): 206-208. DOI:10.3969/j.issn.1673-629X.2010.02.054 (  0) 0) |
| [19] |
AHMAD K, WARIS M, HAYAT M. Prediction of protein submitochondrial locations by incorporating dipeptide composition into chou's general pseudo amino acid composition[J]. The Journal of Membrane Biology, 2016, 249(3): 293-304. DOI:10.1007/s00232-015-9868-8 (  0) 0) |
| [20] |
CHOU K C. Using amphiphilic pseudo amino acid composition to predict enzyme subfamily classes[J]. Bioinformatics, 2005, 21(1): 10-19. DOI:10.1093/bioinformatics/bth466 (  0) 0) |
| [21] |
SHEN J W, ZHANG J, LUO X M, et al. Predicting protein-protein interactions based only on sequences information[J]. Proceedings of the National Academy of Sciences of The United States of America, 2007, 104(11): 4337-4341. DOI:10.1073/pnas.0607879104 (  0) 0) |
| [22] |
SIBLEY A B, COSMAN M, KRISHNAN V V. An empirical correlation between secondary structure content and averaged chemical shifts in proteins[J]. Biophysical Journal, 2003, 84: 1223-1227. DOI:10.1016/S0006-3495(03)74937-6 (  0) 0) |
| [23] |
高晓伟. 革兰氏阳性菌蛋白质亚细胞定位的特征提取及预测算法研究[D]. 呼和浩特: 内蒙古农业大学, 2020. DOI: 10.27229/d.cnki.gnmnu.2020.000810. GAO Xiaowei. Study on feature extraction and prediction algorithm for subcellular localization of gram-positive bacterial protein[D]. Hohhot: Inner Mongolia Agricultural University, 2020. DOI: 10.27229/d.cnki.gnmnu.2020.000810. (  0) 0) |
| [24] |
GIROSI F. An equivalence between sparse approximation and support vector machines[J]. Neural Computation, 1998, 10(6): 1445-1480. DOI:10.1162/089976698300017269 (  0) 0) |
| [25] |
JING X Y, LI F M. Identifying heat shock protein families from imbalanced data by using combined features[J]. Computational and Mathematical Methods in Medicine, 2020, 2020: 8894478. DOI:10.1155/2020/8894478 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


