癌症通常是由遗传变异的累积所驱动,包括单核苷酸变异、小的插入或缺失和拷贝数变异等[1]。基因突变会导致基因活化或基因失活,促使癌症发生和转移,癌症驱动基因的突变会使肿瘤细胞获得抗免疫细胞清除及药物治疗的选择性生长优势[1]。因此,开发识别癌症驱动基因的方法并确定驱动基因,对癌症病理研究及癌症诊断、治疗、靶向药物的研发都具有十分重要的意义。
新一代测序技术的最新进展帮助研究人员生成了大量的癌症基因组数据,并对常见和罕见癌症类型的体细胞突变进行了分类[2]。不同癌症类型体细胞突变数据的获得,为探索癌症不同子类驱动基因提供了数据基础。
在2018年,脑和中枢神经系统癌症是第17位最常见的癌症类型,据估计全球新增病例为29.7万例[3]。由于脑和中枢神经系统肿瘤的发病率相对较低,且肿瘤的异质性较高,因此对脑肿瘤病因学的研究尤其具有挑战性。而癌症基因组的突变率变化又很大。在不同的癌症类型中,它的差异大到1000倍[4]。大多数实体瘤基因组都含有数百种序列水平的基因改变,这些改变中的大多数预计是乘客突变(对肿瘤细胞的选择性生长优势没有直接或间接影响的突变),而很少是驱动突变(对肿瘤细胞有选择性生长优势的突变)[5]。尽管很容易定义生理作用中的“驱动突变”(赋予选择性肿瘤生长优势),从大规模人类癌症基因组数据中系统地识别驱动突变仍然是一个巨大的挑战[6-7]。
为了解决这一重要的任务,在过去的几年中发展了许多方法和计算工具。如Lawrence等人开发的MutSigCV算法根据突变信息建立一个突变背景模型,根据模型判断每个基因的突变是否比偶然突变更显著,由此确定突变基因[4]; Tamborero等人开发的OncodriveCLUST算法利用驱动突变在位点上具有形成突变簇的偏好性以及利用同义突变无偏分布的特点构建背景突变模型,寻找可能的驱动突变[8]; Reimand等人开发的ActiveDriver检测位于翻译后修饰位点,如磷酸化、乙酰化或泛素化位点等,其体细胞突变富集的基因,它使用logistic回归法确定驱动基因[9]。上述研究是通过将基因组区域中观察到的突变率与背景突变率(BMR)预期的突变率进行比较或功能影响测试来检测阳性选择信号,从而识别驱动基因,但所识别出的驱动基因其本质是存在异常的基因,关系图见图 1a。所以本文提出关联规则算法,其基于基因突变频率,将患癌结果作为必要出现的前提,通过算法将基因突变与患癌结果联系起来,关系图见图 1b,再通过支持度、置信度和提升度这三个指标来判断该基因是否是驱动基因,可以有效发现驱动基因,且一定程度上降低了假阳性率。此外,在以往对驱动基因的探索中,更侧重于关注癌症整体,而并非考虑已有明确分类癌症的类间驱动基因差异,本文中给出的驱动基因是针对于不同子类的驱动基因。
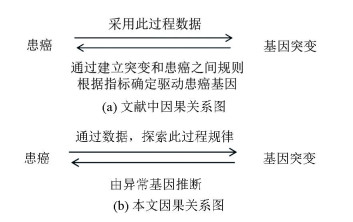
|
图 1 充分必要关系图 Figure 1 Sufficient and necessary conditions diagram |
从TCGA平台下载经Varscan2[10-11]预处理且注释过的外显子低级别脑胶质瘤体细胞突变信息注释(Mutation Annotation Format, MAF)文件,从中提取出全部12 440个突变基因、506样本及其对应的30 447条突变记录,将样本中存在碱基置换突变、移码突变、插入突变、缺失突变等不同基因突变类型均记为该基因突变,且记为1,野生型,即未发生突变的基因记为0,进行离散化矩阵构建,行为样本名,列为突变基因,形成关联算法可识别的低级别脑胶质瘤样本基因突变数据矩阵(见表 1)。
| 表 1 低级别脑胶质瘤样本基因突变数据矩阵(部分) Table 1 Gene mutation data matrix of low-grade glioma samples (part) |
世界卫生组织(World Health Organization, WHO)将胶质瘤按其组织病理学类型以及其生物学行为分为I、II、III和IV级,并定义I级、II级胶质瘤为低级别脑胶质瘤。其指出使用基因型(即IDH突变和1p/19q编码缺失状态)和表型(肿瘤形状、位置等)诊断低级别脑胶质瘤是兼容的[12],故根据IDH1是否突变及1p/19q是否缺失分为三个子类,其中,IDH1突变且1p/19q联合缺失记为IDHmut-codel,IDH1突变且1p/19q不联合缺失记为IDHmut-non-codel,IDH1无突变的野生型记为IDHwt。Tumor Map[13]平台下载低级别脑胶质瘤分类信息数据,将该文件内的分类信息数据与低级别脑胶质瘤样本基因突变数据集合并,最终获得包含491例样本的低级别脑胶质瘤肿瘤样本基因突变及子类信息数据集,其中,IDHmut-codel、IDHmut-non-codel、IDHwt三个子类对应的样本数量分别为163、240和88。
2 方法本文引入关联规则方法,通过该方法将基因突变与患癌结果进行关联,筛选出有效规则并确定驱动基因。
2.1 关联规则关联规则主要反映了事物之间的关联性。若反应同一事物的一条记录既具有特征属性A也具有特征属性B,则称特征属性A和B是关联的[14-15],在本文中将A定义为发生突变的基因,B定义为患低级别脑胶质瘤,结果中若既存在A又存在B则定义为该基因发生突变与患该子类低级别脑胶质瘤存在关联。
若A和B是关联的,则可以记为:
| $ A \rightarrow B $ | (1) |
| $ \text { 基因突变 } \rightarrow \text { 易患该子类低级别脑胶质瘤 } $ | (2) |
其中A在本文中定义为基因或基因集发生突变,关联结果B定义为患该子类低级别低级别脑胶质瘤,A→B表示发生该基因突变会诱使病人更易患该子类低级别脑胶质瘤,将其作为候选驱动基因。
关联规则支持度表达了关联规则在总体发生概率,反应了规则出现的频繁程度,即该基因突变导致患该子类低级别脑胶质瘤在数据中出现的频繁程度:
| ${Support}(A \rightarrow B)=P(A B) $ | (3) |
其中P(AB)表示候选基因或候选基因集发生突变且关联结果为患该子类低级别脑胶质瘤的概率。
关联规则置信度表示构成关联规则的一个特征属性A发生时,另一个特征属性B的发生概率,反映了这两个特征属性之间的关联强度,即该基因突变导致患该子类低级别脑胶质瘤之间关联强度:
| $ { Confidence }(A \rightarrow B)=P(A B) / P(A) $ | (4) |
其中P(A)表示基因或特征基因集发生突变的概率。
关联规则提升度反应了关联规则的重要性及研究者对其感兴趣的程度,即“该基因突变更易导致患该子类低级别脑胶质瘤”这条规则的重要性:
| $ { Lift }(A \rightarrow B)= { Confidence }(A \rightarrow B) / P(B)= \\ P(A B) /(P(A) P(B)) $ | (5) |
其中P(B)表示患该子类癌症的概率。
当提升度为1时,表示基因突变与患该子类癌症之间未存在关联,所以此条规则不予采用。如果提升度小于1,说明该规则表现为负关联,涉及的特征属性是相互排斥的。反之,提升度大于1,则表现为正关联,反应所涉及的特征属性是共生的,即该基因突变可能会导致患该子类低级别脑胶质瘤。
2.2 低级脑胶质瘤驱动基因筛选对低级别脑胶质瘤样本基因突变及子类信息数据集利用改良后的布尔矩阵Apriori方法寻找规则,通过调节支持度和置信度,筛选有效规则并定义有效规则中出现的突变基因为驱动基因,流程见图 2。
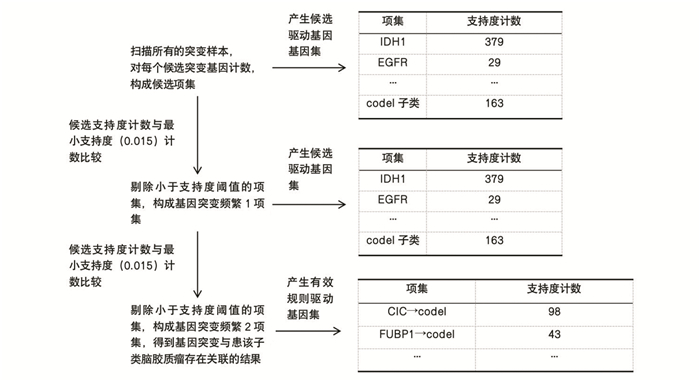
|
图 2 关联规则算法流程 Figure 2 Association rule algorithm flow |
(1) 规定有效规则前项A表示基因是否发生突变,若发生突变计算其支持度,将其作为候选驱动基因集; (2)规定有效规则支持度≥0.015(0.03),筛选出满足支持度阈值的候选驱动基因集; (3)规定有效规则后项B表示是否患低级别脑胶质瘤,筛选出满足“A→B”规则的项集,作为候选驱动基因; 值得注意的是,由于IDHwt子类样本较少,所以适当提升支持度,以保证“发生该基因突变则会诱使病人患低级别脑胶质瘤”此条规则出现的频率不会过低,驱动基因具体筛选参数见表 2。
| 表 2 筛选标准 Table 2 Selection criteria |
使用3.2提供的算法对低级别脑胶质瘤样本基因突变及子类信息数据集进行挖掘,筛选得到有效规则见表 3。其中,IDHmut-codel子类得到10条有效规则,这10个驱动基因突变所驱动的结果更倾向于IDHmut-codel这一子类。值得注意的是,虽然IDH1作为该子类分类标准的一项,预测结果并不是直接与结果相关,而是与CIC、PIKC3CA、TTN、FUBP1等基因联合形成有效规则。子类IDHmut-non-codel得到9条有效规则,子类IDHwt得到3条有效规则。
| 表 3 驱动基因筛选结果 Table 3 Driver gene screening results |
Matthew H. Bailey等人利用TCGA公开的MC3工作组处理的MAF文件通过跨越9 423个肿瘤体的PanCancer和Pansoftware分析,并使用26种计算工具,其工具涵盖基于基因突变频率算法、基因功能算法、通路算法、以及机器学习算法多维度初步筛选驱动基因,再通过实验验证最终筛选出驱动基因,该研究代表了迄今为止对癌症基因和突变最全面的发现[16],我们将此结果做为肿瘤驱动基因预测的金标准。并与通过将基因组区域中观察到的突变率与BMR预期的突变率进行比较或功能影响测试来识别驱动基因的方法进行比较,评判本预测方法的优劣。
文献[16]中给出了经实验验证的24个低级别脑胶质瘤的驱动基因,其与本算法给出的22个驱动基因共有17个基因重合,见表 2,说明关联规则算法对预测驱动基因是有效的和可行的。但值得注意的是,本文所得出的驱动基因与金标准并不完全相同,是因为本文仅仅利用了外显子基因突变信息,给出的驱动基因仅仅是外显子突变驱动基因,并没有包含文献[16]中基于基因功能、通路等其它途径信息预测得到的驱动基因和非编码区域突变信息。
从DriverDBv3分别下载利用MutsigCV、Activedriver方法识别驱动基因的结果,这两种方法使用数据相同,其利用TCGA、ICGC等数据库中低级别脑胶质瘤体细胞突变数据识别驱动基因[17],以及利用R中Oncodriverclust算法利用本研究所用数据计算获得识别驱动基因结果,将这三种算法与本文算法对比,结果如图 3,将四种算法与金标准对比,算法识别驱动基因比对结果见图 2,算法识别驱动基因具体结果见表 4。

|
图 3 四种算法的结果比较 Figure 3 Comparison of results of four algorithms |
| 表 4 四种算法与金标准驱动基因对比结果 Table 4 Comparison results of four algorithms with gold standard drive gene |
在从DriverDBv3获取到使用MutsigCV算法的数据中关于低级别脑胶质瘤共预测出41个驱动基因,其中有3个驱动基因与金标准重合,未有驱动基因与本文算法重合; 使用Activedriver算法的数据中关于低级别脑胶质瘤共预测出225个驱动基因,其中有9个驱动基因与金标准重合,有7个驱动基因与本文算法重合; 在利用R包Oncoclust算法计算获得低级别脑胶质瘤共预测出23个驱动基因,其中有17个驱动基因与金标准重合,有17个驱动基因与本文算法预测的基因重合。这四种算法的准确率分别为7.3%、4%、73.91%以及77.27%。进一步说明相较于其他算法,利用Apriori算法预测低级别脑胶质瘤的驱动基因更准确,假阳性率更低,同时可以预测出不同分子分型中的驱动基因。
3.2.2 基于驱动基因的分类目前检测1p/19q联合缺失的方法应用最多的是荧光原位杂交技术,但其Fish探针并不能直接证明是整条染色体臂的缺失,存在较高的假阳性率[18],所以通过驱动基因作为特征准确区分三个子类也是十分有必要的。而如果只考虑IDH1基因作为特征因子,采用五折交叉验证的随机森林方法对获取到的低级别脑胶质数据进行三子类验证,模型总体准确率仅有64.95%。
若将筛选出的22个驱动基因作为特征因子,用五折交叉验证的随机森林方法对获取到的低级别脑胶质瘤数据进行三个子类的分类验证,最终获取到的三子类分类结果敏感性、特异性、平衡准确性结果见表 5。模型总体准确率达98.99%,结果说明:基于关联规则算法获得的22个驱动基因对分类是有效性,利用关联规则算法预测驱动基因是可行的。
| 表 5 随机森林分类结果 Table 5 Random forest classification results |
筛选出的驱动基因具有多种较为重要的生物学功能,将其映射到GO(Gene Ontology)数据库, 获取这些基因目前已知的功能和参与的过程及细胞组件, 结果见表 6。
| 表 6 GO数据库查询结果(部分) Table 6 GO database query results (Part) |
与金标准相比有17个驱动基因预测结果相同,其中CIC[19]、NOTCH1[20]、ARID1A[21]、TP53[22]、SMARCA4[23]、NIPBL[24]等基因被认为参与RNA聚合酶II启动子转录的负调控; IDH1[18]和IDH2基因参与乙醛酸循环、异柠檬酸代谢过程和三羧酸循环; EGFR[25]、PTEN[26]、TP53和NOTCH1基因被认为参与细胞增殖的正向调控; NOTCH1、ATRX[27]、FUBP1[28]、TP53和SMARCA4基因被认为参与转录调控; NF1[29]、PIK3CA[30]基因被认为参与基因表达调控; PIK3R1[31]基因和PIK3CA基因与磷脂酰肌醇3-激酶信号转导的调控有关; ZBTB20是转录抑制因子家族POK的成员,该家族通过保守的C2H2 Kruppel-type zinc finger和BTB/POZ结构域与DNA相互作用[32]; TCF12基因编码的蛋白是基本螺旋-环-螺旋(bHLH) E-蛋白家族的一员,该家族识别一致结合位点(E-box) CANNTG,该编码蛋白在许多组织中表达,其中包括骨骼肌、胸腺、B-和t细胞,并可能通过与其他bHLH E-蛋白形成异二聚体,参与调节谱系特异性基因的表达,该基因的几种选择性剪接转录变体已被描述,但其中一些变体的全长性质尚未确定[33]。
与金标准相比预测结果不同的驱动基因共有5个,其中ZNF292被预测为一种生长激素端依赖转录因子[34]; MUC16基因编码一种粘蛋白家族成员的蛋白质,这种蛋白质被认为在形成屏障中发挥作用,保护上皮细胞免受病原体的侵袭,这种基因的产物已被用作不同癌症的标记物,表达水平越高,预后越差[35]; PKHD1基因编码的蛋白质预计有一个单一跨膜(TM)结构域和免疫球蛋白样丛状蛋白转录因子结构域的多个副本[36]; HMCN基因编码免疫球蛋白超家族的一个大的细胞外成员[37]; FCGBP基因功能尚不明确。
4 结论1) 运用关联规则算法,通过定义“发生该基因突变则会诱使病人患低级别脑胶质瘤”这一条规则,将基因突变与患癌结果直接联系起来。通过支持度、置信度和提升度这三个指标来判断该基因是否是驱动基因。最终在获取到的491例低级别脑胶质瘤体细胞突变数据中,得到22个与结果存在关联的基因,其中有17个基因在金标准中被列为是驱动基因。22个驱动基因分别对应低级别脑胶质瘤的不同子类,IDHmut-codel子类预测出10个驱动基因,IDHmut-non-codel子类预测出9个驱动基因,针对IDHwt子类预测出3个驱动基因。且经GO数据库查询,找到的驱动基因均具有重要的生物学功能。
2) 用得到的22个驱动基因对已有标签的低级别脑胶质瘤进行分类,通过随机森林方法验证驱动基因关键性,总体模型准确率达98.99%,即得到的22个驱动基因可有效区分低级别脑胶质瘤的三个子类。
3) 基于基因突变频率预测驱动基因提供了新的建立规则的思路,且该规则识别驱动基因的准确率高于目前已有的部分方法,此外方法可用于识别有确定分类标准癌症的不同亚型的驱动基因。
| [1] |
CHENG Feixiong, ZHAO Junfei, ZHAO Zhongming. Advances in computational approaches for prioritizing driver mutations and significantly mutated genes in cancer genomes[J]. Briefings in Bioinformatics, 2016, 17(4): 642-656. DOI:10.1093/bib/bbv068 (  0) 0) |
| [2] |
EISENSTEIN M. Startups use short-read data to expand long-read sequencing market[J]. Nature Biotechnology, 2015, 33: 433-435. DOI:10.1038/nbt0515-433 (  0) 0) |
| [3] |
WILD C P, WEIDERPASS E, STEWART B W. World Cancer Report: Cancer research for cancer prevention[M]. Lyon: International Agency for Research on Cancer, 2020. DOI:10.1007/s00401-016-1545-1
(  0) 0) |
| [4] |
LAWRENCE MS, STOJANOV P, POLAK P, et al. Mutational heterogeneity in cancer and the search for new cancer associated genes[J]. Nature, 2013, 499: 214-218. DOI:10.1038/nature12213 (  0) 0) |
| [5] |
VOGELSTEIN B, PAPADOPOULOS N, VELCULESCU V E, et al. Cancer genome landscapes[J]. Science, 2013, 339(6127): 1546-1558. DOI:10.1126/science.1235122 (  0) 0) |
| [6] |
RAPHAEL B J, DOBSON J R, OESPERET L, et al. Identifying driver mutations in sequenced cancer genomes: computational approaches to enable precision medicine[J]. Genome Medicine, 2014, 6(5): 2-17. DOI:10.1186/gm524 (  0) 0) |
| [7] |
LI Ding, MICHAEL C, MCMICHAEL J F, et al. Expanding the computational toolbox for mining cancer genomes[J]. Nature Reviews Genetics, 2014, 15: 556-570. DOI:10.1038/nrg3767 (  0) 0) |
| [8] |
TAMBORERO D, GONZALEZ-PEREZ A, KANDOTH C, et al. OncodriveCLUST: exploiting the positional clustering of somatic mutations to identify cancer genes[J]. Bioinformatics, 2013, 29(18): 2238-2244. DOI:10.1093/bioinformatics/btt395 (  0) 0) |
| [9] |
REIMAND J, BADER G D. Systematic analysis of somatic mutations in phosphorylation signaling predicts novel cancer drivers[J]. Molecular Systems Biology, 2013, 9(637): 1-18. DOI:10.1038/msb.2012.68 (  0) 0) |
| [10] |
DANIEL C K, CHEN Ken, WYLIE T, et al. VarScan: variant detection in massively parallel sequencing of individual and pooled samples[J]. Bioinformatics, 2009, 25(17): 2283-2285. DOI:10.1093/bioinformatics/btp373 (  0) 0) |
| [11] |
DANIEL C K, ZHANG Qunyuan, SHEN Dong, et al. VarScan 2: Somatic mutation and copy number alteration discovery in cancer by exome sequencing[J]. Genome Research, 2012, 22: 568-576. DOI:10.1101/gr.129684.111 (  0) 0) |
| [12] |
LOUIS D N, PERRY A, REIFENBERGER G, et al. The 2016 World Health Organization Classification of Tumors of the Central Nervous System: a summary[R]. Acta Neuropathologica, 2016, 131: 803-820. DOI: 10.1007/s00401-016-1545-1.
(  0) 0) |
| [13] |
NEWTON Y, NOVAK A M, SWATLOSKI T, et al. TumorMap: exploring the molecular similarities of cancer samples in an interactive portal[J]. Cancer Research, 2017, 77(21): 111-114. DOI:10.1158/0008-5472.CAN-17-0580 (  0) 0) |
| [14] |
华琳, 李林. 医学数据挖掘[M]. 北京: 清华大学出版社, 2016: 156-157. HUA Lin, LI Lin. Medical data mining[M]. Beijing: Tsinghua University Press, 2016: 156-157. (  0) 0) |
| [15] |
TAN Pangning, STEINBACH M, KUMAR V. 数据挖掘导论[M]. 范明, 范宏建, 等. 北京: 人民邮电出版社, 2016: 201-244. TAN Pangning, STEINBACH M, KUMAR V. Introduction to data mining[M]. FAN Ming, FAN Hongjian, et al. Beijing: Posts and Telecommunications Press, 2016: 201-244. (  0) 0) |
| [16] |
BAILEY M H, TOKHEIM C, LI Ding, et al. Comprehensive characterization of cancer drivergenes and mutations[J]. Cell, 2018, 173: 371-385. DOI:10.1016/j.cell.2018.02.060 (  0) 0) |
| [17] |
CHENG Weichong, CHUNG I F, CHEN Chenyang, et al. DriverDB: an exome sequencing database cancer driver gene identification[J]. Nucleic Acids Research, 2014, 42: 1048-1054. DOI:10.1093/nar/gkt1025 (  0) 0) |
| [18] |
王皓. 脑胶质瘤相关分子特征分析[D]. 济南: 山东大学, 2019. WANG Hao. Analysis of molecular characteristics of glioma[D]. Jinan: Shandong University, 2019. (  0) 0) |
| [19] |
LU H C, TAN Qiuming, WANG Wei, et al. Disruption of the ATXN1-CIC complex causes a spectrum of neurobehavioral phenotypes in mice and humans[J]. Nature Genetics, 2017, 49: 527-536. (  0) 0) |
| [20] |
AGRAWAL N, FREDERICK M J, FAKHRY C, et al. Exome sequencing of head and neck squamous cell carcinoma reveals inactivating mutations in NOTCH1[J]. Science, 2011, 333(6046): 1154-1157. DOI:10.1158/1055-9965.disp-11-pr3 (  0) 0) |
| [21] |
NIE Zuqiu, XUE Yutong, YANG Dafei, et al. A specificity and targeting subunit of a human SWI/SNF family-related chromatin remodeling complex[J]. Biochemistry and Molecular Biology, 2000, 20(23): 8879-8888. DOI:10.1128/MCB.20.23.8879-8888.2000 (  0) 0) |
| [22] |
LI Le, MAO Youxiang, ZHAO Lina, et al. p53 regulation of ammonia metabolism through urea cycle controls polyamine biosynthesis[J]. Nature, 2019, 567: 253-256. DOI:10.1038/s41586-019-0996-7 (  0) 0) |
| [23] |
JELINIC P, MUELLER J J, JELINIC N O, et al. Recurrent SMARCA4 mutations in small cell carcinoma of the ovary[J]. Nature Genetics, 2014, 46(2922): 424-426. DOI:10.1038/ng.2922 (  0) 0) |
| [24] |
WATRIN E, SCHLEIFFER A, TANAKA K, et al. Human Scc4 is required for cohesin binding to chromatin, sister-chromatid cohesion, and mitotic progression[J]. Current Biology, 2006, 16(9): 863-874. DOI:10.1016/j.cub.2006.03.049 (  0) 0) |
| [25] |
WANG Ke, YAMAMOTO H, CHIN J R, et al. Epidermal growth factor receptor-deficient mice have delayed primary endochondral ossification because of defective osteoclast recruitment[J]. Journal of Biological Chemistry, 2004, 279(51): 53848-53856. DOI:10.1074/jbc.M403114200 (  0) 0) |
| [26] |
PEZZOLESI M G, ZBUK K M, WAITE K A, et al. Comparative genomic and functional analyses reveal a novel cis-acting PTEN regulatory element as a highly conserved functional E-box motif deleted in Cowden syndrome[J]. Human Molecular Genetic, 2007, 16(9): 1058-1071. DOI:10.1093/hmg/ddm053 (  0) 0) |
| [27] |
BRADLEY E W, DUDAKOVIC A, CARLSON W S, et al. Histone deacetylase H3.3 is required for maintenance of bone mass during aging[J]. Nature, 52(1): 296-307. DOI: 10.1038/nature14345.
(  0) 0) |
| [28] |
BETTEGOWDA C, AGRAWAL N, JIAO Yuchen, et al. Mutations in CIC and FUBP1 contribute to human oligodendroglioma[J]. Science, 2011, 333(6048): 1453-1455. DOI:10.1126/science.1210557 (  0) 0) |
| [29] |
KOCZKOWSKA M, CALLENS T, GOMES A, et al. Expanding the clinical phenotype of individuals with a 3-bp in-frame deletion of the NF1 gene (c.2970_2972del): an update of genotype-phenotype correlation[J]. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2019, 21(3): 867-876. DOI:10.1038/s41436-018-0326-8 (  0) 0) |
| [30] |
WU Guojun, MAMBO E, GUO Zhongming, et al. Uncommon mutation, but common amplifications, of the PIK3CA gene in thyroid tumors[J]. The Journal of Clinical Endocrinology and Metabolism, 2005, 90(8): 4688-4693. DOI:10.1210/jc.2004-2281 (  0) 0) |
| [31] |
PETROVSKI S, PARROTT R E, ROBERTS J L, et al. Dominant splice site mutations in PIK3R1 cause hyper IgM syndrome, lymphadenopathy and short stature[J]. Journal of Clinical Immunology, 2016, 36(5): 462-471. DOI:10.1007/s10875-016-0281-6 (  0) 0) |
| [32] |
SUTHERLAND A P R, ZHANG Hai, ZHANG Ye, et al. Zinc finger protein Zbtb20 is essential for postnatal survival and glucose homeostasis[J]. Molecular and Cellular Biology, 2009, 29(10): 2804-2815. DOI:10.1128/MCB.01667-08 (  0) 0) |
| [33] |
SHARMA V P, FENWICK A L, BROCKOP M S, et al. Mutations in TCF12, encoding a basic helix-loop-helix partner of TWIST1, are a frequent cause of coronal craniosynostosis[J]. Nature Genetics, 2013, 45(10): 304-307. DOI:10.1038/ng.2531 (  0) 0) |
| [34] |
FLYNN M P, HURLEY D L. Growth hormone transcription factor ZN-16 genomic coding regions are composed of a single exon and are evolutionarily conserved in mammals[J]. Gene, 2006, 368(1): 78-83. DOI:10.1038/ng1309 (  0) 0) |
| [35] |
ARGUESO P, SPURR-MICHAUD S, RUSSO C L, et al. MUC16 mucin is expressed by the human ocular surface epithelia and carries the H185 carbohydrate epitope[J]. Investigative Ophthalmology and Visual Science, 003, 44(6): 2487-2495. DOI: 10.1167/iovs.02-0862.
(  0) 0) |
| [36] |
WARD C J, WU Yanhong, JOHNSON R A, et al. Germline PKHD1 mutations are protective against colorectal cancer[J]. Human Genetics, 2011, 129: 345-349. (  0) 0) |
| [37] |
PRAS E, KRISTAL D, SHOHANY N, et al. Rare genetic variants in Tunisian Jewish patients suffering from age-related macular degeneration[J]. Journal of Clinical and Experimental Ophthalmology, 2015, 52(7): 484-492. DOI:10.1136/jmedgenet-2015-103130 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


