慢性阻塞性肺病(Chronic Obstructive Pulmonary Disease,COPD)是一种典型的具有进行性、不可逆的气流限制和反复呼吸道症状的慢性呼吸系统疾病,致残率和病死率很高,全球40岁以上发病率已高达9-10%[1-2]。COPD并非单一疾病,它不仅损伤肺组织,同时还会损伤包括心脏、骨骼、免疫系统、消化系统和中枢神经系统在内的肺外器官和组织,COPD目前已成为全球第三大死因[3-4]。
COPD患者肺部的慢性炎症反应可导致肺实质组织受损,并破坏正常的修复功能和防御机制,导致小气道纤维化,最终产生气体陷闭和进行性气流受限,成为导致气道阻力增加的主要因素[5-6]。由此可见,COPD与小气道病变息息相关,尽早干预轻中度COPD患者的小气道病变,对于延缓病程具有重要意义。研究表明,被诊断为肺癌的患者中40%-70%患有COPD[7],而COPD患者的肺癌发病率为16.7‰,且最长见的病理类型是肺鳞状细胞癌(Lung Squamous Cell Carcinoma,LUSC)[8]。对于吸烟者来说,COPD患者患肺癌的几率是非COPD患者的4~6倍[9],并且轻中度COPD患者患肺癌的几率较重度COPD更高,这可能与轻中度COPD患者存在炎症和氧化应激增高有关[10]。目前,COPD与肺癌共存机制尚不清楚,如果能够探寻COPD合并肺癌的预测因子,研究两者共同的发病机制,发现新的治疗靶点,可为COPD的预防、治疗和肺癌的早期诊断、降低死亡率提供理论依据。
目前,关于COPD的生物信息学研究还较少,王小乐等通过对COPD患者肺泡巨噬细胞的芯片数据集进行生物信息学分析,共获得43个差异表达基因,且与免疫和炎症的发生和发展有关[11]。谷雨等对COPD患者肺组织的芯片数据进行了生物信息学分析,找到了FGG, FGA, IL-6, SERPINE1和SPP1 5个Hub基因,根据调控途径的进一步分析发现差异基因表达可能与代谢及细胞生物功能异常有关[12]。
本研究采用生物信息学方法,从GEO(Gene Expression Omnibus)数据库中筛选3个COPD芯片数据集,挖掘COPD患者小气道上皮细胞(Small airway epithelium,SAEC)的差异表达基因(Differentially Expressed Genes,DEGs)以及潜在的生物标记物,并通过基因本体(Gene Ontology,GO)和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)富集分析预测DEGs的功能及参与的代谢途径。通过对DEGs进行COPD的蛋白质网络互作分析,构建PPI(Protein-Protein Interaction)网络,并使用Cytoscape软件中的CytoHubba和MCODE筛选子模块和Hub基因。最后将10个Hub基因通过TCGA和GTEx数据库验证其在LUSC样本中的差异表达情况及差异基因间的相关性,为确定COPD的分子标记物及COPD与LUSC共存的DEGs奠定一定的基础。
1 材料与方法 1.1 材料使用“COPD”作为关键词,在GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)中搜索并选择了3个芯片数据集:GSE11784、GSE11906和GSE20257,均来自于GPL570平台(Affymetrix Human Genome U133 Plus 2.0 Array,Affymetrix,Inc)。由于芯片数据都出自威尔·康奈尔医学院遗传医学系实验室,其中的部分样本在不同芯片间存在重复,去除重复样本后,最终GSE11784纳入53例正常对照和14例COPD患者,GSE11906纳入46例正常对照和15例COPD患者,GSE20257纳入22例正常对照和9例COPD患者(见表 1),3个芯片共纳入159例样本,其中包含121例正常对照和38例COPD患者。
| 表 1 GEO数据库COPD芯片数据集 Table 1 COPD microarray datasets from GEO database |
分别对3个数据集探针的矩阵文件取log2,然后使用Perl 5.0软件(http://www.perl.org)对数据进行注释、合并,再通过R软件(4.0.3版本)的sva和limma包对合并后的数据进行批次校正。
1.2.2 DEGs分析使用R软件中的Limma包对正常样本和COPD样本进行比较,在校正后的P值(AdjP) < 0.05的条件下鉴定DEGs。
1.2.3 Go和KEGG富集分析使用R软件中的ClusterProfiter和org.Hs.eg.db包进行GO和KEGG富集分析,以P < 0.05定义差异具有统计学意义。
1.2.4 PPI网络分析使用在线网站STRING(http://string-db.org)分析DEGs的相互作用关系,选择最小交互作用得分>0.4的蛋白质对构建PPI网络。然后分别使用Cytoscape中的MCODE(degree cut≥2,node score cut≥0.2,K-core≥2,max depth=100)和CytoHubba插件筛选重要的子模块和Hub基因。
1.2.5 Hub基因在TCGA和GTEx数据库中的显著性验证使用在线网站GEPIA(Gene Expression Profiling Interactive Analysis) (http://gepia.cancer-pku.cn/index.html),选择TCGA和GTEx数据库中的LUSC样本(486例)和正常样本(338例)对Hub基因进行差异性验证(|Log2FC|>2,p-value < 0.05,Jitter Size=0.4)。
1.2.6 Hub基因的相关性分析使用在线网站GEPIA网站,选择TCGA数据库中的LUSC样本(486例)和正常样本(50例)对Hub基因进行差异分析。
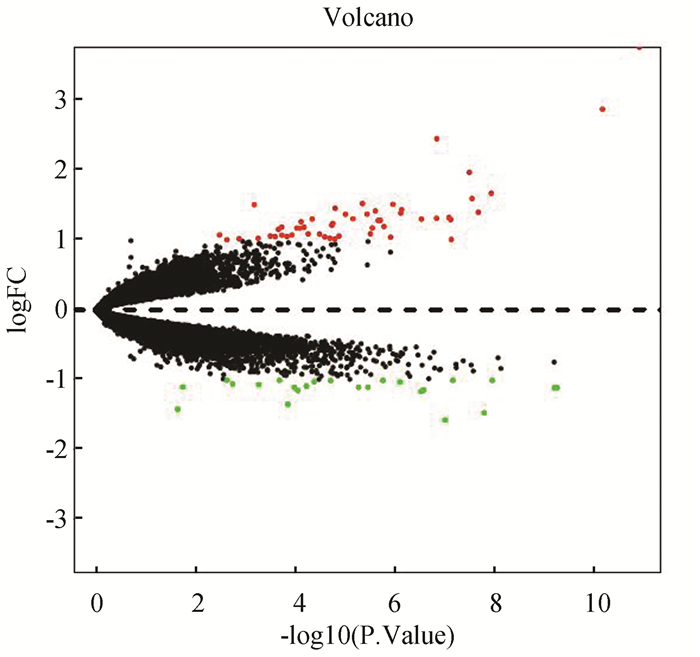
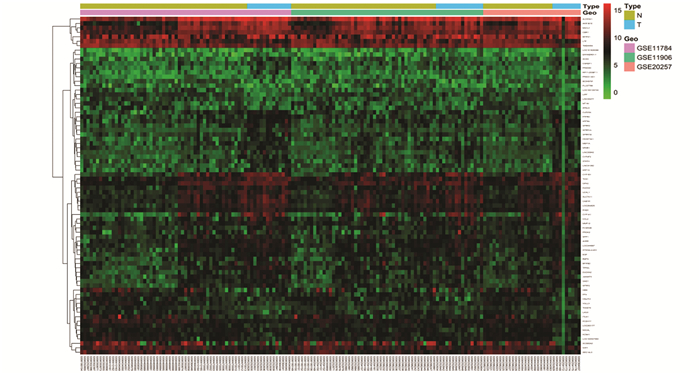
2 结果与分析 2.1 COPD患者的DEGs通过对GSE11784、GSE11906和GSE20257进行RNA水平上正常样本和COPD样本分析,共获得76个DEGs,包括52个上调基因和24个下调基因(见表 2),DEGs的火山图和热图(见图 1和图 2)。
| 表 2 三个GEO数据集中的76个DEGs Table 2 A total of 76 DEGs in three GEO datasets |
|
图 1 DEGs的火山图 Figure 1 Volcano plot for DEGs 注:图中红点代表显著上调基因,绿点代表显著下调基因,黑点代表非显著表达基因,所有差异表达基因符合矫正后p值< 0.05. |
|
图 2 COPD和正常样本DEGs热图 Figure 2 Heat map for DEGs between COPD and normal samples 注:图中从红色到绿色,基因表达水平逐渐下降,所有差异表达基因符合矫正后p值< 0.05;N代表正常样本,T代表肿瘤样本. |
通过R软件的ClusterProfiter和org.Hs.eg.db包进行DEGs的GO富集分析(见表 3),DEGs主要富集在受体-配体活性、信号受体激活剂活性、四吡咯结合、内肽酶活性、血红素结合和G蛋白偶联受体结合等功能上。
| 表 3 COPD差异表达基因的GO富集分析 Table 3 GO enrichment analysis of DEGs in COPD samples |
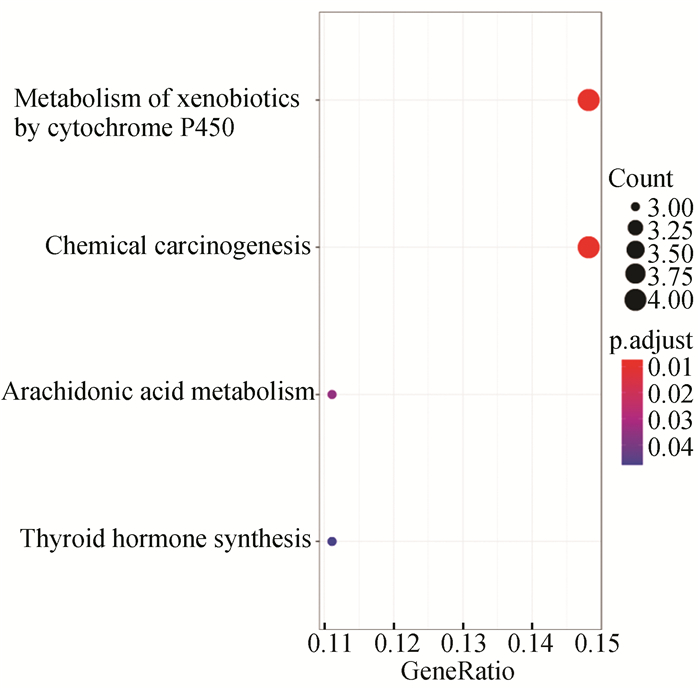
通过R软件的ClusterProfiter包进行DEGs的KEGG代谢通路分析, 结果(见如图 3和表 4),DEGs主要富集在细胞色素P450对外源物质的代谢、化学致癌、花生四烯酸代谢和甲状腺激素合成4条代谢途径中。
|
图 3 COPD差异表达基因的KEGG代谢通路富集分析 Figure 3 KEGG enrichment analysis of DEGs in COPD 注:X轴代表差异基因中与该通路相关的基因数与整个差异基因总数的比值,Y轴代表KEGG富集通路. |
| 表 4 COPD差异表达基因的KEGG代谢通路富集分析 Table 4 KEGG pathway analysis of DEGs in COPD samples |
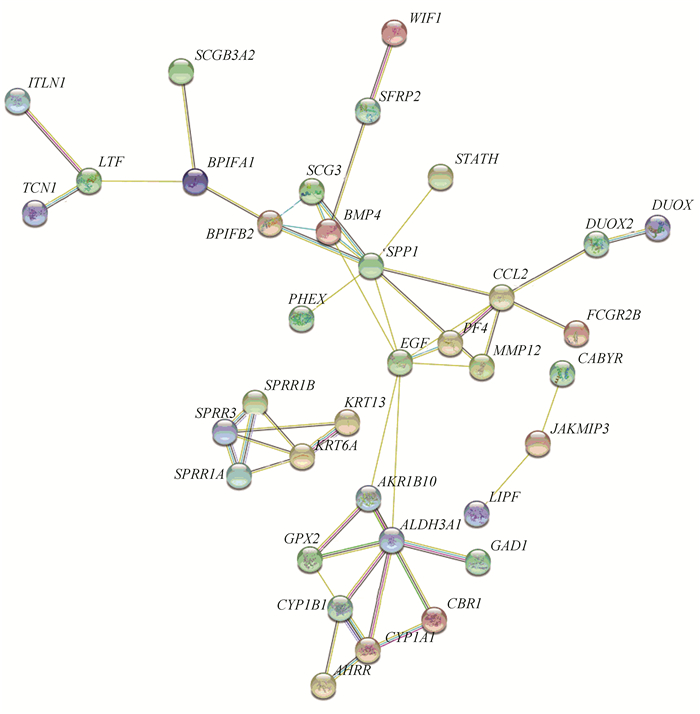
使用在线网站STRING (http://string-db.org) 对52个上调基因和24个下调基因进行COPD的蛋白质互作分析,构建PPI网络(见图 4),每个基因代表一个节点,每2个蛋白质之间的连线代表一个关系对,总共有36个节点和51个关系对。
|
图 4 COPD差异基因的PPI网络 Figure 4 PPI network of DEGs in COPD samples 注:图中圆圈代表基因,连线代表基因间蛋白质的相互作用. |
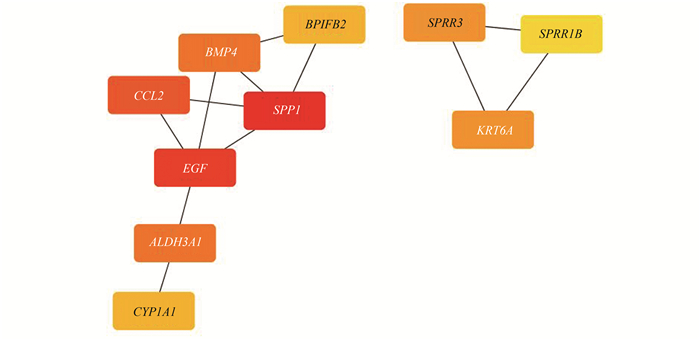
使用Cytoscape软件中的MCODE从PPI网络中筛选得到2个功能模块(见图 5),进一步使用CytoHubba来确定Hub基因(见图 6),评分前十位的基因分别是SPP1、EGF、CCL2、ALDH3A1、BMP4、SPRR3、KRT6A、BPIFB2、CYP1A1和SPRR1B,且都属于上调基因。与图 5比较发现,这10个基因有7个存在于两个功能模块中,因此将这10个基因作为Hub基因做进一步分析对于COPD患病机理及分子机制具有一定的研究意义。

|
图 5 用MCODE插件从PPI网络中筛选获得两个关键功能模块(A和B) Figure 5 PPI network of two key modules obtained by MCODE in cytoscape(A and B) |
|
图 6 通过PPI网络筛选10个Hub基因 Figure 6 Ten hub genes screened by PPI network 注:图中颜色越深,基因评分越高. |
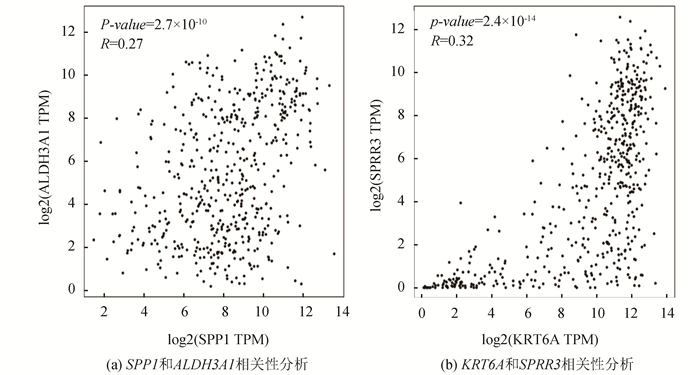
TCGA (The Cancer Genome Atlas)是一个涵盖33种癌症及对应正常样本的数据库,GTEx (Genotype Tissue Expression)数据库仅包含正常人的样本,通常与TCGA数据库结合使用,以弥补TCGA正常样本的不足。本研究中,为探寻COPD与LUSC共同的生物标记物,将GEO数据库中获得的COPD的10个Hub基因在TCGA和GTEx数据库的LUSC样本中进行差异性验证。基因SPP1、ALDH3A1、SPRR3、KRT6和SPRR1B均具有显著性差异(见图 7),表明这5个基因是COPD和LUSC共同的DEGs。进一步分析发现,SPP1和ALDH3A1、SPRR3和KRT6A呈显著正相关(见图 8),其它基因间无显著性关系。

|
图 7 Hub基因SPP1(a)、ALDH3A1(b)、SPRR3(c)、KRT6A(d)、SPRR1B(e)在TCGA和GTEx数据库中LUSC样本中的差异性验证 Figure 7 Significance verification of SPP1(a), ALDH3A1(b), SPRR3(c), KRT6A(d), and SPRR1B(e) in LUSC samples in TCGA and GTEx databases |
|
图 8 Hub基因相关性分析 Figure 8 Correlation analysis of hub genes 注:A: SPP1和ALDH3A1相关性分析,B: KRT6A和SPRR3相关性分析. |
COPD是一种常见的以持续气流受限为特征的疾病,气流受限进行性发展,与气道和肺脏对有毒颗粒或气体的慢性炎性反应增强有关[13]。小气道管腔狭窄,空气阻力将成倍增加,周围气道阻力增高是小气道病变的重要病理生理学特征,故当小气道有炎症或痰液阻塞,很容易造成闭合、萎陷,COPD病变也多从小气道开始[14]。
本研究以小气道上皮细胞为研究对象,筛选3个COPD的GEO芯片数据集进行生物信息学分析,获得了52个上调基因和24个下调基因。GO分析发现DEGs主要富集在受体-配体活性、信号受体激活剂活性、四吡咯结合、内肽酶活性、血红素结合和G蛋白偶联受体结合等功能上,KEGG代谢通路主要集中在细胞色素P450对外源物质的代谢、化学致癌、花生四烯酸代谢和甲状腺激素合成4条途径上。进一步分析发现参与GO和KEGG代谢通路的DEGs均为上调基因,这些功能和代谢通路均与COPD发病机理有密切关系。程序性细胞死亡受体1(PD-1)通过与其配体PD-L1结合,负调控T淋巴细胞活化及效应功能,参与免疫耐受的维持,从而促进了COPD的发生和发展过程[15]。乙酰胆碱受体M3是引起气道平滑肌收缩的主要受体,激活后发生构象改变,并进一步激活磷脂酶C,催化二磷酸磷脂酰肌醇水解为三磷酸肌醇,促进钙离子通道开放,引起气道平滑肌收缩,气流阻力增大[16]。花生四烯酸是在炎性反应中由细胞膜磷脂在磷脂酶A2催化下产生的,并经过酶促反应生成多种代谢物,它们可以通过多种途径刺激呼吸道中的伤害性感受器,导致炎性细胞聚集而发生局部效应[17]。由此可见筛选得到的差异表达基因在COPD的发病机制中占有重要地位,可为后续研究奠定良好的基础。
进一步取交互作用得分>0.4的蛋白质构建PPI网络,发现共有36个基因参与到网络构建中,为获得关键基因,使用Cytoscape软件中的MCODE和CytoHubba插件对PPI网络图进行分析,分别获得了两个重要的子模块和10个Hub基因,比较发现有7个Hub基因位于子模块中,其中ALDH3A1和CYP1A1为细胞色素P450对外源物质的代谢和化学致癌途径中的重要基因(见表 4),因此可初步认为筛选得到的Hub基因对于进一步研究COPD患病机理及分子机制具有一定的研究意义。为寻求COPD与LUSC共存的DEGs,将10个Hub基因通过TCGA和GTEx数据库验证其在LUSC中的差异表达情况,从而确定SPP1、ALDH3A1、SPRR3、KRT6A和SPRR1B为COPD与LUSC共存的DEGs。进一步分析其相关性发现仅SPP1和ALDH3A1、SPRR3和KRT6A间呈显著正相关(见图 8),目前尚未有该基因对间作用机制的相关报道,是否存在共同的调节因子还有待进一步研究。
早期研究并没有发现SPP1与COPD之间的联系[18],血液分析结果显示癌症分期和SPP1表达水平相互矛盾[19],而最新报道表明SPP1在COPD和肺癌患者中均高表达,并且与肺癌较低的生存率密切相关[20],这与本研究结果一致,可以初步认为COPD中SPP1的高表达与患肺癌风险增加有关,SPP1可能是一个治疗靶点,用于延缓COPD患者肺癌的发展,提高其生存时间。
ALDH3A1属于乙醛脱氢酶家族3中的成员A1,在人体的胃、食道和肺中含量较高。通过前列腺肿瘤异种移植的动物实验发现ALDH3A1可以促进前列腺肿瘤细胞肺转移的发生[21]。ALDH3A1基因表达还与大肝癌早期复发转移关系密切,并且该基因表达越高,复发转移时间越长[22]。目前关于ALDH3A1对COPD和LUSCD之间的关系还鲜有报道,通过KEGG代谢通路分析我们可以看到ALDH3A1参与了细胞色素P450对外源物质的代谢和化学致癌两个途径(见表 4)。细胞色素P450是人体内主要的多环芳烃代谢酶,参与内外源性物质的代谢,对于化学致癌物来说,可把无活性的前致癌物激活转变为电子化合物,与DNA或蛋白质结合,导致癌变。由于ALDH3A1在COPD和LUSC中均上调,可以认为高表达的ALDH3A1加速了细胞色素P450对外源物质尤其是化学致癌物质的代谢,从而促进了COPD和LUSC的发生和发展。
SPRRs为一类富含脯氨酸蛋白的基因家族,该家族共有4个成员:SPRR1、SPRR2、SPRR3和SPRR4。它们蛋白产物的中央片段均由串联重复的氨基酸(XKXPEPXX(X为任意氨基酸))序列组成:SPRR1、SPRR3和SPRR4为8个,SPRR2为9个。本研究涉及到的Hub基因为SPRR1B和SPRR3。研究表明这两个基因参与多种癌症的发生和发展。SPRR1B在口腔鳞癌组织中过表达,从而影响细胞增长[23],它还参与黑色素瘤的转移[24],并且是气道上皮细胞鳞状细胞分化的早期生物标志物,其表达下调伴随细胞的恶性转化[25]。通过meta和Kaplan-Meier分析,发现SPRR1B在肺腺癌组织中表达增强,敲除该基因可抑制肺腺癌细胞增殖、迁移和侵袭;此外,通过火山图发现肺癌组织中SPRR3的表达水平也明显增高[26]。同样,SPRR1B和SPRR3在肺癌H1299细胞系中均过表达[27]。本研究与前人结果一致,SPRR1B和SPRR3在COPD和LUSC中均表达上调,提示这两个基因在COPD和LUSC发生机制的探讨中具有潜在的研究价值,建议SPRR家族基因与COPD和肺癌的关系应进一步深入研究。
4 结论综上所述,SPP1、ALDH3A1、SPRR3、KRT6A和SPRR1B可作为COPD的分子标记物及LUSC的DEGs,尤其是ALDH3A1和SPRR家族基因与COPD和LUSC间的关系及作用机制,有望做进一步深入的研究,为找到COPD和LUSC新的治疗靶点提供理论依据。
| [1] |
WANG C, XU J Y, YANG L, et al. Prevalence and risk factors of chronic obstructive pulmonary disease in China (the China Pulmonary Health[CPH] study): a national cross-sectional study[J]. Lancet, 2018, 391(10131): 1706-1717. DOI:10.1016/S0140-6736(18)30841-9 (  0) 0) |
| [2] |
ZHANG Q, SONG W, AYIDAERHAN N, et al. PTPLAD2 and USP49 involved in the pathogenesis of smoke-induced COPD by integrative bioinformatics analysis[J]. International Journal of COPD, 2020, 15: 2515-2526. DOI:10.2147/COPD.S250576 (  0) 0) |
| [3] |
SINGH D, AGUSTI A, ANZUETO A, et al. Global strategy for the diagnosis, management, and prevention of chronic obstructive lung disease: the GOLD science committee report 2019[J]. European Respiratory Journal, 2019, 53(5): 1900164. DOI:10.1183/13993003.00164-2019 (  0) 0) |
| [4] |
LOZANO R, NAGHAVI M, FOREMAN K, et al. Global and regional mortality from 235 causes of death for 20 age groups in 1990 and 2010: a systematic analysis for the Global Burden of Disease Study 2010[J]. Lancet, 2016, 380(9859): 2095-2128. DOI:10.1016/S0140-6736(12)61728-0 (  0) 0) |
| [5] |
DEEPAK D. Recognition of small airways obstruction in asthma and COPD-the road less travelled[J]. Journal of Clinical & Diagnostic Research, 2017, 11(3): TE01-TE05. DOI:10.7860/JCDR/2017/19920.9478 (  0) 0) |
| [6] |
王晓娟, 方向阳. 慢性阻塞性肺疾病全球倡议2019: 慢性阻塞性肺疾病诊断、治疗与预防全球策略解读[J]. 中国全科医学, 2019, 22(18): 2141-2149. WANG Xiaojuan, FANG Xiangyang. Interpretation of the global strategy for the diagnosis, management, and prevention of COPD 2019 by GOLD[J]. Chinese General Practice, 2019, 22(18): 2141-2149. DOI:10.12114/j.issn.1007-9572.2019.00.064 (  0) 0) |
| [7] |
LOGANATHAN R S, STOVER D E, SHI W, et al. Prevalence of COPD in women compared to men around the time of diagnosis of primary lung cancer[J]. Chest, 2006, 129(5): 1305-1312. DOI:10.1378/chest.129.5.1305 (  0) 0) |
| [8] |
DE TORRES J, MARFN J M, CASANOVA C, et al. Lung cancer in patients with chronic obstructive pulmonary disease—incidence and predicting factors[J]. American Journal of Respiratory and Critical Care Medicine, 2011, 184(8): 913-919. DOI:10.1164/rccm.201103-0430OC (  0) 0) |
| [9] |
YOUNG R P, HOPKINS R J, CHRISTMAS T, et al. COPD prevalence is increased in lung cancer, independent of age, sex and smoking history[J]. European Respiratory Journal, 2009, 34(2): 380-386. DOI:10.1183/09031936.00144208 (  0) 0) |
| [10] |
BRETT A S, AL-HASAN M N. COPD exacerbations—A target for antibiotic stewardship[J]. New England Journal of Medicine, 2019, 381(2): 174-185. DOI:10.1056/NEJMe1905520 (  0) 0) |
| [11] |
王小乐, 朱洁, 童佳兵, 等. 慢性阻塞性肺疾病肺泡巨噬细胞差异表达基因的生物信息学分析[J]. 细胞与分子免疫学杂志, 2020, 36(11): 961-966. WANG Xiaole, ZHU Jie, TONG Jiabing, et al. Bioinformatical analysis of differentially expressed genes in alveolar macrophages of chronic obstructive pulmonary disease(COPD)[J]. Chinese Journal of Cellular and Molecular Immunology, 2020, 36(11): 961-966. DOI:10.13423/j.cnki.cjcmi.009091 (  0) 0) |
| [12] |
谷雨, 王文俊, 胡悦, 等. 基于芯片数据的慢性阻塞性肺疾病患者的生物信息学分析[J]. 国际呼吸杂志, 2021, 41(10): 736-743. GU Yu, WANG Wenjun, HU Yue, et al. Bioinformatics analysis in chronic obstructive pulmonary disease patients based on microarraydata[J]. International Journal of Respiration, 2021, 41(10): 736-743. DOI:10.3760/cma.j.cn131368-20210128-00078 (  0) 0) |
| [13] |
王志华, 谢俊刚. 慢性阻塞性肺疾病的表观遗传学机制研究进展[J]. 中华结核和呼吸杂志, 2017, 40(6): 472-474. WANG Zhihua, XIE Jungang. Research progress on epigenetic mechanism of COPD[J]. Chinese Journal of Tuberculosis and Respiratory Diseases, 2017, 40(6): 472-474. DOI:10.3760/cma.j.issn.1001-0939.2017.06.015 (  0) 0) |
| [14] |
朱金源, 马莉琼, 张锦. 家族序列相似性13A基因干扰对人气道上皮细胞凋亡和增殖的影响及与慢性阻塞性肺疾病患者小气道重塑的关系[J]. 中华医学杂志, 2020, 100(32): 2481-2487. ZHU Jinyuan, MA Liqiong, ZHANG Jin. Effect of family with sequence similarity 13 member A gene interference on apoptosis and proliferation of human airway epithelial cells and its relationship with small airway remodeling in patients with chronic obstructive pulmonary disease[J]. National Medical Journal of China, 2020, 100(32): 2481-2487. DOI:10.3760/cma.j.cn112137-20200326-00945 (  0) 0) |
| [15] |
周建鹏, 邓静敏, 段敏超. 程序性细胞死亡受体1与慢性阻塞性肺疾病中T淋巴细胞免疫[J]. 中华微生物学和免疫学杂志, 2021, 41(3): 237-241. ZHOU Jianpeng, DENG Jingmin, DUAN Minchao. Programmed cell death 1 and T lymphocyte immunity in chronic obstructive pulmonary disease[J]. Chinese Journal of Microbiology and Immunology, 2021, 41(3): 237-241. DOI:10.3760/cma.j.cn112309-20200713-00352 (  0) 0) |
| [16] |
GOSENS R, ZAAGSMA J, MEURS H, et al. Muscarinic receptor signaling in the pathophysiology of asthma and COPD[J]. Respiratory Research, 2006, 7: 73. DOI:10.1186/1465-9921-7-73 (  0) 0) |
| [17] |
林树新, 於峻. 花生四烯酸代谢物对呼吸道感受器的作用[J]. 生理学报, 2007(2): 141-149. LIN Shuxin, YU Jun. Effects of arachidonic acid metabolites on airway sensors[J]. Acta Physiologica Sinica, 2007(2): 141-149. DOI:10.13294/j.aps.2007.02.005 (  0) 0) |
| [18] |
PAPAPORFYRIOU A, LOUKIDES S, KOSTIKAS K, et al. Increased levels of osteopontin in sputum supernatant in patients with COPD[J]. Chest, 2014, 146(4): 951-958. DOI:10.1378/chest.13-2440 (  0) 0) |
| [19] |
RUD A K, BOYE K, OIJORDSBAKKEN M, et al. Osteopontin is a prognostic biomarker in non-small cell lung cancer[J]. BMC Cancer, 2013, 13(1): 540. DOI:10.1186/1471-2407-13-540 (  0) 0) |
| [20] |
MIAO T, XIAO W, DU L, et al. High expression of SPP1 in patients with chronic obstructive pulmonary disease (COPD) is correlated with increased risk of lung cancer[J]. FEBS Open Bio, 2021, 11(4): 1237-1249. DOI:10.1002/2211-5463.13127 (  0) 0) |
| [21] |
YAN J, MELO J D, CUTZ J C, et al. Aldehyde dehydrogenase 3A1 associates with prostate tumorigenesis[J]. British Journal of Cancer, 2014, 110(10): 2593-2603. DOI:10.1038/bjc.2014.201 (  0) 0) |
| [22] |
魏复群. ALDH3A1基因表达与大肝癌早期复发转移关系研究[D]. 福州: 福建医科大学, 2014. WEI Fuqun. Study on the relationship between the expression of ALDH3A1 gene and early recurrence and metastasis of large hepatocellular carcinoma[D]. Fuzhou: Fujian Medical University, 2014. (  0) 0) |
| [23] |
MICHIFURI Y, HIROHASHI Y, TORIGOE T, et al. Small proline-rich protein-1B is overexpressed in human oral squamous cell cancer stem-like cells and is related to their growth through activation of MAP kinase signal[J]. Biochemical and Biophysical Research Communications, 2013, 439(1): 96-102. DOI:10.1016/j.bbrc.2013.08.021 (  0) 0) |
| [24] |
SHENG Z, HAN W, HUANG B, et al. Screening and identification of potential prognostic biomarkers in metastatic skin cutaneous melanoma by bioinformatics analysis[J]. Journal of Cellular and Molecular Medicine, 2020, 24(19): 11613-11618. DOI:10.1111/jcmm.15822 (  0) 0) |
| [25] |
TESFAIGZI J, CARLSON D M. Expression, regulation, and function of the SPR family of proteins[J]. Cell Biochemistry & Biophysics, 1999, 30(2): 243-265. DOI:10.1007/BF02738069 (  0) 0) |
| [26] |
ZHANG Z, SHI R, XU S, et al. Identification of small proline-rich protein 1B (SPRR1B)as a prognostically predictive biomarker for lung adenocarcinoma by integrative bioinformatic analysis[J]. Thoracic Cancer, 2021, 2(4): 796-806. DOI:10.1111/1759-7714.13836 (  0) 0) |
| [27] |
XIONG Y, LI M, ZHANG P, et al. Study on genetype in lung squamous carcinoma by high-throughput of transcriptomesequence[J]. Chinese Journal of Lung Cancer, 2017, 20(11): 727-731. DOI:10.3779/j.issn.1009-3419.2017.11.01 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


