2. 广西壮族自治区人民医院 耳鼻咽喉头颈科, 南宁 530000;
3. 广西百色市人民医院 耳鼻咽喉头颈外科, 广西 百色 533000;
4. 广西百色市人民医院 病理科, 广西 百色 533000
2. Department of Otorhinolaryngology, Head and Neck, The People's Hospital of Guangxi Zhuang Autonomous Region, Nanning 530000, China;
3. Department of Otorhinolaryngology, Head and Neck Surgery, People's Hospital of Guangxi Baise, Baise 533000, Guangxi, China;
4. Department of Pathology, People's Hospital of Guangxi Baise, Baise 533000, Guangxi, China
头颈部鳞状细胞癌(HNSC)是全球第八大常见癌症[1],主要起源于口腔、口咽、下咽、喉和鼻咽内的亚解剖部位[2]。在全球范围内,2020年报告的HNSC新病例约880 000例,死亡病例约440 000例[1]。在台湾和东南亚,口腔鳞状细胞癌占HNSC的主要比例[3]。根据TNM分期和原发部位,HNSC主要采用不同组合的手术、放疗和化疗的方法[4]。尽管技术和支持治疗的进步提高了HNSC患者的生活质量,但由于术后局部复发和远处转移,整体预后仍然较差[5]。接受同步放化疗的HNSC患者的5年总生存(Overall survival,OS)率约为50%[6],中位OS时间为66.3个月[7]。
免疫治疗作为一种新的治疗方法已经应用于多种恶性肿瘤的研究,并在一些恶性肿瘤取得较好疗效。尽管免疫检查点抑制剂在癌症治疗中显示出巨大的应用前景,但不同肿瘤和同一肿瘤不同患者中的治疗效果存在较大的异质性[8]。恶性肿瘤的免疫逃避机制和肿瘤微环境免疫抑制状态,构成了免疫治疗的障碍。HNSC患者的不良临床结果可能取决于其肿瘤免疫浸润的特征[9]。因此,迫切需要了解影响HNSC肿瘤免疫微环境的分子标志物,以进行个体化治疗及改善预后。
分泌型磷蛋白1(Secreted Phosphoprotein 1,SPP1),也称为骨桥蛋白(Osteopontin,OPN),是由SPP1基因编码的一种钙结合糖磷蛋白,存在于许多正常组织中[10]。SPP1既可以充当细胞粘附蛋白,又可以充当细胞因子,通过多种细胞表面受体(包括多种整联蛋白和CD44)将信号传递至细胞内[11]。SPP1与这些细胞表面受体的结合可引起细胞功能的广泛变化,例如移动性和粘附性增强、生长和分裂加速、细胞存活时间延长和血管生成[12]。SPP1参与许多生理和病理过程,包括骨和骨基质重塑、血管生成、癌变、炎症和自身免疫[13]。Zhang等[14]研究发现SPP1可能促进膀胱癌的增殖,抑制细胞凋亡并加速侵袭,从而促进膀胱癌的发展和转移。临床研究表明,在许多类型的人类癌症中,SPP1水平与恶性程度或患者存活率之间存在相关性[15]。Overgaard等[16]对320例头颈癌病例进行了研究,报告称SPP1高表达的患者局部复发率比低表达患者高2.85倍。目前SPP1在HNSC中的免疫浸润相关性研究较少,通过TCGA的HNSC数据,分析了SPP1在HNSC中对免疫浸润的影响及其与临床预后的相关性,以明确SPP1在HNSC预后和个体化治疗中的作用。
1 材料和方法 1.1 数据来源及数据处理数据来源自UCSC官网(https://xenabrowser.net/datapages)下载的TCGA HNSC的RNA测序数据,以及对应样本的临床、预后数据和批次效应注释文件。使用R语言读取HNSC的counts数据,并根据TCGA官网(https://portal.gdc.cancer.gov)下载的泛癌样本注释文件过滤样本,将不合格样本剔除(活检前有放化疗病史、合并多种肿瘤,以及为复发或转移的肿瘤患者),同时提取其中的mRNA表达矩阵。根据过滤的样本,筛选对应样本的临床数据及预后数据。
1.2 表达差异分析使用R语言的DESeq2[17]包对mRNA表达矩阵进行表达差异分析,分析过程纳入批次效应,以lgFC绝对值>0.5, 校正后P值< 0.05为界值,以获得差异基因用于下游分析。
1.3 临床相关性分析使用R语言将TCGA HNSC的mRNA表达矩阵counts数据转换为TPM格式数据,以用于后续分析。使用UCSC官网下载的HNSC的临床数据,结合TCGA HNSC的mRNA表达矩阵,将TCGA HNSC患者按SPP1表达量的4分位数分组,高于表达量75%分位数为高SPP1表达组,低于表达量25%分位数为低SPP1表达组,分析SPP1表达与临床病理特征的相关性,使用Cox回归模型进行单变量及多变量分析,并绘制Kaplan-Meier生存曲线。
1.4 相关性分析及基因富集分析基因富集分析是分析基因功能的一种方法,主要包括基因本体论(Gene Ontology,GO)[18]分析和信号通路分析。其中GO分析包括3个部分,分别为分子功能(Molecular Function,MF)分析、生物过程(Biological Process,BP)分析和细胞组成(Cellular Components,CC)分析。KEGG[19]的全称为京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG),是从分子水平信息,特别是基因组测序和其他高通量实验技术产生的大规模分子数据集,了解细胞、生物体和生态系统等生物系统的高级功能和实用性的综合性数据库。GSEA[20]全称是基因集富集分析(Gene set enrichment analysis, GSEA),利用测序或芯片获得的全基因组表达谱进行分析,不需要指定差异基因阈值,得出的结果更加可靠。使用R语言的cor.test函数对SPP1与差异基因中其他mRNA分子进行相关性分析,计算Pearson相关系数,并将与SPP1最正相关的前200个基因用于下游分析。使用R语言clusterProfiler[21]包中的EnrichGO及EnrichKEGG函数对SPP1最正相关的前200个基因进行GO及KEGG分析,使用gseKEGG函数对整个mRNA表达矩阵进行基于GSEA算法的KEGG分析。
1.5 肿瘤浸润免疫细胞分析CIBERSORT (https://cibersort.stanford.edu)是一种准确且稳健的算法,用于根据基因表达谱计算组织的细胞组成[22]。使用R语言的CIBERSORT函数对HNSC的mRNA的表达矩阵进行肿瘤浸润免疫细胞分析,获得所有样本的肿瘤浸润免疫细胞表达比例矩阵,并将肿瘤免疫浸润矩阵结合HNSC的mRNA的表达矩阵中SPP1的表达量数据进行SPP1表达与肿瘤浸润免疫细胞关联性分析。
1.6 统计分析对测序数据的清洗和分析使用R语言(版本4.0.3)进行,对临床数据的统计分析使用IBM SPSS 23.0版本进行。绘图使用R语言及GraphPad Prism 8.0软件。使用卡方检验分析SPP1表达与患者临床病理特征之间的关联。使用单变量和多变量Cox回归分析评估生存数据。生存曲线使用Kaplan-Meier方法绘制,并使用log-rank检验进行比较。在所有分析中,小于0.05的双尾P值被认为具有统计学意义(*P < 0.05, **P < 0.01, ***P < 0.001)。
2 结果 2.1 数据来源及数据处理为了获取清洁的数据来源,使用R语言读取HNSC的RNA测序counts格式数据,并根据TCGA官网的样本注释文件过滤样本,将不合格样本剔除(活检前有放化疗病史、合并多种肿瘤,以及为复发或转移肿瘤患者),同时提取其中的mRNA表达矩阵,从而获得了包含466个肿瘤组织样本,39个正常组织样本,以及18 498个编码基因的mRNA表达矩阵。使用R语言读入并合并下载自UCSC官网的相应样本的临床及预后数据,剔除临床资料缺失的样本后,获得433个具有完整临床病理分期及预后数据的肿瘤组织样本信息。处理后的数据将用于后续分析。
2.2 表达差异分析为了获得TCGA HNSC mRNA表达矩阵中的差异基因,使用R语言的DESeq2包对mRNA表达矩阵进行表达差异分析,分析过程纳入批次效应,以lgFC绝对值> 0.5, 校正后P值< 0.05为界值,通过肿瘤组织样本与正常组织样本比较,获得了8 146个差异基因,其中上调基因3 631个,下调基因4 515个,并以火山图展示基因集差异表达情况(见图 1a)。在获得的差异基因中,SPP1在肿瘤组织样本表达较正常组织样本显著增高(见图 1b,P < 0.001),lgFC值为2.53。进一步通过GEPIA网站[23]的生存曲线绘制,发现SPP1在HNSC中具有预后价值。因此,推测SPP1可能在HNSC发生与发展过程起重要作用。我们在表达差异分析中以lgFC绝对值> 0.5为界值,获取了8 146个差异基因,目的是为了获取更多的与SPP1相关性高的差异基因用于后续分析。

|
图 1 TCGA HNSC数据集的基因差异分析 Figure 1 Analysis of gene differential expression in TCGA HNSC data set |
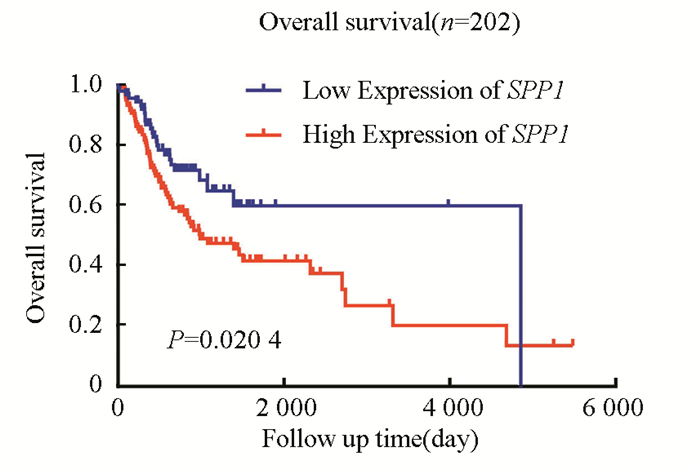
为了探索SPP1表达与HNSC患者临床病理特征的相关性及其预后价值,将TCGA HNSC患者按SPP1表达量的4分位数分组,高于表达量75%分位数为高SPP1表达组,低于表达量25%分位数为低SPP1表达组,两组共包含202例肿瘤样本(见表 1)。分析了SPP1与临床病理特征的相关性,结果(见表 2),SPP1表达与年龄(P = 0.003)、临床分期(P = 0.013)及T分期(P= 0.001)显著相关。然而,SPP1的表达与性别(P = 0.555)、N分期(P = 0.191)及M分期(P= 0.507)无关。Cox回归分析用于确定SPP1表达水平是否可以作为影响生存的危险因素。通过单变量Cox回归分析,与SPP1低表达者相比,SPP1高表达患者死亡风险显著增加(P=0.022)(见表 3)。将单变量分析中对生存影响有统计学意义(P < 0.05)的变量纳入多变量Cox回归分析,结果显示当SPP1表达(P=0.037)、性别(P=0.024)及M分期(P=0.004)包括在内时,SPP1可能是预测不良生存的独立危险因素(见表 4)。Kaplan-Meier生存曲线表明SPP1高表达患者的总生存期明显短于SPP1低表达患者(见图 2,P=0.020 4),SPP1高表达患者中位生存时间为993 d,而低表达患者中位生存时间为4 856 d。总的来说,这些结果表明,SPP1在HNSC患者中的高表达与肿瘤的进展及较短的存活期相关。
| 表 1 TCGA HNSC患者基线资料表 Table 1 Baseline data of TCGA HNSC patients |
| 表 2 SPP1表达与TCGA HNSC患者临床病理特征的相关性 Table 2 Correlation between SPP1 expression and clinicopathological characteristics of TCGA HNSC patients |
| 表 3 TCGA HNSC患者各种预后参数的单变量Cox回归分析 Table 3 Univariate Cox regression analysis of various prognostic parameters of TCGA HNSC patients |
| 表 4 TCGA HNSC患者各种预后参数的多变量Cox回归分析 Table 4 Multivariate Cox regression analysis of various prognostic parameters of TCGA HNSC patients |
|
图 2 按SPP1的高表达和低表达分组,202名HNSC患者的Kaplan-Meier总生存曲线 Figure 2 Kaplan-Meier overall survival curve of 202 HNSC patients grouped by high and low expressions of SPP1 |
为了解SPP1在HNSC中的生物学功能,包括相关通路,使用R语言的cor.test函数对SPP1与差异基因中其他mRNA分子进行相关性分析(见图 3a),图中展示了相关系数最高的15个正相关及15个负相关的差异基因。选择与SPP1最正相关的前200个基因进行富集分析。使用R语言的clusterProfiler包进一步探索了基于这200个基因的潜在功能途径。GO分析显示SPP1最正相关的前200个基因主要富集于细胞外基质组成、胶原纤维组成、胶原蛋白结合、中性粒细胞脱颗粒、免疫反应中涉及的中性粒细胞活化、三级颗粒、IgG结合、清道夫受体活性及免疫球蛋白结合等条目(见图 3b-3d)。此外,KEGG通路分析表明SPP1最正相关的前200个基因主要富集于吞噬体、补体凝血级联、中性粒细胞胞外陷阱形成等免疫相关信号通路(见图 3e)。GSEA分析用于探索KEGG通路,结果显示补体凝血级联、吞噬体、细胞粘附分子、中性粒细胞胞外陷阱形成及细胞因子-细胞因子受体相互作用信号通路显著富集(见图 3f)。这些结果表明SPP1在HNSC中与细胞外基质及胶原纤维形成等生物学过程有关,并与免疫学功能及免疫相关通路有关联。

|
图 3 SPP1在TCGA HNSC中与其他差异表达基因的相关性分析及功能和通路富集分析 Figure 3 Correlation analysis of SPP1 with other differentially expressed genes and function and pathway enrichment analysis in TCGA HNSC |
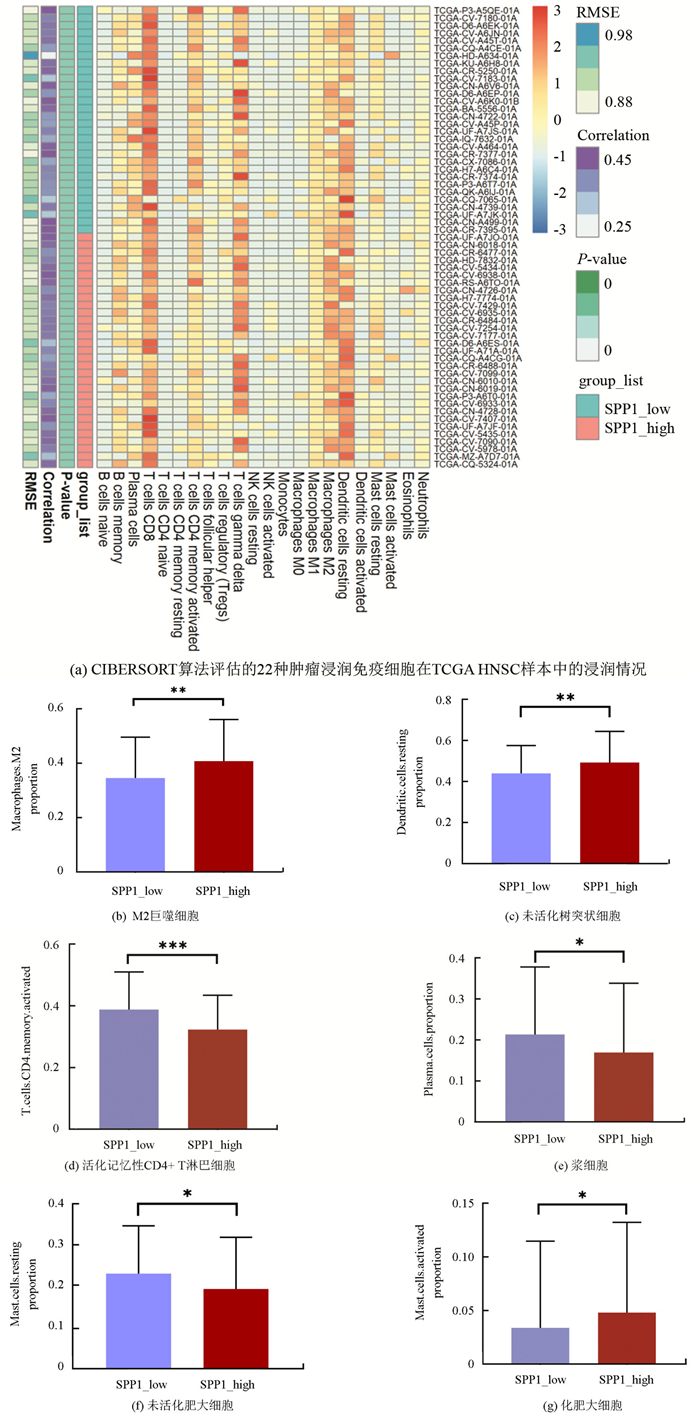
为了明确SPP1与肿瘤浸润免疫细胞的关系,进一步使用R语言的CIBERSORT函数评估了TCGA HNSC的免疫细胞浸润比例,获得了TCGA HNSC样本中包含22种肿瘤浸润免疫细胞的浸润比例矩阵(见图 4a)。通过结合TCGA HNSC mRNA表达矩阵中SPP1表达量进行分析,发现M2巨噬细胞在SPP1高表达组中浸润比例高于SPP1低表达组(见图 4b,P=0.001 1),未活化树突状细胞在SPP1高表达组中浸润比例高于SPP1低表达组(见图 4c,P=0.005 5),活化肥大细胞在SPP1高表达组中浸润比例高于SPP1低表达组(图 4g,P=0.048 8),而活化记忆性CD4+ T淋巴细胞在SPP1高表达组中浸润比例低于SPP1低表达组(见图 4d,P < 0.001),浆细胞在SPP1高表达组中浸润比例低于SPP1低表达组(见图 4e,P=0.026 6),未活化肥大细胞在SPP1高表达组中浸润比例低于SPP1低表达组(见图 4f,P=0.038 6)。这些结果表明SPP1的表达与HNSC中肿瘤浸润免疫细胞状态密切相关。
|
图 4 TCGA HNSC中与SPP1相关的肿瘤浸润免疫细胞分析 Figure 4 Analysis of tumor infiltrating immune cells related to SPP1 in TCGA HNSC |
SPP1是一种糖磷蛋白,主要与整合素结合发挥作用,它在多种肿瘤中高表达,例如肝癌、肺癌、前列腺癌、乳腺癌、结直肠癌及头颈部鳞状细胞癌等[24]。Staibano等研究发现,在喉鳞状细胞癌中,SPP1表达与异型增生程度呈正相关,与生存率呈负相关[25]。通过对TCGA的HNSC数据分析,发现SPP1在HNSC中高表达,与之前的研究一致。推测,SPP1在HNSC中的高表达,可能与HNSC的发生与发展相关。分析了SPP1与HNSC患者的临床病理特征的关系,发现SPP1与年龄、T分期及临床分期有关,且高表达SPP1患者具有较短的生存期,通过Cox回归模型,发现SPP1表达水平是影响患者生存的独立危险因素。因此,研究结果表明SPP1的高表达,促进HNSC的进展,并且预示较差的预后,可能成为潜在的预测HNSC预后的生物标志物。
作为一种基质细胞蛋白,SPP1充当细胞-细胞外基质相互作用的调节剂,通过与细胞表面受体、生长因子、结构基质蛋白及蛋白酶结合,使它们成为细胞外基质环境的重要组成部分[26]。SPP1调节金属蛋白酶的分泌以及成纤维细胞的增殖和分化,在伤口愈合中发挥着重要作用[27]。成纤维细胞也与炎症密切相关,它们在肿瘤微环境中产生胶原蛋白和其他细胞外基质成分,刺激癌细胞增殖和血管生成[28]。通过GO分析发现,SPP1与细胞外基质组成、胶原纤维组成及胶原蛋白结合等生物学过程及分子功能有关,与此前的研究一致。上述结果表明SPP1涉及多种生物学功能,包括细胞外基质形成、组织重塑和修复、炎症反应及肿瘤进展等。随着免疫治疗方法的兴起,SPP1在免疫学方面的功能研究也逐渐开展。SPP1在多种免疫性疾病,如系统性红斑狼疮、多发性硬化症和类风湿性关节炎中高表达,具有免疫调节特性[29]。在肿瘤方面,Wei等[30]研究发现,SPP1是一种有效的巨噬细胞趋化因子,沉默SPP1明显削弱了胶质瘤细胞募集巨噬细胞的能力。在结肠癌中,SPP1通过与CD44结合负调节T细胞活化并促进癌症进展[29]。但目前SPP1在HNSC中免疫学功能研究较少,尤其在肿瘤免疫微环境方面。通过GO分析显示SPP1与中性粒细胞脱颗粒、免疫反应中涉及的中性粒细胞活化、三级颗粒、IgG结合、清道夫受体活性及免疫球蛋白结合等免疫相关条目有关。此外,KEGG通路分析表明SPP1与吞噬体、补体凝血级联、中性粒细胞胞外陷阱形成等免疫相关信号通路有关。基于GSEA算法的KEGG分析还显示SPP1与细胞粘附分子及细胞因子-细胞因子受体相互作用信号通路有关。这些结果表明SPP1在HNSC中与免疫学功能及免疫相关通路有关联。但SPP1在这些免疫相关功能及通路中的具体分子机制还有待进一步研究。
SPP1对多种免疫细胞具有趋化性,包括巨噬细胞、树突细胞和T细胞等,SPP1主要通过与这些免疫细胞表面的整合素及CD44分子相互作用,通过不同的细胞信号通路,调节免疫细胞的存活、极化与迁移等;根据细胞所处的病理环境不同,SPP1可能会表现为促炎或抗炎作用(免疫抑制)[28, 31]。巨噬细胞被识别为两种不同的极化状态,经典激活的M1型(促炎)和选择性活化的M2型(免疫抑制)[32]。M1巨噬细胞倾向于促进抗肿瘤免疫反应,而M2巨噬细胞发挥促肿瘤作用。树突状细胞(Dendritic cells, DCs)通过加工和呈递肿瘤抗原给T细胞,在抗肿瘤免疫反应中发挥关键作用。诱导瘤内DCs的成熟以激活T细胞介导的抗癌免疫可能是一种有效的抗肿瘤策略[33]。肿瘤内CD4+ T细胞负责抗肿瘤免疫反应。Al-Shibli等[34]研究发现,CD4+T细胞数量的增加与非小细胞肺癌疾病特异性存活率的改善显著相关,并且高密度的基质CD4+ T细胞是非小细胞肺癌患者的有利独立预后因素。浆细胞分泌对特异性抗原具有高亲和力的抗体,在介导特异性细胞免疫和体液免疫中起重要作用[35]。浆细胞还可以通过细胞因子和免疫调节因子的表达发挥其抗肿瘤作用,创造更有效的抗肿瘤微环境[35]。肥大细胞传统上与过敏和炎症有关,但现在已经认识到肥大细胞对肿瘤细胞和肿瘤微环境具有重要影响[36]。肥大细胞在一些肿瘤中与不良预后有关,如胃癌[37]、肺癌[38]和乳腺癌[39]。通过CIBERSORT算法评估的22种肿瘤浸润免疫细胞在TCGA HNSC样本中的浸润情况,发现SPP1表达与巨噬细胞、树突状细胞、肥大细胞、浆细胞及记忆性CD4+ T淋巴细胞浸润水平显著相关,在SPP1高表达组M2巨噬细胞、未活化树突状细胞及活化肥大细胞浸润比例显著增加,而活化记忆性CD4+ T淋巴细胞、浆细胞及未活化肥大细胞浸润比例显著减少。因此,认为SPP1可能通过影响HNSC中各种肿瘤浸润免疫细胞的浸润水平以及它们的活化状态,从而影响免疫抑制微环境的形成,进而导致HNSC的进展,并影响患者的预后。但SPP1在HNSC中影响肿瘤浸润免疫细胞浸润水平的具体分子机制尚未清楚,有待进一步研究。
4 结论总之,SPP1具有免疫调节的特性,在肿瘤免疫微环境中起重要作用,并且可能作为HNSC有价值的预后生物标志物及免疫治疗靶标。为深入研究HNSC的发生发展机制、治疗和预后靶点提供了理论依据。但目前的研究主要基于数据库生物信息学分析,SPP1与肿瘤浸润免疫细胞的直接关系仍需要组织样本及细胞实验进一步研究。靶向SPP1改善免疫抑制微环境以增强免疫治疗效果的方法需要在未来的基础及临床试验中进一步验证。
| [1] |
SUNG H, FERLAY J, SIEGEL R L, et al. Global Cancer Statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 (  0) 0) |
| [2] |
MARUR S, FORASTIERE A A. Head and neck cancer: changing epidemiology, diagnosis, and treatment[J]. Mayo Clinic Proceedings, 2008, 83(4): 489-501. DOI:10.4065/83.4.489 (  0) 0) |
| [3] |
CHIEN C Y, SU CY, CHUANG H C, et al. Comprehensive study on the prognostic role of osteopontin expression in oral squamous cell carcinoma[J]. Oral Oncology, 2009, 45(9): 798-802. DOI:10.1016/j.oraloncology.2008.12.006 (  0) 0) |
| [4] |
MARUR S, FORASTIERE A A. Head and neck squamous cell carcinoma: Update on epidemiology, diagnosis, and treatment[J]. Mayo Clinic Proceedings, 2016, 91(3): 386-396. DOI:10.1016/j.mayocp.2015.12.017 (  0) 0) |
| [5] |
SACCO A G, COHEN E E. Current treatment options for recurrent or metastatic head and neck squamous cell carcinoma[J]. Journal of Clinical Oncology, 2015, 33(29): 3305-3313. DOI:10.1200/JCO.2015.62.0963 (  0) 0) |
| [6] |
LIN S S, MASSA S T, VARVARES M A. Improved overall survival and mortality in head and neck cancer with adjuvant concurrent chemoradiotherapy in national databases[J]. Head Neck, 2016, 38(2): 208-215. DOI:10.1002/hed.23869 (  0) 0) |
| [7] |
STOKES W A, AMINI A, JONES B L, et al. Survival impact of induction chemotherapy in advanced head and neck cancer: A National Cancer Database analysis[J]. Head Neck, 2017, 39(6): 1113-1121. DOI:10.1002/hed.24739 (  0) 0) |
| [8] |
ZHANG Y, LIN A, LI Y, et al. Age and mutations as predictors of the response to immunotherapy in head and neck squamous cell cancer[J]. Frontiers in Cell and Developmental Biology, 2020, 8: 608969. DOI:10.3389/fcell.2020.608969 (  0) 0) |
| [9] |
FENOGLIO D, BELGIOIA L, PARODI A, et al. Development of exhaustion and acquisition of regulatory function by infiltrating CD8+CD28- T lymphocytes dictate clinical outcome in head and neck cancer[J]. Cancers (Basel), 2021, 13(9). DOI:10.3390/cancers13092234 (  0) 0) |
| [10] |
GIACHELLI C M, STEITZ S. Osteopontin: A versatile regulator of inflammation and biomineralization[J]. Matrix Biology, 2000, 19(7): 615-622. DOI:10.1016/s0945-053x(00)00108-6 (  0) 0) |
| [11] |
DENHARDT D T, GIACHELLI C M, RITTLING S R. Role of osteopontin in cellular signaling and toxicant injury[J]. Annual Review of Pharmacology and Toxicology, 2001, 41: 723-749. DOI:10.1146/annurev.pharmtox.41.1.723 (  0) 0) |
| [12] |
BELLAHCÈNE A, CASTRONOVO V, OGBUREKE K U, et al. Small integrin-binding ligand N-linked glycoproteins (SIBLINGs): multifunctional proteins in cancer[J]. Nature Reviews Cancer, 2008, 8(3): 212-226. DOI:10.1038/nrc2345 (  0) 0) |
| [13] |
DENHARDT D T, NODA M, O'REGAN A W, et al. Osteopontin as a means to cope with environmental insults: Regulation of inflammation, tissue remodeling, and cell survival[J]. The Journal of Clinical Investigation, 2001, 107(9): 1055-1061. DOI:10.1172/JCI12980 (  0) 0) |
| [14] |
ZHANG N, LI F, GAO J, et al. Osteopontin accelerates the development and metastasis of bladder cancer via activating JAK1/STAT1 pathway[J]. Genes Genomics, 2020, 42(4): 467-475. DOI:10.1007/s13258-019-00907-6 (  0) 0) |
| [15] |
WEBER G F, LETT G S, HAUBEIN N C. Osteopontin is a marker for cancer aggressiveness and patient survival[J]. British Journal of Cancer, 2010, 103(6): 861-869. DOI:10.1038/sj.bjc.6605834 (  0) 0) |
| [16] |
OVERGAARD J, ERIKSEN J G, NORDSMARK M, et al. Plasma osteopontin, hypoxia, and response to the hypoxia sensitiser nimorazole in radiotherapy of head and neck cancer: Results from the DAHANCA 5 randomised double-blind placebo-controlled trial[J]. Lancet Oncology, 2005, 6(10): 757-764. DOI:10.1016/S1470-2045(05)70292-8 (  0) 0) |
| [17] |
LOVE M I, HUBER W, ANDERS S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biology, 2014, 15(12): 550. DOI:10.1186/s13059-014-0550-8 (  0) 0) |
| [18] |
The Gene Ontology Consortium. The Gene Ontology resource: Enriching a GOld mine[J]. Nucleic Acids Research, 2021, 49(D1): D325-D334. DOI:10.1093/nar/gkaa1113 (  0) 0) |
| [19] |
KANEHISA M, SATO Y, FURUMICHI M, et al. New approach for understanding genome variations in KEGG[J]. Nucleic Acids Research, 2019, 47(D1): D590-D595. DOI:10.1093/nar/gky962 (  0) 0) |
| [20] |
SUBRAMANIAN A, TAMAYO P, MOOTHA V K, et al. Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(43): 15545-15550. DOI:10.1073/pnas.0506580102 (  0) 0) |
| [21] |
YU G, WANG L G, HAN Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. OMICS: A Journal of the Integrative Biology, 2012, 16(5): 284-287. DOI:10.1089/omi.2011.0118 (  0) 0) |
| [22] |
NEWMAN A M, LIU C L, GREEN M R, et al. Robust enumeration of cell subsets from tissue expression profiles[J]. Nature Methods, 2015, 12(5): 453-457. DOI:10.1038/nmeth.3337 (  0) 0) |
| [23] |
LI C, TANG Z, ZHANG W, et al. GEPIA2021: Integrating multiple deconvolution-based analysis into GEPIA[J]. Nucleic Acids Research, 2021, 49(W1): W242-W246. DOI:10.1093/nar/gkab418 (  0) 0) |
| [24] |
WEI T, BI G, BIAN Y, et al. The significance of secreted phosphoprotein 1 in multiple human cancers[J]. Frontiers in Molecular Biosciences, 2020, 7: 565383. DOI:10.3389/fmolb.2020.565383 (  0) 0) |
| [25] |
STAIBANO S, MEROLLA F, TESTA D, et al. OPN/CD44v6 overexpression in laryngeal dysplasia and correlation with clinical outcome[J]. British Journal of Cancer, 2007, 97(11): 1545-1551. DOI:10.1038/sj.bjc.6604070 (  0) 0) |
| [26] |
LOK Z, LYLE A N. Osteopontin in vascular disease[J]. Arteriosclerosis, Thrombosis, and Vascular Biology, 2019, 39(4): 613-622. DOI:10.1161/ATVBAHA.118.311577 (  0) 0) |
| [27] |
ICER M A, GEZMEN-KARADAG M. The multiple functions and mechanisms of osteopontin[J]. Clinical Biochemistry, 2018, 59: 17-24. DOI:10.1016/j.clinbiochem.2018.07.003 (  0) 0) |
| [28] |
IIDA T, WAGATSUMA K, HIRAYAMA D, et al. Is osteopontin a friend or foe of cell apoptosis in inflammatory gastrointestinal and liver diseases?[J]. International Journal of Molecular Sciences, 2017, 19(1): 7. DOI:10.3390/ijms19010007 (  0) 0) |
| [29] |
SHURIN M R. Osteopontin controls immunosuppression in the tumor microenvironment[J]. Journal of Clinical Investigation, 2018, 128(12): 5209-5212. DOI:10.1172/JCI124918 (  0) 0) |
| [30] |
WEI J, MARISETTY A, SCHRAND B, et al. Osteopontin mediates glioblastoma-associated macrophage infiltration and is a potential therapeutic target[J]. Journal of Clinical Investigation, 2019, 129(1): 137-149. DOI:10.1172/JCI121266 (  0) 0) |
| [31] |
WANG K X, DENHARDT D T. Osteopontin: Role in immune regulation and stress responses[J]. Cytokine and Growth Factor Reviews, 2008, 19(5/6): 333-345. DOI:10.1016/j.cytogfr.2008.08.001 (  0) 0) |
| [32] |
DENARDO D G, RUFFELL B. Macrophages as regulators of tumour immunity and immunotherapy[J]. Nature Reviews Immunology, 2019, 19(6): 369-382. DOI:10.1038/s41577-019-0127-6 (  0) 0) |
| [33] |
GARDNER A, RUFFELL B. Dendritic cells and cancer immunity[J]. Trends in Immunology, 2016, 37(12): 855-865. DOI:10.1016/j.it.2016.09.006 (  0) 0) |
| [34] |
AL-SHIBLI K I, DONNEM T, AL-SAAD S, et al. Prognostic effect of epithelial and stromal lymphocyte infiltration in non-small cell lung cancer[J]. Clinical Cancer Research, 2008, 14(16): 5220-5227. DOI:10.1158/1078-0432.CCR-08-0133 (  0) 0) |
| [35] |
KROEGER D R, MILNE K, NELSON B H. Tumor-infiltrating plasma cells are associated with tertiary lymphoid structures, cytolytic t-cell responses, and superior prognosis in ovarian cancer[J]. Clinical Cancer Research, 2016, 22(12): 3005-3015. DOI:10.1158/1078-0432.CCR-15-2762 (  0) 0) |
| [36] |
LICHTERMAN J N, REDDY S M. Mast cells: A new frontier for cancer immunotherapy[J]. Cells, 2021, 10(6): 1270. DOI:10.3390/cells10061270 (  0) 0) |
| [37] |
SAMMARCO G, VARRICCHI G, FERRARO V, et al. Mast cells, angiogenesis and lymphangiogenesis in human gastric cancer[J]. International Journal of Molecular Sciences, 2019, 20(9). DOI:10.3390/ijms20092106 (  0) 0) |
| [38] |
TAKANAMI I, TAKEUCHI K, NARUKE M. Mast cell density is associated with angiogenesis and poor prognosis in pulmonary adenocarcinoma[J]. Cancer, 2000, 88(12): 2686-2692. (  0) 0) |
| [39] |
REDDY S M, REUBEN A, BARUA S, et al. Poor response to neoadjuvant chemotherapy correlates with mast cell infiltration in inflammatory breast cancer[J]. Cancer Immunology Research, 2019, 7(6): 1025-1035. DOI:10.1158/2326-6066.CIR-18-0619 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


