宫颈癌(Cervical cancer, CC)的发病率和死亡率在女性恶性肿瘤中均排名第二位,仅次于乳腺癌[1]。在中国,每年死于宫颈癌的女性有5万多名[2]。宫颈癌的临床治疗方法包括手术、化疗和放疗,这些治疗手段有效降低了早期宫颈癌患者的死亡率,但晚期宫颈癌患者的治疗效果不佳[3]。因此,迫切需要识别新的肿瘤标志物,对宫颈癌患者的预后进行准确判断,以期指导临床治疗。
有研究证实基因组不稳定性是癌症的驱动因素之一[4]。基因组不稳定性已被确定为一个重要的预后因素,基因组不稳定性的积累与肿瘤进展和生存相关[5]。尽管基因组不稳定性的分子机制尚未完全了解,但异常转录和转录后调控与基因组不稳定性有关,显示了分子标记作为基因组不稳定性定量测量的潜力[6]。例如,Christine How[7]等研究发现基因组不稳定性在宫颈癌中具有重要作用,且与宫颈癌患者的预后显著相关。长链非编码RNA (lncRNA)是一类无编码蛋白质能力的、长度大于200 nt的转录本[8]。近年来,越来越多的体内和体外实验证明,lncRNA在不同的生物学过程中发挥着重要的作用[9-10]特别是lncRNA的异常表达可能影响细胞增殖、肿瘤进展或转移[11-12]。目前已发现大量的lncRNA在各种癌症中异常表达[13-14],部分在宫颈癌中异常表达的lncRNA与宫颈癌患者的预后密切相关[15-17]。新的研究证明lncRNA在维持基因组不稳定性方面起到关键作用[18-19]。Mendell[20]等研究发现一种特定的lncRNA即被DNA损伤(NORAD)激活的非编码RNA,与参与DNA复制和修复的蛋白质相互作用,有助于基因组的稳定性。虽然一些lncRNA已被证实参与了基因组不稳定性,但基因组不稳定性相关的lncRNA及其在癌症中的临床意义仍有很大部分未被探索。
本研究基于TCGA数据库中宫颈癌的lncRNA表达谱和体细胞突变谱,设计了一个基于突变假设的预后模型,以研究lncRNA标记作为基因组稳定性指标的可能性,为宫颈癌的预后提供新的研究思路。
1 材料和方法 1.1 数据收集女性宫颈癌患者的临床数据、转录组数据和体细胞突变信息来自癌症基因组图谱(TCGA)数据库(https://portal.gdc.cancer.gov)。我们保留337例具有配对lncRNA和mRNA表达谱、生存信息、体细胞突变信息和常见临床病理特征的女性样本,以供进一步研究。本研究中所有的宫颈癌患者按照批次分为两个组,分别命名为Train组和Test组。临床和病理特征的简要总结(见表 1)。
| 表 1 两组宫颈癌患者的临床信息 Table 1 Clinical information of two groups of patients with cervical cancer |
结合TCGA数据库中宫颈癌的lncRNA表达谱和体细胞突变谱,鉴定基因组不稳定性相关的lncRNA:1)计算每个患者的累积体细胞突变数量;2)患者按体细胞突变累积数量降序排列;3)将前25%的患者定义为基因组不稳定(GU-like)组,最后25%定义为基因组稳定(GS-like)组; 4)使用微阵列显著性分析(SAM)方法比较GU-like组和GS-like组之间lncRNA表达谱;5)两组间差异表达的lncRNA(logFC>0或logFC < 0, P < 0.05)被定义为基因组不稳定性相关的lncRNA。
1.3 统计分析采用欧氏距离法和沃德连锁法进行层次聚类分析。采用单因素和多因素Cox分析来评估基因组不稳定性相关lncRNA的表达水平与总生存率之间的关系。我们构建了一个预后风险模型用于结果预测,公式如下:
| $\begin{gathered}\operatorname{GILncSig}(\text { patient })=\sum\limits_{i=1}^n \operatorname{coef}(lncRNAi) * \\ \operatorname{expr}(lncRNAi)\end{gathered}$ | (1) |
其中,GILncSig(patient)是宫颈癌患者的预后风险值。lncRNAi代表第i个预后lncRNA, expr (lncRNAi)代表患者lncRNAi的表达水平,coef(lncRNAi)表示lncRNAi对预后风险值的贡献,这些评分由多变量Cox分析的回归系数获得。利用预后模型分别计算Train组与Test组各样本的预后风险值。将各组样本的风险值从低到高排序,以Train组中患者的中位风险值作为分界点,将患者分为高GILncSig分值的高风险组和低GILncSig分值的低风险组。采用Kaplan-Meier法计算各预后危险组的生存率和中位生存期,采用log-rank检验评估高风险组和低风险组的生存期差异,其显著性水平为5%。采用多变量Cox回归和分层分析来评估GILncSig是否独立于其他关键临床因素。利用survival ROC包输出所有样本的ROC曲线对GILncSig的预测能力进行评估。采用R-version 4.0.3进行所有统计分析[21]。
1.4 功能富集分析通过皮尔逊相关系数来衡量配对的lncRNA和mRNA的表达是否具有相关性,前10个mRNA被认为是lncRNA共同表达的相关伴侣。为了预测lncRNA的潜在功能,使用R语言的clusterProfiler数据包对lncRNA共表达的mRNA进行GO和KEGG的功能注释、分类或通路分析。
2 结果 2.1 基因组不稳定性相关lncRNA的鉴定为了识别与基因组不稳定性相关的lncRNA,计算每个患者的体细胞突变累积数量并按降序排列。根据体细胞突变的累积数量,将前25%(n=73)患者分配到GU-like组和最后25%(n=74)名患者分配到GS-like组。然后比较GU-like组73例患者和GS-like组74例患者的lncRNA表达谱,发现差异显著的lncRNA。通过Wilcoxon检验,总共36个lncRNA具有显著差异(logFC>0或logFC < 0, P < 0.05)。其中,在GU-like组中发现9个lncRNA上调, 27个lncRNA下调(见图 1)。
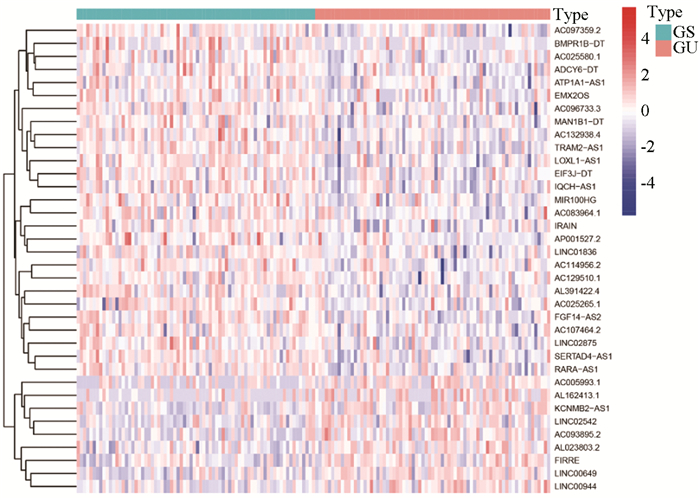
|
图 1 差异表达的基因组不稳定性相关lncRNA Figure 1 Differentially expressed genomic instability-related lncRNAs 注:表达越高或越低,颜色越深(红色上调,蓝色下调). |
利用36个差异表达lncRNA的集合,对来自TCGA数据库的337名宫颈癌患者进行了无监督层次聚类分析,结果(见图 2a)。根据36个差异表达的lncRNA的表达水平,将337个样本分为两组。两组样本的体细胞突变模式有显著差异。体细胞突变累积数量较多的组命名为GU-like组,另一组命名为GS-like组。GU-like组体细胞突变累积数量中位数显著高于GS-like组(P < 0.01, Mann-Whitney U检验; 见图 2b)。接下来,比较了UBQLN4基因(一个新发现的基因组不稳定性驱动因子)在两组中的表达水平。结果(见图 2c),GU-like组中UBQLN4的表达水平与GS-like组无明显差异(P=0.51, Mann-Whitney U检验)。为了确定36个lncRNA的潜在功能和通路是否与基因组不稳定性相关,通过GO和KEGG富集分析来预测其潜在功能。首先筛选出36个差异表达的lncRNA相关的蛋白编码基因(protein coding genes, PCGs)PCGs,挑选出与每个lncRNA相关性最高的前10个PCGs。构建了一个lncRNA-mRNA共表达网络,其中节点为lncRNA和mRNA,如果它们相互关联,则lncRNA和mRNA连接在一起(见图 2d)。对lncRNA相关的PCGs进行GO富集分析,以确定PCGs的功能。关于生物过程,该网络中的PCGs主要与2-氧代戊二酸代谢过程显著相关。细胞组分中GO含量最高的是运动纤毛。此外,在分子功能方面,GO含量最高的是转移酶活性,转移含氮基团(见图 2e)。对lncRNA相关的PCGs进行KEGG通路分析,我们发现其与2-氧代羧酸代谢通路显著相关(见图 2e)。
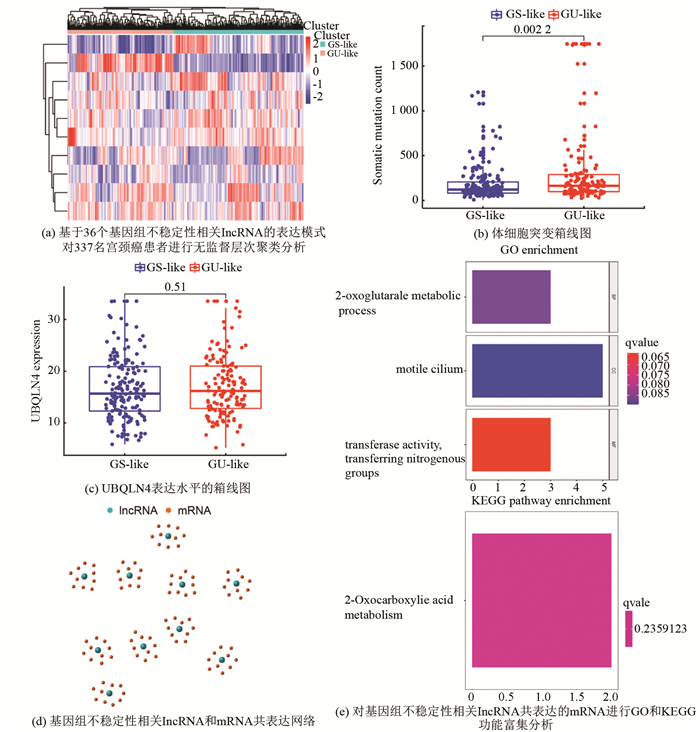
|
图 2 宫颈癌患者基因组不稳定性相关lncRNA的鉴定和功能注释 Figure 2 Identification and functional annotation of lncRNAs related to genomic instability in patients with cervical cancer *注: (a)左边为GU-like组,右边为GS-like组,表达越高或越低,颜色越深(红色上调,蓝色下调),(b)红色簇代表GU-like组,蓝色代表GS-like组,P=0.002 2,(c)红色簇代表GU-like组,蓝色代表GS-like组,P=0.51, (d)显示基因组不稳定性相关lncRNA共表达相关性排名前十的PCGs(红色点代表mRNA, 蓝色点代表LncRNA),(e)分析了基因组不稳定性相关lncRNA共表达基因的GO注释和KEGG途径的显著富集。 |
为了进一步研究这些lncRNA与宫颈癌患者生存预后之间的关系,将TCGA数据库下载的337例宫颈癌患者分为Train组(n=169)和Test组(n=168)。为了筛选与预后相关的lncRNA,采用单因素Cox分析分析36个基因组不稳定性相关lncRNA的表达水平与Train组中病人生存时间和生存状态的关系,2个基因组不稳定性相关lncRNA (AC107464.2和AP001527.2)被确定为宫颈癌预后相关lncRNA(P < 0.05)。根据单因素Cox分析系数和两个预后相关lncRNA的表达水平来构建预后风险模型(GILncSig)评估宫颈癌患者的预后风险值:GILncSig分值=(-1.201 8×AC107 464.2的表达量)+(0.091 6×AP001527.2的表达量)。在GILncSig中,AP001527.2的系数是正值,说明它可能是宫颈癌生存预后相关危险因素,其高表达与预后不良有关,AC107464.2的系数是负值,说明它可能是宫颈癌预后相关保护性因素,其高表达与更长的生存期相关。根据预后风险模型得到Train组中每个患者的风险值,然后以中位风险值作为分界点将这些患者分为不同的预后组。风险值大于等于中位风险值的组命名为高风险组,风险值小于中位风险值的组命名为低风险组。Kaplan-Meier分析显示低风险组患者的生存结局明显优于高风险组患者(P < 0.001;见图 3a)。高风险组5年生存率为11.8%,低风险组为15.7%(见图 3a)。对GILncSig进行ROC曲线分析,得出曲线下面积(AUC)为0.762(见图 3b)。我们根据风险值对训练集中的患者进行排序,观察两个预后相关lncRNA的表达水平、患者体细胞突变数和UBQLN4的表达水平如何随着风险值的增加而变化(见图 3c)。在低风险患者中,风险lncRNA AP001527.2表达水平下调,而保护性lncRNA AC107464.2表达水平上调,与高风险组患者的表达模式相反(见图 3c)。比较高风险组和低风险组患者体细胞突变模式和UBQLN4表达模式,高风险组患者躯体突变的数量与低风险组患者无明显差异(P = 0.078, Mann-Whitney U检验; 见图 3d),此外,UBQLN4在两组患者中表达水平无明显差异(P = 0.28, Mann-Whitney U test; 见图 3e)。

|
图 3 在Train组中识别基因组不稳定性衍生的GILncSig得分用于预测结果 Figure 3 Identifying GILncSig score derived from genomic instability in Train group for results prediction *注:(c)左边为低风险组,右边为高风险组,表达越高或越低,颜色越深(红色上调,蓝色下调), (d) 红色簇代表GU-like组,蓝色代表GS-like组,P=0.078, (e)红色簇代表GU-like组,蓝色代表GS-like组,P=0.28. |
为了评估GILncSig的准确性,在Test组中验证其预后作用。将Train组中得到的GILncSig分值和中位风险值应用到Test组中,将患者分为两组,低风险组72例,高风险组80例(见图 4a)。高风险组和低风险组患者生存率具有明显差异(P < 0.05),与Train组趋势一致。
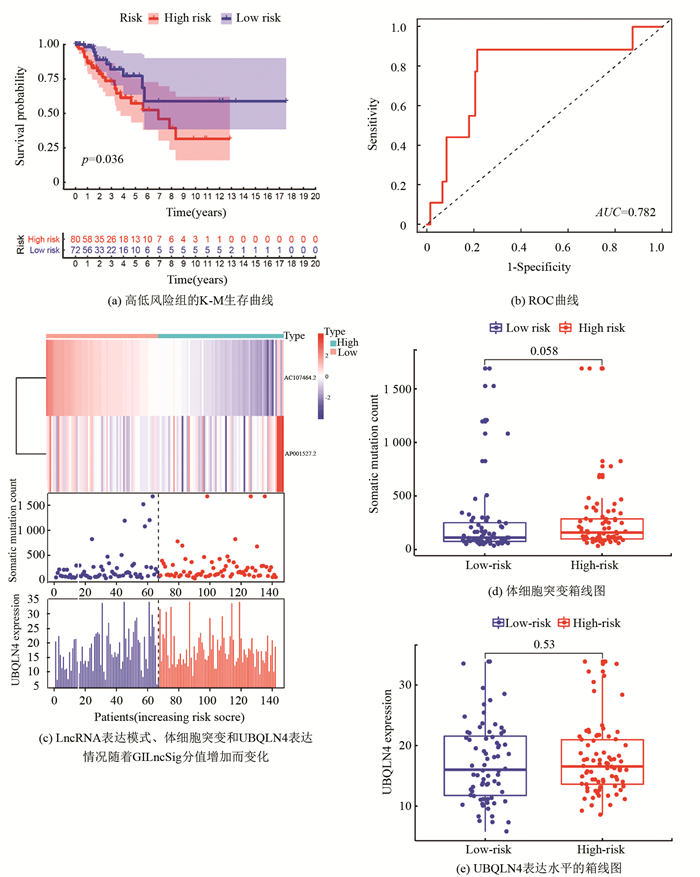
|
图 4 在Test组中GILncSig的独立验证 Figure 4 Independent validation of GILncSig in test group *注:(c) 左边为低风险组,右边为高风险组,表达越高或越低,颜色越深(红色上调,蓝色下调), (d) 红色簇代表GU-like组,蓝色代表GS-like组,P=0.058, (e)红色簇代表GU-like组,蓝色代表GS-like组,P=0.53. |
对GILncSig进行时间依赖性ROC曲线分析,得出曲线下面积(AUC)为0.782 (见图 4b)。随后,根据评分对Test的患者进行排序,观察基因组不稳定性相关lncRNA、患者体细胞突变数和UBQLN4的表达水平随着风险值增加而变化的情况(见图 4c)。与Train组结果相似的是,在低风险患者中,风险lncRNA AP001527.2表达水平下调,保护性lncRNA AC107464.2表达水平上调,而高风险患者中两者表达情况正好相反(见图 4c)。同样,体细胞突变模式在高风险组中和低风险组中无明显差异(P=0.058, 见图 4d),UBQLN4表达模式在两组中也无明显差异(P=0.53, 见图 4e)。
2.4 独立预后分析为了评估GILncSig的预后作用是否独立于常见的临床变量,对年龄、病理分级和GILncSig进行了多变量Cox回归分析。结果显示,在调整年龄、病理分级后,GILncSig与各组的总生存率显著相关(见表 2)。说明GILncSig是与宫颈癌患者的整体生存相关的独立预后因子。
| 表 2 单因素和多因素COX回归分析GILncSig与不同病人组的总生存率 Table 2 Univariate and multivariate COX regression analyses of GILncSig and overall survival in different patient groups |
统计了每个基因在样本中的突变情况,其中发生TTN基因突变的样本数最多。进一步分析显示,在Train组、Test组和TCGA组中,高风险组TTN突变患者的比例与低风险组无明显差异(见图 5a)。进一步检验GILncSig与TTN突变状态相比有更好的预测结果。当GILncSig应用于TTN野生型(TTN-wild)患者时,GILncSig将TTN-wild患者分为TTN Wild/GS-like组和TTN Wild/GU-like组,当GILncSig应用于TTN突变型(TTN-Mutation)患者时,TTN-Mutation患者被GILncSig分为TTN Mutation/GS-like组和TTN Mutation/GU-like组(见图 5b)。GILncSig对TTN Wild/GS-like组、TTN Wild/GU-like组、TTN Mutation/GS-like组和TTN Mutation/GU-like组4种风险组的生存曲线无明显差异(P=0.581)。

|
图 5 GilncSig与TTN体细胞突变之间的关系 Figure 5 Relationship between GilncSig and TTN somatic mutation *注:(a)红色代表存在TTN突变,绿色代表不存在TTN突变. |
在近几十年中,随着HPV疫苗接种和早期联合筛查的应用,宫颈癌的发病率和死亡率有下降趋势,但发展中国家的宫颈癌发病率仍然很高,且患者往往预后不佳[22]。因此,确定可靠的新生物标志物来预测宫颈癌的生存预后至关重要。基因组不稳定性被认为是大多数癌症所共有的特征[23-24]。基因组不稳定性在癌症进展和复发中起着重要的主导作用,表明基因组不稳定性的模式和程度具有重要的诊断和预后意义[25-26]。近几年,lncRNA作为一种新型的ncRNA,已被证实是肿瘤生物学过程的重要组成部分,其在癌症中的异常表达与疾病发生发展密切相关,可能有作为患者预后标志物的潜力[27-29]。随着对lncRNA功能机制研究的不断深入,认识到lncRNA对基因组稳定性也具有至关重要的作用[30-31]。已经做了一些研究,但全基因组识别基因组不稳定性相关的lncRNA以及系统探索其在癌症中的临床意义仍处于初阶阶段。因此,开发了一个结合lncRNA表达和肿瘤突变表型来识别基因组不稳定性相关lncRNA的预测模型。随后我们结合lncRNA表达谱和宫颈癌体细胞突变谱作为研究对象,鉴定了36个新的基因组不稳定性相关的lncRNA。通过对36个与基因组不稳定性相关lncRNA共表达的基因的功能分析,我们的观察发现,与36个lncRNA共表达的基因在2-氧代戊二酸代谢过程和2-氧羧酸代谢通路中富集。2-氧代戊二酸的代谢异常可能会影响2-氧代戊二酸依赖的加氧酶(2OGXs)的活性。2-氧代戊二酸依赖的加氧酶(2OGXs) 的活性改变可能会影响其核酸修复、转录/蛋白生物合成调节等生物功能,并与某些肿瘤的基因组不稳定性相关[32]。接下来,我们研究了基因组不稳定性相关的lncRNA是否可以预测宫颈癌的临床结果,并产生了包含两个基因组不稳定性相关的lncRNA(AC107464.2和AP001527.2) 的预后风险模型(GILncSig)。GILncSig将Train组患者分成两个风险组,两组患者存活率有显著差异,这在Test组中得到了验证。高风险组TTN突变患者的比例与低风险组无明显差异,这说明GILncSig的预后意义与单独的TTN突变状态无明显差异。虽然我们的研究为更好地评估宫颈癌患者的基因组不稳定性和预后提供了重要的见解。此外,GILncSig是基于突变假设的计算框架来识别的,因此,还需要进一步的生物学功能研究,以了解GILncSig在维持基因组不稳定性方面的调节机制。
4 结论提出了一个基于突变假设的计算框架来识别与基因组不稳定性相关的lncRNA,为进一步研究lncRNA在基因组不稳定性中的作用提供了重要的途径和资源。通过将lncRNA表达谱、体细胞突变谱和宫颈癌临床信息结合在一起研究,确定了一个由基因组不稳定性衍生的预后风险模型(GILncSig)作为一个独立的预后标志物来对宫颈癌患者的危险亚组进行分层,在独立的患者队列中成功验证。通过进一步的前瞻性研究,GILncSig可能对宫颈癌患者的基因组不稳定性和制定治疗策略方面具有重要意义。
| [1] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 (  0) 0) |
| [2] |
LIU X, SONG J, ZHANG Y, et al. ASF1B promotes cervical cancer progression through stabilization of CDK9[J]. Cell Death, Disease, 2020, 11(8): 705. DOI:10.1038/s41419-020-02872-5 (  0) 0) |
| [3] |
张科, 杜国波, 谭榜宪. 长链非编码RNA与宫颈癌预后关系研究进展[J]. 医学综述, 2020, 26(2): 274-279. ZHANG Ke, DU Guobo, TAN Bangxian. Research progress in relationship between long non-coding RNA and prognosis of cervical cancer[J]. Medical Recapitulate, 2020, 26(2): 274-279. DOI:10.3969/j.issn.1006-2084.2020.02.013 (  0) 0) |
| [4] |
NEGRINI S, GORGOULIS V G, HALAZONETIS T D. Genomic instability—an evolving hallmark of cancer[J]. Nature Reviews Molecular Cell Biology, 2010, 11(3): 220-228. DOI:10.1038/nrm2858 (  0) 0) |
| [5] |
KOICHI S, SUMIKO O, CHIKAKO T, et al. The genomic damage estimated by arbitrarily primed PCR DNA fingerprinting is useful for the prognosis of gastric cancer[J]. Gastroenterology, 2003, 125(5): 1330-1340. DOI:10.1016/j.gastro.2003.07.006 (  0) 0) |
| [6] |
OTTINI L, FALCHETTI M, LUPI R, et al. Patterns of genomic instability in gastric cancer: clinical implications and perspectives[J]. Annals of Oncology Official Journal of the European Society for Medical Oncology, 2006, 17(S7): vii97. DOI:10.1093/annonc/mdl960 (  0) 0) |
| [7] |
CHRISTINE H, JEFF B, JONATHAN S, et al. Chromosomal instability as a prognostic marker in cervical cancer[J]. BMC Cancer, 2015, 15(1). DOI:10.1186/s12885-015-1372-0 (  0) 0) |
| [8] |
MATTICK J S, RINN J L. Discovery and annotation of long noncoding RNAs[J]. Nature Structural & Molecular Biology, 2015, 22(1): 5. DOI:10.1038/nsmb.2942 (  0) 0) |
| [9] |
KOZIOL M J, RINN J L. RNA traffic control of chromatin complexes[J]. Current Opinion in Genetics & Development, 2010, 20(2): 142. DOI:10.1016/j.gde.2010.03.003 (  0) 0) |
| [10] |
WANG L, ZHOU D, TU J, et al. Exploring the stability of long intergenic non-coding RNA in K562 cells by comparative studies of RNA-Seq datasets[J]. Biology Direct, 2014, 9(1): 1-15. DOI:10.1186/1745-6150-9-15 (  0) 0) |
| [11] |
ANNA S C, YUMI K, YUSUKE Y, et al. The emerging roles of long non-coding RNA in cancer[J]. Cancer Science, 2018, 109. DOI:10.1111/cas.13642 (  0) 0) |
| [12] |
吴茵, 赵兰静, 潘杰. 长链非编码RNA在宫颈癌发生发展及预后中作用的研究进展[J]. 中国肿瘤临床与康复, 2020, 27(10): 1278-1280. WU Yin, ZHAO Lanjing, PAN Jie. Research progress on the role of long non-coding RNAs in the development and prognosis of cervical cancer[J]. Chinese Journal of Clinical Oncology and Rehabilitation, 2020, 27(10): 1278-1280. DOI:10.13455/j.cnki.cjcor.2020.10.34 (  0) 0) |
| [13] |
BARTONICEK N, MAAG J, DINGER M E. Long noncoding RNAs in cancer: mechanisms of action and technological advancements[J]. Molecular Cancer, 2016, 15(1): 43. DOI:10.1186/s12943-016-0530-6 (  0) 0) |
| [14] |
HUARTE M. The emerging role of lncRNAs in cancer[J]. Nature Medicine, 2015, 21(11): 1253. DOI:10.1038/nm.3981 (  0) 0) |
| [15] |
邢瑶, 孟碧, 刘阳晨. lncRNA在宫颈癌预后中的研究进展[J]. 现代肿瘤医学, 2019, 27(11): 2018-2021. XING Yao, MENG Bi, LIU Yangchen. Research progress of lncRNA in prognosis of cervical cancer[J]. Journal of Modern Oncology, 2019, 27(11): 2018-2021. DOI:10.3969/j.issn.1672-4992.2019.11.040 (  0) 0) |
| [16] |
张妍, 李娟, 齐丽荣, 等. 利用生物信息学分析方法识别子宫颈癌患者预后相关长链非编码RNA[J]. 临床与实验病理学杂志, 2020, 36(10): 1222-1226. ZHANG Yan, LI Juan, QI Lirong, et al. Identification of prognostic lncRNA in patients with cervical cancer by bioinformatics analysis[J]. Chinese Journal of Clinical and Experimental Pathology, 2020, 36(10): 1222-1226. DOI:10.13315/j.cnki.cjcep.2020.10.021 (  0) 0) |
| [17] |
底斐瑶, 王一鹤, 底泽亚, 等. 基于TCGA数据库确定宫颈癌预后免疫相关性长链非编码RNA并构建预后模型[J]. 世界最新医学信息文摘, 2020, 20(87): 34-37+39. DI Feiyao, WANG Yihe, DI Zeya, et al. Identifying immune-related lncRNA and constructing prognostic model in cervical cancer patients based on TCGA database[J]. World Latest Medicine Information, 2020, 20(87): 34-37+39. DOI:10.3969/j.issn.1671-3141.2020.87.011 (  0) 0) |
| [18] |
LIU Hong. Linking lncRNA to genomic stability[J]. Science China(Life Sciences), 2016, 59(3): 119-120. DOI:10.1007/s11427-016-5009-6 (  0) 0) |
| [19] |
D'ALESSANDRO G, FAGAGNA F. Long non-coding RNA in the control of genome stability and cancer phenotypes[J]. Non-coding RNA Investigation, 2018, 2: 13-13. DOI:10.21037/ncri.2018.03.01 (  0) 0) |
| [20] |
MUNSCHAUER M, NGUYEN C T, SIROKMAN K, et al. Publisher Correction: The NORAD lncRNA assembles a topoisomerase complex critical for genome stability[J]. Nature, 2018, 563. DOI:10.1038/s41586-018-0584-2 (  0) 0) |
| [21] |
YU G, WANG L G, HAN Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. Omics-A Journal of Integrative Biology, 2012, 16(5): 284-287. DOI:10.1089/omi.2011.0118 (  0) 0) |
| [22] |
ARBYN M, CASTELLSAGUE X, SARAIYA M, et al. Worldwide burden of cervical cancer in 2008[J]. Annals of Oncology, 2011, 22(12). DOI:10.1093/annonc/mdr015 (  0) 0) |
| [23] |
BARTKOVA J, HOREJSI Z, KOED K, et al. DNA damage response as a candidate anti-cancer barrier in early human tumorigenesis[J]. Nature, 2005, 434(7035): 864-870. DOI:10.1038/nature03482 (  0) 0) |
| [24] |
GORGOULIS V, VASSILIOU L, KARAKAIDOS P, et al. Activation of the DNA damage checkpoint and genomic instability in human precancerous lesions[J]. Nature, 2005, 434(7035): 907-913. DOI:10.1038/nature03485 (  0) 0) |
| [25] |
KRONENWETT U, PLONER A, ZETTERBERG A, et al. Genomic instability and prognosis in breast carcinomas[J]. Cancer Epidemiol Biomarkers Preview, 2006, 15(9): 1630-1635. DOI:10.1158/1055-9965.EPI-06-0080 (  0) 0) |
| [26] |
METTU R, WAN Y W, HABERMANN J K, et al. A 12-gene genomic instability signature predicts clinical outcomes in multiple cancer types[J]. The International of Biological Markers, 2010, 25(4): 219-228. DOI:10.5301/JBM.2010.6079 (  0) 0) |
| [27] |
RAJNISH A G, NILAY S, KEVIN C W, et al. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis[J]. Nature, 2010, 464(7291): 1071-1076. DOI:10.1038/nature08975 (  0) 0) |
| [28] |
HUARTE M. Large non-coding RNAs: missing links in cancer?[J]. Human Molecular Genetics, 2010, 19(R2): 152-61. DOI:10.1093/hmg/ddq353 (  0) 0) |
| [29] |
PRENSNER J R, IYER M K, BALBIN O A, et al. Transcriptome sequencing across a prostate cancer cohort identifies PCAT-1, an unannotated lincRNA implicated in disease progression[J]. Nature Biotechnology, 2011. DOI:10.1038/nbt.1914 (  0) 0) |
| [30] |
MUNSCHAUER M, NGUYEN C T, SIROKMAN K, et al. The NORAD lncRNA assembles a topoisomerase complex critical for genome stability[J]. Nature, 2018, 561(7721): 132-136. DOI:10.1038/s41586-018-0453-z (  0) 0) |
| [31] |
WANG L H, LEI J, AN X, et al. GUARDIN is a p53-responsive long non-coding RNA that is essential for genomic stability[J]. Nature Cell Biology, 2018, 20(4): 492-502. DOI:10.1038/s41556-018-0066-7 (  0) 0) |
| [32] |
ISLAM M S, LEISSING T M, CHOWDHURY R, et al. 2-Oxoglutarate-dependent oxygenases[J]. Annual Review of Biochemistry, 2018, 87: 585-620. DOI:10.1146/annurev-biochem-061516-044724 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


