2. 上海市分子影像重点实验室,上海 201200
2. Shanghai Key Laboratory of Molecular Imaging, Shanghai 201200, China
肺癌是全球死亡率最高的癌症之一,非小细胞肺癌(Non-small cell lung cancer,NSCLC)是肺癌中最常见的类型,约占所有肺癌病例的80%[1]。肺腺癌(Lung adenocarcinoma,LUAD)是非小细胞肺癌的主要亚型之一,对全球不吸烟者而言是致死率最高的疾病[2-3]。由于LUAD在早期容易转移复发,LUAD患者的预后效果很差,平均5年生存率低于20%[4]。在临床实践中,肿瘤分期系统已广泛应用于癌症患者的指导治疗和预后评估。然而,预后的判断通常只基于固有的解剖学信息,由于肺腺癌的异质性,很难预测疾病的发展。因此,迫切需要寻找有效的预后生物标志物来帮助临床医生做出准确的肺腺癌诊断,预测临床结果,为个体化医学提供参考。
过去几年里,研究发现自噬在肿瘤的发生过程中发挥了重要的作用[6-9]。自噬是一个复杂的生理病理过程,自噬的溶酶体降解功能在细胞生理学中起着至关重要的作用,如适应代谢应激、清除危险物质(如蛋白质聚集体、受损或老化的细胞器、细胞内病原体)、细胞分化和发育过程中的更新等[10]。在癌症中,自噬具有双重作用,它既能够抑制良性肿瘤的生长,也可以促进晚期癌症的发展[11]。目前,许多研究小组已经确定把自噬作为癌症治疗的潜在靶点。
本项研究构建了一个结合多个自噬相关基因和临床参数的模型来预测LUAD患者的预后。从TCGA数据库的LUAD数据中筛选出表达具有显著差异的自噬相关基因,对差异自噬相关基因进行单因素Cox回归分析来确定与LUAD患者生存相关的候选基因,然后使用lasso回归模型筛选出预后相关基因,对预后相关基因进行多因素Cox分析,构建风险评分模型,并对风险评分模型进行内部验证和外部验证。最后将风险评分与临床参数结合,构建了预测患者生存概率的列线图模型,使用一致性指数(Concordance index, C-index)、校准曲线和ROC曲线来评估模型的性能。
1 材料和方法 1.1 数据收集和数据预处理在人类自噬专用数据库HADb (http://www.autophagy.lu/)、ARN数据库(http://autophagyregulation.org)、自噬数据库(http://www.tanpaku.org/autophagy/index.html)上下载了自噬相关基因共1 417个。从TCGA(https://portal.gdc.cancer.gov/)下载LUAD的COUNT数据和FPKM数据各585例,包含526例肺腺癌样本和59例癌旁正常肺组织。从UCSC Xena(https://xenabrowser.net/)下载TCGA-LUAD的生存信息641例和临床数据706例。对于TCGA数据,过滤生存信息、肿瘤分期信息、年龄、性别和复发信息不完整的样本,保留了TCGA的417例肺腺癌样本和48例正常样本。在TCGA数据中筛选出自噬相关基因的信息,并将癌症样本(n=417)随机分配为训练组和测试组,比例为1:1。
同时,在GEO(https://www.ncbi.nlm.nih.gov/geo/)数据库下载了GSE50081数据集用于外部验证,该数据集包括了127例肺腺癌样本和54例正常样本。
1.2 预后相关基因的筛选使用“limma”包对自噬相关基因进行差异分析,差异基因筛选标准为:|logFC|>1.5,P < 0.05。
对表达具有显著差异的自噬相关基因使用单因素Cox比例风险回归分析筛选出候选基因,筛选阈值为:风险比HR≠1,p < 0.05。
Lasso是一种高维预测回归方法,并已被广泛应用于高维数据生存分析的Cox比例风险回归模型中[12]。为了进一步筛选出与LUAD生存显著相关的基因,在训练集(n = 209)中使用Lasso回归模型对候选基因进行筛选,并进行十折交叉验证,以确定最佳的预后相关基因。
1.3 多基因预后标志的建立和评估对预后相关基因进行多因素Cox比例风险回归分析,获得预后相关基因的回归系数。然后,采用predict函数将基因的表达水平和回归系数进行组合算出每个患者的风险评分。
使用“survminer”包计算出最优cutoff值,以cutoff为临界值,将训练组分为高风险组和低风险组。为了确定风险评分在预测肺腺癌患者临床预后中的作用,采用对数秩检验对训练组进行了生存分析,比较高风险组和低风险组之间的生存差异。绘制了与时间相关的ROC曲线来进一步评估风险评分的预后性能,并计算了其3年和5年的AUC值。
此外,为了探讨多基因预后标志在其他临床参数中的诊断能力,进行了一项分层分析,以cutoff值为分界点进行分组,使用Kaplan-Meier曲线比较了stage亚组、年龄、性别亚组中高低风险组的生存差异。
1.4 多基因预后标志的验证使用内部验证集(n=208),外部验证集GSE50081(n=127),以及全集(n=417)来验证风险评分的预测能力和适用性。在验证集中,使用训练集中获得的回归系数计算每个样本的风险评分,然后根据cutoff值将患者分为高风险组和低风险组,采用对数秩检验进行生存分析,绘制与时间相关的ROC曲线。
1.5 列线图的构建和验证对风险评分和一些临床参数(stage、T期、N期、年龄、性别、复发)进行了单因素Cox回归分析,以比较风险评分与临床参数的预后能力。然后,使用多因素Cox回归模型来确定风险评分是否具有临床独立性,其中,在单因素Cox回归分析中具有显著统计学差异(p < 0.05)的临床参数也被纳入多因素Cox回归模型中。
基于上述单因素和多因素Cox回归分析,筛选出具有统计学差异的参数作为独立预后参数,用于列线图的构建,以预测患者3年、5年的生存概率。
为了评价模型的预测能力,计算出列线图模型的C-index,并绘制其3年、5年的ROC曲线,同时绘制了3年时stage、风险评分和列线图的ROC曲线,比较三者的预测能力。然后,使用校准曲线,通过500次重采样,以3年、5年的观察速率来可视化列线图的性能,列线图的预测结果和实际结果都能够在校准曲线中进行比较,其中,45°线为最佳预测结果。在内部验证集和全集中使用上述相同的办法来验证结果。
2 结果 2.1 预后相关基因的筛选在HADb数据库、ARN数据库和自噬数据库中共下载了1 417个自噬相关基因,其中938个基因在TCGA数据中有表达。对938个自噬基因进行差异分析,获得了38个上调基因和44个下调基因(见图 1a),筛选条件为|logFC|>1.5,P < 0.05。
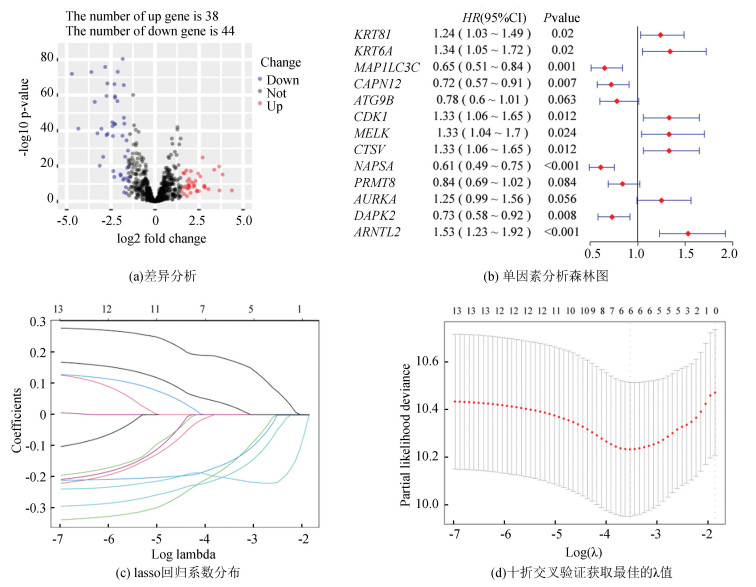
|
图 1 回归分析筛选与LUAD预后相关的自噬相关基因 Figure 1 Regression analysis for screening autophagy-related genes associated with LUAD prognosis |
在全集中,对差异基因进行单因素Cox回归分析,发现有13个候选基因与肺腺癌生存相关(见图 1b)。为进一步确定与LUAD患者预后相关的基因,使用“glmnet”R包对候选基因进行了LASSO回归分析以及十折交叉验证,其结果显示,当λmin=0.029时,模型性能达到最佳,此时筛选出了6个预后相关基因(见图 1c, 1d),即ARNTL2、NAPSA、ATG9B、CAPN12、MAP1LC3C、KRT81,这些基因中有4个(NAPSA、ATG9B、CAPN12、MAP1LC3C)的风险比小于1,表明它们的低表达与预后不良有关,而ARNTL2和KRT81的风险比大于1,表明它们的过度表达与低生存率有关。
2.2 多基因预后标志的建立和评估对6个预后相关基因进行多因素Cox回归分析(见图 2),然后,使用predict函数结合多基因的回归系数和表达量构建风险评分,通过“survminer”R包获取风险评分的最优cutoff值,以cutoff值为分界点,将患者分为高风险组和低风险组,并展示了训练集中患者的生存状态和6个预后相关基因的热图(见图 3a)。对训练组进行生存分析,结果显示,与低风险组相比,高风险组的预后结果更差(见图 3b)。然后,我们构建了一个与时间相关的ROC曲线(见图 3c),其3年、5年的AUC值分别为0.852、0.868,这表明这个多基因预后标志具有较好的预测能力。
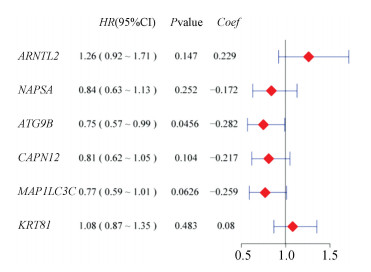
|
图 2 预后相关基因的多因素Cox回归分析 Figure 2 Multivariate Cox regression analysis of prognosis-associated genes |
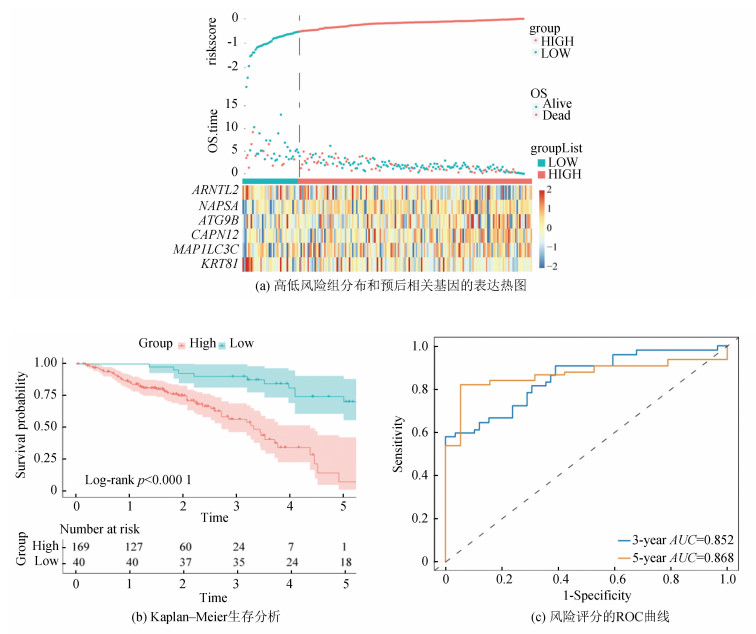
|
图 3 训练集中多基因特征的预后分析 Figure 3 Prognostic analysis of polygenic traits in training set |
此外,对stage、年龄和性别进行了风险分层,以cutoff值为分界点,将训练组的患者分为高风险组和低风险组,进行Kaplan-Meier生存分析(见图 4)。在stage Ⅰ/Ⅱ、stageⅢ/Ⅳ、男性、女性、年龄大于65岁和年龄小于65岁的亚组中,高风险组的生存率都显著低于低风险组(p < 0.05)。
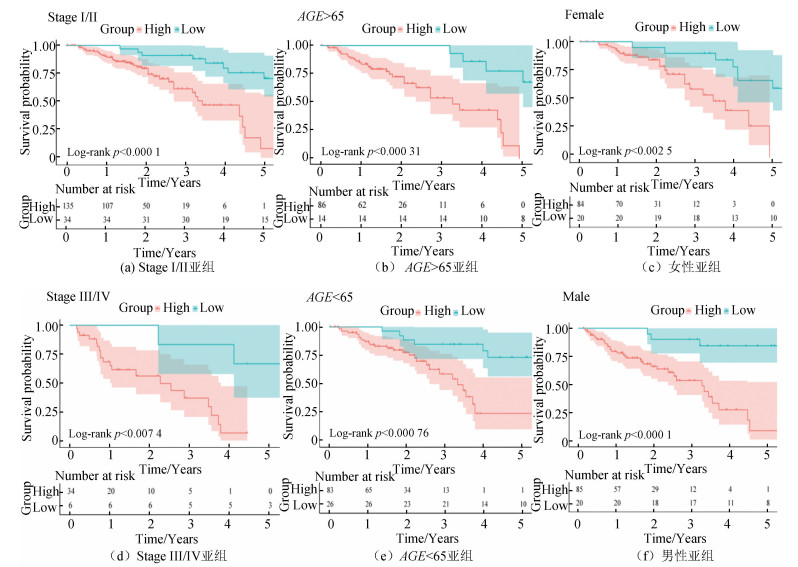
|
图 4 风险评分在不同亚组中的生存分析 Figure 4 Survival analysis of riskscore in different subgroups |
使用内部测试集(n=208)、外部测试集(n=127)和全集(n=417)来验证风险评分的预测能力。与训练集中的结果一致,测试集的生存分析曲线都显示高风险组的预后结果比低风险组的差(见图 5a-5c)。ROC曲线显示,内部测试集的3年、5年AUC值为0.863、0.938(见图 5d),外部测试集的3年、5年AUC值为0.939、0.852(见图 5e),全集的3年、5年AUC值为0.861、0.905(见图 5f),以上结果都显示风险评分在预测LUAD患者的预后方面表现良好。
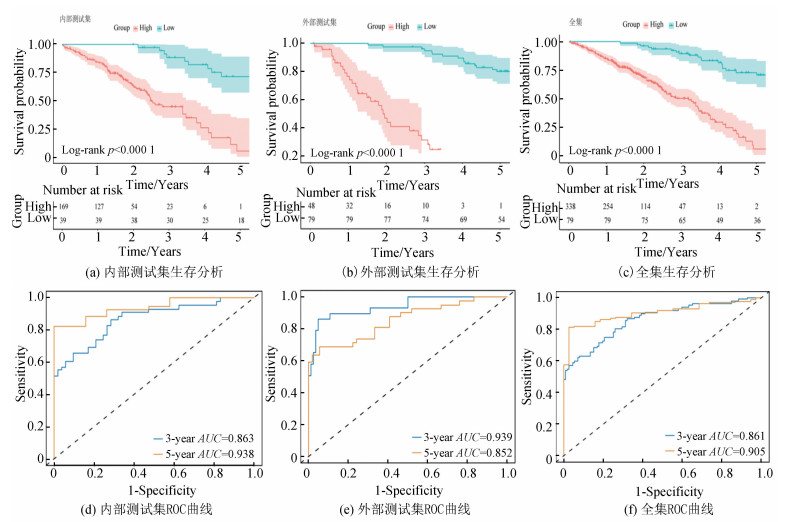
|
图 5 风险评分的内部验证和外部验证 Figure 5 Internal validation and external validation of riskscore |
对风险评分和一些临床参数(stage、T期、N期、年龄、性别、复发)进行了单因素和多因素Cox比例风险回归分析,其结果显示风险评分可以作为预测LUAD预后的独立参数,而在传统临床参数中,stage和复发也可以作为独立预后参数(见图 6a, 6b)。我们将传统临床风险因素和风险评分相结合,构建一种能够有效预测患者3年、5年生存率的列线图(见图 6c)。列线图的C-index指数为0.807,表明列线图有较好的预测能力。校准曲线显示,列线图的预测结果与实际结果较为一致(见图 7a)。ROC曲线显示,列线图 3年、5年的AUC值分别为0.898、0.88(见图 7d)。三年时,列线图生存的AUC值远高于风险评分模型和stage的AUC值(见图 7g),这表明列线图可能是预测LUAD预后生存的最佳方式。
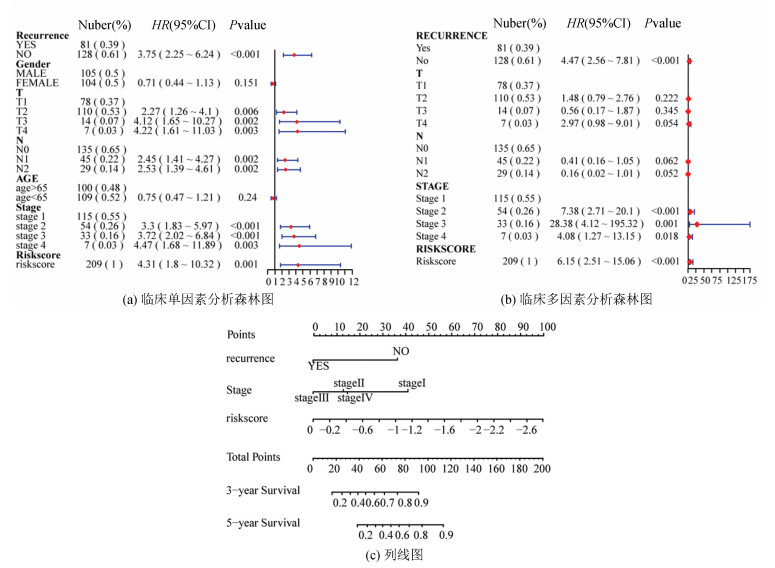
|
图 6 临床单、多因素Cox分析以及列线图的构建 Figure 6 Clinical univariate and mutlivariate Cox analyses and construction of nomogram |
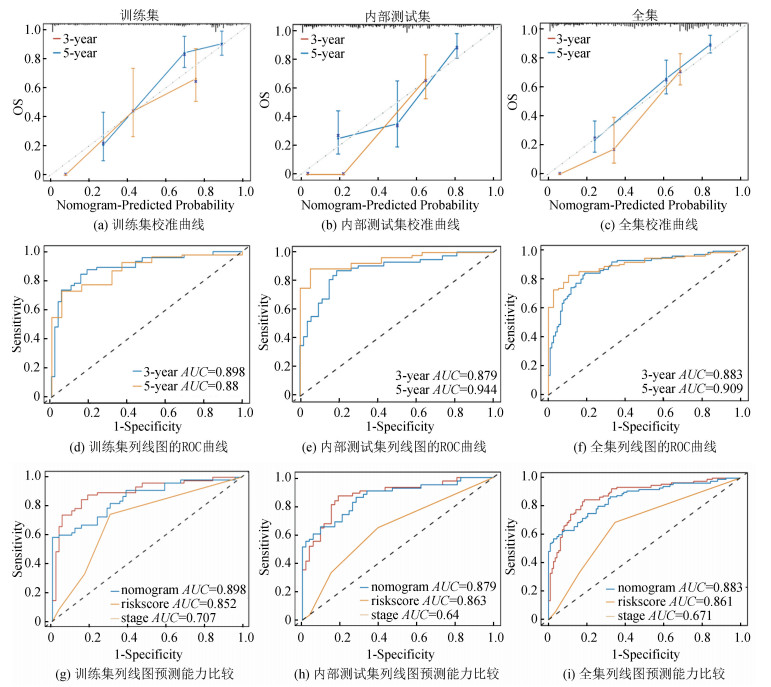
|
图 7 列线图预测LUAD生存率的性能以及列线图、风险评分和Stage预测能力的比较 Figure 7 Performance of nomogram for predicting survival in LUAD and comparison of prediction ability of nomogram, riskscore, and Stage |
为了验证列线图的预测价值,使用内部测试集(n=208)和全集(n=417)来检验上述的发现。内部测试集和全集的列线图的C-Index指数分别为0.8和0.792,校准曲线也显示两个测试集列线图的3年、5年生存预测结果与实际结果有良好的一致性(见图 7b, 7c)。列线图的ROC曲线显示,两个测试集具有较好的预测准确度(见图 7e, 7f)。同时,在3年期的生存预测中,列线图无论在哪组都比风险评分和stage有更好的预测准确度(见图 7h, 7i)。
3 讨论自噬是高度保守的代谢过程,在循环代谢能量以维持细胞内稳态方面起着关键作用[13]。有研究表明了多个自噬相关基因与肺癌的发生发展密切相关[14-16],因此,决定把自噬相关基因作为肺腺癌治疗的潜在靶点。通过对TCGA肺腺癌数据中的938个自噬相关基因进行差异分析,获得了82个差异基因,然后对差异自噬基因进行单因素Cox回归分析,筛选出了13个与LUAD生存相关的候选基因,然后使用lasso回归进一步筛选出6个与LUAD预后相关的基因。通过多因素Cox回归分析获得每个预后相关基因的回归系数,通过每个基因的表达量和回归系数计算出每个患者的风险评分。在训练集中,风险评分能够很好地将高风险患者和低风险患者区分开,并且其预测性能也在内部、外部测试集中得到了验证。同时,在分层分析中,风险评分在stage,年龄和性别亚组中的风险分层表现也很好,这意味着此风险评分模型可以根据亚组将LUAD患者分为高低风险组,帮助临床医生进行临床决策。
用于构建风险评分的6个基因包括ARNTL2、NAPSA、ATG9B、CAPN12、MAP1LC3C和KRT81。ARNTL2属于PAS超家族,在昼夜节律和缺氧过程中起着重要的作用,其在乳腺癌、肾细胞癌等人类恶性肿瘤中具有致癌作用[17-19],目前已有研究报道ARNTL2的高表达与肺腺癌的低生存期相关,并且能够影响肺腺癌的免疫浸润水平[20-21]。NAPSA是天冬氨酸肽酶,其编译的蛋白酶能够参与肺表面活性物质蛋白B在肺中的蛋白水解过程,目前它已被证实是肺腺癌的生物标记物,并且已被用作识别原发性肺腺癌的免疫组化染色剂[22-24]。ATG9B是自噬相关基因,在自噬过程中起调节作用,与肝癌[25]、肾细胞癌[26]、胃癌[27]等多种癌症的发生发展有关,但其在肺腺癌中的作用还尚未阐明。CAPN12是一种钙蛋白酶,钙蛋白酶能够调节多种细胞生理过程,包括细胞增殖、细胞迁移、细胞侵袭、细胞自噬等,各种癌症的发病机制也需要钙蛋白酶系统,其可能起到促进癌症发展的作用,最新研究也确定了CAPN12是新的结直肠癌易感基因[28-30]。MAP1LC3C是自噬蛋白ATG8的同源物,被用作自噬机制的生物标志物,有研究发现,MAP1LC3C介导了MET/HGF-RTK信号通道在癌症中的作用,MAP1LC3C和MET复合物招募HGF并且激活MET-RTK信号通路从而进行自噬降解,进而影响肿瘤转移[31]。在肺癌方面,有研究证明其与肺腺癌氧化磷酸化过程十分相关[32]。KRT81是一种角蛋白,相关研究发现其与肺腺癌肿瘤转移相关[33]。上述结果都表明风险评分模型具有潜在的临床应用价值。
最后,单因素和多因素Cox回归分析表明,风险评分可以作为预后评估的独立因素。为了提高风险评分的预测能力,结合临床参数和风险评分构建了一个基于多基因预后标志的列线图来预测患者生存率。通过比较,列线图的预测性能高于风险评分和stage的预测性能,并在验证集中得到同样的结果。这表明,与单一的临床参数相比,列线图模型更能帮助临床医生预测LUAD患者的生存状态,并为临床医生提供治疗指导。然而,我们的研究还有一些不足之处,我们的数据只包含TCGA数据库的mRNA数据,未来还可以从单核苷酸多态性、拷贝数变异数据、DNA甲基化等突变数据中进一步分析这6种新的生物标志物是否与上述突变相关。
4 结论基于6个基因的多基因预后标志来预测LUAD患者的生存风险,在训练集和测试集中都表现出良好的准确率,并且独立于其他临床特征。然后,结合多基因预后标志和临床特征构建了列线图模型以预测LUAD患者的预后生存率,与单一临床特征相比,列线图模型具有更好的预测性能。因此,这6个基因很可能是LUAD的潜在生物标志物,基于多基因预后标志和临床特征的列线图模型很有可能用于评估LUAD患者的生存率,并帮助临床医生进行个体化治疗的临床决策。
| [1] |
YOU S, GAO L. Identification of NMU as a potential gene conferring alectinib resistance in non-small cell lung cancer based on bioinformatics analyses[J]. Gene, 2018, 678: 137-142. DOI:10.1016/j.gene.2018.08.032 (  0) 0) |
| [2] |
WU C, XU B, ZHOU Y, et al. Correlation between serum IL-1β and miR-144-3p as well as their prognostic values in LUAD and LUSC patients[J]. Oncotarget, 2016, 7(52): 85876. DOI:10.18632/oncotarget.13042 (  0) 0) |
| [3] |
YAN L, JIAO D, HU H, et al. Identification of lymph node metastasis-related microRNAs in lung adenocarcinoma and analysis of the underlying mechanisms using a bioinformatics approach[J]. Experimental Biology and Medicine, 2017, 242(7): 709-717. DOI:10.1177/1535370216677353 (  0) 0) |
| [4] |
LIN J J, CARDARELLA S, LYDON C A, et al. Five-year survival in EGFR-mutant metastatic lung adenocarcinoma treated with EGFR-TKIs[J]. Journal of Thoracic Oncology, 2016, 11(4): 556-565. DOI:10.1016/j.jtho.2015.12.103 (  0) 0) |
| [5] |
HOU H, ZHANG C, QI X, et al. Distinctive targetable genotypes of younger patients with lung adenocarcinoma: A cBioPortal for cancer genomics data base analysis[J]. Cancer Biology & Therapy, 2020, 21(1): 26-33. DOI:10.1080/15384047.2019.1665392 (  0) 0) |
| [6] |
ONRATI A V, DYCZYNSKI M, OJHA R, et al. Targeting autophagy in cancer[J]. Cancer, 2018, 124(16): 3307-3318. DOI:10.1002/cncr.31335 (  0) 0) |
| [7] |
WHITE E, MEHNERT J M, CHAN C S. Autophagy, metabolism, and cancer[J]. Clinical Cancer Research, 2015, 21(22): 5037-5046. DOI:10.1158/1078-0432.CCR-15-0490 (  0) 0) |
| [8] |
LI Y J, LEI Y H, YAO N, et al. Autophagy and multidrug resistance in cancer[J]. Chinese Journal of Cancer, 2017, 36(1): 1-10. DOI:10.1186/s40880-017-0219-2 (  0) 0) |
| [9] |
夏少怀, 李文才, 夏学巍, 等. 自噬基因CTSL表达与胶质母细胞瘤患者预后相关[J]. 生物信息学, 2021, 19(2): 136-148. XIA Shaohuai, LI Wencai, XIA Xuewei, et al. Autophagy gene CTSL expression correlates with prognosis of patients with glioblastoma[J]. Chinese Journal of Bioinformatics, 2021, 19(2): 136-148. DOI:10.12113/202001011 (  0) 0) |
| [10] |
LEVINE B, KROEMER G. Biological functions of autophagy genes: A disease perspective[J]. Cell, 2019, 176(1/2): 11-42. DOI:10.1016/j.cell.2018.09.048 (  0) 0) |
| [11] |
ONORATI A V, DYCZYNSKI M, OJHA R, et al. Targeting autophagy in cancer[J]. Cancer, 2018, 124(16): 3307-3318. DOI:10.1002/cncr.31335 (  0) 0) |
| [12] |
ZHANG H H, LU W. Adaptive Lasso for Cox's proportional hazards model[J]. Biometrika, 2007, 94(3): 691-703. DOI:10.1093/biomet/asm037 (  0) 0) |
| [13] |
SAHIB Z, JIN S H, MAHOUD A, et al. Cross talk between autophagy and oncogenic signaling pathways and implications for cancer therapy[J]. Biochimica et Biophysica Acta (BBA) - Reviews on Cancer, 2021, 1876(1): 188565. DOI:10.1016/j.bbcan.2021.188565 (  0) 0) |
| [14] |
ZHANG P, LING L, ZHENG Z, et al. ATG7-dependent and independent autophagy determine the type of treatment in lung cancer[J]. Pharmacological Research, 2021, 163: 105324. DOI:10.1016/j.phrs.2020.105324 (  0) 0) |
| [15] |
CAI J, LI R, XU X, et al. CK1α suppresses lung tumour growth by stabilizing PTEN and inducing autophagy[J]. Nature Cell Biology, 2018, 20(4): 465-478. DOI:10.1038/s41556-018-0065-8 (  0) 0) |
| [16] |
YAN R, JIANG Y, LAI B, et al. The positive feedback loop FOXO3/CASC11/miR-498 promotes the tumorigenesis of non-small cell lung cancer[J]. Biochemical and Biophysical Research Communications, 2019, 519(3): 518-524. DOI:10.1016/j.bbrc.2019.08.136 (  0) 0) |
| [17] |
HOGENESCH J B, GU Y Z, MORAN S M, et al. The basic helix-loop-helix-PAS protein MOP9 is a brain-specific heterodimeric partner of circadian and hypoxia factors[J]. Journal of Neuroscience, 2000, 20(13): RC83-RC83. DOI:10.1523/JNEUROSCI.20-13-j0002.2000 (  0) 0) |
| [18] |
HA N H, LONG J, CAI Q, et al. The circadian rhythm gene Arntl2 is a metastasis susceptibility gene for estrogen receptor-negative breast cancer[J]. PLoS Genetics, 2016, 12(9): e1006267. DOI:10.1371/journal.pgen.1006267 (  0) 0) |
| [19] |
WANG S, MA X, YING Y, et al. Upregulation of ARNTL2 is associated with poor survival and immune infiltration in clear cell renal cell carcinoma[J]. Cancer Cell International, 2021, 21(1): 1-13. DOI:10.1186/s12935-021-02046-z (  0) 0) |
| [20] |
SONG C, WU Z, WANG Q, et al. A combined two-mRNA signature associated with PD-L1 and tumor mutational burden for prognosis of lung adenocarcinoma[J]. Frontiers in Cell and Developmental Biology, 2021, 9(1): 1-14. DOI:10.3389/fcell.2021.634697 (  0) 0) |
| [21] |
SUN S, GUO W, WANG Z, et al. Development and validation of an immune-related prognostic signature in lung adenocarcinoma[J]. Cancer Medicine, 2020, 9(16): 5960-5975. DOI:10.1002/cam4.3240 (  0) 0) |
| [22] |
GIORDANO G, CAMPANINI N, VAROTTI E. Immunohistochemical expression of Napsin A in normal human fetal lungs at different gestational ages and in acquired and congenital pathological pulmonary conditions[J]. Virchows Archiv, 2020, 477(4): 557-563. DOI:10.1007/s00428-020-02809-5 (  0) 0) |
| [23] |
SALAMA M E M. Role of Napsin A and survivin immunohistochemical expression in bronchogenic adenocarcinoma[J]. Asian Pacific Journal of Cancer Prevention(APJCP), 2020, 21(11): 3345. DOI:10.31557/APJCP.2020.21.11.3345 (  0) 0) |
| [24] |
ROBERTS E A, MORRISON L E, BEHMAN L J, et al. Chromogenic immunohistochemical quadruplex provides accurate diagnostic differentiation of non-small cell lung cancer[J]. Annals of Diagnostic Pathology, 2020, 45: 151454. DOI:10.1016/j.anndiagpath.2019.151454 (  0) 0) |
| [25] |
WANG N, TAN H Y, LI S, et al. Atg9b deficiency suppresses autophagy and potentiates endoplasmic reticulum stress-associated hepatocyte apoptosis in hepatocarcinogenesis[J]. Theranostics, 2017, 7(8): 2325. DOI:10.7150/thno.18225 (  0) 0) |
| [26] |
MA Z, QI Z, SHAN Z, et al. The role of CRP and ATG9B expression in clear cell renal cell carcinoma[J]. Bioscience Reports, 2017, 37(6): BSR20171082. DOI:10.1042/BSR20171082 (  0) 0) |
| [27] |
CHEN J F, WU P, XIA R, et al. STAT3-induced lncRNA HAGLROS overexpression contributes to the malignant progression of gastric cancer cells via mTOR signal-mediated inhibition of autophagy[J]. Molecular Cancer, 2018, 17(1): 1-16. DOI:10.1186/s12943-017-0756-y (  0) 0) |
| [28] |
GRIEVE S, GAO Y, HALL C, et al. Calpain genetic disruption and HSP90 inhibition combine to attenuate mammary tumorigenesis[J]. Molecular and Cellular Biology, 2016, 36(15): 2078-2088. DOI:10.1128/MCB.01062-15 (  0) 0) |
| [29] |
NIAN H, MA B. Calpain-calpastatin system and cancer progression[J]. Biological Reviews, 2021, 96(3): 961-975. DOI:10.1111/brv.12686 (  0) 0) |
| [30] |
GUO X, LIN W, WEN W, et al. Identifying novel susceptibility genes for colorectal cancer risk from a transcriptome-wide association study of 125, 478 subjects[J]. Gastroenterology, 2021, 160(4): 1164-1178. DOI:10.1053/j.gastro.2020.08.062 (  0) 0) |
| [31] |
BELL E S, COELHO P P, PARK M. LC3C mediates selective autophagy of the MET RTK, inhibiting cancer cell invasion[J]. Autophagy, 2020, 16(5): 959-961. DOI:10.1080/15548627.2020.1728099 (  0) 0) |
| [32] |
XU Z, WU Z, ZHANG J, et al. Development and validation of an oxidative phosphorylation-related gene signature in lung adenocarcinoma[J]. Epigenomics, 2020, 12(15): 1333-1348. DOI:10.2217/epi-2020-0217 (  0) 0) |
| [33] |
YAO X, ZHANG H, TANG S, et al. Bioinformatics analysis to reveal potential differentially expressed long non-coding RNAs and genes associated with tumour metastasis in lung adenocarcinoma[J]. OncoTargets and Therapy, 2020, 13: 3197. DOI:10.2147/OTT.S242745 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


