肺癌是近年来全球范围内发病率和死亡率最高的恶性疾病[1],其中85%以上为非小细胞肺癌(NSCLC)[2], 包括肺腺癌(LUAD),肺鳞状细胞癌(LUSC)和大细胞肺癌(LCLC),以及其他不常见的类型,其中肺腺癌最常见。目前,在肺腺癌的分子病理学、临床肿瘤学、靶向治疗等研究方面取得了较好进展,但肺腺癌患者的死亡率没有显著降低[3-4]。因此,需要寻找肺腺癌的早期诊断生物标志以提高患者的生存率。
近年,肿瘤与免疫的相关性受到越来越多的重视,肿瘤中免疫细胞浸润程度与肿瘤生长、进展和患者结局有关,其不仅对患者的生存具有预测价值,还可影响肿瘤的治疗效果[5-6]。肺癌、乳腺癌等实体肿瘤组织中存在免疫细胞浸润,免疫细胞浸润类型与这些实体肿瘤的临床特征有较强的相关性且免疫细胞浸润情况可用于肿瘤风险分层[7-9],免疫细胞包括B细胞,NK细胞、T细胞、DC细胞等等,而这些细胞通常会表达一些特定基因。
从TCGA和GEO数据库中下载肺腺癌mRNA表达数据,用生物信息学方法筛选肺腺癌差异表达基因,对差异基因进行系统性分析,并利用Cibersort计算肺腺癌和正常肺组织样本中不同种类免疫细胞的浸润程度,探讨肺腺癌早期诊断的生物标志物,为肺腺癌的靶向治疗研究提供新思路。
1 材料与方法 1.1 数据下载通过GEO(https://www.ncbi.nlm.nih.gov/GEO31210)和TCGA数据库(https://portal.gdc.cancer.gov/)下载肺腺癌基因表达及临床病理数据。包括mRNA和clinical;GSE31210数据集基于GPL570([HG-U133_Plus_2] Affymetrix Human Genome U133 Plus 2.0 Array)平台,包括226例肺腺癌肿瘤样本和20例正常肺组织样本;TCGA数据集包括526例肺腺癌样本和59例正常肺组织样本。
1.2 差异基因筛选(DEGs)利用R语言limma包,以P < 0.05及|log 2 FC|>2为条件,筛选正常组织以及肺腺癌样本之间的差异表达基因[10],再将GEO和TCGA的差异基因取交集,得到234个差异基因,使用(http://PSB.ugent.be/webtools/Venn/)在线绘制Venn图,然后用R语言ggscatter包绘制差异基因的火山图。
1.3 GO和KEGG富集分析利用DAVID网站(https://david.ncifcrf.gov/tools.jsp,对DEGs进行GO注释及KEGG富集分析,研究DEGs的生物功能,包括生物过程(BP)、分子功能(MF)和细胞成分(CC);KEGG用于通路富集分析,P<0.05和FDR<0.05被标记为有效项。
1.4 PPI网络构建及hub gene基因的选择通过STRING数据库(https://string-db.org/)构建PPI网络,Cytoscape软件将PPI网络可视化[11],并利用cytoHubba插件选择前20个基因作为hub基因。
1.5 预后生存分析按照表达量高低将肺腺癌样本分为: 高表达和低表达两组,利用R语言survival包在GSE31210和TCGA数据库里分别做预后生存分析;并使用卡普兰-迈耶(Kaplan-Meier)(http://kmplot.com/analysis/index.php) 在线工具对20个关键基因进行生存分析,并对有预后价值的基因用GraphPad Prism 5软件绘制生存图。
1.6 诊断生物标志物的筛选用R语言pROC包绘制肺腺癌预后生存分析中有统计学意义的基因的ROC曲线[12],并根据AUC值对肺腺癌有诊断价值的hub基因进行评估,按AUC>0.7筛选肺腺癌诊断生物标志物。
1.7 GSEA基因富集分析为提高分析结果的准确性,采用GSEA软件(https://www.gsea-msigdb.org/gsea/index.jsp)分析已筛选的肺腺癌诊断生物标志物等基因是否在所选数据集中富集分子通路[13-14],计算富集分数并进行显著性检验分析。
1.8 免疫细胞浸润的评估为了评估肺腺癌中免疫细胞浸润情况,以及筛选的肺腺癌诊断生物标志物等基因表达与肺腺癌组织中免疫细胞浸润情况间的关系,用CIBERSORT算法(https://cibersort.stanford.edu/)对GSE31210数据进行分析,根据P<0.05筛选合适的样本并计算样本中每种免疫细胞的百分比,用ggplot2包绘制22种免疫细胞浸润可视化小提琴图,并分析其22种免疫细胞浸润的差异。
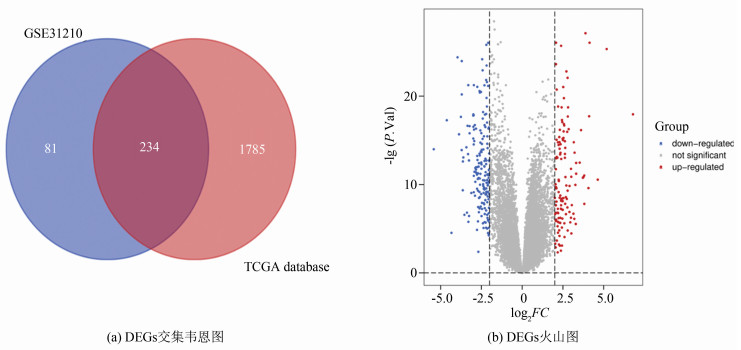
2 结果分析 2.1 肺腺癌差异表达基因(DEGs)结果显示:TCGA数据库中肺腺癌差异基因有2 019个,其中有1 195个表达上调,824个表达下调;GEO数据库中肺腺癌差异基因为315个,129个上调,186个下调;由TCGA和GEO共得到234个肺腺癌差异表达基因DEGs(见图 1)。
|
图 1 TCGA和GEO数据库中LUAD差异基因的筛选 Figure 1 Screening of LUAD differential genes in TCGA and GEO databases 注:(a)中蓝色为GEO数据集DEGs,粉色为TCGA数据集DEGs;(b)中红色代表上调基因,蓝色代表下调基因;横坐标表示倍数变化,纵坐标表示P值。 |
GO分析发现,DEGs在分子功能(MF)方面主要富集在:血清型內肽酶活性、氧运输功能、肝素结合、金属内肽酶活性;在细胞组分(CC)方面主要富集在:质膜的组成成分、质膜、细胞外基质、胞外、细胞黏附等;在生物过程(BP)方面主要富集在胶原代谢、蛋白水解、血小板脱粒、免疫反应等(见图 2a)。KEGG富集分析发现DEGs主要涉及免疫、蛋白、胶原、细胞外成分等一系列与微环境相关的通路,如:PPAR信号通路、与疟疾、补体和凝血级联反应、PI3K AKT信号通路、病毒蛋白与细胞因子和细胞因子受体的相互作用通路, 以及蛋白质消化和吸收相互作用通路、趋化因子信号通路及细胞周期信号通路(见图 2b)。

|
图 2 肺腺癌差异基因的功能富集分析 Figure 2 Functional enrichment analysis of LUAD differential genes |
用STRING构建的PPI网络(见图 3a);利用cytoHubba插件选择的前20个hub基因分别是:SPP1、CLDN5、BDNF、TEK、IL6、PPBP、CXCL13、MMP9、CCNA2、EGF、CAV1、MMP7、CDH5、SELE、MMP3、MMP13、MMP1、HMMR、TOP2A、DLGAP5等基因(见图 3b)。

|
图 3 PPI网络构建和Hub基因 Figure 3 PPI network construction and hub genes 注:(a)中高表达基因标记为红色,低表达基因标记为蓝色;(b)中红色表示富集分数高,连接线表示基因间的相互作用. |
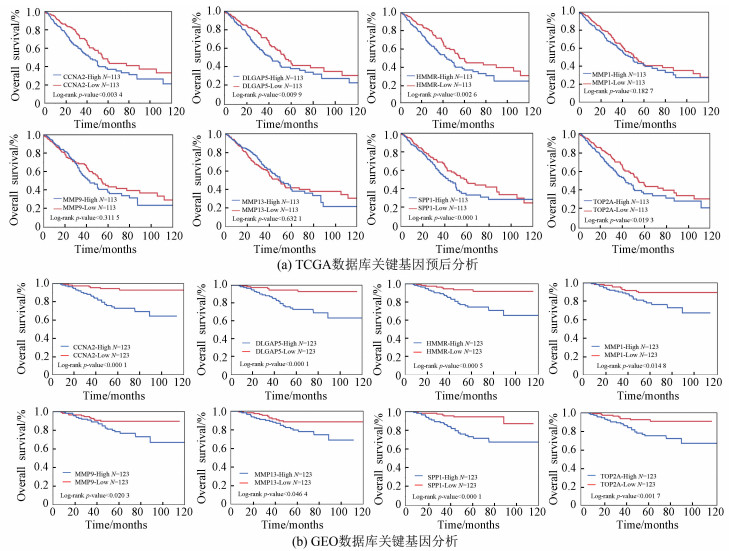
采用卡普兰-迈耶曲线和对数秩检验分析了以上20个肺腺癌关键基因对总生存期的影响,结果显示:CCNA2、DLGAP5、HMMR、MMP1、MMP9、MMP13、SPP1、TOP2A等8个基因对肺腺癌生存期有影响(P<0.05),其中CCNA2、DLGAP5、HMMR、MMP1、SPP1、TOP2A等6个基因对肺腺癌生存期有显著影响(P<0.01)(见图 4)。
|
图 4 肺腺癌关键基因的预后分析 Figure 4 Prognostic analysis of key genes in LUAD 注:A.TCGA数据库关键基因预后分析;B.GEO数据库关键基因分析;横坐标表示时间(月),纵坐标表示患者总生存比率,蓝线表示基因高表达,红线表示基因低表达。 |
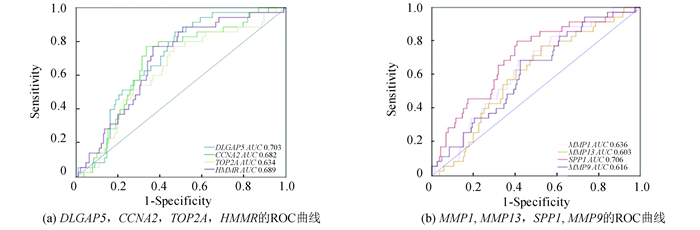
对以上与肺腺癌预后生存相关的8个关键基因进行ROC分析,结果显示:DLGAP5 (AUC=0.703)、CCNA2(AUC=0.682)、TOP2A(AUC=0.634)、HMMR(AUC=0.689)、MMP1(AUC=0.636)、MMP13(AUC=0.603)、SPP1(AUC=0.706)、MMP9(AUC=0.616),其中DLGAP5、SPP1的AUC>0.7,提示它们具有较高的诊断价值(见图 5)。
|
图 5 肺腺癌预后生存相关的8个hub基因的ROC曲线 Figure 5 ROC curves of eight hub genes associated with prognostic survival in LUAD 注:横坐标表示特异性,纵坐标表示敏感性。 |
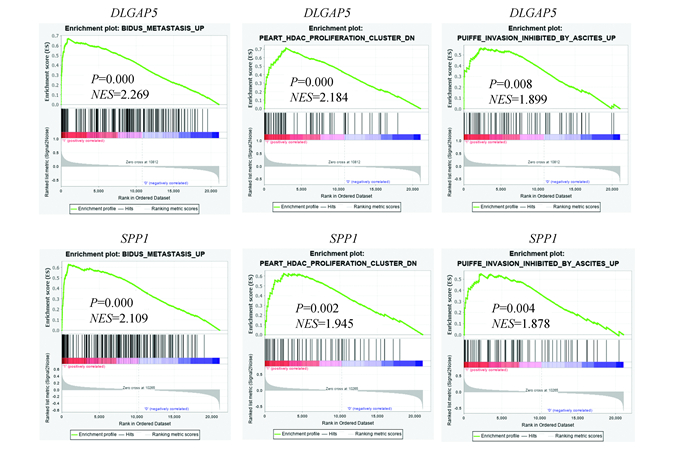
通过上述分析,发现DLGAP5及SPP1与其它hub基因相比更具有作为诊断标志物与预后标志物的潜力,因此验证肺腺癌中DLGAP5、SPP1这2个关键基因的富集相关通路及其免疫相关功能,用GSEA根据DLGAP5、SPP1在肺腺癌组织表达的高低,验证其是否富集在列表的顶部或底部并进行相关功能注释[15],结果发现高表达DLGAP5、SPP1的肺腺癌样本中富集了转移、增殖、侵袭等通路,说明DLGAP5、SPP1等基因在肺癌转移、增殖、侵袭过程中起到促进作用(见图 6,表 1)。
|
图 6 GSEA分析DLGAP5、SPP1基因富集通路 Figure 6 Gene enrichment pathway of DLGAP5 and SPP1 by GSEA analysis |
| 表 1 DLGAP5、SPP1基因在转移、增殖、侵袭等通路的GSEA富集分析结果 Table 1 Results of GSEA analysis of DLGAP5 and SPP1 in metastasis, proliferation, and invasion |
利用GSE31210数据进行分析,使用Cibersort软件“反卷积算法”,分析了数据库中所有肺腺癌样本组织中免疫细胞构成情况(见图 7);然后对正常肺组织与肺腺癌组织中免疫细胞浸润情况进行分析,结果显示:肺腺癌组织免疫细胞情况与正常肺组织存在明显差异,且肺腺癌组织中免疫细胞数量较多的分别是未活化的CD4+记忆性T细胞、记忆性B细胞、滤泡辅助性T细胞、调节性T细胞、嗜酸性粒细胞、M0巨噬细胞(P<0.05)(见图 8)。
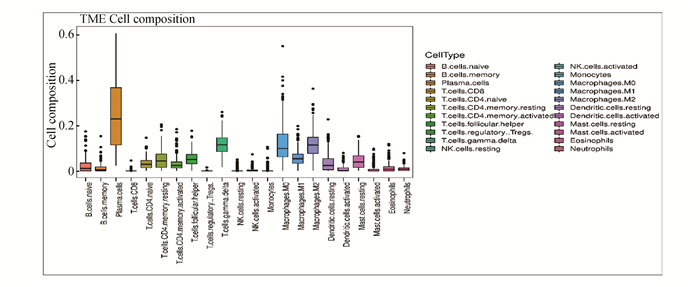
|
图 7 肺腺癌组织样本中22种免疫细胞构成图 Figure 7 Composition of 22 types of immune cells in LUAD tissue samples |
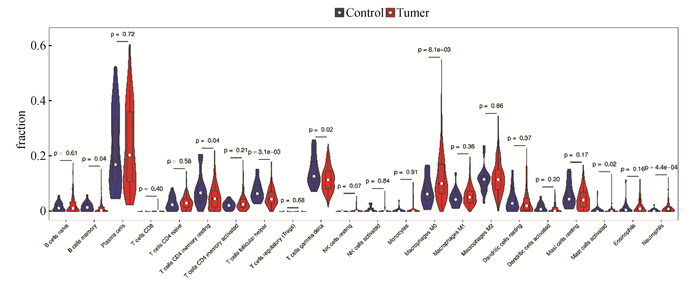
|
图 8 肺腺癌及正常组织中免疫细胞占比小提琴图 Figure 8 Violin plot of percentage of immune cells in LUAD and normal tissues 注:红色为肺腺癌样本,蓝色为正常肺组织样本;横坐标表示免疫细胞,纵坐标表示免疫细胞的百分比。 |
使用Cibersort分析GSE31210数据库中肺腺癌样本的DLGAP5、SPP1基因表达与免疫细胞浸润的关系,结果(见图 9),肺腺癌组织中浆细胞、未活化的CD4+记忆细胞、调节T细胞、巨噬细胞(M0、M1、M2)及中性粒细胞等免疫细胞的数量与DLGAP5、SPP1基因表达水平显著相关(P<0.05),肺腺癌组织中DLGAP5基因高表达时浆细胞、M0巨噬细胞、中性粒细胞等细胞数量较多(P<0.05),而DLGAP5低表达时记忆B细胞、未活化的记忆CD4+T细胞、滤泡辅助性T细胞、未活化肥大细胞分布较多。肺腺癌组织中SPP1基因高表达时巨噬细胞、静息树突细胞、中性粒细胞分布较多,而SPP1基因低表达时肺腺癌组织中浆细胞、未活化记忆CD4+T细胞、调节T细胞、M2巨噬细胞、肥大细胞分布高。以上结果表明DLGAP5、SPP1的表达水平与肺腺癌组织中浆细胞、未活化的CD4+T记忆细胞、调节T细胞、巨噬细胞、巨噬细胞M0、M1、中性粒细胞浸润密切相关。

|
图 9 DLGAP5、SPP1基因表达与肺腺癌组织免疫细胞浸润的关系 Figure 9 Relationship between DLGAP5 and SPP1 gene expression and immune cell infiltration in LUAD 注:(a) DLGAP5基因; (a) SPP1基因;红色代表高表达样本组,蓝色代表低表达样本组。 |
肺腺癌在世界各地发病率和死亡率都很高[16]。肺腺癌的高死亡率在很大程度上归因于诊断不及时,因此寻找特异性早期诊断生物标志物对改善肺腺癌的预后至关重要。本研究利用生物信息学工具分析肺腺癌的mRNA表达谱及其肺腺癌中免疫细胞浸润情况。本研究共筛选出234个肺腺癌DEGs,通过构建PPI网络和富集分析及生存分析,共筛选出20个关键基因,其中CCNA2、DLGAP5、HMMR、MMP1、MMP9、MMP13、SPP1、TOP2A等8个基因对肺腺癌有预后价值。在生物过程方面,DEGs主要涉及胶原代谢、蛋白水解、免疫反应等生物过程,介导血清型内肽酶活性、金属内肽酶活性等分子功能,DEGs基因产物主要富集于细胞外基质、胞外、细胞黏附。研究证明,细胞外基质与受体相互作用参与细胞黏附、细胞周期以及细胞增殖,而这些是导致肺癌中肿瘤增殖和细胞凋亡的关键[17-18]。本研究中肺腺癌关键基因富集的通路主要与PPAR信号通路、PI3K-AKT信号通路、TGF-β信号通路及细胞周期信号通路密切相关,该结果与Tang等人的研究结果相符[19]。
肿瘤中发生浸润的免疫细胞是肿瘤微环境的重要组成部分,其浸润模式与临床结果存在一定相关性,并可能成为药物靶标来提高生存率。本研究中GSEA结果显示DLGAP5、SPP1基因在肺腺癌中主要涉及增殖、迁移、侵袭等有关通路,与肺腺癌的发生发展有关,可作为肺腺癌诊断及预后评估的指标。SPP1(分泌型磷酸蛋白1),也称为骨桥蛋白(OPN)或早期T淋巴细胞活化1蛋白,是一种多功能分泌型酸性糖蛋白,参与体内诸多生理和病理过程[20],其在淋巴细胞、巨噬细胞、内皮细胞、上皮细胞、骨细胞及肿瘤细胞中高表达[21]。SPP1可能参与细胞基质的形成,与肺癌的转移、免疫逃逸等有关[22],在乳腺癌中SPP1与乳腺癌恶性程度呈正相关;在肺癌中SPP1的表达与肿瘤分期、淋巴结侵袭和肿瘤生长有关[23-24]。DLGAP5是细胞周期调控基因的表达产物[25],在肝细胞癌、脑膜瘤和肾上腺皮质瘤等癌症中的表达水平随疾病侵袭性升高而升高[26]。
DLGAP5是细胞周期调控基因的表达产物[25],在肝细胞癌、脑膜瘤和肾上腺皮质瘤等癌症中的表达水平随疾病侵袭性升高而升高[26],因此推测周期调控基因DLGAP5可能在肺癌的免疫浸润方面存在一定作用。另一方面,DLGAP5与视网膜母细胞瘤的发生、浸润有关[27],且已在肺腺癌中被证实为生物诊断标志物。因此,DLGAP5和SPP1基因作为肿瘤微环境中浸润性免疫细胞重要组成部分,可有效预测患者预后[28]。
浸润性免疫细胞是肿瘤微环境的重要组成部分,可有效预测患者预后。本研究用“反卷积算法”分析了数据库中肺腺癌样本组织中免疫细胞构成及正常肺组织与肺腺癌组织中免疫细胞浸润情况,发现肺腺癌组织免疫细胞构成情况与在正常肺组织有明显差异,肺腺癌组织中免疫细胞数量较多主要是未活化的CD4+记忆性T细胞、记忆性B细胞、滤泡辅助性T细胞、调节性T细胞、嗜酸性粒细胞、M0巨噬细胞,且巨噬细胞M0、中性粒细胞数量与肺腺癌浸润程度有关,提示这些免疫细胞参与了肺腺癌的发生与发展。巨噬细胞是肿瘤中主要的免疫浸润细胞,是连接炎症和癌症的关键细胞类型[29],主要为巨噬细胞M1和巨噬细胞M2。巨噬细胞M1可激活细胞因子的产生,募集前免疫刺激白细胞TME,导致肿瘤细胞的吞噬作用,而M2型巨噬细胞可通过基底膜破裂、白细胞募集、血管生成和免疫促进肿瘤的发展[30-31]。有研究表明,巨噬细胞M1水平的增加肿瘤患者预后较好[32],而巨噬细胞M2水平的增加预后较差[33]。在免疫细胞浸润分析中发现,DLGAP5和SPP1低表达的样品中M2巨噬细胞增多。M2巨噬细胞具有激活肿瘤细胞增殖的作用,更重要的是可释放多种细胞因子抑制淋巴T细胞功能,成为影响T淋巴细胞功能和促进肿瘤细胞免疫逃逸的重要因素[34]。前期研究发现,SPP1过表达可参与肺腺癌A549细胞诱导的巨噬细胞的M2极化,进而减弱了T淋巴细胞活性,促进A549细胞增殖、迁移和侵袭[35]。在A549细胞上清液促进了THP-1巨噬细胞向M2的极化,而敲除巨噬细胞中SPP1的可逆转这一过程,以上均表明SPP1在A549细胞和肿瘤微环境中起重要作用[36]。
4 结论研究发现DLGAP5及SPP1与肺腺癌患者的预后生存相关,DLGAP5、SPP1表达越高,则肺腺癌患者预后生存越差;同时,DLGAP5、SPP1基因表达水平与肺腺癌组织免疫细胞浸润密切相关。因此,DLGAP5、SPP1有望成为肺腺癌潜在的诊断和预后生物标志物以及免疫相关治疗靶点,尚需进一步研究证实。
| [1] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:Globocan estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 (  0) 0) |
| [2] |
MITCHELL P L, JOHN T. Lung cancer in 2016: Immunotherapy comes of age[J]. The Lancet. Respiratory Medicine, 2016, 4(12): 947-949. DOI:10.1016/S2213-2600(16)30379-4 (  0) 0) |
| [3] |
GETTINGER S, LYNCH T. A decade of advances in treatment for advanced non-small cell lung cancer[J]. Clinics in Chest Medicine, 2011, 32(4): 839-851. DOI:10.1016/j.ccm.2011.08.017 (  0) 0) |
| [4] |
BOOLELL V, ALAMGEER M, WATKINS D N, et al. The evolution of therapies in non-small cell lung cancer[J]. Cancers (Basel), 2015, 7(3): 1815-1846. DOI:10.3390/cancers7030864 (  0) 0) |
| [5] |
施春波, 董长征, 陈磊, 等. 肿瘤浸润性免疫细胞对肺腺癌预后影响的研究[J]. 浙江医学, 2020, 42(15): 1594-1597+1682. SHI Chunbo, DONG Changzheng, CHEN Lei, et al. Impact of tumor infiltrating immune cells on prognosis in lung adenocarcinoma[J]. Zhejiang Medical Journal, 2020, 42(15): 1594-1597+1682. DOI:10.12056/j.issn.1006-2785.2020.42.15.2019-3701 (  0) 0) |
| [6] |
HAINAUT P, PLYMOTH A. Targeting the hallmarks of cancer: Towards a rational approach to next-generation cancer therapy[J]. Current Opinion in Oncology, 2013, 25(1): 50-51. DOI:10.1097/CCO.0b013e32835b651e (  0) 0) |
| [7] |
KARN T, JIANG T, HATZIS C, et al. Association between genomic metrics and immune infiltration in triple-negative breast cancer[J]. JAMA Oncology, 2017, 3(12): 1707-1711. DOI:10.1001/jamaoncol.2017.2140 (  0) 0) |
| [8] |
LIU X, WU S, YANG Y, et al. The prognostic landscape of tumor-infiltrating immune cell and immunomodulators in lung cancer[J]. Biomedicine & Pharmacotherapy, 2017, 95: 55-61. DOI:10.1016/j.biopha.2017.08.003 (  0) 0) |
| [9] |
YANG S, LIU T, CHENG Y, et al. Immune cell infiltration as a biomarker for the diagnosis and prognosis of digestive system cancer[J]. Cancer Science, 2019, 110(12): 3639-3649. DOI:10.1111/cas.14216 (  0) 0) |
| [10] |
ROBINSON M D, McCarthy D J, SMYTH G K, et al. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data[J]. Bioinformatics (Oxford, England), 2010, 26(1): 139-140. DOI:10.1093/bioinformatics/btp616 (  0) 0) |
| [11] |
柯帅, 王文波, 廖红展, 等. 生物信息学方法筛选胶质母细胞瘤的核心基因[J]. 生物信息学, 2020, 18(1): 56-64. KE Shuai, WANG Wenbo, LIAO Hongzhan, et al. Identifying core genes of glioblastoma by bioinformatics analysis[J]. Chinese Journal of Bioinformatics, 2020, 18(1): 56-64. DOI:10.12113/201907004 (  0) 0) |
| [12] |
ROBIN X, TURCK N, HAINARD A, et al. pROC: An open-source package for R and S+ to analyze and compare ROC curves[J]. BMC Bioinformatics, 2011, 12: 77. DOI:10.1186/1471-2105-12-77 (  0) 0) |
| [13] |
MOOTHA V K, LINDGREN C M, ERIKSSON K F, et al. PGC-1alpha-responsive genes involved in oxidative phosphorylation are coordinately downregulated in human diabetes[J]. Nature Genetics, 2003, 34(3): 267-273. DOI:10.1038/ng1180 (  0) 0) |
| [14] |
SUBRAMANIAN A, TAMAYO P, MOOTHA V K, et al. Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(43): 15545-15550. DOI:10.1073/pnas.0506580102 (  0) 0) |
| [15] |
WANG Y, CHEN L, JU L, et al. Novel biomarkers associated with progression and prognosis of bladder cancer identified by co-expression analysis[J]. Frontiers in Oncology, 2019, 9: 1030. DOI:10.3389/fonc.2019.01030 (  0) 0) |
| [16] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018: Globocan estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 (  0) 0) |
| [17] |
CAVALLARO U, CHRISTOFORI G. Cell adhesion and signalling by cadherins and Ig-CAMs in cancer[J]. Nature Reviews. Cancer, 2004, 4(2): 118-132. DOI:10.1038/nrc1276 (  0) 0) |
| [18] |
李昂, 谢俞宁, 仵红娇, 等. 肺腺癌预后关键基因的筛选、验证及其调控通路分析[J]. 山东医药, 2020, 60(23): 1-5. LI Ang, XIE Yuning, WU Hongjiao, et al. Screening, validation and regulatory pathway analysis of key prognostic genes in lung adenocarcinoma[J]. Shandong Medical Journal, 2020, 60(23): 1-5. DOI:10.3969/j.issn.1002-266X.2020.23.001 (  0) 0) |
| [19] |
TANG Q, ZHANG H, KONG M, et al. Hub genes and key pathways of non-small lung cancer identified using bioinformatics[J]. Oncology Letters, 2018, 16(2): 2344-2354. DOI:10.3892/ol.2018.8882 (  0) 0) |
| [20] |
LISBERG A, CUMMINGS A, GOLDMAN J W, et al. A Phase Ⅱ study of pembrolizumab in egfr-mutant, pd-l1+, tyrosine kinase inhibitor naïve patients with advanced NSCLC[J]. Journal of Thoracic Oncology : Official Publication of the International Association for the Study of Lung Cancer, 2018, 13(8): 1138-1145. DOI:10.1016/j.jtho.2018.03.035 (  0) 0) |
| [21] |
YOSHIDA H, KIM Y H, OZASA H, et al. Nivolumab in non-small-cell lung cancer with EGFR mutation[J]. Annals of Oncology: Official Journal of the European Society for Medical Oncology, 2018, 29(3): 777-778. DOI:10.1093/annonc/mdx745 (  0) 0) |
| [22] |
ZHANG Y, DU W, CHEN Z, et al. Upregulation of PD-L1 by SPP1 mediates macrophage polarization and facilitates immune escape in lung adenocarcinoma[J]. Experimental Cell Research, 2017, 359(2): 449-457. DOI:10.1016/j.yexcr.2017.08.028 (  0) 0) |
| [23] |
TUCK A B, CHAMBERS A F, ALLAN A L. Osteopontin overexpression in breast cancer: Knowledge gained and possible implications for clinical management[J]. Journal of Cellular Biochemistry, 2007, 102(4): 859-868. DOI:10.1002/jcb.21520 (  0) 0) |
| [24] |
HU Z, LIN D, YUAN J, et al. Overexpression of osteopontin is associated with more aggressive phenotypes in human non-small cell lung cancer[J]. Clinical Cancer Research: An Official Journal of the American Association for Cancer Research, 2005, 11(13): 4646-4652. DOI:10.1158/1078-0432.CCR-04-2013 (  0) 0) |
| [25] |
WANG Q, CHEN Y, FENG H, et al. Prognostic and predictive value of HURP in nonsmall cell lung cancer[J]. Oncology Reports, 2018, 39(4): 1682-1692. DOI:10.3892/or.2018.6280 (  0) 0) |
| [26] |
GOMEZ C R, KOSARI F, MUNZ J M, et al. Prognostic value of discs large homolog 7 transcript levels in prostate cancer[J]. PLoS One, 2013, 8(12): e82833. DOI:10.1371/journal.pone.0082833 (  0) 0) |
| [27] |
HUANG J, ZHANG L, LI Z, et al. Screening and identification of key biomarkers for retinoblastoma: Evidence from bioinformatics analysis[J]. Medicine (Baltimore), 2020, 99(20): e19952. DOI:10.1097/MD.0000000000019952 (  0) 0) |
| [28] |
CHEN D S, MELLMAN I. Elements of cancer immunity and the cancer-immune set point[J]. Nature, 2017, 541(7637): 321-330. DOI:10.1038/nature21349 (  0) 0) |
| [29] |
CONWAY E M, PIKOR L A, KUNG S H, et al. Macrophages, inflammation, and lung cancer[J]. American Journal of Respiratory and Critical Care Medicine, 2016, 193(2): 116-130. DOI:10.1164/rccm.201508-1545CI (  0) 0) |
| [30] |
WANG H W, JOYCE J A. Alternative activation of tumor-associated macrophages by IL-4: Priming for protumoral functions[J]. Cell Cycle, 2010, 9(24): 4824-4835. DOI:10.4161/cc.9.24.14322 (  0) 0) |
| [31] |
QUAIL D F, JOYCE J A. Microenvironmental regulation of tumor progression and metastasis[J]. Nature Medicine, 2013, 19(11): 1423-1437. DOI:10.1038/nm.3394 (  0) 0) |
| [32] |
MEI J, XIAO Z, GUO C, et al. Prognostic impact of tumor-associated macrophage infiltration in non-small cell lung cancer: A systemic review and meta-analysis[J]. Oncotarget, 2016, 7(23): 34217-34228. DOI:10.18632/oncotarget.9079 (  0) 0) |
| [33] |
YUAN Z Y, LUO R Z, PENG R J, et al. High infiltration of tumor-associated macrophages in triple-negative breast cancer is associated with a higher risk of distant metastasis[J]. Onco Targets and Therapy, 2014, 7: 1475-1480. DOI:10.2147/OTT.S61838 (  0) 0) |
| [34] |
OSTRAND-ROSENBERG S, SINHA P, BEURY D W, et al. Cross-talk between.myeloid-derived suppressor cells (MDSC), macrophages, and dendritic cells enhances tumor-induced immune suppression[J]. Seminars in Cancer Biology, 2012, 22(4): 275-281. DOI:10.1016/j.semcancer.2012.01.011 (  0) 0) |
| [35] |
OXNARD G R, YANG J C, YU H, et al. TATTON: A multi-arm, phase Ib trial of osimertinib combined with selumetinib, savolitinib, or durvalumab in EGFR-mutant lung cancer[J]. Annals of Oncology: Official Journal of the European Society for Medical Oncology, 2020, 31(4): 507-516. DOI:10.1016/j.annonc.2020.01.013 (  0) 0) |
| [36] |
ZHANG Y, DU W, CHEN Z, et al. Upregulation of PD-L1 by SPP1 mediates macrophage polarization and facilitates immune escape in lung adenocarcinoma[J]. Experimental Cell Research, 2017, 359(2): 449-457. DOI:10.1016/j.yexcr.2017.08.028 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


