2. 吉首大学第一附属医院/湘西自治州人民医院神经外科 湖南 吉首 416000;
3. 吉首大学临床转化医学研究中心 湖南 吉首 416000
2. Department of Neurosurgery, Xiangxi Autonomous Prefecture People's Hospital/First Affiliated Hospital of Jishou University, Jishou 416000, Hunan, China;
3. Centre for Clinical and Translational Medicine Research, Jishou University, Jishou 416000, Hunan, China
胶质瘤是颅内最常见的、最具侵袭性的原发性肿瘤,弥漫性胶质瘤(DGs)是最常见类型,它包括Ⅱ-Ⅲ级星形细胞瘤和少突细胞瘤以及Ⅳ级胶质母细胞瘤(GBM)。胶质瘤内部的异质性很复杂,即使是同一组织学分级,患者的分子和临床特征仍然存在较大差别,例如,低级别胶质瘤(Ⅱ级)IDH野生型患者中位生存期只有20.4月;而IDH突变型合并1p/19q共缺失与否,其中位生存期分别为96月和76.8月;GBM的预后最差,中位生存期仅为12-15月,其中15%的患者超过72月。因此单纯的组织学分类已无法满足胶质瘤的分型,甚至可能在临床上产生错误的指导作用。将胶质瘤分子分型纳入分型标准是大势所趋,但是,胶质瘤高度异质性和预后差异性背后的分子基础还不完全清楚。
IDH突变是DGS中最常见、最早可检测到的遗传变异之一,证据支持该突变为神经胶质瘤发生的驱动力,其突变状态也是最早被引入胶质瘤分子病理的。虽然IDH突变具有良好的对患者预后进行分层的能力,但还不足以解释一切肿瘤特征。与野生型相比,IDH突变型DGs具有一些独特的特征(如其好发于低级别胶质瘤(LGG),具有更好的预后),但是它也对现有一些治疗方式不敏感。IDH1/2突变和其突变产物D2HG可能使胶质瘤产生免疫抑制,从而导致IDH突变型免疫应答水平明显低于野生型胶质瘤[1],这也是导致胶质瘤对很多治疗不敏感的原因之一。所以进一步挖掘IDH突变胶质瘤的分子遗传学特征有助于深入了解其发病机制和发现新治疗靶点,并联合其它分子特征改善对患者的分层,从而指导治疗。本研究拟开发一种反应患者免疫浸润相关的预后预测标签,以便从肿瘤免疫微环境的角度阐明DGs内部的分子异质性。
1 材料与方法 1.1 数据收集利用R的TCGAbiolinks包从GDC下载和处理TCGA胶质瘤基因测序原始数据,包括拷贝数变异数据,突变数据及对应的临床信息。原始数据用DESeq2包进行数据标准化,用preprocessCore包消除系统偏移。从数据库(http://cgga.org.cn)获取CGGA数据。从GEO数据库获得GSE16011数据集(https://www.ncbi.nlm.nih.gov/geo)。TCGA数据集作为训练集,CGGA325数据集作为交叉验证集,CGGA301和GSE16011数据集作为外部验证集。所有数据集中的非DGs样本不纳入本研究;当多个探针对应同一基因名时取平均值。从nCounter © PanCancer Immune Profiling Panel (Human)(https://www.nanostring.com/)获取了770个免疫相关癌基因。选取已经证实的胶质瘤突变基因,数据来源于mutational cancer drivers database<https://www.intogen.org/search>,通过检索共获得75个与GBM相关,50个与LGG相关的候选基因。用maftools包分析这些基因的突变情况,然后只选择有潜在破坏性的突变(包括错义突变、无义突变、缺失性移码突变、插入性移码突变)作为有意义的分子事件进行后续分析。该项目获得吉首大学伦理审查委员会批准。
1.2 建立DGs预后模型首先,用单因素COX风险回归分析从这770个免疫相关基因中筛选生存相关变量,P<0.001的基因作为候选分子标签进入(LASSO)回归分析。用glmnet包进行LASSO回归分析,对候选基因进行惩罚回归,以排除相对不重要的独立变量减少过度拟合。通过cv.glmnet函数交叉验证筛选最优的λ值,构建预测模型。采用基于COX回归线性协变量加权法和靶基因加权值计算风险评分,每个患者均获得免疫风险评分(IMRS), 公式如下:
| $ IMRS = \sum\limits_{i = 1}^N {{\beta _i} * \exp i} $ |
N表示预后基因的数目,expi表示基因的表达量,βi表示基因在单因素COX风险回归分析中的系数。得出评分后用surv_cutpoint函数计算最佳阈值将患者分成高、低风险组。用年龄、性别、等级等临床特征作为协变量,对联合风险评分用survivalanalysis包的analyse_multivariate函数进行多变量COX回归分析,判断其对胶质瘤患者独立预后作用。用survivalROC包行时间依赖性受试者工作特征(ROC)分析,比较联合风险评分对DGs患者1-10年的预后预测能力。随后,将这个公式应用于另外两个外部验证集来验证IMRS的稳定性和可靠性。采用survival包进行风险评分和临床特征(P值<0.05)构建多变量COX比例风险回归模型,然后用regplot包绘制诺莫图,DynNom和rsconnect包生成动态列线图。
1.3 签名相关免疫细胞浸润分析建立风险分子签名预测模型后,我们进一步用TCGA胶质瘤测序表达数据,针对该风险模型包含的临床特征、基因突变、基因拷贝数变异等的变化进行分析,以期更好的发现和解释其影响患者预后的机制。按TCGAbiolinks的用户指南进行基因突变和基因拷贝数变异的数据整理和分析,然后与患者的风险评分进行匹配。ComplexHeatmap包用于热图的绘制。用PROGENy包从基因表达谱推断每个样本中的11个信号通路的通路活性评分,摄动反应基因信号途径反应了癌症基因表达中的信号足迹[2]。GSEA在GSEA4.0.3 (http://www.broadinstitute.org/gsea/index.jsp)中进行。基于分子签名数据库搜索了v7.1 H(hallmark gene sets),以确定与高危人群生存不良相关的具有代表性的明确定义的生物学过程或状态,|NES|> 2和FDR < 0.001被认为有统计学意义。通过CIBERSORT算法(https://cibersort.stanford.edu/)推导出22个浸润免疫细胞的绝对比例。
1.4 统计学分析所有的统计分析均使用R进行(版本3.6.2, http://www.r-project.org)。使用survminer包的surv_cutpoint函数选取基因表达量或评分的最佳阈值对患者进行风险分组。采用Kaplan-Meier评估法评估各组低风险组和高风险组之间的生存差异,并采用log-rank检验进行比较。除特别说明,所有统计检验均为双面检验,P值<0.05,被视为有统计学意义。用ggstatsplot包进行IMRS与检查点基因的表达值等的相关性分析和作图,用ggpubr和ggplot2包进行多组间的比较和作图,使用Mann-Whitney U或Kruskal-Wallis检验来评估箱线图的统计学意义。
2 结果分析 2.1 免疫签名的发展共调查了770个免疫相关基因,其中运用单变量COX回归分析进行筛选初步确定了52个基因为与IDH突变生存相关的候选标签基因。为了减少初次筛查后过度拟合的风险,进一步运用LASSO回归分析。经过1 000次迭代后,最终有6个基因(包括TRAF3, ATG10, BID, TAB1(也叫MAP3K7IP1), MAP3K1, RPS6)被纳入模型。对这6个基因进行线性加权来构建了IMRS公式(见图 1a)。ROC分析对患者1至10年的存活预测,显示该模型对不同时间均有较强的预测能力(AUC=0.673~0.853)(见图 1b)。用临床特征作为协变量,对风险评分进行COX多因素分析,发现联合评分仍具有强烈的独立预后预测能力(HR=9.59, P < 0.000 1)(见图 1c)。Kaplan-Meier分析显示,在训练集中,高危组患者的总生存期(OS)显著低于低危组(见图 1d),同时在交叉验证集中证实了这一差异。进一步分析发现,风险评分与患者的OS线性相关,如按上下四分位法将患者分成高,中,低三个风险组,结果发现组间OS亦存在显著差异(见图 1e)。
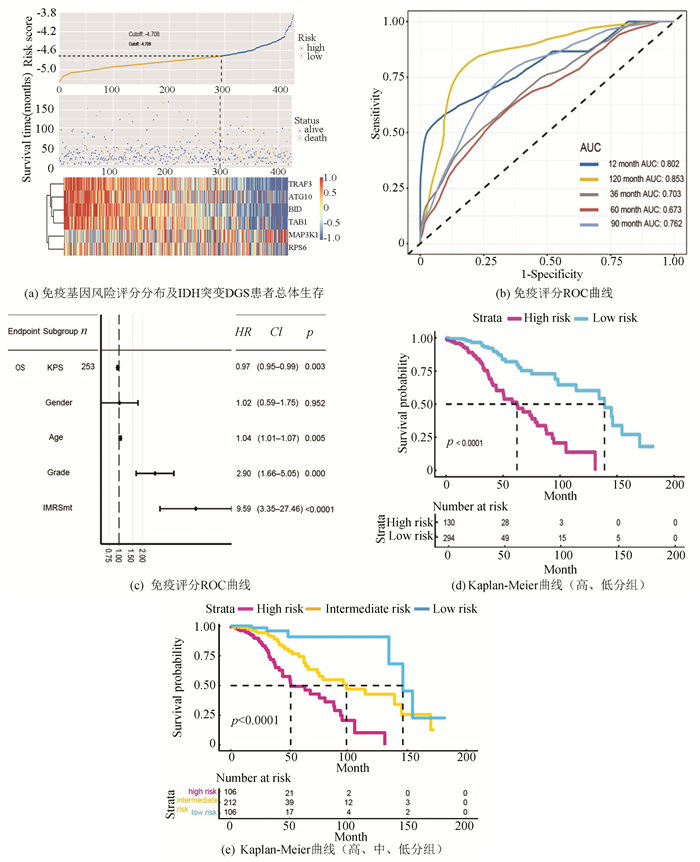
|
图 1 免疫风险评分模型(IMRS)的开发和验证 Figure 1 Development and validation of IMRS model |
在另外两个独立的外部验证数据集中评估IMRS的普适性能力。结果显示,在验证数据集中,低风险组患者的OS均明显延长(见图 2a, 2b)。根据1p19q杂合性丢失状态(LOH),IDH突变胶质瘤通常又可以分为两种亚型:即IDH突变合并1p19q共缺失或IDH突变合并1p19q无缺失亚型[3],每一种分型都有各自的基因组特点。IMRS按表达高低可以将不同亚型进一步进行预后预测分层(见图 2c, 2d),结果表明该风险评分的预测不依赖于胶质瘤的上述变量,具有相对的独立性,同时也表明该预后签名具有良好的预后表现。还分析了IMRS是否对治疗反应具有预测作用,因TCGA数据中只有8例有替莫唑胺(TMZ)或放疗且均属高风险组,故我们利用CGGA325数据(81例)进行分析。结果发现在TMZ治疗组中,获得更长存活时间的患者具有更低的IMRS评分(见图 2e, 2f),所以该评分可以预测患者对治疗的反应性。同时在CGGA301中证实了这一结果(见图 2g, 2h)。
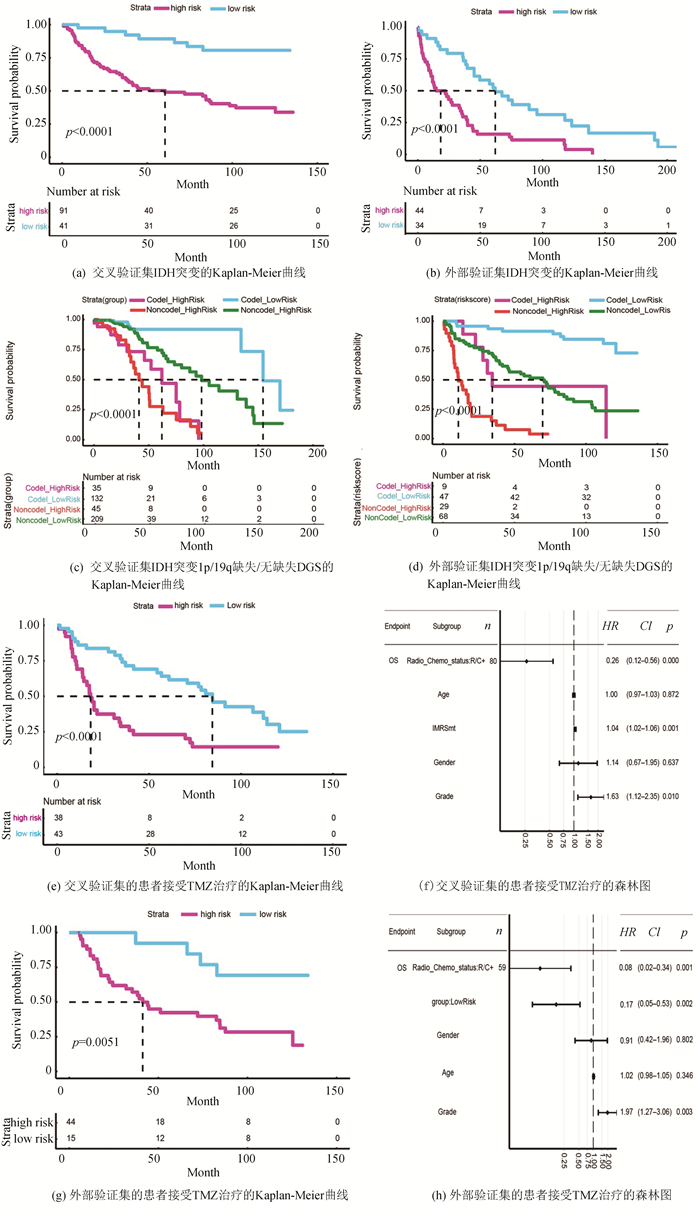
|
图 2 验证集IDH突变状态下的生存分析 Figure 2 Survival analysis of validation sets under IDH mutation state |
按TMIT分类发现IDH突变型只有一例属于Ⅰ型,即所谓“热肿瘤”,其它均属“冷肿瘤”(见图 3a)。但是它们内部免疫状态仍然存在很大差别,用CIBERSORT(http://cibersort.stanford.edu/)分析肿瘤免疫细胞浸润情况,结果显示巨噬细胞M2占据了所有样本的最大浸润比例(见图 3b)。为了比较肿瘤内的抗癌和促癌免疫浸润状态, 定义了两个总体评分:(1)肿瘤相关巨噬细胞浸润评分(TAM),包括单核细胞、巨噬细胞M1、巨噬细胞M2等免疫细胞;(2)肿瘤相关T细胞浸润评分(TIS),由四个T细胞评分得到(CD8T细胞、幼稚型CD4T细胞、沉默记忆CD4+T细胞、和滤泡帮助T细胞)。删除了信号活性小于50%的细胞类型(记忆活性的CD4T细胞,调节性T细胞,伽马delta-T细胞,巨噬细胞M0)后,计算两个评分与IMRS的相关性,发现TIS与IMRS呈明显的负相关;而TAM与IMRS呈现与TIS相反的趋势。表明IDH突变型胶质瘤内巨噬细胞M的高度浸润总体效应是发挥促癌作用的(见图 3c, 3d)。风险分组间的卡方检验也印证了此结果(见图 3e, 3f)。以上结果说明高IMRS富集了促肿瘤免疫浸润,而低IMRS则富集了相对较多的抗肿瘤免疫浸润。各类型免疫细胞浸润与IMRS密切相关,主要有记忆B细胞(r=0.36, p=2.53×10-14),幼稚型CD4T细胞(r=0.38, p=5.51×10-16),巨噬细胞M2 (r=-0.33, p=1.88×10-12),在TIS浸润中幼稚型CD4T细胞作出了主要贡献,而在TAM浸润中巨噬细胞M2作出了主要贡献(见图 3g)。利用TCGA数据我们开发了一个诺莫图(见图 3h),同时制作了在线(https://huangchunhai.shinyapps.io/DynNomapp/)预测App。校准曲线显示,预测的2年、5年、10年生存率与实际观察比值密切相关(见图 3i)。
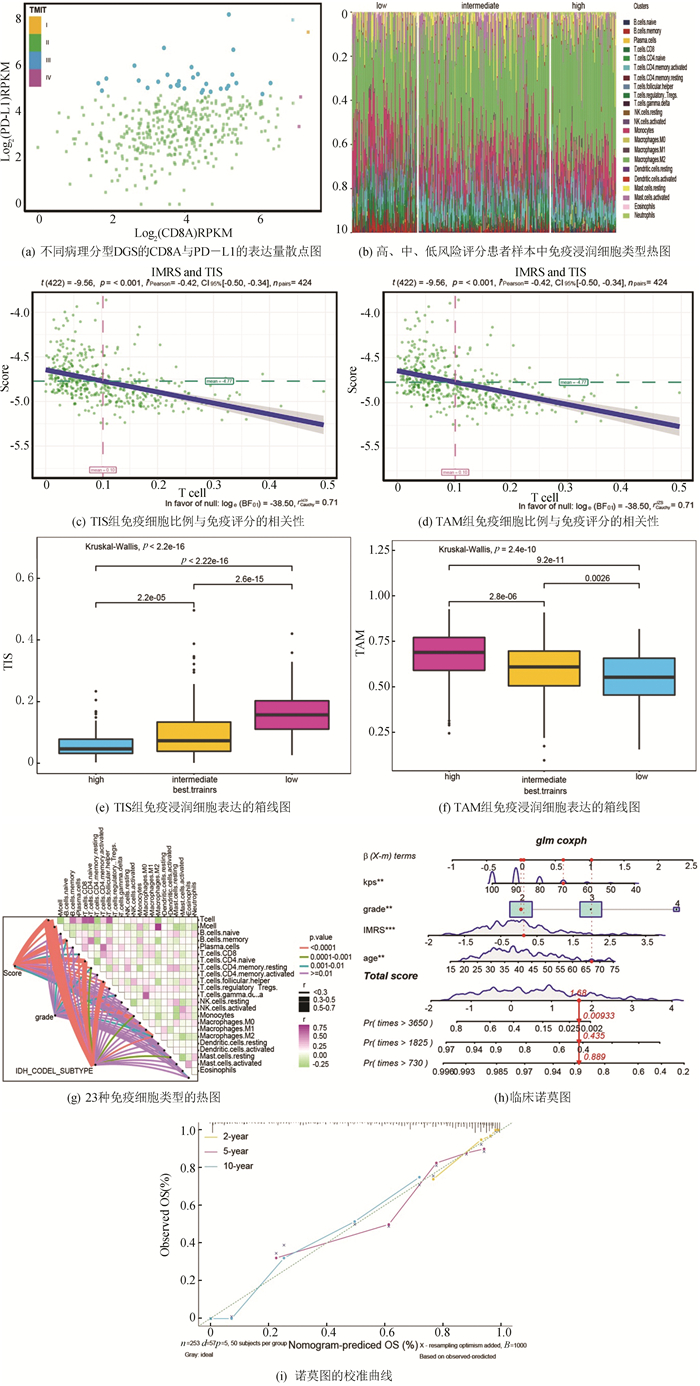
|
图 3 免疫细胞浸润景观和临床诺莫图的建立 Figure 3 Establishment of landscape of immune cell infiltration and clinical nomograms |
DGS是一种具有不同恶性程度和异质性的疾病,IDH突变是常见的一种类型,IDH表型与基因组稳定性有关。IDH突变与胶质瘤的免疫微环境显著相关,然而,IDH突变与胶质瘤的免疫浸润状态之间的关系尚不完全清楚,因此,了解IDH突变与免疫微环境的关系对预测弥漫性胶质瘤的预后有重要意义。在本文中,利用R语言算法通过对TCGA、CGGA和GEO数据库的数据进行研究,发现了一些可以反应IDH突变型胶质瘤内不同免疫状态的免疫基因标签,并证明免疫浸润程度与患者预后密切相关。
在本研究中对IDH突变在DGs免疫浸润状态中的作用进行了全面研究。首先用TCGA数据库中的胶质瘤基因测序数据结合癌症免疫相关基因通过LASSO回归分析筛选出6个差异表达的基因,并建立了免疫风险评分IMRS模型。利用分子特征和临床特征的互补价值,并将它们整合到一个新的诺莫图中,研究发现联合评分比单一的生物标志物具有更强烈的预后预测能力。同样在CGGA和GEO的验证数据集中也能反映相同的结论。IMRS模型由6个基因组成,分别是TRAF3、ATG10、BID、TAB1、MAP3K1、RPS6。TRAF3作为TRAF家族的重要成员,在哺乳动物的抗病毒免疫过程中发挥着重要的作用[5],是参与RIP2诱导的神经胶质瘤细胞生长的负调节剂,有研究证实TRAF3在胶质瘤中表达下调。ATG10是一种与自噬相关的特殊蛋白相关基因,通过编码自噬酶E2,并与自噬相关基因7(ATG7)相互作用,招募泛素样分子ATG12,与ATG12-ATG5产生共轭反应,在癌症的增值和侵袭中发挥作用[6-7]。BID是一个只含有BH3结构域的促凋亡bcl-2家族成员,即可以参与凋亡的外部信号转导,又可以介导DNA损伤反应来调节细胞死亡程序[6-7]。当细胞暴露于含凋亡因子的环境时,形成tBID, tBID在线粒体中积累,使细胞色素C释放到细胞质中发挥调节细胞凋亡的作用,或tBID从细胞质转运到线粒体外膜激活Bix和Bak,导致线粒体外膜通透化(MOMP),从而激活caspase-3导致细胞凋亡[9]。近来研究表明,在胶质瘤细胞中小分子Mcl-1抑制剂可诱导细胞凋亡,为胶质瘤的治疗提供了一种新的治疗策略。TAB 1是一种与转化生长因子β活化激酶1(TAK1)相互作用的特异蛋白,在应激条件下通过增加TAK1的激活调节TAK1介导的体外细胞因子释放,导致下游信号诱导TAB 1的糖基化[11]。研究表明,NF-кB信号通路的激活可以自主的提高HIF-1α的活性并增强糖酵解代谢,TAB 1可能通过NF-кB/HIF-1α参与巨噬细胞糖酵解和活化,是DNA发生和进化的主要环节,HIF-1α被认为是DNA治疗的一个新的靶点。MAP3K1[12]是MAPK家族的一个成员,具有调节细胞凋亡、生存、迁移、分化等多重作用,可以被多种刺激激活,几乎在所有细胞中参与重要调节功能,意味着其可能是控制癌症的靶点。近期的大量基因组学研究表明,不同类型癌症中均发现MAP3K1基因拷贝数异常、染色体突变、基因无效突变。认识MAP3K1基因及其蛋白功能在不同类型癌症中的改变,为研究肿瘤细胞药物治疗靶点提供指导意义。RPS6在细胞增殖和DNA修复中发挥重要作用,最近的一项研究报道[13]在高级别胶质瘤中RPS6显著上调。RPS6下调显著抑制了胶质瘤干细胞(GSCs)的特征,RPS6上调与GBM细胞中干细胞特征的诱导和维持有关,RPS6敲除可以抑制GBM细胞的球形成电位和GSC标记物的表达,这些发现是靶向治疗核糖体蛋白下调胶质母细胞瘤干细胞特性的突破。这些基因有望作为新的分子靶点,为免疫浸润在胶质瘤研究中提供了新的方向。研究中,观察到在高免疫评分危险组的6个基因过表达,患者的总生存期较差,提示TRAF3、ATG10、BID、TAB1、MAP3K1、RPS6的高表达与IDH突变DGS患者预后较差有关。
为了更好地了解这些IDH突变相关免疫预后基因的生物学功能,依据1p19q LOH状态将IDH突变胶质瘤分为IDH突变合并1p19q缺失和未缺失两种不同免疫状态的亚组,进行了进一步的生物信息学分析。发现1p/19q未缺失组主要富集于免疫评分的高风险组,提示1p/19q未缺失可能包含致癌基因。
在肿瘤细胞微环境中,T细胞的激活与抑制通常处于异常状态,其正常化被广泛视为治疗肿瘤的重要手段。在对CGGA325数据中的81例TMZ治疗组分析发现:IMRS评分较低的患者具有更长的存活时间,说明IMRS评分在一定程度上可以反映对化疗的敏感性,同时也表明该预后签名具有可靠的预测能力。免疫细胞浸润分析发现高IMRS富集了以巨噬细胞M2为主的促肿瘤免疫浸润细胞,而低IMRS却富集了以幼稚型CD4+T细胞为主的抗肿瘤免疫浸润细胞。最后开发一组反应患者免疫浸润相关的预后预测标签,以便从肿瘤免疫微环境的角度阐明IDH突变胶质瘤肿瘤内部的分子异质性。基因免疫标签可以反应IDH突变型胶质瘤内不同免疫浸润状态,并表明浸润程度影响着患者的预后,此外,该签名将可能是识别受益于免疫疗法的患者的有用预测工具。
4 结论通过分析TCGA、CGGA和GEO队列中DGs的基因突变信息、基因表达谱和免疫浸润情况,构建IMRS公式,将患者分为高危组和低危组;建立免疫微环境相关基因的IDH突变DGs预后模型,证明该模型可作为IDH突变DGs患者独立的预后因素,低风险组患者的OS较高风险组均明显延长。签名相关免疫细胞浸润分析显示高IMRS富集了促肿瘤免疫浸润,而低IMRS则富集了相对较多的抗肿瘤免疫浸润。将免疫评分与临床因素结合构建诺莫图和在线预测App,可以定量预测DGs患者的OS,有利于临床医生(在IDH突变的弥漫性胶质瘤的诊治中)做出临床决策。
| [1] |
KOHANBASH G, CARRERA D A, SHRIVASTAV S, et al. Isocitrate dehydrogenase mutations suppress STAT1 and CD8+ T cell accumulation in gliomas[J]. Journal of Clinical Investigation, 2017, 127(4): 1425-1437. DOI:10.1172/JCI90644 (  0) 0) |
| [2] |
SCHUBERT M, KLINGER B, KLUNEMANN M, et al. Perturbation-response genes reveal signaling footprints in cancer gene expression[J]. Nature Communications, 2018, 9(1): 20. DOI:10.1038/s41467-017-02391-6 (  0) 0) |
| [3] |
WAITKUS M S, DIPLAS B H, YAN H. Isocitrate dehydrogenase mutations in gliomas[J]. Neuro Oncology, 2016, 18(1): 16-26. DOI:10.1093/neuonc/nov136 (  0) 0) |
| [4] |
OCK C Y, KEAM B, KIM S, et al. Pan-cancer immunogenomic perspective on the tumor microenvironment based on PD-L1 and CD8 T-cell infiltration[J]. Clinical Cancer Research, 2016, 22(9): 2261-2270. DOI:10.1158/1078-0432.CCR-15-2834 (  0) 0) |
| [5] |
CAI X, YANG Y, XIA W, et al. RIP2 promotes glioma cell growth by regulating TRAF3 and activating the NFkappaB and p38 signaling pathways[J]. Oncology Reports, 2018, 39(6): 2915-2923. DOI:10.3892/or.2018.6397 (  0) 0) |
| [6] |
KONGARA S, KARANTZA V. The interplay between autophagy and ROS in tumorigenesis[J]. Frontiers in Oncology, 2012, 2: 171. DOI:10.3389/fonc.2012.00171 (  0) 0) |
| [7] |
JO Y K, KIM S C, PARK I J, et al. Increased expression of ATG10 in colorectal cancer is associated with lymphovascular invasion and lymph node metastasis[J]. PLoS One, 2012, 7(12): e52705. DOI:10.1371/journal.pone.0052705 (  0) 0) |
| [8] |
KANTARI C, WALCZAK H. Caspase-8 and bid: Caught in the act between death receptors and mitochondria[J]. Biochimica Biophysica Acta, 2011, 1813(4): 558-563. DOI:10.1016/j.bbamcr.2011.01.026 (  0) 0) |
| [9] |
LEMKE J, VON KARSTEDT S, ZINNGREBE J, et al. Getting TRAIL back on track for cancer therapy[J]. Cell Death Differ, 2014, 21(9): 1350-1364. DOI:10.1038/cdd.2014.81 (  0) 0) |
| [10] |
LIU J W, ZHU Z C, LI K, et al. UMI-77 primes glioma cells for TRAIL-induced apoptosis by unsequestering Bim and Bak from Mcl-1[J]. Molecular and Cellular Biochemistry, 2017, 432(1/2): 55-65. DOI:10.1007/s11010-017-2997-x (  0) 0) |
| [11] |
PATHAK S, BORODKIN V S, ALBARBARAWI O, et al. O-GlcNAcylation of TAB1 modulates TAK1-mediated cytokine release[J]. EMBO J, 2012, 31(6): 1394-1404. DOI:10.1038/embij.2012.8 (  0) 0) |
| [12] |
WANG J, ZUO J, WAHAFU A, et al. Combined elevation of TRIB2 and MAP3K1 indicates poor prognosis and chemoresistance to temozolomide in glioblastoma[J]. CNS Neuroscience & Therapeutics, 2020, 26(3): 297-308. DOI:10.1111/cns.13197 (  0) 0) |
| [13] |
SHIRAKAWA Y, HIDE T, YAMAOKA M, et al. Ribosomal protein S6 promotes stem-like characters in glioma cells[J]. Cancer Science, 2020, 111(6): 2041-2051. DOI:10.1111/cas.14399 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


