2. 甘肃省牦牛繁育工程重点实验室, 兰州 730050;
3. 青海大通种牛场, 西宁 810000
2. Key Laboratory of Yak Breeding Engineering, Lanzhou 730050, China;
3. Datong Cattle Farm in Qinghai Province, Xining 810000, China
NLRP蛋白家族最早在人上被发现,共有14个家族成员,可以分为免疫相关蛋白和生殖相关蛋白。免疫相关蛋白主要在先天免疫系统发挥功能,包括NLRP1、NLRP3、NLRP6、NLRP10和NLRP12,生殖相关蛋白主要在哺乳动物生殖系统中起作用,包括NLRP2、NLRP4、NLRP5、NLRP7、NLRP8、NLRP9、NLRP11、NLRP13和NLRP14[1]。NLRP蛋白的基本结构主要由3个保守的结构域组成,第一个是氨基端的PYD(Pyrin domain),该结构域包含92个氨基酸,主要功能是介导蛋白之间的相互作用,第二个是中间的NACHT结构域,该结构域是以4个相近的含核苷水解酶域的蛋白合起来命名的, 包括神经元凋亡抑制蛋白(Neuronal apoptosis inhibitor protein, NAIP)、Ⅱ型主要组织相容性复合体转录激活子(Class Ⅱ major histocompatibility complex trans-activator, CⅡTA)、来源于柄孢霉的非亲和位点蛋白(HET-E)、哺乳动物端粒酶相关蛋白(Telomerase associated protein 1, TP1)。最后是羧基端的LRRs(Leucine-rich repeats)结构域[2],该结构域含有9-12个亮氨酸重复,主要功能是介导自身调节、蛋白间的相互作用和病原相关分子模式的传感。
近年来对NLRP基因的研究主要集中在炎症小体上。NLRP家族中的炎症体包含NLRP1和NLRP3炎症小体。炎症小体是由胞浆内模式识别受体参与组装的多蛋白复合物。炎症小体活化后可促进炎症因子的成熟和释放,从而参与先天免疫过程。炎症小体募集天冬氨酸蛋白水解酶-1(Caspase-1),引起capase-1蛋白水解pro-IL-1b和pro-IL-18,产生成熟的活性细胞因子[3]。NLRP1炎症体由感受器NLRP1、结合蛋白ASE和效应蛋白Caspase-1三个部分组成,能够促进IL-18,IL-1b及IL-33的成熟,在炎症发生时起作用[3]。在NLRP蛋白中,NLRP10是唯一一个缺乏LRR结构域的家族成员。有研究表明,该蛋白是炎症和细胞凋亡的多功能负调节剂,能激活促炎性半胱天冬酶、半胱天冬酶-1和半胱天冬酶-5[4]。另外,与生殖相关的NLRP基因也备受关注。小鼠中Nlrp2是卵母细胞质量的关键调节因子,敲除掉该基因的雌性小鼠的繁殖率会随着年龄的增长而显著下降[5]。Nlrp4和Nlrp9在小鼠上具有特异性复制的特点,在小鼠中Nlrp4有7个拷贝基因,Nlrp9有3个拷贝基因[6]。Nlrp4g参与了小鼠早期胚胎的发育,在卵母细胞中特异性表达[7]。Nlrp9的复制基因可能没有具体的功能,有研究发现同时敲除Nlrp9a和Nlrp9e或在合子中沉默Nlrp9b对小鼠早期胚胎发育没有明显的影响,表明在小鼠早期胚胎发育过程中可能不需要Nlrp9的参与[8]。啮齿类动物中没有Nlrp7,被认为是在物种进化过程中由Nlrp2复制而形成的,二者基因位点相近,由Nlrp2执行Nlrp7的功能[9]。NLRP7不仅在免疫活动中发挥作用,还是一种母源印记基因,相关研究表明NLRP7印记状态的异常会导致人胚胎发育的异常[10],从而造成葡萄胎或者流产现象的发生。
牦牛是生活在海拔2 500-6 000 m的青藏高原及其邻近地区的一种独特的家畜,自古以来,牦牛是青藏高原地区牧民必不可缺的家畜,为高原地区的牧民提供了吃、住、行等基本生存保证[11]。牦牛能适应高海拔地区的严寒、缺氧、缺草等恶劣自然条件,是哺乳动物中研究高海拔适应性的理想物种[12]。与大部分一年一胎的牛科动物相比,牦牛的性成熟时间晚[13],生殖性能差,每两年生育一胎或每三年生育两胎。其次,牦牛体外受精的囊胚发育率低,影响牦牛生产。近年来,多个NLRP基因在牦牛生殖相关的研究中被报道,如陈虹[14]在牦牛卵巢蛋白质分析中鉴定到NLRP5在卵母细胞和受精卵中高表达;杨芳[15]通过对牦牛和犏牛睾丸的比较转录组分析发现NLRP14的上调可能会造成原始生殖细胞或未分化精原细胞的积累,进而加剧生殖细胞凋亡,表明该家族基因在牦牛上也发挥重要作用。目前,NLRP基因家族在人、小鼠基因组上被完整鉴定,但在牦牛上尚无报道。本研究对牦牛NLRP基因家族进行系统性分析,可为进一步研究牦牛NLRP基因家族的功能奠定基础。
1 材料与方法 1.1 牦牛NLRP基因家族的鉴定从UniProt数据库[16]下载已知人类NLRP蛋白序列(见表 1),使用BLSAT+软件[17]构建本地数据库,同源比对牦牛蛋白序列。牦牛参考基因组、蛋白序列和注释文件来源于NCBI(https://www.ncbi.nlm.nih.gov/genome/?term=wild+yak)。同时利用HMMER软件对人NLRP蛋白序列建立HMM模型,在牦牛全基因组蛋白序列中搜索同源序列,筛选阈值设置为E-value<1×10-5,去除可变剪切后,同时满足两种鉴定方法的序列被认为是牦牛NLRP基因家族成员,基因名字以基因组注释文件给定的名称命名。
| 表 1 人类NLRP蛋白信息 Table 1 Human NLRP proteins |
采用ClustalW[18]对鉴定出的牦牛NLRP蛋白进行序列比对,利用MEGA-X软件[19]对牦牛、小鼠、大鼠、狗、牛、人NLRP蛋白序列进行多重序列比对,采用最大似然法构建系统进化树。最后使用Evolview软件(https://evolgenius.info//evolview-v2/#login)对进化树进行美化处理。
1.3 牦牛NLRP蛋白理化性质分析利用在线生物信息学软件Expasy Protoparam[20]、SignaIP 5.0 Server[21]、TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/TMHMM/)、Wolf PSORT[22]分别预测NLRP蛋白长度、分子量、等电点、不稳定系数、信号肽、跨膜结构和蛋白亚细胞定位情况。采用在线工具GSDS[23]对牦牛NLRP基因结构进行可视化分析。
1.4 基因结构和保守结构域分析使用NCBI在线工具CD-search[24]和PFAM数据库[25]对鉴定出的牦牛NLRP蛋白进行保守结构域鉴定。利用MEME[26]分析NLRP保守基序(默认参数),并用TBtools [27]进行展示。运用NetPhos 3.1 Server(http://www.cbs.dtu.dk/services/NetPhos/)分析NLRP蛋白的磷酸化位点,并且统计各个位点的个数。采用SOPMA[28]预测牦牛NLRP蛋白的二级结构。使用STRING数据库[29]查询牦牛NLRP的蛋白互作网络。
1.5 牦牛NLRP基因家族的表达分析牦牛不同组织的转录组数据下载自NCBI数据库(PRJNA362606)[30]。首先,利用fastq-dump软件(https://ncbi.github.io/sra-tools/fastq-dump.html)将下载得到的SRA文件转换为fastq格式文件, 对于上述所有过滤后的RNA-Seq数据,使用fastp[31]对下载的原始数据需要进行质控和预处理,得到的clean data用于后续分析。在Hisat2(2.1.0版)中使用Hisat2-build命令[32],默认参数下构建基因组索引。随后利用featureCounts计数, 得到每个基因在各个样本中的raw counts。在这项研究中,我们使用FPKM作为一个标准化的转录丰度(Fragments Per Kilobase of exon model per Million mapped fragments),利用R脚本根据各个样本的raw counts计算出基因的表达量构建出基因表达矩阵,共18个转录组样本。最后提取NLRP基因的表达数据,利用R软件对其进行分析、聚类和可视化。
2 结果分析 2.1 牦牛NLRP基因家族成员的鉴定及蛋白理化特性分析从牦牛基因组中共鉴定出8个NLRP基因家族成员(见表 2),按照注释文件中的信息命名。蛋白基本理化性质分析结果显示:牦牛NLRP蛋白分子量介于113 117.63-124 957.17 Da之间,蛋白长度介于996-1 098之间,等电点介于5.52-8.27之间,消光系数介于70 340-130 560之间,不稳定系数介于44.74-57.41之间,脂肪族指数介于88.57-98.02之间。8个NLRP蛋白均为不稳定、无信号肽、无跨膜结构域的疏水蛋白。亚细胞定位结果显示,不同的NLRP蛋白定位有所差异,NLRP3、NLRP6和NRLP12均定位于细胞质膜,NLRP5和NLRP14定位于细胞质基质或者细胞核,NLRP8定位于细胞核中,NLRP2和NLRP9定位于细胞质基质中。所有的NLRP蛋白都有丝氨酸、苏氨酸、酪氨酸磷酸化位点(见表 3),磷酸化位点数依次为丝氨酸>苏氨酸>酪氨酸,每个家族成员的磷酸化位点个数介于86~102个之间。其中,NLRP14含有的磷酸化位点最多(102个),NLRP2最少(83个)。
| 表 2 牦牛NLRP蛋白基本信息和编码蛋白理化性质分析结果 Table 2 Basic information of NLRP proteins and physiological and chemical properties of their encoded proteins |
| 表 3 牦牛NLRP蛋白的磷酸化位点预测 Table 3 Phosphorylation site prediction of NLRPs of yak |
通过ClustalW对8个NLRP蛋白进行多重序列比对,结果显示NLRP蛋白的氨基酸序列相似度低(见图 1)。两两比对分数在17.1%到38.4%不等。其中NLRP3和NLRP12的比对分数最高(38.4%),NLRP2和NLRP5的比对分数最低(17.1%)。序列中部分位点Y、R、E、V、F在所有蛋白序列中完全一致。

|
图 1 牦牛NLRP蛋白氨基酸序列比对情况 Figure 1 Amino acid sequence alignment of NLRP proteins in yak |
利用MEGA X对8个牦牛、8个黄牛、14个人、10个狗、12个大鼠和19个小鼠NLRP蛋白氨基酸序列进行多重序列比对和构建系统进化树(见图 2)。从进化树的结构可以看出不同家族成员在进化上已经发生了分离,同一家族成员氨基酸序列相似度高,在6个物种中都聚在一起。免疫相关和生殖相关的NLRP蛋白已分开(Group Ⅰ和Group Ⅱ),生殖相关的NLRP蛋白之间相似度要高于免疫相关蛋白。通过与已验证的人、小鼠等物种的NLRP蛋白序列比对,进一步确认注释文件的准确性。

|
图 2 牦牛NLRP蛋白的系统进化分析 Figure 2 Phylogenetic analysis of NLRP proteins in yak |
基因结构分析结果显示(见图 3),NLRP基因内含子数量介于8-12个之间,外显子数目介于9-13个之间。5′-UTR和3′-UTR的长短差异较大,3′-UTR长度要大于5′-UTR。NLRP8和NLRP2没有5′-UTR和3′-UTR结构,NLRP9、NLRP5、NLRP12和NLRP6没有5′-UTR结构。从基因结构预测的结果上看,免疫相关蛋白和生殖相关蛋白没有显著区别。

|
图 3 牦牛NLRP家族成员基因结构 Figure 3 Gene structure of NLRP family members in yak |
MEME预测结果显示牦牛NLRP蛋白存在10种保守基序(motif 1-10)(见图 4),长度分别为34、49、49、48、21、48、50、41、39、29 bp。NLRP5、NLRP12和NLRP14没有motif 4。NLRP14的motif数量最多(16个),NLRP6数量最少(10个)。10个motif中,C、D、L、N、G、D位点的一致性较好(见图 5)。牦牛NRLP蛋白与其他物种相同,都具有该家族代表性保守结构域:NACHT、PYRIN和LRR(见图 6)。

|
图 4 牦牛NLRP蛋白保守基序分布 Figure 4 Conserved motifs of NLRP proteins in yak |
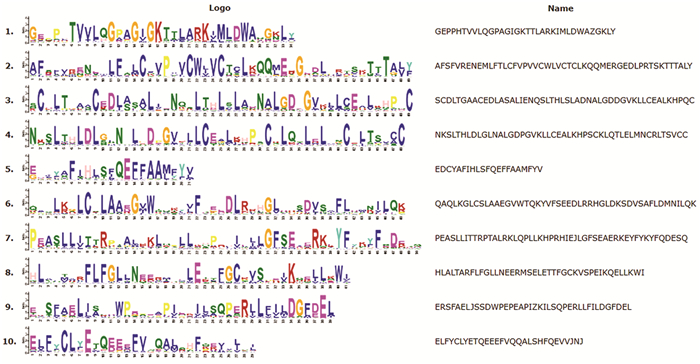
|
图 5 牦牛NLRP蛋白典型保守基序序列图 Figure 5 Typical conserved motif sequences of NLRP proteins in yak 注:每个字母代表一种氨基酸,字母的大小代表该位点氨基酸的个数,与氨基酸一致性有关,字母越大个数越少,说明该位点的一致性越好,保守性也越好,反之说明该位点的一致性较差,保守性低. |
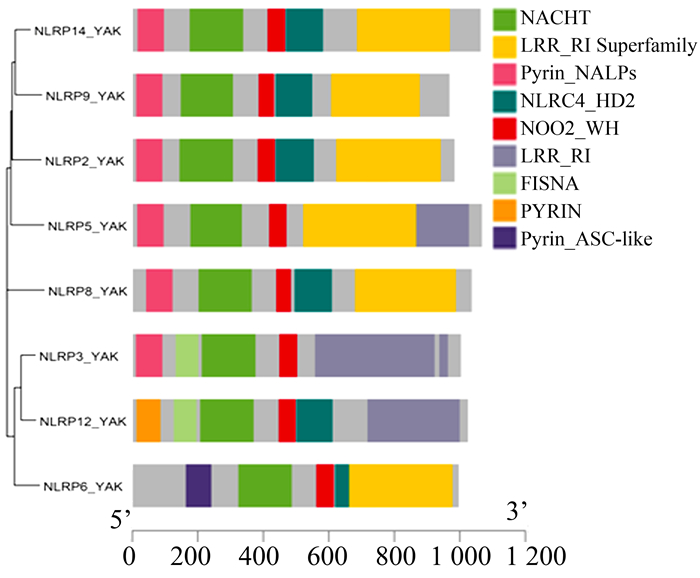
|
图 6 牦牛NLRP蛋白保守结构域分布 Figure 6 Conserved domains of NLRP proteins in yak |
对牦牛NLRP蛋白的氨基酸序列进行二级结构预测,结果(见表 4),牦牛NLRP蛋白二级结构主要元件为α-螺旋,其次依次为无规则卷曲、延伸链和β-转角。为了解蛋白之间的功能联系和在生物系统中的工作原理,在蛋白互作网络数据库中查询了8个NLRP蛋白的互作网络(见图 7)。NLRP2和NLRP14的调控网络相似,都与PYCARD、1L1B、1L18、NEK7和MAVS互作。NLRP3、NLRP6和NLRP12也与PYCARD互作。NLRP5、NLRP8和NLRP9具有独立的调控网络。NLRP5与BMP15、ZAR1、RAD16等蛋白存在相互作用。NRLP8和MEFV、TAF8、TAF3等蛋白互作。NLRP9与TBK1、UBC、TTC12等蛋白互作。
| 表 4 NLRP蛋白二级结构 Table 4 Secondary structure of NLRP proteins in yak |
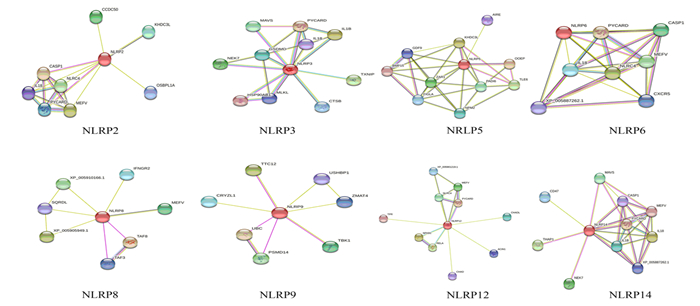
|
图 7 牦牛NLRP家族成员蛋白互作网络 Figure 7 Protein-protein interaction network of NLRP family members in yak |
为了研究NLRP基因家族在牦牛不同组织中的表达情况,本研究从公共数据库NCBI下载了牦牛转录组测序数据[30],获得了8个牦牛NLRP基因在6种组织中的表达模式。结果显示,牦牛NLRP基因在6种组织中的表达较为微弱或者不表达(见图 8)。NLRP2只在肺脏和脾脏中检测到表达。NLRP3、NLRP5在6种组织中均有表达,二者均在脾脏中的表达量最高。NLRP6在肾脏中的表达量要高于脾脏和肺脏。NLRP8在脾脏和肝脏中检测到表达。NLRP9仅在脾脏中检测到。NLRP12在心脏中不表达,在其他组织中表达。NLRP14只在肺和肾脏中检测到。
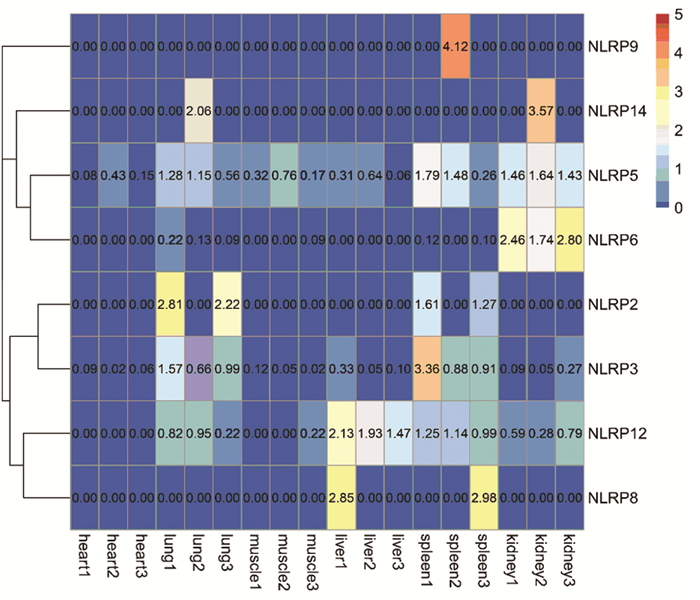
|
图 8 NLRP基因在牦牛不同组织的表达模式 Figure 8 Expression patterns of NLRP genes in different tissues of yak 注:基因表达热图中的每个小方块代表每个基因,基因的表达量由小方块的颜色深度来表示。行表示每个基因在不同样本中的表达,列表示每个样本中所有基因的表达。表达量值按照行进行标准化,并基于平均连接算法进行聚类. |
随着基因组学的发展,动物基因组的测定以及注释信息的完善,使得我们能够通过生物信息学的方法分析基因的相关信息,从序列的角度直接解析基因的各种信息,对目标基因可能存在的生物学功能进行预测[33]。本研究共鉴定出牦牛NLRP基因家族的8个成员,其中免疫相关的有NLRP3、NLRP6和NLRP12,生殖相关的有NLRP2、NLRP5、NLRP8、NRLP9和NLRP14。
NLRP基因在很多物种中被鉴定出来,但是在物种之间存在很大差异。例如,在灵长类上已鉴定出14个NLRP基因[34],在黄牛上已鉴定出8个NLRP基因[35],在小鼠上已鉴定出20个NLRP基因[36],在狗上已鉴定出11个NLRP基因[35]。小鼠基因组上缺失了NLRP7、NLRP8、NLRP11和NLRP13,但在NLRP1、NRLP4和NLRP9上发生了复制,其发生复制和平行扩张的都是与生殖相关的基因。与小鼠不同的是,牦牛NLRP家族中免疫相关基因缺失NLRP1和NLRP10,生殖相关基因缺失NLRP4、NLRP7、NLRP11和NLRP13,同时也没有鉴定出平行扩张的基因。物种间的比对结果显示,NLRP家族部分基因在进化过程中是保守的,如NLRP3(牦牛与人有84%相同,牦牛与小鼠有80%相同,人与小鼠有82%相同,牦牛与黄牛有99%相同);而部分生殖相关基因NLRP5(牦牛与人有71%相同,牦牛与小鼠有63%相同,人与小鼠有50%相同,牦牛与黄牛有99%相同)与NLRP14(牦牛与人有83%相同,牦牛与小鼠有70%相同,人与小鼠有62%相同,牦牛与黄牛有99%相同)序列在物种间的差异较大,这可能是生殖相关基因进化更为快速导致的。牦牛NLRP蛋白除了都含有NLRP蛋白家族独有的PYRIN、NACHT和LRR保守结构域,还预测出NOD2_WH和FISNA结构域(见图 6)。NOD2_WH这种有翼的螺旋结构在NOD2蛋白中被发现[37],但它的分子功能尚不清楚。FISNA结构域在鱼类和其他脊椎动物中存在,该域经常与NACHT域相关联[38],具体功能尚无报道。
一些蛋白在基因表达调控、生长发育、信号因子传递及细胞周期调控等生命过程中存在相互作用,这种不同蛋白之间的相互作用构成了蛋白互作网络。结果显示,牦牛NLRP2、NLRP3、NLRP6、NLRP12、NLRP14与均与参与炎症反应的蛋白PYCARD存在相互作用。PYCARD是凋亡相关斑点样蛋白,能够被NLRP3募集,并且与天冬氨酸蛋白水解酶-1(caspase-1)形成多蛋白聚合的炎症小体,它的作用是激活caspase-1,通过促进炎症因子IL-1β和IL-18的成熟和分泌,从而促进细胞凋亡,调节固有免疫应答[39]。NLRP2、NLRP6、NLRP14均与MLKL互作。MLKL是混合谱系激酶结构域样蛋白,相关研究表明在没有凋亡蛋白抑制剂(IAPs)和半胱氨酸的天冬氨酸蛋白水解酶-8(caspase-8)情况下,MLKL对于TLR诱导的NRLP3激活有重要作用[40]。NLRP5与TLE6、BMP15、FIGLA、NPM2、GDF9等参与生殖过程的蛋白互作,BMP15蛋白由卵母细胞衍生生长因子基因BMP15编码,在绵羊中发现该基因的自然突变可以以剂量敏感的方式导致排卵率和不育表型的增加[41]。另外,绵羊GDF9基因突变以类似于BMP15失活突变的方式导致排卵率增加和不孕,表明GDF9对绵羊正常卵泡发育至关重要[42]。
转录组分析结果显示牦牛NRLP基因家族成员在6个组织中的表达存在差异。免疫相关基因NLRP3在脾脏中表达较高,可能与脾脏作为最大的免疫器官有关。NLRP5在6个组织中表达,在脾脏中的表达量要高于其他组织(见图 8)。相关研究表明NLRP5在猪上的同源物在早期胚胎发育中起作用,受精卵中NLRP5的表达被抑制会导致胚胎的早期发育受到抑制[43]。在合子基因组激活前,NLRP5的转录物在母猪植入前胚胎中显著下调直到盒子基因组激活后检测不到任何表达,表明NLRP5基因是母猪早期胚胎发生所必需的,提示该基因可能在合子基因组激活中发挥重要作用。蛋白互作网络揭示NLRP5也与生殖细胞标记蛋白GDF9、BMP15、ZAR1等存在相互作用(见图 7)。NLRP8在牦牛脾脏和肝脏中检测到微弱表达,该基因在啮齿类动物基因组中缺失,在其他物种中其研究尚无报道。NLRP14只在肺脏和肾脏中检测到,相关研究表明其在卵子发生和早期胚胎发育中发挥重要作用,该基因的沉默会导致胚胎发育阻滞[45]。
从蛋白互作网络以及基因表达谱的结果来看,部分NLRP基因及蛋白的功能很难完全划分为免疫和生殖两部分,它们的功能是复杂的。从文献报道的结果来看,NLRP基因既可以在免疫方面发挥作用,又能够参与动物的生殖过程。NLRP7是与动物生殖相关的基因,但是也有研究发现该基因在先天免疫系统中也起作用,编码的蛋白同样能够招募ASC促进caspase-1激活和促进IL-1调节因子和IL-18的成熟[46]。NLRP9在此前研究中被认为是生殖相关基因,在小鼠卵巢中表达,参与合子基因组的激活[47]。此外,玉香等[48]在对NLRP9在仔猪组织中的表达、定位及在猪小肠上皮细胞(IPEC-J2)细胞中的功能研究中发现NLRP9参与了肠粘膜免疫过程,其表达主要定位于小肠上皮、睾丸和卵巢中。
4 结论通过同源比对和保守结构域搜索,在牦牛基因组中鉴定出牦牛8个NLRP家族成员,并且通过系统进化分析确定牦牛NLRP基因家族的分类和进化关系,并且通过生物信息学对牦牛NLRP基因家族的基因结构、理化性质、蛋白保守结构域和二级结构进行了预测分析。通过STRING数据库查询蛋白互作关系,进一步了解牦牛NLRP蛋白与其他蛋白之间的功能联系,为确定基因功能提供参考。最后利用转录组测序数据,初步展示了NLRP基因在牦牛不同组织中的表达情况,上述分析将为后续牦牛NLRP基因功能研究提供理论基础。
| [1] |
吴比. 小鼠Nlrp14基因敲除模型的建立[D]. 哈尔滨: 东北农业大学, 2013. WU Bi. Establishing Nlrp14 knock-out mouse model[D]. Harbin: Northeast Agricultural University, 2013. (  0) 0) |
| [2] |
CAO J Y, ZHOU L T, LI Z L, et al. Dopamine D1 receptor agonist A68930 attenuates acute kidney injury by inhibiting NLRP3 inflammasome activation[J]. Journal of Pharmacological Sciences, 2020, 143(3): 226-233. DOI:10.1016/j.jphs.2020.04.005 (  0) 0) |
| [3] |
HARRIS P A, DURAISWAMI C, FISHER D T, et al. High throughput screening identifies ATP-competitive inhibitors of the NLRP1 inflammasome[J]. Bioorganic & Medicinal Chemistry Letters, 2015, 25(14): 2739-2743. DOI:10.1016/j.bmcl.2015.05.032 (  0) 0) |
| [4] |
HA H J, KIM D S, HAHN Y. A 2.7-kb deletion in the human NLRP10 gene exon 2 occurred after the human-chimpanzee divergence[J]. Biochemical Genetics, 2009, 47(9/10): 665-670. DOI:10.1007/s10528-009-9262-2 (  0) 0) |
| [5] |
KUCHMIY A A, D'HONT J, HOCHEPIED T, et al. NLRP2 controls age-associated maternal fertility[J]. Journal of Experimental Medicine, 2016, 213(13): 2851-2860. DOI:10.1084/jem.20160900 (  0) 0) |
| [6] |
陈静, 刘芳, 霍建超, 等. NLRP家族中生殖相关基因的研究进展[J]. 畜牧与兽医, 2017, 49(6): 200-203. DOI: CNKI:SUN:XMYS.0.2017-06-04. CHEN Jing, LIU Fang, HUO Jianchao, et al. Research progress of NLRP reproduction related genes[J]. Animal Husbandry & Veterinary Medicine, 2017, 49(6): 200-203. DOI: CNKI:SUN:XMYS.0.2017-06-04. (  0) 0) |
| [7] |
彭辉. Nlrp基因家族的表达及Nlrp2和Nlrp4g在小鼠早期胚胎发育过程中的功能[D]. 杨凌: 西北农林科技大学, 2012. PENG Hui. Expression analysis of Nlrp gene family and function of Nlrp2 and Nlrp4g in early embryonic development in the mouse[D]. Yangling: Northwest A & F University, 2012. (  0) 0) |
| [8] |
林秀娇. Nlrp9b在小鼠早期胚胎发育中的表达、定位及功能[D]. 福州: 福建农林大学, 2015. LIN Xiujiao. The expression, localization and function of Nlrp8b gene in the early mouse embyro[D]. Fuzhou: Fujian Agriculture and Forestry University, 2015. (  0) 0) |
| [9] |
SLIM R, WALLACE E P. NLRP7 and the genetics of hydatidiform moles: Recent advances and new challenges[J]. Frontiers in Immunology, 2013, 4: 242. DOI:10.3389/fimmu.2013.00242 (  0) 0) |
| [10] |
李广栋, 崔炜, 田秀芝, 等. NLRP7基因在母源印记中的研究进展[J]. 中国畜牧兽医, 2018, 45(4): 1002-1008. LI Guangdong, CUI Wei, TIAN Xiuzhi, et al. Research progress on NLRP7 gene in maternal imprinting[J]. China Animal Husbandry & Veterinary Medicine, 2018, 45(4): 1002-1008. DOI:10.16431/j.cnki.1671-7236.2018.04.021 (  0) 0) |
| [11] |
胡泉军. 牦牛基因组数据库建设[D]. 兰州: 兰州大学, 2014. HU Quanjun. Database construction of yak genome[D]. Lanzhou: Lanzhou University, 2014. (  0) 0) |
| [12] |
石仙, 熊显荣, 兰道亮, 等. 牦牛CCND2基因的克隆及其在不同发情时期卵巢中的表达[J]. 中国农业科学, 2017, 50(13): 2604-2613. SHI Xian, XIONG Xianrong, LAN Daoliang, et al. Cloning and expression analysis of CCND2 gene in yak ovaries during different periods of estrus[J]. Scientia Agricultura Sinica, 2017, 50(13): 2604-2613. DOI:10.3864/j.issn.0578-1752.2017.13.018 (  0) 0) |
| [13] |
闫振龙. 牦牛睾丸促性腺激素受体和性激素受体增龄性变化与免疫调控研究[D]. 兰州: 甘肃农业大学, 2014. YAN Zhenlong. The inmine regulation study of gonadotropin receptor and hormone receptor in yak testis(Bos grunniens) at different ages[D]. Lanzhou: Gansu Agricultural University, 2014. (  0) 0) |
| [14] |
陈虹. 牦牛卵巢Label-free蛋白组学分析及相关蛋白在生殖系统的表达[D]. 兰州: 甘肃农业大学, 2018: 2604-2613. CHEN Hong. Analysis of label-free quantification proteomics in yak's ovary and expression of related proteins in the reproductive system[D]. Lanzhou: Gansu Agricultural University, 2018. (  0) 0) |
| [15] |
杨芳. 基于RNA-Seq对牦牛和犏牛睾丸转录组的比较分析[D]. 成都: 四川农业大学, 2017. YANG Fang. Comparative analysis of testis transcriptome for yak and cattleyak based on RNA-seq[D]. Chengdu: Sichuan Agricultural University, 2017. (  0) 0) |
| [16] |
CONSORTIUM U P. UniProt: a hub for protein information[J]. Nucleic Acids Research, 2015(D1): 204-212. DOI:10.1093/nar/gku989 (  0) 0) |
| [17] |
CAMACHO C, COULOURIS G, AVAGYAN V, et al. BLAST+: architecture and applications[J]. BMC Bioinformatics, 2009, 10(1): 421. DOI:10.1186/1471-2105-10-421 (  0) 0) |
| [18] |
LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. ClustalW and ClustalX version 2[J]. Bioinformatics, 2007, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 (  0) 0) |
| [19] |
SUDHIR K, GLEN S, LI M, et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology & Evolution, 2018(6): 6. DOI:10.1093/molbev/msy096 (  0) 0) |
| [20] |
GASTEIGER E, GATTIKER A, HOOGLAND C, et al. ExPASy: the proteomics server for in-depth protein knowledge and analysis[J]. Nucleic Acids Research, 2003, 31(13): 3784-3788. DOI:10.1093/nar/gkg563 (  0) 0) |
| [21] |
SignalP 5. 0 improves signal peptide predictions using deep neural networks[J]. Nature Biotechnology, 2019, 37: 420-423. DOI:10.1038/s41587-019-0036-z (  0) 0) |
| [22] |
PAUL H, KEUN-JOON P, TAKESHI O, et al. WoLF PSORT: protein localization predictor[J]. Nucleic Acids Research, 2007, 35(Web Server issue): 585-587. DOI:10.1093/nar/gkm259 (  0) 0) |
| [23] |
HU B, JIN J, GUO A Y, et al. GSDS 2.0: an upgraded gene feature visualization server[J]. Bioinformatics, 2014, 31(8): 1296. DOI:10.1093/bioinformatics/btu817 (  0) 0) |
| [24] |
MARCHLER-BAUER A, BRYANT S H. CD-Search: protein domain annotations on the fly[J]. Nucleic Acids Research, 2004, 32(S2): W327-W331. DOI:10.1093/nar/gkh454 (  0) 0) |
| [25] |
BATEMAN A, COIN L, DURBIN R, et al. The Pfam protein families database[J]. Nucleic Acids Research, 2004, 32(S1): D138-D141. DOI:10.1093/nar/gkh121 (  0) 0) |
| [26] |
BAILEY T L, MIKAEL B, BUSKE F A, et al. MEME Suite: tools for motif discovery and searching[J]. Nucleic Acids Research, 2009, 37: W202-W208. DOI:10.1093/nar/gkp335 (  0) 0) |
| [27] |
CHEN C, CHEN H, ZHANG Y, et al. TBtools - an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 (  0) 0) |
| [28] |
Geourjon C, Deleage G. SOPMA: significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J]. Computer Applications in the Biosciences, 1995, 11(6): 681-684. DOI:10.1093/bioinformatics/11.6.681 (  0) 0) |
| [29] |
SZKLARCZYK D, MORRIS J H, COOK H, et al. The STRING database in 2017: quality-controlled protein-protein association networks, made broadly accessible[J]. Nucleic acids research, 2016, D362-D368. DOI:10.1093/nar/gkw937 (  0) 0) |
| [30] |
TANG Q, GU Y, ZHOU X, et al. Comparative transcriptomics of five high-altitude vertebrates and their low-altitude relatives[J]. GigaScience, 6(12): 1-9. DOI:10.1093/gigascience/gix105 (  0) 0) |
| [31] |
CHEN S, ZHOU Y, CHEN Y, et al. fastp: an ultra-fast all-in-one FASTQ preprocessor[J]. Bioinformatics, 2018, 34(17): i884-i890. DOI:10.1093/bioinformatics/bty560 (  0) 0) |
| [32] |
WEN G. A simple process of RNA-sequence analyses by Hisat2, Htseq and DESeq2[C]// Proceedings of the 2017 International Conference on Biomedical Engineering and Bioinformatics. New York: ACM, 2017. DOI: 10.1145/3143344.3143354.
(  0) 0) |
| [33] |
罗荣松, 冯郁, 宋明民, 等. 小鼠Nlrp基因家族生物信息学分析[J]. 基因组学与应用生物学, 2018, 37(3): 1168-1176. LUO Rongsong, FENG Yu, SONG Mingmin, et al. Bioinformatics analysis of Nlrp gene family in rat[J]. Genomics and Applied Biology, 2018, 37(3): 1168-1176. DOI:10.13417/j.gab.037.001168 (  0) 0) |
| [34] |
TSCHOPP J, MARTINON F, BURNS K. NALPs: A novel protein family involved in inflammation[J]. Nature Reviews Molecular Cell Biology, 2003, 4(2): 95-104. DOI:10.1038/nrm1019 (  0) 0) |
| [35] |
彭辉, 张涌. NLRP蛋白家族的研究进展[J]. 中国畜牧杂志, 2014, 50(3): 90-93. PENG Hui, ZHANG Yong. Research progress of NLRP protein family[J]. China Academic Journal, 2014, 50(3): 90-93. DOI:10.3969/j.issn.0258-7033.2014.03.022 (  0) 0) |
| [36] |
彭辉, 张文昌, 肖天放, 等. Nlrp基因家族部分免疫相关基因在小鼠上的表达[J]. 福建农林大学学报(自然科学版), 2013, 42(4): 398-402. PENG Hui, ZHANG Wenchang, XIAO Tianfang, et al. Expression of immunization-related Nlrp genes in mouse[J]. Journal of Fujian Agriculture and Forestry University(Natural Science Edition), 2013, 42(4): 398-402. DOI:10.3969/j.issn.1671-5470.2013.04.013 (  0) 0) |
| [37] |
MAEKAWA S, OHTO U, SHIBATA T, et al. Crystal structure of NOD2 and its implications in human disease[J]. Nature Communications, 2016, 7: 11813. DOI:10.1038/ncomms11813 (  0) 0) |
| [38] |
STEIN C, CACCAMO M, LAIRD G, et al. Conservation and divergence of gene families encoding components of innate immune response systems in zebrafish[J]. Genome Biology, 2007, 8(11): R251. DOI:10.1186/gb-2007-8-11-r251 (  0) 0) |
| [39] |
李艳蝶, 卢美萍. NLRP3炎症小体与儿童自身炎症性疾病研究进展[J]. 浙江大学学报(医学版), 2017, 46(4): 449-453. LI Yandie, LU Meiping. Progress on the study of NLRP3 inflammasome in autoinflammatory disease of children[J]. Journal of Zhejiang University(Medical Sciences), 2017, 46(4): 449-453. DOI:10.3785/j.issn.1008-9292.2017.08.17 (  0) 0) |
| [40] |
LAWLOR K E, KHAN N, MILDENHALL A, et al. RIPK3 promotes cell death and NLRP3 inflammasome activation in the absence of MLKL[J]. Nature Communications, 2015, 6(1): 1-19. DOI:10.1038/ncomms7282 (  0) 0) |
| [41] |
GALLOWAY S M, MCNATTY K P, CAMBRIDGE L M, et al. Mutations in an oocyte-derived growth factor gene (BMP15) cause increased ovulation rate and infertility in a dosage-sensitive manner[J]. Nature Genetics, 2000, 25(3): 279-283. DOI:10.1038/77033 (  0) 0) |
| [42] |
HANRAHAN J P. Mutations in the genes for oocyte-derived growth factors GDF9 and BMP15 are associated with both increased ovulation rate and sterility in Cambridge and Belclare sheep (Ovis aries)[J]. Biology of Reproduction, 2004, 70(4): 900-909. DOI:10.1095/biolreprod.103.023093 (  0) 0) |
| [43] |
PENG Hui, LIU Fang, LI Wenhao, et al. Knockdown of NLRP5 arrests early embryogenesis in sows[J]. Animal Reproduction Science, 2015, 163: 151-156. DOI:10.1016/j.anireprosci.2015.11.004 (  0) 0) |
| [44] |
MICHIHARU H, KIRKMAN N J, MAYO K E, et al. The mouse germ-cell-specific leucine-rich repeat protein NALP14: a member of the NACHT nucleoside triphosphatase family[J]. Biology of Reproduction, 2005(4): 879. DOI:10.1095/biolreprod.104.033753 (  0) 0) |
| [45] |
XIN T, PASCAL G, MONGET P. Evolution and functional divergence of NLRP genes in mammalian reproductive systems[J]. BMC Evolutionary Biology, 2009, 9(1): 202. DOI:10.1186/1471-2148-9-202 (  0) 0) |
| [46] |
KHARE S, DORFLEUTNER A, BRYAN N B, et al. An NLRP7-containing inflammasome mediates recognition of microbial lipopeptides in human macrophages[J]. Immunity, 2012, 36(3): 464-476. DOI:10.1016/j.immuni.2012.02.001 (  0) 0) |
| [47] |
刘芳, 江宁, 肖琼, 等. Nlrp9b在小鼠不同组织中的表达和定位[J]. 中国兽医杂志, 2015, 51(12): 30-32+50. DOI: CNKI:SUN:ZSYZ.0.2015-12-009. LIU Fang, JIANG Ning, XIAO Qiong, et al. Expression and localization of NLRP9b gene in mouse different tissues[J]. Chinese Journal of Veterinary Medicine, 2015, 51(12): 30-32+50. DOI: CNKI:SUN:ZSYZ.0.2015-12-009. (  0) 0) |
| [48] |
玉香. NLRP9在仔猪组织中的表达、定位及在IPEC-J2细胞中的功能研究[D]. 福州: 福建农林大学, 2018. YU Xiang. Expression and localization of NLRP9 in piglet tissues and its function in IPEC-K2 cells[D]. Fuzhou: Fujian Agriculture and Forestry University, 2018. (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


