2. 战略支援部队特色医学中心 普通外科,北京 100101;
3. 清华长庚医院 肝胆胰治疗中心,北京 102218;
4. 战略支援部队特色医学中心 病理科,北京 100101
2. Department of General Surgery, Strategic Support Force Medical Center, Beijing 100101, China;
3. The Center for Hepatopancreatobiliary Diseases, Beijing Tsinghua Changgung Hospital, Beijing 102218, China;
4. Department of Pathology, Strategic Support Force Medical Center, Beijing 100101, China
胰腺癌是消化系统中一种高度恶性的肿瘤,胰腺导管腺癌(Pancreatic ductal adenocarcinoma, PDAC)是最常见的病理类型,预后极差。近年来随着检测和治疗手段的进步,病人的确诊时间提前,预后略有改善,但五年生存率仍低于8%[1-2]。根据美国癌症协会2019年发布的数据,胰腺癌相关的死亡率在恶性肿瘤中排第四位[2]。亦有数据估计,PDAC将在2030年成为第二大引起死亡的肿瘤疾病[3]。手术切除是实现胰腺癌患者治愈和长期生存的唯一有效途径。然而,由于胰腺癌发病隐匿,起始阶段往往缺乏特定的临床症状和指标,且恶性程度高、进展迅速,大多数患者在入院时即处于晚期阶段。大量的研究表明,基因突变和异常表达与PDAC的癌变和进展密切相关,包括KRAS基因的突变、抑癌基因(TP53, CDKN2A, SMAD4)的异常激活、以及诸如表皮生长因子受体等基因的异常表达[4]。然而,这些异常表达的基因在胰腺癌的发生和发展过程中的确切机制仍不清楚。随着人类基因组研究的不断进展,越来越多的具有独特功能的RNA被发现。其中非编码RNA(No coding RNAs, ncRNAs)作为多种疾病发生中不容忽视的重要因素,近年来已成为基因组研究的热点。常见的ncRNA包括miRNAs、lncRNAs、pseudogenes和circRNAs[5]。竞争性内源RNA(ceRNA)假说认为ncRNAs与mRNAs之间存在调控机制,具体表现为lncRNAs、假基因和circRNAs可以与mRNA竞争性结合共享的miRNAs结合位点而形成闭环联系[6]。越来越多的证据表明,ncRNAs可作为肿瘤基因的启动子或抑制子来发挥致癌作用[7]。此外,ncRNAs的研究价值还在于其是否可作为肿瘤疾病诊断和预后的有效标志物[8]。如果能对新的指标进行监测并作出早期诊断和干预,胰腺癌患者的预后将得到很大的改善。本文拟通过生物信息学方法并基于ceRNA的调控机制分析与胰腺导管腺癌临床预后的相关的调控网络,以期为胰腺癌的诊治提供新的思路。
1 材料和方法 1.1 微阵列数据胰腺癌mRNA的微阵列数据是从GEO数据库获得的,具体的检索式为:((("genes"(MeSH Terms) OR gene(All Fields)) OR ("rna, messenger"(MeSH Terms) OR mRNA(All Fields))) AND ("pancreatic neoplasms"(MeSH Terms) OR pancreatic cancer(All Fields))) OR ("Pancreatic Carcinoma"(All Fields) OR pancreatic carcinoma (All Fields))。对数据集的标题和摘要进行详细的阅读和筛选后,选择(GSE62165, GSE55643和GSE62452) 3个数据集进行后续的数据处理。这3个数据集的具体信息为:
(1) GSE62165, Affymetrix GPL13667 platform, Affymetrix Human Genome U219 Array。该数据集共包含131个组织样本,其中有13个正常样本和118个PDAC样本。
(2) GSE55643, Affymetrix GPL6480 platform, Agilent-014850 Whole Human Genome Microarray 4×44K G4112F (Probe Name version)。共包含53个组织样本,其中有8个正常样本和45个PDAC样本。
(3) GSE62452, Affymetrix GPL6244 platform, Affymetrix Human Gene 1.0 ST Array (transcript (gene) version)。共包含130个组织样本,其中有61个正常样本和69个PDAC样本。
1.2 差异表达分析使用GEO2R的在线工具对PDAC样本与健康对照样本基因表达数据进行处理并筛选出DEGs。首先,将三个数据集中的样本分为两组:正常组(“Nor”,正常组织样本)和癌症组(“Can”,癌症组织样本)。然后,对“Nor”和“Can”组的基因表达数据进行差异表达分析。|log2 FC|>1(FC=Fold change)和P < 0.05作为设定值来确定基因是否出现差异表达。为了进一步减少因测序平台的不同带来的影响,对3个数据集中的差异表达基因取交集并绘制Venn图。
1.3 蛋白互作网络的构建使用STRING(Search Tool for the Retrieval of Interacting Genes)来对差异基因之间的关联性进行预测和分析(score > 0.4 (medium confidence)被认为是基因之间有关联)[9]。然后使用Cytoscape来实现蛋白互作网络可视化绘制[10]。Cytoscape软件中的MCODE(Molecular Complex Detection)插件可基于拓扑学和给定的数据进行网络聚类分析。具体参数为:MCODEscores>5, degree cut-off=2, node score cut-off=0.2, Max depth=100, and k-score=2.
1.4 DEGs基因富集分析基因本体数据库(Gene Ontology, GO)是目前可以对特定基因进行功能注释的重要生物信息学数据库。同时,京都基因和基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes, KEGG)作为可对基因进行代谢通路富集分析的数据库而被广泛使用[11]。在本研究中对DEGs进行了GO功能注释和KEGG代谢通路富集分析。在GO功能注释中,在生物学过程(BP)、细胞成分(CC)和分子功能(MF)层面进行功能注释。在GO功能注释和KEGG代谢通路富集分析中得分前10位的数据在图中进行了展示。
1.5 GEPIA数据库分析GEPIA数据库可对来自TCGA和GTEx的9 736个肿瘤和8 587个正常样本的RNA测序数据进行分析[12]。本研究中使用GEPIA数据库来进行验证GEO数据库中筛选获得的DEGs在TCGA数据库中的表达情况。
1.6 Kaplan-Meier生存分析本研究中使用了Kaplan-Meier生存数据来评估候选基因是否可作为胰腺癌患者的预后指标[13]。首先,在Kaplan-Meier数据库中检索DEGs。然后根据指定基因的中位表达值将所有胰腺癌患者分为低表达组和高表达组,并在生成的生存曲线图中对比两个分组的患者的生存状况。同时计算出危险比(hazard ratio, HR)、95%置信区间(95% confidence interval, CI)、log-rank P值。
1.7 PROGgene数据库分析PROGgene数据库常被用于研究不同基因在不同类型的癌症中的预后价值[14]。在数据库中对DEGs和候选miRNAs进行检索并指定癌症类型为PDAC。然后根据标题和摘要进行筛选,以进一步评估特定基因的生存价值。
1.8 miRNAs和lncRNAs在ENCORI数据库预测和分析利用ENCORI数据库(也称StarBase数据库)预测LAMA3上游潜在的miRNAs和lncRNAs[15]。该数据库可同时使用七种数据库(PITA、RNA22、miRmap、microT、miRanda、PicTar和Targetscan)的数据来对潜在的miRNAs和lncRNAs进行预测。将同时被三个以上的数据库中预测到的miRNAs作为进一步研究的对象。此外,该数据库还可对miRNAs和基因在32种癌症的表达情况进行泛癌分析。因此,还使用StarBase数据库对miRNA和特定基因之间的相关性进行分析以进一步筛选miRNA-gene pairs。R < -0.1和P < 0.05被设定为具有统计学意义的参考值。
1.9 miRNet数据库分析鉴于lncRNAs、假基因和circRNAs可以与mRNA竞争性结合共享的miRNAs结合位点而形成闭环联系[6]。试图对hsa-miR-20b-5p上游的lncRNAs进行筛选,以解释其通过ceRNA机制在胰腺导管腺癌的发病于进展中的作用。miRNet数据库被用来预测并筛选预测hsa-miR-20b-5p的上游的潜在lncRNAs[16]。然后,把结果与从StarBase数据库获得的lncRNAs取交集,以更准确预测可能对hsa-miR-20b-5p起到调节作用的lncRNAs。
1.10 Lnc ATLAS数据库分析为了进一步对筛选获得的lncRNAs进行亚细胞定位,在本研究中将从不同数据库中筛选获得的lncRNAs输入到lncATLAS数据库进行检索[17]。只有定位于细胞质中lncRNAs并在五种以上不同的细胞类型中表达才可被选择用于后续的分析。
2 结果分析 2.1 筛选与胰腺癌相关的差异表达基因在筛选与PDAC相关的关键差异表达基因的过程中,使用GEO数据库的GEO2R在线分析工具对GSE62165、GSE55643和GSE62452数据集的数据进行了处理和差异表达分析。3个数据集的组织样本均被分为两组:“Nor”组和“Can”组。筛选的DEGs(见图 1a、1b和1c)。在数据集GSE62165中,共计获得了6 887个DEGs,包括4 697个上调基因和2 190个下调基因。在对数据集GSE55643的对照组和癌症组的比较中,1 968个和716个基因分别出现上调和下调。同时数据集GSE62452共计有211个上调基因和120个下调基因。为了进一步筛选与胰腺导管腺癌发生或进展相关的潜在基因,对在三个数据集中获得的DEGs取了交集。最终获得了83个上调的基因和25个下调的基因,共计108个基因在上述三个数据集中均出现差异表达(见图 1d)。这108个DEGS也被筛选确定为关键差异表达基因,并进行后续的处理和分析。
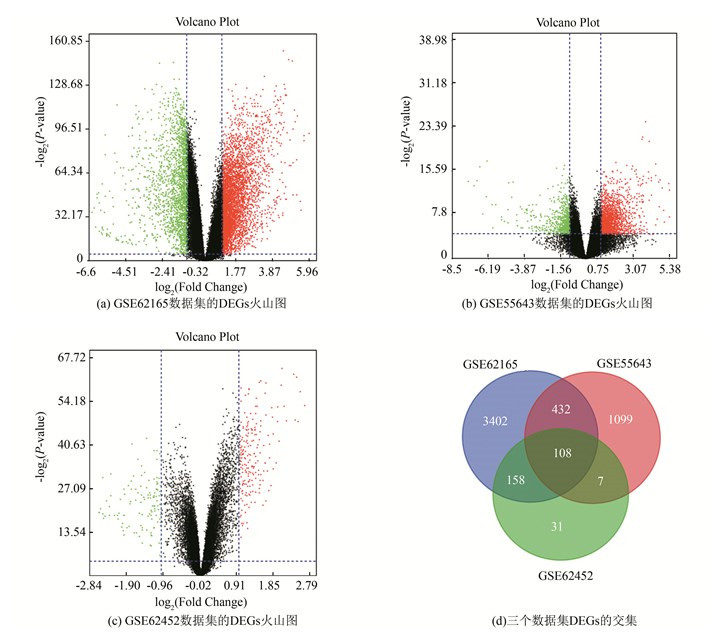
|
图 1 PDAC的差异表达基因的鉴定 Figure 1 Screening of differentially expressed genes (DEGs) in PDAC 注:黑点代表癌症组织与正常组织之间的表达无显著差异,绿点和红点分别代表三个数据集中下调和上调的基因(胰腺癌vs.正常样本). |
为进一步探究关键DEGs在胰腺癌中的作用,首先使用STRING软件构建了DEGs的蛋白互作网络(Protein-Protein Interaction network, PPI)(见图 2a),并且在Cytoscape中使用MCODE插件对相互作用的蛋白进一步筛选并对结果进行优化。最终对检测到的两个关键集团(分别涉及7个基因和3个基因)进行了蛋白互作网络数据的可视化(见图 2c, 2d)。然后,又对DEGs进行了GO功能注释和KEGG通路富集分析,以进一步分析这些差异基因的潜在功能。在生物过程(BP)、细胞成分(CC)、分子功能(MF)三个层面中富集程度最高的10种GO功能(见图 2e~2g)。结果表明,在BP分类中细胞与基质间的连接组装、半桥粒的装配和中胚层形成等功能出现显著变化。金属氨基肽酶的活性、阴离子-阳离子同向转运体的活性以及NAD(P)H的活动度等在MF分类中均出现明显变化。而在CC分类中,功能的的变化主要集中于质膜的内容物、质膜外侧面的固有成分等方面。KEGG通路富集分析表明,DEGs主要富集在细胞外基质-受体相互作用、黏着斑、PI3K-Akt信号通路和癌症通路等通路中(见图 2h)。为了进一步对候选的差异表达基因进行筛选,对其具有的多重功能进行分析和整理并通过KEGG通路进行展示(见图 2b)。有6个基因(LAMA3、LAMB3、LAMC2、ITGA2、ITGA3和ITGB4)在参与到多种代谢通路中。考虑到它们在PDAC的肿瘤发生中所起的潜在关键作用,这些基因被视为候选基因在3个数据集中的表达量及其变化(见表 1)。

|
图 2 PPI网络构建及DEGs的GO功能和KEGG通路富集分析 Figure 2 Construction of PPI network and GO function and KEGG pathway enrichment analyses of DEGs 注:(a)中浅红色节点为上调基因,浅蓝色结节为下调的基因. |
| 表 1 关键候选基因在胰腺癌组织和正常组织之间的表达变化 Table 1 Expression variation of key candidate genes in pancreatic cancer and normal controls |
为进一步发掘这些关键候选基因的价值并提高分析的准确性,引入了TCGA和GTEx数据库中胰腺癌及健康对照的样本的数据对候选基因进行验证,并在GEPIA数据库中确认了6个候选基因的表达。结果表明:与健康对照组相比,肿瘤组织中六个候选基因(LAMA3、LAMB3、LAMC2、ITGA2、ITGA3和ITGB4)的表达量均升高(见图 3)。这与从GEO数据库三个数据集获得的结果一致。然后,对六个候选基因进行了生存分析。首先使用了TCGA泛癌数据中的胰腺癌来评估候选基因作为预后指标的价值(包括Overall Survival, OS和Recurrent-free Survival, RFS)。结果表明:高表达的LAMA3和ITGA3的PDAC患者的生存率较差(见图 4a),但没有证据表明其他基因的高表达可以提供更好的生存。对于PDAC患者的RFS,LAMA3、LAMC2和ITGA3与生存恶化有关,而没有任何基因可以作为改善预后的有前途的生物标志物(见图 4b)。通过上述数据的相互验证,可以认为在所有差异表达的候选基因中,LAMA3和ITGA3在癌症组织中的高表达可作为患者具有较差的预后的有效的基因标志物(生存分析结果详见图 4c~4f)。随后,又使用PROGgeneV2数据库的数据扩大样本量,进一步评估LAMA3和ITGA3的预后价值。高表达水平的LAMA3仅在数据集GSE21501中提示患者具有较差的预后(见图 4g)。然而,GSE21501以及GSE57495数据集中的数据均无法支持ITGA3可作为预后不良的指标(见图 4h~4j)。综上所述,高表达水平的LAMA3可能是胰管腺癌发生和进展的关键因素,导致较差的预后。
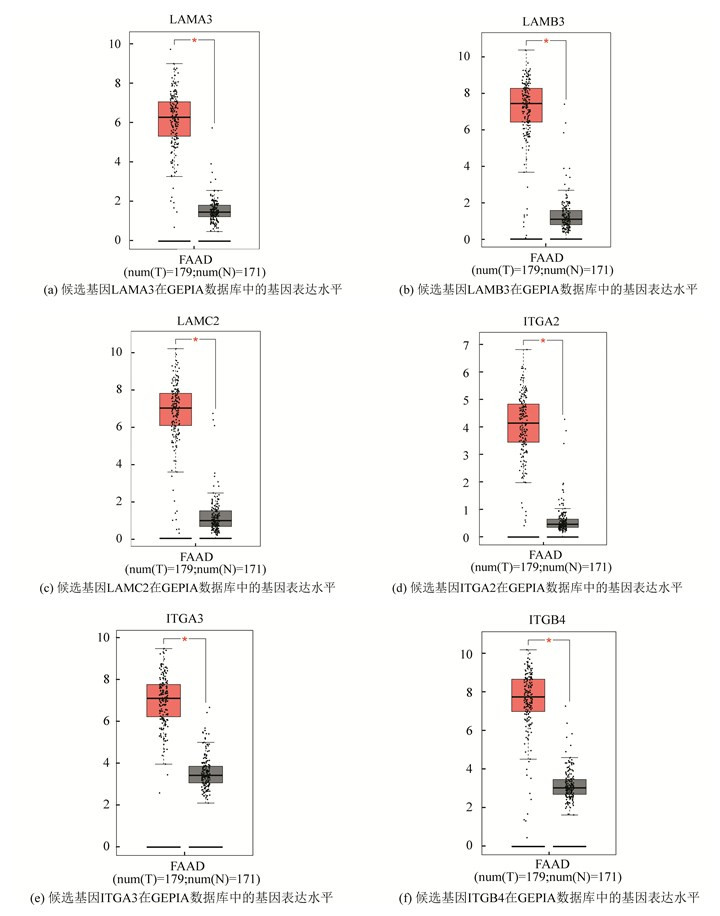
|
图 3 筛选获得的6个关键候选基因在GEPIA数据库中的基因表达水平 Figure 3 Expression levels of six key genes in pancreatic cancer calculated and plotted by GEPIA database 注:红色背景代表肿瘤组织,灰色背景代表正常对照组织; “*”表示“P-value < 0.05”,Y轴为基因表达量,TPM=Transcript per million. |
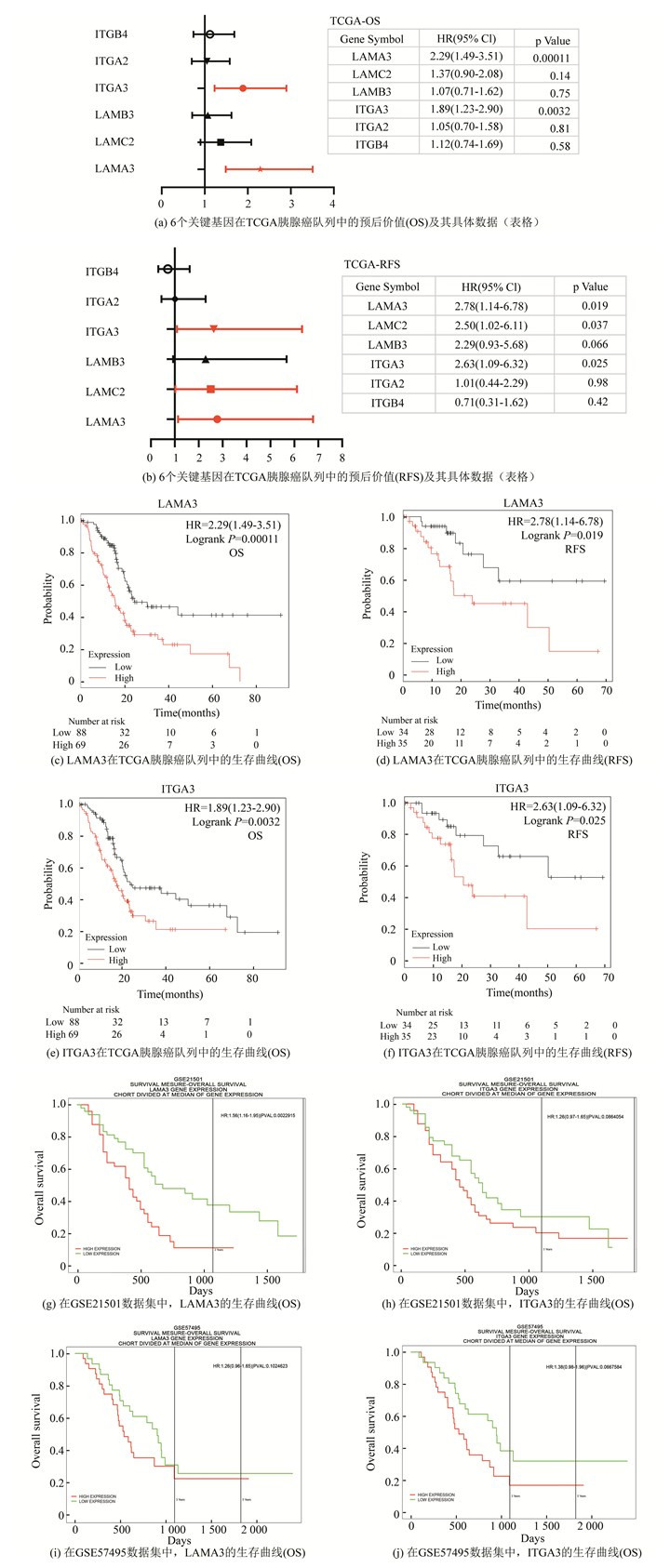
|
图 4 Kaplan-Meier数据库和PROGgeneV2数据库对6个候选基因在胰腺癌中的预后价值的评估 Figure 4 Assessment of prognostic values of six key genes in pancreatic cancer using Kaplan-Meier database and PROGgeneV2 database 注:(a)中红色条带表示预后不良;黑色条带表示没有统计学意义;(g~j)中红色线条表示基因高表达水平患者的生存数据,绿色线条代表基因低表达水平患者的生存数据;Log-rank P < 0.05代表具有显著性差异. |
越来越多的证据表明,miRNAs通过负调控相关基因的表达在癌症的发生和发展等生物学过程中起着关键作用。因此,使用Starbase数据库对LAMA3的上游miRNAs进行了预测和筛选。各数据库对LAMA3上游miRNAs的预测情况(见表 2)。最终筛选获得了21个可能对LAMA3起到调控作用的miRNAs。为了更好地可视化,miRNA-LAMA3关联网络(见图 5a)。然后, 使用TCGA的泛癌数据进一步评估这些miRNAs的预后价值。在所有预测到的miRNAs中,hsa-miR-520a-3p和hsa-miR-20b-5p的高表达提示PDAC患者具有良好的OS。其余的miRNAs的高表达均提示PDAC患者的不良预后,而hsa-miR-93-5p、hsa-miR-20a-5p、hsa-miR-17-5p和hsa-miR-106b-5p则无统计学意义(见图 5b)。根据LAMA3的功能和其可能的致癌作用,推测其上游的miRNAs应当属于抑制性miRNA。因此,我们又对hsa-miR-520a-3p-LAMA3和hsa-miR-20b-5p-LAMA3之间的相关性进行了分析。在PDAC中只有hsa-miR-20b-5p与LAMA3呈显著的负相关(见图 5c~5f)。根据上述分析,hsa-miR-20b-5p-LAMA3轴可能在PDAC的发生和进展过程中起到关键作用。
| 表 2 LAMA3上游潜在miRNAs的预测 Table 2 Results of miRNAs targeting to LAMA3 using different prediction programs |

|
图 5 结合miRNA的生存数据及其与LAMA3的相关性,筛选LAMA3的上游潜在的miRNAs Figure 5 Screening of potential miRNAs targeting to LAMA3 based on survival-associated miRNAs and correlation analysis 注:(b)中绿色条带表示预后良好;红色条带表示预后不良;黑色条带表示没有统计学意义。OS的详细信息在(b)中的表格中进行展示。 |
ncRNA之一的lncRNAs与miRNAs之间存在的竞争机制使其可以在细胞质中mRNA结合而影响其正常功能的行使。因此首先利用Starbase和miRNet数据库来预测hsa-miR-20b-5p上游潜在的lncRNAs。miRNet和StarBase数据库分别预测出了74种和79种lncRNAs(见图 6a)。对这两个数据库预测的lncRNAs取交集后,共有12个lncRNAs被两个数据库同时预测到(SNHG16、LINC00839、XIST、HCP5、RP11-226L15.5、HOTAIR、FGD5-AS1、EPB41L4A-AS1、HCG18、MALAT1、H19和AC105760.2)。然后,使用GEPIA数据库来验证这些lncRNAs在PDAC患者中的表达水平。12种lncRNAs中,只有3种(HCP5、FGD5-AS1和H19)在肿瘤组织中的表达水平明显高于健康对照组(见图 6c~6e)。考虑到lncRNAs与miRNA之间的相互作用的场所应当是细胞质,使用lncATLAS数据库来实现上述三种lncRNA的细胞内定位。只有FGD5-AS1定位于多种细胞系的细胞质中(见图 6f)。综上所述,可以推测FGD5-AS1通过与hsa-miR-20b-5p的竞争性结合导致LAMA3的表达升高,从而在胰腺癌的发生和进展中发挥重要作用。FGD5-AS1-hsa-miR-20b-5p-LAMA3的竞争机制(见图 6b)。
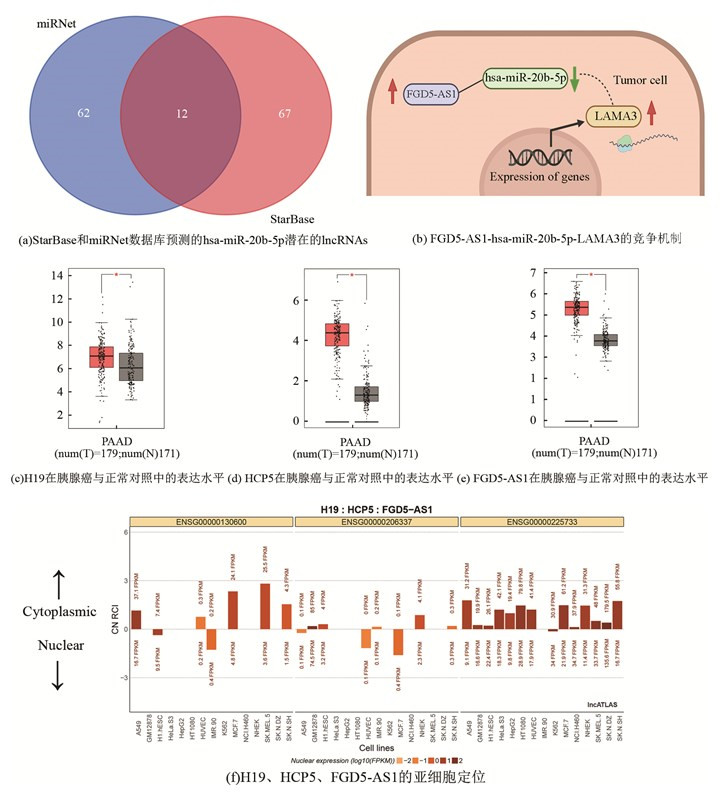
|
图 6 胰腺癌中hsa-miR-20b-5p上游潜在的lncRNAs的鉴定 Figure 6 Screening of upstream potential lncRNAs of hsa-miR-20b-5p in pancreatic cancer 注:(b~d)中“*”表示“P-value < 0.05”,Y轴为基因表达量,TPM=Transcript per million. |
根据美国癌症协会2019年公布的数据,胰腺癌引起的死亡率在恶性肿瘤中排名第四[2]。到2030年,PDAC的发病率将会持续增加并有可能成为所有肿瘤疾病中的第二大死因[4]。现有的临床和流行病学研究已然证实吸烟、慢性胰腺炎、肥胖和糖尿病等是胰腺癌的发病过程中重要的独立危险因素[18]。但不幸的是这些危险因素在胰腺癌发生过程中的具体分子机制仍不清楚。此外,胰腺癌由于进展迅速、早期即可出现转移,因此多数病人确诊时已失去手术干预的机会,因此预后极差。当前,临床工作迫切地需要新的有效的生物标志物来为诊断或治疗提供思路。微阵列技术的出现和运用已在多种疾病中识别出独特的生物标志物,这也进一步明确了PADC的基因改变。
在本研究中,通过对三个GEO数据集中胰腺癌和癌旁正常组织的基因表达数据的处理和分析,筛选得到了108个DEGs,其中83个基因上调、25个基因下调。通过对上述DEGs进行GO功能和KEGG通路富集分析后发现,这些异常表达的基因主要集中在细胞-基质间的连接、细胞外基质(ECM)及其受体之间的相互作用和黏着斑等方面。肿瘤微环境在肿瘤的进展过程中起着至关重要的作用,这一点已被广泛接受。更重要的是,ECM作为肿瘤微环境的重要成员,已被证实在多种肿瘤发展的各个阶段发挥着重要的作用。此外,由于驻留在ECM的细胞之间可以相互交流并作出相应的改变,使得ECM的成分和状态经常出现波动。与ECM中胶原纤维密度小于5%的患者相比,胶原纤维增加的患者罹患乳腺癌的风险更高[19-20]。此外,细胞外基质成分的不稳定会导致ECM刚度的变化,而ECM刚度的增加会导致基质重塑和细胞迁移[21]。ECM重塑在多种上皮来源的恶性肿瘤中进展过程中的不利影响已经被证实[22-23]。换言之,ECM的成分的异常表达可能会导致ECM的刚度增加,进而使ECM出现重塑并促进细胞迁移和肿瘤的进展。为了进一步获得在胰腺癌发生和进展过程中的关键基因,对上述DEGs在TCGA数据库中的表达水平进行了验证。最终获得了6个高表达的关键候选基因。然后对这6个关键基因进行了生存分析。结果表明LAMA3的高表达往往提示PDAC患者较差的预后。因此,LAMA3被视为可能与胰腺癌的发生和进展有关的最重要的基因。LAMA3是编码层粘连蛋白的基因,已被证实参与多种肿瘤的进展。层粘连蛋白是ECM分子和基底膜的主要成分,与Ⅳ型胶原纤维连接蛋白共同形成。LAMA3的低表达会导致基底膜的形成受阻,导致肿瘤细胞黏附减少,最终肿瘤细胞的转移[24]。同时,LAMA3的过表达已被证明影响多种肿瘤细胞的侵袭和转移[25]。此外,周等人证实LAMA3在结直肠癌组织中出现异常表达,并提出LAMA3的检测有助于结直肠癌的早期诊断[26]。陈等人进一步研究发现胰腺导管腺癌患者过表达的层粘连蛋白α3, β3和γ2链往往提示患者较差的预后[27]。综上所述,LAMA3可能是胰腺癌发生和发展的关键的基因。
ncRNA中的miRNA可通过调控靶基因的表达而在多种生物过程中发挥关键作用[28]。根据miRNA的作用机制,其缺失会导致相关蛋白表达的增加。使用在线数据库预测到了靶向LAMA3的21个潜在的miRNAs。鉴于miRNAs是通过与mRNA的3’端非翻译区域(3’-UTR)的互补序列相结合来发挥调控基因表达的作用,因此这些潜在的miRNAs应当在胰腺癌中发挥抑制作用。生存分析的结果提示hsa-miR-520a-3p-LAMA3和hsa-miR-20b-5p-LAMA3可能在胰腺癌中起重要作用。对他们在胰腺癌中表达量的相关性分析的结果表明,hsa-miR-20b-5p与LAMA3具有显著的负相关。许多研究表明,hsa-miR-20b-5p可能是多种癌症进展中的重要抑制因子。Cheung等证实人宫颈上皮内瘤变(CIN)患者hsa-miR-20b-5p的有助于宫颈癌的发展和进展,并可作为区分晚期CIN和正常宫颈上皮的生物标志物[29]。Huang等人还提示hsa-miR-20b-5p可作为食管鳞状细胞癌的新的血清生物学指标[30]。在Ulivi等人的一项研究中经贝伐单抗治疗后,血清hsa-miR-20b-5p水平升高可以改善转移性结直肠癌的OS和RFS[31]。这些研究结果侧面印证了hsa-miR-20b-5p-LAMA3轴可能在胰腺癌发展中的关键作用。为此,进一步探索了miRNAs的上游lncRNA,并通过两个在线数据库获得了12个潜在的lncRNA。利用GEPIA数据库测定胰腺癌组与健康对照组间差异表达基因。与健康对照组相比,12种潜在的lncRNAs中只有3种(HCP5、FGD5-AS1、H19)显著升高。亚细胞定位分析表明,只有FGD5-AS1定位于细胞质中。FGD5-AS1已被多项研究证实参与到多种类型的癌症的发展。例如,FGD5-AS1通过hsa-miR-302e促进CDCA7的表达,促进大肠癌细胞的增殖、迁移和侵袭[32]。通过hsa-miR-107增强非小细胞肺癌细胞增殖,促进FGFRL1的表达[33]。此外,在口腔癌患者中FGD5-AS1更是被视作精准治疗的靶向分子[34]。更重要的是,现有的研究发现FGD5-AS1可能通过调控miR-520a-3p/KIAA1522轴使得胰腺癌细胞增殖能力增强[35]。这些结果支持FGD5-AS1在胰腺癌中可能起关键作用的假设。结合ceRNA的机制我们提出了FGD5-AS1-hsa-miR-20b-5p-LAMA3调控轴可能在胰腺癌的发生和发展过程中具有关键作用。
4 结论基于多重生物信息学的分析表明,hsa-miR-20b-5p-LAMA3的相互作用可能会通过增加细胞外基质的刚度在胰腺癌的进展中发挥重要作用。此外,成功地鉴定了has-miR-20b-5p-LAMA3轴的潜在上游lncRNA(FGD5-AS1)。更重要的是,结果表明FGD5-AS1-has-miR-20b-5p-LAMA3调控网络可能作为胰腺癌临床治疗的新靶点和预后的生物标志物。
5 数据使用声明本研究分析的数据集获取自数据库The Cancer Genome Atlas (TCGA, https://portal.gdc.cancer.gov/) 和The Gene Expression Omnibus (GEO, https://www.ncbi.nlm.nih.gov/geo/)。
| [1] |
OETTLE H, NEUHAUS P, HOCHHAUS A, et al. Adjuvant chemotherapy with gemcitabine and long-term outcomes among patients with resected pancreatic cancer: The CONKO-001 randomized trial[J]. JAMA, 2013, 310(14): 1473-1481. DOI:10.1001/jama.2013.279201 (  0) 0) |
| [2] |
SIEGEL R L, MILLER K D, JEMAL A. Cancer statistics, 2019[J]. A Cancer Journal for Clinicians, 2019, 69(1): 7-34. DOI:10.3322/caac.21551 (  0) 0) |
| [3] |
RAHIB L, SMITH B D, AIZENBERG R, et al. Projecting cancer incidence and deaths to 2030: The unexpected burden of thyroid, liver, and pancreas cancers in the United States[J]. Cancer Research, 2014, 74(11): 2913-2921. DOI:10.1158/0008-5472.Can-14-0155 (  0) 0) |
| [4] |
ARDITO C M, GRVNER B M, TAKEUCHI K K, et al. EGF receptor is required for KRAS-induced pancreatic tumorigenesis[J]. Cancer Cell, 2012, 22(3): 304-317. DOI:10.1016/j.ccr.2012.07.024 (  0) 0) |
| [5] |
EDDY S R. Non-coding RNA genes and the modern RNA world[J]. Nature Reviews. Genetics, 2001, 2(12): 919-929. DOI:10.1038/35103511 (  0) 0) |
| [6] |
SALMENA L, POLISENO L, TAY Y, et al. A ceRNA hypothesis: The Rosetta Stone of a hidden RNA language?[J]. Cell, 2011, 146(3): 353-358. DOI:10.1016/j.cell.2011.07.014 (  0) 0) |
| [7] |
TAN L, MAI D, ZHANG B, et al. PIWI-interacting RNA-36712 restrains breast cancer progression and chemoresistance by interaction with SEPW1 pseudogene SEPW1P RNA[J]. Molecular Cancer, 2019, 18(1): 9. DOI:10.1186/s12943-019-0940-3 (  0) 0) |
| [8] |
WANG W, LOU W, DING B, et al. A novel mRNA-miRNA-lncRNA competing endogenous RNA triple sub-network associated with prognosis of pancreatic cancer[J]. Aging (Albany NY), 2019, 11(9): 2610-2627. DOI:10.18632/aging.101933 (  0) 0) |
| [9] |
SZKLARCZYK D, GABLE A L, LYON D, et al. STRING v11: Protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Research, 2019, 47(D1): D607-D613. DOI:10.1093/nar/gky1131 (  0) 0) |
| [10] |
SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks[J]. Genome Research, 2003, 13(11): 2498-2504. DOI:10.1101/gr.1239303 (  0) 0) |
| [11] |
KANEHISA M, FURUMICHI M, TANABE M, et al. KEGG: New perspectives on genomes, pathways, diseases and drugs[J]. Nucleic Acids Research, 2017, 45(D1): D353-D361. DOI:10.1093/nar/gkw1092 (  0) 0) |
| [12] |
TANG Z, LI C, KANG B, et al. GEPIA: A web server for cancer and normal gene expression profiling and interactive analyses[J]. Nucleic Acids Research, 2017, 45(W1): W98-W102. DOI:10.1093/nar/gkx247 (  0) 0) |
| [13] |
NAGY Á, LÁNCZKY A, MENYHÁRT O, et al. Validation of miRNA prognostic power in hepatocellular carcinoma using expression data of independent datasets[J]. Scientific Report, 2018, 8(1): 9227. DOI:10.1038/s41598-018-27521-y (  0) 0) |
| [14] |
GOSWAMI C P, NAKSHATRI H. PROGgeneV2: Enhancements on the existing database[J]. BMC Cancer, 2014, 14: 970. DOI:10.1186/1471-2407-14-970 (  0) 0) |
| [15] |
LI J H, LIU S, ZHOU H, et al. starBase v2.0: Decoding miRNA-ceRNA, miRNA-ncRNA and protein-RNA interaction networks from large-scale CLIP-Seq data[J]. Nucleic Acids Research, 2014, 42(Database issue): D92-D97. DOI:10.1093/nar/gkt1248 (  0) 0) |
| [16] |
FAN Y, HABIB M, XIA J. Xeno-miRNet: A comprehensive database and analytics platform to explore xeno-miRNAs and their potential targets[J]. PeerJ, 2018, 6: e5650. DOI:10.7717/peerj.5650 (  0) 0) |
| [17] |
MAS-PONTE D, CARLEVARO-FITA J, PALUMBO E, et al. LncATLAS database for subcellular localization of long noncoding RNAs[J]. Rna, 2017, 23(7): 1080-1087. DOI:10.1261/rna.060814.117 (  0) 0) |
| [18] |
MOORE A, DONAHUE T. Pancreatic cancer[J]. JAMA, 2019, 322(14): 1426. DOI:10.1001/jama.2019.14699 (  0) 0) |
| [19] |
MCCORMACK V A, DOS SANTOS SILVA I. Breast density and parenchymal patterns as markers of breast cancer risk: A meta-analysis[J]. Cancer Epidemiology, Biomarkers & Prevention, 2006, 15(6): 1159-1169. DOI:10.1158/1055-9965.Epi-06-0034 (  0) 0) |
| [20] |
BOYD N F, GUO H, MARTIN L J, et al. Mammographic density and the risk and detection of breast cancer[J]. The New England Journal of Medicine, 2007, 356(3): 227-236. DOI:10.1056/NEJMoa062790 (  0) 0) |
| [21] |
BARRIGA E H, FRANZE K, CHARRAS G, et al. Tissue stiffening coordinates morphogenesis by triggering collective cell migration in vivo[J]. Nature, 2018, 554(7693): 523-527. DOI:10.1038/nature25742 (  0) 0) |
| [22] |
MORIGGI M, GIUSSANI M, TORRETTA E, et al. ECM remodeling in breast cancer with different grade: Contribution of 2D-DIGE proteomics[J]. Proteomics, 2018, 18(24): e1800278. DOI:10.1002/pmic.201800278 (  0) 0) |
| [23] |
BURGESS J K, MAUAD T, TJIN G, et al. The extracellular matrix - the under-recognized element in lung disease?[J]. The Journal of Pathology, 2016, 240(4): 397-409. DOI:10.1002/path.4808 (  0) 0) |
| [24] |
JIN L, LIM M, ZHAO S, et al. The metastatic potential of triple-negative breast cancer is decreased via caloric restriction-mediated reduction of the miR-17~92 cluster[J]. Breast Cancer Research and Treatment, 2014, 146(1): 41-50. DOI:10.1007/s10549-014-2978-7 (  0) 0) |
| [25] |
MOLLER-LEVET C S, BETTS G N, HARRIS A L, et al. Exon array analysis of head and neck cancers identifies a hypoxia related splice variant of LAMA3 associated with a poor prognosis[J]. PLoS Computational Biology, 2009, 5(11): e1000571. DOI:10.1371/journal.pcbi.1000571 (  0) 0) |
| [26] |
ZHOU D, YANG L, ZHENG L, et al. Exome capture sequencing of adenoma reveals genetic alterations in multiple cellular pathways at the early stage of colorectal tumorigenesis[J]. PLoS One, 2013, 8(1): e53310. DOI:10.1371/journal.pone.0053310 (  0) 0) |
| [27] |
CHEN J, ZHANG H, LUO J, et al. Overexpression of α3, β3 and γ2 chains of laminin-332 is associated with poor prognosis in pancreatic ductal adenocarcinoma[J]. Oncology Letters, 2018, 16(1): 199-210. DOI:10.3892/ol.2018.8678 (  0) 0) |
| [28] |
LEE Y S, DUTTA A. MicroRNAs in cancer[J]. Annual Review of Pathology, 2009, 4: 199-227. (  0) 0) |
| [29] |
CHEUNG T H, MAN K N, YU M Y, et al. Dysregulated microRNAs in the pathogenesis and progression of cervical neoplasm[J]. Cell Cycle, 2012, 11(15): 2876-2884. DOI:10.4161/cc.21278 (  0) 0) |
| [30] |
HUANG Z, ZHANG L, ZHU D, et al. A novel serum microRNA signature to screen esophageal squamous cell carcinoma[J]. Cancer Medicine, 2017, 6(1): 109-119. DOI:10.1002/cam4.973 (  0) 0) |
| [31] |
ULIVI P, CANALE M, PASSARDI A, et al. Circulating plasma levels of miR-20b, miR-29b and miR-155 as predictors of bevacizumab efficacy in patients with metastatic colorectal cancer[J]. International Journal of Molecular Sciences, 2018, 19(1): 307. DOI:10.3390/ijms19010307 (  0) 0) |
| [32] |
FAN Y, LI H, YU Z, et al. Long non-coding RNA FGD5-AS1 promotes non-small cell lung cancer cell proliferation through sponging hsa-miR-107 to up-regulate FGFRL1[J]. Bioscience Reports, 2020, 40(1): BSR20193309. DOI:10.1042/bsr20193309 (  0) 0) |
| [33] |
LI D, JIANG X, ZHANG X, et al. Long noncoding RNA FGD5-AS1 promotes colorectal cancer cell proliferation, migration, and invasion through upregulating CDCA7 via sponging miR-302e[J]. In Vitro Cellular & Developmental Biology. Animal, 2019, 55(8): 577-585. DOI:10.1007/s11626-019-00376-x (  0) 0) |
| [34] |
LIU L, ZHAN Y, HUANG Y, et al. LncRNA FGD5-AS1 can be predicted as therapeutic target in oral cancer[J]. Journal of Oral Pathology & Medicine, 2020, 49(3): 243-252. DOI:10.1111/jop.12989 (  0) 0) |
| [35] |
LIN J, LIAO S, LIU Z, et al. LncRNA FGD5-AS1 accelerates cell proliferation in pancreatic cancer by regulating miR-520a-3p/KIAA1522 axis[J]. Cancer Biology Therapy, 2021, 22(3): 257-266. DOI:10.1080/15384047.2021.1883184 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


