2. 黑龙江省医院,哈尔滨 150036
2. Heilongjiang Provincial Hospital, Harbin 150036, China
背景:WHO国际癌症研究机构发布的2020年全球最新癌症负担的数据显示,全世界有超过193万人被新确诊为结直肠癌,占全球新确诊癌症人数的9.7%;在2020年,中国有超过55万人新患结直肠癌,占中国新确诊癌症人数的12.2%[1]。因此,深人了解结直肠癌的研究新进展,对结直肠癌的早诊断、早治疗、早预防具有重要意义[1]。结直肠癌主要是由良性腺瘤息肉引起的肿瘤,然后通过多步突变发展成腺癌;每位患者的肿瘤表现出2至8个驱动程序突变[2]。
腺瘤性息肉病大肠杆菌基因(Adenomatous polyposis coli,APC)突变是腺瘤形成的关键分子步骤[2]。从腺瘤到结直肠癌的发展是一个多步骤过程,涉及k-ras和p53基因的突变以及DNA甲基化使DNA表达沉默等。许多环境因素可能会通过调节这些分子途径来增加患结直肠癌的风险。处于晚期且无法治愈的阶段的结直肠癌通常可产生临床发现,而癌前腺瘤性息肉和早期可高度治愈的结直肠癌通常无症状。这种现象使腺瘤或早期癌症难以通过临床表现进行检测。近年来,NGS在临床肿瘤学中的应用不断拓宽。结合循环肿瘤DNA (CirculatingTumor DNA,ctDNA),NGS逐渐成为肿瘤研究治疗中的强力工具,在癌症的早筛早诊、分子分型、预后预测、疗效监测起着重要指导作用。在临床实践中通过单次测序即可同时获得与结直肠癌有关的多个基因序列或全基因组信息,从而有助于临床决策的制定。在这篇综述中,总结了结直肠癌的驱动突变基因,重点是在结直肠癌患者中发现基因突变与临床表型的关系。
1 NGS技术与结直肠癌中的驱动突变基因癌变过程中,细胞内调控网络受到干扰,使得细胞重编程,最终驱动不受控制的细胞增殖,许多重要的细胞进程被干扰,从而导致癌变,其中,体细胞和生殖系突变是最主要的原因。调查显示:在癌症中,大约90%的癌症基因发生体细胞突变,而20%的发生种系突变,有10%同时有体细胞和种系突变[3]。体细胞突变是致癌过程中的关键,也是致癌过程的核心。不同类型的突变通常是由于暴露于不同类型的环境致癌物而引起的,识别体细胞突变使我们能够更多地了解结直肠癌的发生和发展,但更重要的是,它有助于我们优化结直肠癌患者的治疗方法,甚至找到新的治疗方法[4]。
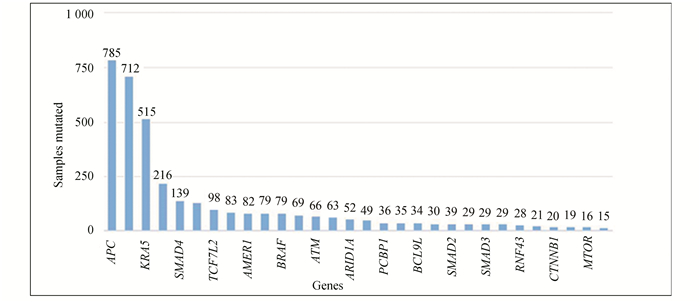
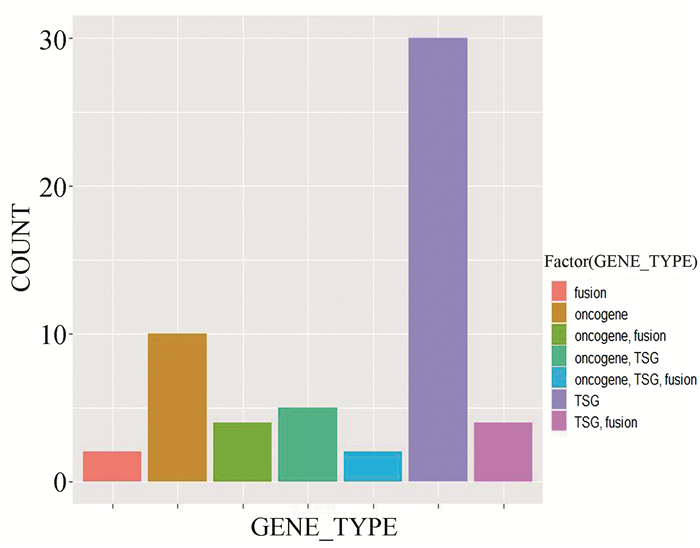
目前美国国家综合癌症网络(National Comprehensive Cancer Network,NCCN)指南已明确认可使用NGS技术检测结直肠癌患者中KRAS/NRAS/BRAF三基因的突变情况;所有转移性结直肠癌患者均应进行RAS(KRAS和NRAS)和BRAF突变的基因分型,可以单独检测,也可以作为一个NGSpanel的一部分进行检测[5]。另外整合癌症基因组数据库(IntOgen,https://www.intogen.org/search)提供识别到的各种肿瘤类型中的驱动突变,其中已明确72个结直肠癌驱动突变基因,最常见的15个驱动突变基因在数据库中所有结直肠癌患者中的突变频数(见图 1)。癌基因数据库(Cancer Gene Census,https://cancer.sanger.ac.uk/census)数据库中每个基因根据其体细胞突变特征以及其在致癌过程中的作用被分为原癌基因,抑癌基因以及融合基因三类。如果一个基因被划分为致癌基因,则表明该基因产物能够驱动肿瘤,肿瘤样本中的该基因突变会导致功能获得;如果一个基因被确定为抑癌基因,则表明野生型的基因产物具有肿瘤抑制功能,而肿瘤中该基因的突变会导致功能丧失;对于融合基因,其通常是由两个不同基因基因组结构重排导致产生新的嵌合基因;数据库目前收录的结直肠癌中的体细胞突变基因有59个,其分类情况(见图 2)。在线人类孟德尔遗传数据库(OMIM,https://www.omim.org/),侧重于疾病表型与其致病基因之间的关联,包括了现在所有已知的遗传病和超过15000个基因的信息,其中已收录55个与结直肠癌表型相关的体细胞突变基因(见表 1)。
|
图 1 IntOgen数据库中结直肠癌最常见的15个驱动突变基因 Figure 1 Fifteen most common driver mutation genes in colorectal cancer in IntOgen database |
|
图 2 Cancer Gene Census数据库中结直肠癌相关的59个突变基因的分类情况 Figure 2 Classification of 59 mutated genes related to colorectal cancer in Cancer Gene Census database |
| 表 1 OMIM数据库中与结直肠癌表型相关的体细胞突变基因 Table 1 Somatic mutations related to colorectal cancer phenotypes in OMIM database |
NGS逐渐成为肿瘤分子诊断的有力工具, 在结直肠癌基因突变检测中表现出巨大的临床价值[6]。大量研究强调了靶向NGS的重要性,比如研究者使用多重PCR靶向富集结合NGS技术对17例结直肠癌病例和14例正常样本的MMR基因进行检测,在外显子区域共发现15种突变,与Sanger测序验证结果一致,而NGS更节约时间和成本,说明该技术适合MMR基因突变筛查[7]。另外基于NGS技术建立的结直肠癌共识分子亚型(CMS)分型体系,对于结直肠癌的治疗及预后检测具有更加精确的指导意义[8]。随着NGS技术的不断发展,越来越多的研究将用来明确NGS应用于结直肠癌突变基因检测的可行性、准确性和实用性。
2 基因突变作为结直肠癌诊断的肿瘤标志物目前虽然有乙状结肠肠镜检查等用于结肠癌的筛检,但发现较晚,因此不适合结肠癌的早期诊断;血液肿瘤标志物的检测是结肠癌早期诊断中最方便、最常用的方法之一,例如癌胚抗原(Carcinoembryonic antigen,CEA)是目前临床实验室用于诊断结肠癌的重要肿瘤标志物,但仍有40% 左右的患者CEA呈阴性,因此有必要寻找其他的早期诊断结肠癌的较敏感和特异的方法。研究证实在肿瘤形成过程中,一些和细胞恶性转变有关的蛋白,如p62、p53等能诱导机体自身抗体的产生,而且这些自身抗体往往早在临床症状之前出现[9]。因此对这些肿瘤相关性抗原(Tumor-associated antigen,TAA)突变状态和表达水平的检测与肿瘤特异性抗原检测在结直肠癌的诊疗中有着同等重要的作用。
当前普遍研究观点认为结直肠癌是在多种基因的共同作用下发生发展的,例如KRAS、BRAF、PIK3CA、PTEN等基因[10];临床常用的结直肠癌诊断的分子标志物,包括KRAS/NRAS、BRAF突变、微卫星不稳定性(Microsatellite instability,MSI)等[1];NCCN和美国病理学家学院以及其他协会也推荐:MMR /MSI状态、BRAF p.V600 E和RAS突变检测可预测结直肠癌风险和化学疗法的疗效[8]。
此外,一些基因的高突变率也在结直肠癌中出现,如腺瘤性息肉病大肠杆菌(APC)突变是家族性腺瘤性息肉病(FAP)的标志,而FAP是最常见的遗传性结直肠癌综合征;RAS、SRC和MYC等致癌基因的体细胞突变已被证明与散发性结直肠癌的发生有关,且RAS的突变(HRAS,KRAS,NRAS)在50%的结直肠癌散发病例中均被发现;另一方面,肿瘤抑制基因在结直肠癌进程中的作用也不容忽视,其表现为双等位缺失,如APC 5q21基因、TP53 17p基因和DCC/SMAD2-4 18q基因。更为重要的是,错配修复基因(MMR)突变也将失去其间接抑制肿瘤的发生的作用,从而导致结直肠癌发生的高风险,如hMSH2,hMLH1、hPMS1、hPMS2,hMSH6和hMLH3等基因[8]。总之,结直肠癌的发生是一系列基因突变累积的结果,癌症基因组图谱计划显示,结直肠癌不仅在众所周知的肿瘤相关基因(例如APC,TP53,KRAS,PIK3CA,SMAD4)上发生突变,而且在其他基因中也具有突变,包括SMAD2,CTNNB1,FAM123B,SOX9和ARID1A等[11],更有最新的研究结果显示TP53,APC,KRAS,BRAF和ATM突变覆盖了结直肠癌大部分的患者[12]。
目前,随着NGS技术的不断发展,更多的研究者也总结出早期结直肠癌中可能出现的高突变基因,如MYH基因的突变会引起减毒的腺瘤性息肉病综合征,其表型与APC突变引起的减毒的腺瘤性息肉病相似,并且有发生结直肠癌的高风险;P53基因是从晚期腺瘤过渡到早期癌症的关键基因,其突变与结直肠癌的发生密不可分[13]。POLE和POLD1是DNA合成、修复过程中两个重要的关键酶,它们可以确保基因组的正确复制,若POLE和POLD1基因的核酸外切酶区域发生突变将导致DNA错配修复的缺失,进而导致大量突变的累积,同样代表着形成肿瘤的高风险性[10]。
3 不同结直肠癌亚型的基因突变特征 3.1 黏液腺癌与非黏液腺癌结直肠癌按照组织学分型可分为腺癌和腺鳞癌以及未分化癌。大多数结直肠癌是腺癌,约占四分之三;也有研究者按照结直肠癌组织学分型标准将结直肠癌分为非黏液性腺癌(管状腺癌、筛状粉刺型腺癌、髓样癌、锯齿状腺癌、微乳头状癌等)、黏液性腺癌(黏液腺癌、印戒细胞癌)、神经内分泌肿瘤(神经内分泌瘤、神经内分泌癌、混合型腺神经内分泌癌)、鳞癌特殊类型结直肠癌(透明细胞癌、梭形细胞癌等等)和未分化癌[14]。所有结直肠癌的5%至15%符合黏液腺癌的标准;当超过50%的病变由细胞外黏液池组成时,肿瘤被定义为黏液性腺癌。有研究发现,与黏液微卫星稳定(Microsatellite stabilization,MSS)的结直肠腺癌相比,TP53在非黏液MSS结直肠腺癌中的突变频率更高。BRAF、KRAS、SMAD4突变更常见于黏液MSS结直肠腺癌;微卫星不稳定(Microsatellite instability,MSI)黏液和非黏液结直肠腺癌之间的BRAF、KRAS、SMAD4突变率没有差异。与非黏液组相比,黏液组中有五个黏蛋白糖蛋白基因(MUC)的体细胞突变发生率更高,如MUC5B,MUC6,MUC14,MUC16和MUC18基因[4]。这些基因组突变可能在对黏液腺癌治疗和预后的反应差异中起重要作用。
3.2 CMS分型近年来,越来越多的研究发现肿瘤病人的血清中存在有各种类型的抗细胞自身抗原的抗体,也就是与肿瘤发生有关的TAA。其不仅在在监控肿瘤的发生发展中起着重要作用,这些由自身免疫产生的抗体对于肿瘤的免疫治疗以及免疫分型也提供了临床指导。结直肠癌是一种基因异质性疾病,肿瘤的发生、生长和进展可能涉及多种不同的分子途径。研究者通过对不同平台获得的结直肠癌分子基因表达谱进行全面的评估和比较,建立了基于肿瘤和肿瘤周边浸润性基质基因表达的共识分子亚型(CMS)分型体系[8, 10]。根据CMS分型体系,主要将结直肠癌分为4种共识分子亚型。(1) CMS1(微卫星不稳定性免疫分型,约占14%):一种“超级突变”肿瘤,通常具有微卫星高度不稳定(MSI-H)的特性,存在着BRAF突变和明显的免疫浸润,通常与复发后存活率降低有关;(2)CMS2(常见的分型,约占37%):通常表现的特征为WNT/MYC信号通路激活;(3)CMS3 (代谢分型,约占13%):通常表现的特征为代谢通路的失常、KRAS基因突变;(4)CMS4(间叶细胞亚型,约占23%):通常表现的特征为TGF-β通路的激活,血管再生、间质浸润及炎性浸润较其他亚型明显增强[10]。根据肿瘤微环境之间的差异,具有高度免疫细胞浸润的CMS1、CMS4亚型可归为“热”肿瘤,而CMS2、CMS3亚型因缺乏免疫细胞浸润被归为“冷”肿瘤[10]。CMS分类具有较好的预后价值,CMS1的预后最好,CMS4预后相对较差,CMS2 / 3预后介于两者之间[8]。不同的分子亚型具有不同的突变状态和不同的预后,为结直肠癌的诊疗提供了新的肿瘤标志物。
4 基因突变指导结直肠癌治疗手术切除是任何年龄的局部非转移性结直肠癌的主要传统的治疗方式,单独手术可以治愈结直肠癌Ⅰ期的患者,对于Ⅲ期疾病(淋巴结阳性),仅手术切除的5年生存率为20%~50%,因此建议辅助治疗。对于患有Ⅱ期疾病的患者,其5年生存率在50%~65%之间波动,但单纯手术治疗容易再次复发或发生转移[8]。随着NGS技术的不断发展,越来越多的靶向治疗方案已经运用到临床中,特别是在转移性结直肠癌的常规化疗中加入靶向药物可提高疗效。
目前对结直肠癌的治疗新策略,包括细胞毒性化学治疗剂和生物制剂,已导致人类结直肠癌的治疗取得重大进展。了解结直肠癌的遗传特征对于设计未来的治疗方法至关重要。例如,抗表皮生长因子受体(Epidermal growth factor receptor,EGFR)抗体和抗血管内皮生长因子抗体是目前结直肠癌化疗中的关键药物[15]。EGFR激活后引起下游RAS-RAF或PI3K-PTEN-AKT途径等多个信号通路的激活被认为是结直肠癌最常见的致癌机制;抗EGFR抗体能阻断其下游信号通路传导,从而发挥抗肿瘤的作用[15]。结直肠癌常用的靶向药物为EGFR的单克隆抗体西妥昔单抗和帕尼单抗[16]。近年的研究表明,对于抗EGFR抗体药物,野生型KRAS肿瘤的反应率要高于那些KRAS突变类型的肿瘤[16]。此外,具有BRAF(V600E)突变状态的肿瘤即使对KRAS野生型也显示出对抗EGFR药物的不良反应[17]。另一方面,索拉非尼是一种临床批准的针对多种受体酪氨酸激酶(包括BRAF)的多激酶抑制剂,可以潜在地恢复BRAF突变的结直肠癌对抗EGFR抗体治疗的敏感性[18]。EGFR下游信号通路中的其他基因突变如RAS,BRAF或PIK3CA以及无功能的PTEN也可以预测抗EGFR疗法的耐药性[19]。因此,检测与分析结直肠癌患者的KRAS、NRAS、BRAF、PIK3CA等多基因突变,能更准确地预测患者用抗EGFR单抗药物的疗效[15]。近年来也有研究发现,HER-2、AREG、EREG等非RAS通路的基因也与EGFR单克隆抗体治疗转移性结直肠癌的疗效相关[5, 20]。
NCCN指南也推荐在进行西妥昔单抗及帕尼单抗治疗前,需进行RAS及BRAF基因检测。专家组强烈建议所有转移性结直肠癌患者进行肿瘤组织RAS(KRAS/NRAS)基因分型(原发肿瘤或转移)。患有已知KRAS或NRAS突变的患者不应单独使用西妥昔单抗或帕尼单抗或与其他抗癌药联合治疗,因为它们几乎对治疗不敏感,且PIK3CA的突变以及非功能的PTEN基因突变均与上述单抗的疗效差有关[5]。2020年中国临床肿瘤学会更新了结直肠癌诊疗指南,其中也规定结直肠癌转移治疗的原则应该考量KRAS、NRAS、BRAF的基因状态;RAS和BRAF突变型与野生型的治疗方案不同,尤其指出BRAF突变患者使用抗EGFR治疗无显著获益[21-23]。
目前,局部晚期结直肠癌的标准疗法包括新辅助化疗(Neoadjuvant chemotherapy,NACT),但并非所有的患者都对新辅助化疗获益,肿瘤内异质性(Intratumoral heterogeneity,ITH)已被认为是化疗和放疗抵抗的机制。基于突变等位基因肿瘤异质性(Mutant-allele tumor heterogeneity,MATH)的NGS技术可作为衡量ITH的指标,为结直肠癌的靶向治疗提供潜在的生物标志物[24]。应用NGS检测局部晚期直肠癌患者ITH与NACT疗效之间的相关性,证明了较高的MATH评分与NACT较差的治疗反应有关。由此可见,术前使用NGS进行分子筛选,通过MATH值量化肿瘤异质性程度,便于临床医生为患者制定更加合理有效的治疗方案[24]。
5 基因突变作为结直肠癌预后的肿瘤标志物结直肠癌的发展是一个多步骤过程,是由遗传改变(包括染色体异常,基因突变和表观遗传学改变)的积累而引起的,且不同基因突变的结直肠癌接受治疗后可能会表现出不同的预后。
AJCC第八版结直肠癌分期系统规定,KRAS基因突变作为预后和预测因子的证据水平分别为Ⅰ级和Ⅱ级;NRAS基因突变作为预测因子的证据水平为Ⅱ级[25]。KRAS和NRAS基因是生长受体信号转导通路中的重要介导因子,控制细胞的增殖和存活,肿瘤细胞RAS基因突变后,EGFR能在不接收信号的情况下自动激活其下游信号转导通路,引起肿瘤细胞增殖,抑制凋亡;结直肠癌中KRAS基因突变的发生率约为40%,NRAS基因突变的发生率约为7%;KRAS和NRAS基因突变均提示Ⅲ至Ⅳ期结直肠癌预后不良[6, 25-26]。更重要的是,RAS基因突变预示进展期的结直肠癌的抗EGFR靶向治疗反应较差[25-26]。
AJCC第八版结直肠癌分期系统同样也规定BRAF基因突变作为预后和预测因素的证据等级分别为Ⅱ级和Ⅰ级[25]。BRAF蛋白是一种丝氨酸-苏氨酸激酶,负责将增殖信号从KRAS或NRAS蛋白传导到其他酶上,从而介导细胞的生长或增殖;6%~10%结直肠癌中可以检测到BRAF基因V600E位点的突变,BRAF基因突变能够激活其他酶以使肿瘤细胞持续生长,从而抵消抗EGFR靶向治疗阻止细胞增殖和生长的作用;BRAF V600E位点突变与不良的预后相关,尤其是能够阻止EFGR单克隆抗体对Ⅳ期结直肠癌的疗效[6, 25]。因此,BRAF基因突变也是抗EGFR靶向药物疗效的衡量标准之一[25-26]。
另外,最新的研究结果也显示了TP53,APC,KRAS,BRAF,ATM,PIK3CA等基因的突变形成了预测结直肠癌诊断和预后的肿瘤标志物[12]。对基因突变状态的识别有利于发现结直肠癌患者中预后差的人群,更加有针对性的提供临床治疗。
6 展望在这篇综述中,与结直肠癌相关的突变基因为结直肠癌的病理生理学的研究提供了新的见识,这些基因可能可以作为结直肠癌的基因检测标志物以及结直肠癌治疗的新型分子靶标。近年来,基因组学的进展拓宽了NGS技术从基础科研到临床实践中的应用。NGS技术不仅有助于阐明结直肠癌的发病机制,而且能够提供相关基因位点的突变信息,从而用于筛查高危人群,实现早期诊断,指导精准治疗,判断疗效及预后。在精准医学时代,相信随着高通量测序数据库的建立,NGS将会为结直肠癌提供更多更有效的基因突变标志物,为结直肠癌的诊疗提供参考。另外,结直肠癌的发生不仅受基因突变的影响,基因表达紊乱、表观遗传改变也是结直肠癌发生发展的高风险因素,对这些方面的进一步深入研究也将为结直肠癌治疗提供指导性见解。总之,随着实验技术和测序技术的发展,尤其是三代测序技术的出现,为我们提供了更加快速、精准的方法来检测基因状态,相信我们未来将会有更多更好的研究成果,以帮助临床医生提高结直肠癌的治疗效果,预防复发。
| [1] |
苏文雨, 房静远, 刘凯. 2020年结直肠癌的研究新进展[J]. 中华医学信息导报, 2021, 36(3): 12-13. SU Wenyu, FANG Jingyuan, LIU Kai. New progress in colorectal cancer research in 2020[J]. Chinese Medical Information Guide, 2021, 36(3): 12-13. DOI:10.3760/cma.j.issn.1000-8039.2021.03.107 (  0) 0) |
| [2] |
TESTA U, PELOSI E, CASTELLI G. Colorectal cancer: Genetic abnormalities, tumor progression, tumor heterogeneity, clonal evolution and tumor-initiating cells[J]. Medical Sciences(Basel, Switzerland), 2018, 6(2). DOI:10.3390/medsci6020031 (  0) 0) |
| [3] |
FUTREAL P A, COIN L, MARSHALL M, et al. A census of human cancer genes[J]. Nature Reviews. Cancer, 2004, 4(3): 177-183. DOI:10.1038/nrc1299 (  0) 0) |
| [4] |
REYNOLDS I S, O'CONNELL E, FICHTNER M, et al. Mucinous adenocarcinoma of the colon and rectum: A genomic analysis[J]. Journal of Surgical Oncology, 2019, 120(8): 1427-1435. DOI:10.1002/jso.25764 (  0) 0) |
| [5] |
BENSON A B, VENOOK A P, AL-HAWARY M M, et al. Colon cancer, Version 2.2021, NCCN clinical practice guidelines in oncology[J]. Journal of the National Comprehensive Cancer Network, 2021, 19(3): 329-359. DOI:10.6004/jnccn.2021.0012 (  0) 0) |
| [6] |
HIRSCH B, ENDRIS V, LASSMANN S, et al. Multicenter validation of cancer gene panel-based next-generation sequencing for translational research and molecular diagnostics[J]. Virchows Archiv, 2018, 472(4): 557-565. DOI:10.1007/s00428-017-2288-7 (  0) 0) |
| [7] |
黄凯, 陈慧杰, 刘方奇, 等. 多重PCR靶向富集结合高通量测序筛查结直肠癌中MMR基因的突变[J]. 复旦学报(医学版), 2016, 43(2): 205-210. HUANG Kai, CHEN Huijie, LIU Fangqi, et al. Multiple PCR targeted enrichment combined with high-throughput sequencing to screen MMR gene mutations in colorectal cancer[J]. Fudan University Journal of Medical Sciences, 2016, 43(2): 205-210. DOI:10.3969/j.jssn.1672-8467.2016.02.014 (  0) 0) |
| [8] |
RECIO-BOILES A, CAGIR B. Colon cancer[Z]. In: Statpearls: Treasure Island (FL), 2021.
(  0) 0) |
| [9] |
SOUSSI T. P53 antibodies in the sera of patients with various types of cancer: A review[J]. Cancer Research, 2000, 60(7): 1777-1788. (  0) 0) |
| [10] |
丁呈圣, 金志明. 结肠癌免疫检查点抑制剂研究进展[J]. 医学研究杂志, 2021, 50(1): 8-13. DING Chengsheng, JIN Zhiming. Research progress on immune checkpoint inhibitors for colon cancer[J]. Journal of Medical Research, 2021, 50(1): 8-13. DOI:10.11969/j.issn.1673-548X.2021.01.003 (  0) 0) |
| [11] |
GOTO T, MARUSAWA H, CHIBA T. Landscape of genetic aberrations detected in human colorectal cancers[J]. Gastroenterology, 2013, 145(3): 686-688. DOI:10.1053/j.gastro.2013.07.029 (  0) 0) |
| [12] |
ZHUANG Y, WANG H, JIANG D, et al. Multi gene mutation signatures in colorectal cancer patients: Predict for the diagnosis, pathological classification, staging and prognosis[J]. BMC Cancer, 2021, 21(1): 380. DOI:10.1186/s12885-021-08108-9 (  0) 0) |
| [13] |
CAPPELL M S. Pathophysiology, clinical presentation, and management of colon cancer[J]. Gastroenterology Clinics of North America, 2008, 37(1): 1-24. DOI:10.1016/j.gtc.2007.12.002 (  0) 0) |
| [14] |
杨军, 郭睿, 康安静, 等. 一种新的结直肠癌组织学分型、分级-评分方案[J]. 南方医科大学学报, 2014, 34(2): 169-173. YANG Jun, GUO Rui, KANG Anjing, et al. A new histological classification, grading and scoring scheme for colorectal cancer[J]. Journal of Southern Medical University, 2014, 34(2): 169-173. DOI:10.3969/j.issn.1673-4254.2014.02.05 (  0) 0) |
| [15] |
GUO F, GONG H, ZHAO H, et al. Mutation status and prognostic values of Kras, Nras, Braf and Pik3ca in 353 chinese colorectal cancer patients[J]. Scientific Reports, 2018, 8(1): 6076. DOI:10.1038/s41598-018-24306-1 (  0) 0) |
| [16] |
VAN CUTSEM E, KOHNE C H, HITRE E, et al. Cetuximab and chemotherapy as initial treatment for metastatic colorectal cancer[J]. The New England Journal of Medicine, 2009, 360(14): 1408-1417. DOI:10.1056/NEJMoa0805019 (  0) 0) |
| [17] |
LAURENT-PUIG P, CAYRE A, MANCEAU G, et al. Analysis of Pten, Braf, and Egfr status in determining benefit from cetuximab therapy in wild-type kras metastatic colon cancer[J]. Journal of Clinical Oncology, 2009, 27(35): 5924-5930. DOI:10.1200/JCO.2008.21.6796 (  0) 0) |
| [18] |
DI NICOLANTONIO F, MARTINI M, MOLINARI F, et al. Wild-type braf is required for response to panitumumab or cetuximab in metastatic colorectal cancer[J]. Journal of Clinical Oncology, 2008, 26(35): 5705-5712. DOI:10.1200/JCO.2008.18.0786 (  0) 0) |
| [19] |
THERKILDSEN C, BERGMANN T K, HENRICHSEN-SCHNACK T, et al. The predictive value of Kras, Nras, Braf, Pik3ca and Pten for Anti-Egfr treatment in metastatic colorectal cancer: A systematic review and meta-analysis[J]. Acta Oncologica (Stockholm, Sweden), 2014, 53(7): 852-864. DOI:10.3109/0284186X.2014.895036 (  0) 0) |
| [20] |
吴骁, 王冰一, 陈凯, 等. 二代测序法检测结肠癌基因突变与临床病理特征的关系[J]. 广西医科大学学报, 2017, 34(6): 828-831. WU Xiao, WANG Bingyi, CHEN Kai, et al. The relationship between the detection of colon cancer gene mutations and clinicopathological features by the second-generation sequencing method[J]. Journal of Guangxi Medical University, 2017, 34(6): 828-831. DOI:10.16190/j.cnki.45-1211/r.2017.06.008 (  0) 0) |
| [21] |
ZHANG Suzhan, LI Jin, CAI Sanjun, et al. Chinese society of clinical oncology (Csco) diagnosis and treatment guidelines for colorectal cancer 2018 (English Version)[J]. Chinese Journal of Cancer Research, 2019, 31(1): 117-134. DOI:10.21147/j.issn.1000-9604.2019.01.07 (  0) 0) |
| [22] |
YUAN Y, WANG X, CHEN G, et al. Updates in version 2019 of CSCO guidelines for colorectal cancer from version 2018[J]. Chinese Journal of Cancer Research, 2019, 31(3): 423-425. DOI:10.21147/j.issn.1000-9604.2019.03.03 (  0) 0) |
| [23] |
WENG S, YUAN Y, WANG X, et al. Updates in version 2020 of CSCO guidelines for colorectal cancer from version 2019[J]. Chinese Journal of Cancer Research, 2020, 32(3): 403-407. DOI:10.21147/j.issn.1000-9604.2020.03.11 (  0) 0) |
| [24] |
RAJPUT A, BOCKLAGE T, GREENBAUM A, et al. Mutant-allele tumor heterogeneity scores correlate with risk of metastases in colon cancer[J]. Clinical Colorectal Cancer, 2017, 16(3): e165-e170. DOI:10.1016/j.clcc.2016.11.004 (  0) 0) |
| [25] |
TONG G J, ZHANG G Y, LIU J, et al. Comparison of the eighth version of the american joint committee on cancer manual to the seventh version for colorectal cancer: A retrospective review of our data[J]. World Journal of Clinical Oncology, 2018, 9(7): 148-161. DOI:10.5306/wjco.v9.i7.148 (  0) 0) |
| [26] |
ROTH A D, TEJPAR S, DELORENZI M, et al. Prognostic Role of Kras and Braf in stage Ⅱ and Ⅲ resected colon cancer: Results of the translational study on the petacc-3, rortc 40993, sakk 60-00 trial[J]. Journal of Clinical Oncology, 2010, 28(3): 466-474. DOI:10.1200/JCO.2009.23.3452 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


