生物信息学是应用数学、信息学、统计学和计算机科学的方法研究生物学问题的学科。近年来,科学技术的进步极大地增强了我们获取生物学数据的能力,生物信息学也跨过后基因组时代[1],迈入大数据时代[2]。类型、内容、复杂度不断增加的生物学数据迫使生物信息学研究人员思考如何有效整合这些数据,用以研究复杂度更甚的生命系统运作机制。本文将针对大数据时代的整合生物信息学,从数据、方法、系统等不同层面进行梳理和探讨。
1 数据整合 1.1 生物学数据及生物信息学数据库不断扩张生物信息学的发展伴随着传统生物学数据(如物种基础数据、生理生化、性状遗传、环境资料等等)以及各类组学数据(如基因组、转录组、蛋白组、代谢组、表观组、表型组等等)的不断积累。这些数据不断被存放到相关的数据库中。目前已有数以万计的生物信息学数据库针对不同研究对象、领域方向整合了相应的原始/第二手数据。国际学术期刊《核酸研究》(Nucleic Acids Research)每年推出数据库专辑对当年新发表的较有影响力的生物信息学数据库进行收集整理;此外,Database、Bioinformatics等期刊也专门刊登生物信息数据库相关论文。整理过的生物学数据大多可以通过文献查阅与数据库检索挖掘实现数据的收集和整合。生物信息学领域主要的数据库和工具见图 1。

|
图 1 主要生物信息学数据库、工具(根据DaTo的统计分析结果提供,http://bis.zju.edu.cn/dato/) Figure 1 Main bioinformatics databases and tools (according to the statistical results of DaTo: http://bis.zju.edu.cn/dato/) |
除了传统的核酸序列数据库以外,其他各类较有影响力的数据库数据量也在不断增加,如GWAS atlas[3]总结了47 565个项目的数据;KEGG[4]收集的参考Pathway maps数量已达826 189;TCGA[5]数据库已产出了超过2.5 PB的基因组、表观基因组、转录组和蛋白组数据,大大提高了对肿瘤诊断、治疗以及预防的能力;ENCODE[6]收录了19 180种生物样本。我国的NONCODE[7]收录的动植物lncRNA转录本数量已分别达549 813和94 697。
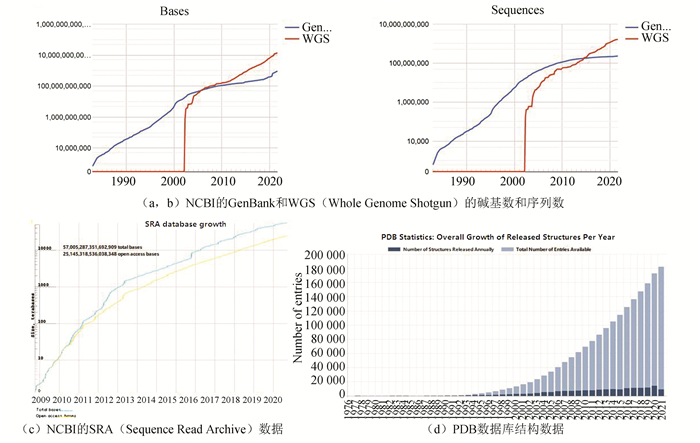
以三大传统核酸序列数据库(NCBI的GenBank[8]、EMBL-EBI的ENA[9]以及DDBJ[10])为代表的生物信息学数据库还在继续扩张中(见图 2)。
|
图 2 主要生物信息学数据库的数据统计(2021-09-18) Figure 2 Data statistics of main bioinformatics databases(2021-09-18) 注:(a, b)NCBI的GenBank和WGS(Whole Genome Shotgun)的碱基数和序列数https://www.ncbi.nlm.nih.gov/genbank/statistics/;(c)NCBI的SRA(Sequence Read Archive)数据https://trace.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?;(d)PDB数据库结构数据https://www.rcsb.org/stats/growth/growth-released-structures。 |
虽然我国的生物信息学数据库建设一开始落后于西方国家,但从2017年开始,我国每年的数据库发表数量(以论文计)已超越美国,位居全球第一。深圳华大生命科学研究院(原深圳华大基因研究院)于2011年承建的国家基因库(CNGB)目前已收录超过5PB的测序数据[11];北京基因组所于2015年建立了国内首个组学原始数据汇交、存储、管理与共享系统GSA(Genome Sequence Archive),目前数据量已突破10PB[12]。令人鼓舞的是,在2019年,由中国科学院北京基因组研究所(国家生物信息中心)作为依托单位,联合中国科学院生物物理研究所和中国科学院上海营养与健康研究所共同建设成立了国家基因组科学数据中心(NGDC),开启了我国国家级生物信息学科学数据汇聚与共享的发展征程。
随着生物信息学迈入大数据时代,飞速增长的生物学数据已远远超出了传统生物信息学方法的能力范围,生物组学大数据的数据挖掘与整合分析已成为当前生物信息学研究的新挑战。
1.2 生物大数据的多模态特性,数据格式及数据有偏处理生物信息学的产生主要是人类基因组计划带来的海量序列数据的结果,可见生物信息学依赖于生物学的发展,也受制于生物学研究所能产生的数据类型。生物信息学进入大数据时代后,生物学数据多模态的特性愈发突出。不同平台和类型的生物学数据的产生与积累涉及到数据的储存读写问题,数据保存在文件或记录中的规则不同就导致了不同的数据格式。主要的生物学数据类型及格式包括:文本型定性数据(如纯文本数据UTF-8,ASCII(.txt)、特定格式的序列相关文档格式(见表 1)、XML格式(.xml、.dtd)、HTML格式(.htm、.html、.xhtml)、JSON(JavaScript Object Notation)格式等);表格型定量数据(如制表符分隔的文件(.tab、.tsv)、电子表格Excel文件(.xls、.xlsx)、逗号分隔文件(.csv)等);图形图像数据(如光栅格式图像:tiff、png、jpeg、dng、bmp、gif等;矢量格式:SVG可缩放矢量图形、AutoCAD绘图交换格式、封装的PostScript、Adobe可移植文档格式等;制图数据geotiff、geopdf、geojpeg2000、shapefile、netcdf等;地理信息系统属性数据格式(.mdb)、mapinfo交换格式(.mif)等);数字音频数据(如免费无损音频编解码器FLAC、波形音频格式WAV、音频交换文件格式AIFF、MPEG音频层3等);数字视频数据(如MPEG-4、MOV、AVI、MXF等);以及各种实验测试观察数据,如光谱数据等表示轮廓以及峰位和强度的图JCAMP格式、测序原始数据(见表 1)、芯片原始数据CEL格式、结构测定格式(.hkl、.sca、.mtz、.pdb)等。如何有效挖掘与整合分析这些不同类型的数据,进而实现生物信息学的理论与应用研究,是整合生物信息学面临的主要问题之一。
| 表 1 序列文件格式 Table 1 File formats ofr sequences |
相较于传统生物学数据(如物种基础数据、生理生化、性状遗传、环境资料等),组学数据特有的大数据量、多尺度、高维度、异质性等特征给整合生物信息学分析带来了很大的挑战。譬如,由于测量技术限制,组学数据往往存在一定程度的“缺陷”(误差、缺失),必须利用数学、计算科学的方法处理。在生物数据整合过程中,需要将由不同来源的数据合并成一致的形式,因而会面临实体识别问题(Entity Identification Problem)和数据去冗余问题[13]。大量的组学数据还带来了存储与数据压缩问题,如文本型格式适用于单个或少量个体的基因组,但对大量个体基因组进行研究时,文本型格式需要的存储空间是海量的,因而需要改进存储方式,常见的方法是生成参考基因组并以SNP的形式存储个体基因组。
另外,对于复杂生物系统的测量总是存在偏差的,因此数据整合时必须要进行变换处理。数据变换包括很多个方面,而最常见就是归一化(Normalization),用于改进涉及距离度量的挖掘算法的精度和有效性。归一化是把数据映射到设定的区间,如(0,1)区间内。假设目前数据中的任一值x,归一化映射后的值为y,则
生物信息学的发展一直伴随着数据标准的提出与应用。以Hadoop文件格式标准为例,其包含文本文件、序列文件、基于行的格式、AVRO数据文件(数据定义以JSON格式存储,数据以二进制格式存储,最大限度地减小文件大小和提高效率)和拼花文件格式(数据按列存储)以及优化的行列(ORC)。JSON是一种简单的文件格式,可以被大多数编程语言简单地读取和处理;XML是一种被广泛应用于数据交换的格式,具有保留原始数据结构和文件构建方式的功能,并允许开发人员在不干扰数据读取的情况下使用数据编写部分文档。相较于XML格式,JSON格式在网络传输上带来了更大的便利,但可读性和查询速度要逊色于XML格式,较长的JSON格式文件会带来繁琐复杂的数据节点查找问题。关系数据格式(RDF)是知识图谱(Knowledge Graph)、语义网络(Semantic Network)、本体库(Ontology)数据的描述形式,描述了实体、属性、关系等,是文件形式的知识库存储方式。RDF数据可以存储在XML和JSON,以及其他序列化的数据结构中。
组学数据的标准也随着技术进步不断改进。以基因表达数据为例,基因芯片时期的数据标注标准MIAME[14](Minimum Information about a Microarray Experiment)规定了微阵列实验六个部分的信息描述,为基因表达数据库和公共仓库的建立,数据分析工具的开发建立基础。而对于测序数据,则有ENCODE[6]制定了数据标准化的分析方案和指南(https://www.encodeproject.org/data-standards/),提供了信息描述、实验指南、统一分析处理要求以及各种分析类型的质量指标应用等。
在系统生物学方面,SBML[15](Systems Biology Markup Language)已被越来越多的生物网络建模工具接受。BioModels数据库(https://www.ebi.ac.uk/biomodels/)收集了大量生物网络模型,基本上支持了SBML、CellML、Matlab等格式。而除了生物反应网络数据标准化交换格式外,SBGN(Systems Biology Graphical Notation)也致力于提供支持SBML的网络图像表示的可视化标准。
然而,上述标准仍不能单独覆盖所有生物数据实体,因此当前急需一个全面的标准化命名体系,用于统一命名方式。
2 方法整合 2.1 生物信息学主要方法(计算生物学)与其他学科一样,生物信息学也是一门寻找规律的学科,许多生物信息学研究可以归结为通过数据分析寻找相似与差异(“同与不同”),并建立预测模型的过程。统计学习(Statistical Learning)和机器学习(Machine Learning)是两类较为常见的模型构建方法,可用于序列比较和模式识别、表达谱聚类和生物标志物识别、生物网络的关联分析、医学图像自动识别及分子分型、人群队列疾病预测等问题[16]。严格来说,统计学习是统计学的子领域,而机器学习则是人工智能的子领域,统计学习强调模型及其可解释性,精度和不确定性;机器学习强调大规模应用和预测准确性。然而,这种区别正变得越来越模糊,许多生物信息学方法已经兼具两者特征。
由于生物大数据具有多模态、高维度的特点,在生物大数据中寻找相似与差异(“同与不同”)的方法不尽相同,而且常常会出现假阳性、假阴性的结果。差异分析除了直接的比较(生物学功能为依据)外,还有各种统计比较分析(p-value为依据),通过观察样本是否落在设定的各种统计分布区间判断其是否为异常值。预测模型的目标是找出实际数据与它们所属的类别之间的关系,当类别未知时,往往可使用无监督学习算法,让算法自行发现数据中的构成模式。此类研究可归结为降维(Dimension Reduction)、聚类(Clustering)、分类(Classification)、回归(Regression)四大问题。
由于生物学大数据,特别是组学数据特征数量庞大,如转录组测序数据基因数量可以万计,生物学大数据的维度非常高,其中包含了大量的冗余和噪声,对数据进行直接计算和存储所消耗的资源极为庞大,因此非常有必要对数据进行降维操作。降维算法可以去除数据中存在的冗余信息和噪声,抽取出最能代表数据分布的特征,可以在一定程度上提高学习算法的准确性和数据的可理解性。将其数据降至为二维或三维还有助于对数据进行可视化,以便于直观发现数据间存在的关系。常用的降维方法包括有基于特征选取的:缺少值比率(Missing Value Ratio)、低方差过滤器(Low Variance Filter)、高相关滤波器(High Correlation Filter)、随机森林(Random Forest)、后向特征消除(Backward Feature Elimination)和前向特征选择(Forward Feature Selection);基于组分因素的:因素分析(Factor Analysis)、主成分分析(Principal Component Analysis)、独立分量分析(Independent Component Analysis);依据基于映射(Projection)的:等距映射ISOMAP(Isometric Mapping)、t-SNE(t-distributed Stochastic Neighbor Embedding)和UMAP(Uniform Manifold Approximation and Projection)等。其中,主成分分析通过识别一组具有最大方差和相互不相关的特征的线性组合来生成低维表示的数据集,有助于理解变量在无监督环境下的潜在的相互作用。
生物信息学迈入大数据时代所带来的一个突出问题就是高昂的数据标注成本,对所有数据集进行人工标注的想法在庞大数据量的冲击下变得不切实际[17]。因此,聚类由于其无需标注的特性得到了广泛的研究和应用。简而言之,聚类就是按数据内在的相似性将未标注的数据集划分为多个类别,使类别内的数据相似度较大而类别间的数据相似度较小的操作。聚类是无监督学习的分类方式。常见的聚类算法包括(1)划分聚类:K-means算法、K-medoids算法、K-pototypes算法、CLARANS算法;(2)层次聚类:BIRCH算法、CURE算法;(3)密度聚类:DBSCAN算法、OPTICS算法、DENCLUE算法;(4)网格聚类:STING算法、CLIQUE算法、WAVE-CLUSTER算法;(5)混合聚类:GMM(高斯混合模型)、CLIQUE(综合密度和网格的算法);(6)其他:如SOM神经网络模型聚类、FCM模糊聚类等。其中,K-means聚类根据聚类中心点的距离将数据分为k个不同的聚簇,层次聚类则是通过创建一棵聚类树来构建多级分层结构。
与聚类不同,分类属于监督学习的范畴,即根据一些给定的已知类别的样本(有标签数据),使计算机能够对未知类别的样本进行分类。分类要求必须事先明确知道各类别的信息,是一种对离散型随机变量建模或预测的监督学习算法。常用的分类算法包括:决策树分类法;基于规则的分类器;朴素的贝叶斯分类算法(Naive Bayesian Classifier);基于支持向量机(Support Vector Machine,SVM)的分类器;神经网络法;k-最邻近法(K-Nearest Neighbor,KNN);模糊分类法。
与分类相似,回归也是监督学习的一种算法,但回归方法是一种对数值型连续随机变量进行预测和建模的监督学习算法,产生的结果一般也是数值型的。回归任务的特点是标注的数据集具有数值型的目标变量。每一个观察样本都有一个数值型的标注真值以监督算法。常用的回归算法有:线性回归、回归树(包括随机森林(RF)或梯度提升树(GBDT))、深度学习(特殊的非线性回归分析方法)、最近邻算法等。
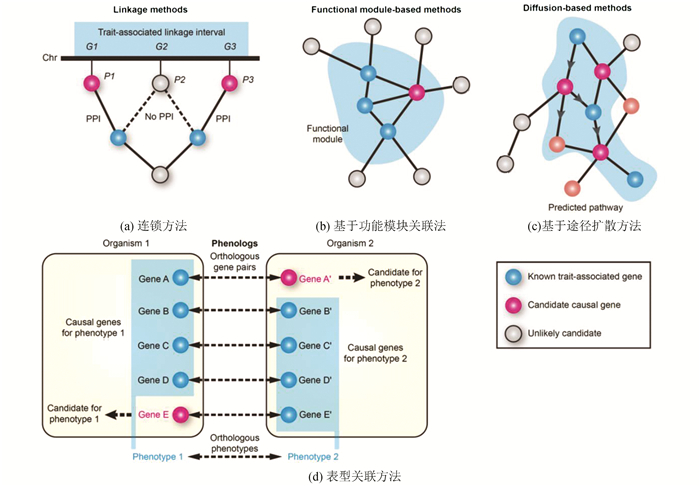
基于统计比较的相关性分析(Correlation Analysis)已大量地被用来判断两个或多个变量(基因、蛋白等)是否相关,进而确定它们的相关性正负和强弱(见图 3)。根据变量的数据类型不同(如连续变量、二分类变量、无序分类变量和有序分类变量),采用的统计检验分析方法也不同。相关性不等于因果性,需要判断区分自变量和因变量,有关因果关系的研究也是生物信息学建模的重要内容[18]。
|
图 3 生物信息学相关性关系网络建立示意图(陈迪俊博士提供[20]) Figure 3 Schematic diagram of correlation networks construction in bioinformatics(provided by Dr.CHEN Dijun[20]) |
在生物数据的统计分析中,统计学显著性与生物学显著性的差异是另一个值得关注的问题。如在筛选差异表达基因列表时,纯粹用p值(p-value)往往不如用倍数法(fold-change)加上非严格的p值得到的结果可重复性强;p值强调限制假阳性,而生物学研究更侧重假阴性;从生物学角度看,潜在的基因可能具有低p值。另外,统计学结果时常会被滥用,忽视样本偏差就是一个常见的问题,只有当样本可以代表总体时,统计结果才是可信、精确的。预防统计滥用要使用合适的图表、规避偏差。柱状统计图可能是最容易使用、最容易理解的图表,但许多人忽视其中的平均数(如均值、中位数、众数)、偏差、误差等[19]。
2.2 各方法的整合在生物信息学领域,针对特定问题的算法并非只有一种,而是十分多样化,因而相应的工具也十分多样化,如针对植物蛋白亚细胞定位的预测工具多达上百个。具体在从事生物学数据分析处理、建模预测等工作中,应根据自己的需求选择适合的方法和工具,整合多种方法对特定问题进行分析,而不建议依赖使用一种方法去完成。比如在单细胞组学研究中,对稀有细胞群体的鉴定是一个广受关注的问题,常规的细胞聚类方法常常会将稀有细胞群体视为噪声,而对边界值容忍度较强、鉴定稀有细胞群体效果较好的方法对于主要细胞群体的聚类效果往往又不尽人意,因此需要联合多种聚类方法进行鉴定[21]。
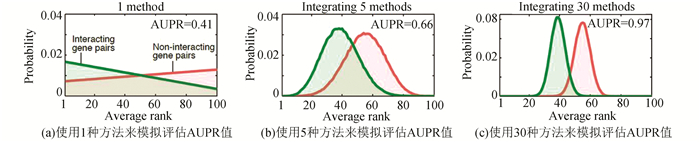
一般来说,每种方法都有其局限性,尤其是对于特定的研究对象,一些常规方法可能并不可靠。因此,整合多种方法进行分析是一个非常自然的想法,如PSI[22]对植物蛋白质的亚细胞定位进行了整合生物信息学的预测,整合了10余种方法,其结果显著提高了预测的准确性。使用1种、5种、30种方法来模拟评估AUPR值,发现整合方法越多,结果越可信(见图 4)。
|
图 4 不同数量方法整合评估AUPR值(刘丽丽博士提供) Figure 4 Assessment of AUPR value by different numbers of methods(provided by Dr.LIU Lili) |
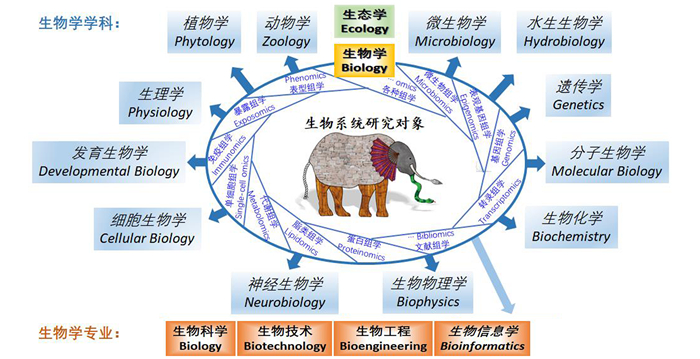
生物信息学的远景是可以基于系统的生物学数据,全面自主发掘生命科学的规律。目前生命科学本身的研究还处于“各自为政、自我突破”(学科方向为中心)的局面(见图 5)。但未来研究的方向已经显现,那就是整合各种学科研究成果,整合各类数据,从系统角度进行研究,这给生物信息学、系统生物学带来的是全新的挑战[23-24]。需要有其他学科发展的新方法,也同时是生物信息学本身要考虑解决的问题。
|
图 5 生物系统各组学研究内容及生物学科、专业的概括 Figure 5 Research contents of different omics in biological systems and summary of different biological subjects and disciplines |
面对大数据时代的挑战,各种数学方法、信息技术应运而生(见图 6),我们已进入了数据密集型科学发展阶段。不断增强的数据获取与存储能力、数学建模与计算能力、人工智能与物联网实时结合能力将进一步推进生命科学与技术的变革式发展。如何整合各种计算与信息资源,发掘数据背后的生物学规律,促进相应应用开发落地,是生物信息学领域面临的另一个层次的挑战。
|
图 6 大数据时代计算科学与信息技术的主要领域 Figure 6 Main areas of computer science and information technology in the big data era |
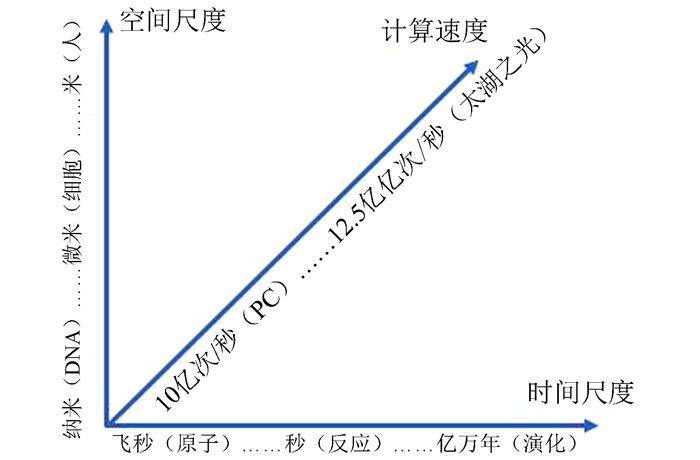
由于生物系统的复杂性,在整合分析过程中,应该注意时空尺度与计算复杂度要素(见图 7),还需要注意:(1)全局与局部的关系:应充分考虑总体特征及局部细节;(2)并列与关联关系:如编码与非编码、表达网络与代谢网络等之间的关系;(3)静态与动态的关系:应加强时间序列动态数据的分析;(4)时空维度的模型;(5)细胞与个体、个体与群体的关系;(6)算法、软件与硬件的整合。
|
图 7 生物系统的时空复杂性与计算速度 Figure 7 Spatiotemporal complexity and computation speed of biological systems |
作为交叉学科,生物信息学的快速发展得益于其他相关学科的不断发展与交叉。整合生物信息学将进一步促进相关学科的深度融合,其发展更是依赖于拥有整合生物信息学知识与技术背景的人员,关键是人才、是教育。生物信息学人才培养的需要充分考虑其他学科的系统理论知识与技术的支撑。相关教材建设也需要充分考虑其多学科交叉的特点,优化整合各方面知识点,突出生物信息学的学科特色以及理论与实践紧密结合的特点[25]。另外,还可建立问题驱动和数据驱动并重、多学科联动的教学体系,建立基因组、表观基因组、转录组、蛋白质组、代谢组以及各种表型组的复杂数据分析技术训练平台,如用于开发综合教育资源,对研究人员进行生物信息学数据分析训练的社区驱动框架[26]。
4.2 工程化研究整合生物信息学的科学研究,其研究范式也可能需要更偏向于整合型、工程化研究,组织整合相关团队力量进行攻关,类似于现行的国家重点专项大科学(工程)计划等,但需要更紧密、整合度更高的组织,否则容易出现项目承接后缺少沟通,难以整合研究资源的情况。
5 问题与展望生物信息学伴随着生命科学与计算机信息技术的快速发展而发展,大量数据库和软件工具被开发出来反馈助力生命科学的研究与发展。
5.1 数据问题:数据库变成垃圾堆生物数据库并非十全十美,囿于测量技术的限制和潜在的操作错误,数据库中存在错误数据是几乎不可避免的。随着时间推移,错误数据会不断积累,而日益庞大的数据量又使得及时的数据校验与更新几乎成为奢望[27]。对于数据库中的错误数据,值得注意的是数据来源本身可能就存在问题,如组学数据的测量值往往与实际值存在较大偏差,许多实际存在的数量关系无法很好地转换到测序结果中[28]。另一个较为典型的例子是,上世纪六七十年代就有人提出,生物学研究中可能存在错误鉴别细胞系的问题,但未能引起科研工作者足够警觉,以至于错认细胞系的问题因为细胞系的以误传误,不断扩大对科研结果的负面影响。2017年PLoS ONE[29]报道有超过3万篇研究论文错认了细胞系,这些错误影响的不仅是论文本身的工作,还影响了超过4万篇引用了这些论文的其他工作,以至于近年来发表相关论文时还必须增加对所用细胞系的审查鉴定工作,也发展了许多针对各类细胞系的鉴定方法。由于生物信息学是一门数据驱动的学科,因此确保数据的准确性是十分重要的。
5.2 方法问题:常用的变成经典的多年来,生物信息学家针对各类生物学问题开发了许多方法与工具,但仅有其中的一小部分被广泛使用,这是因为使用者往往无法比较所有工具,从众选择常用工具就成为最稳妥的选择。不断增长的使用者数量促使了常用工具在易用性上的改进,同时强化了其权威性,成为了经典。然而,这种马太效应也潜在地阻止更适合、更先进的方法被广泛应用。
5.3 假设问题:假设的变成像真的生命系统的复杂性迫使我们在研究时采用“还原论”(Reductionism),使用一系列假设简化条件。基于奥卡姆剃刀原则,我们的目标是在尽可能少的假设下建模真实条件中的生命系统。但整合生物信息学所采用的数据往往是在不同条件下,基于不同的技术手段和假设获得的,如何统一这些不同的假设,是整合生物信息学研究需要着重考虑的问题。
5.4 几点思考生命科学是极其复杂的,探究其规律,依赖于各种手段去描述它,去测量它,去模型它,再去预测它。首先在于数据信息的采集,如同“瞎子摸象”的过程,各类方法,产生了各类数据,但要注意的是,哪些是全局的特征数据,哪些是局部的数据,哪些是常态的数据,哪些是特定状态的数据。即便去描述这些数据,有时也是个问题,所以生物信息学也需要有手段去可视化这些数据。但归根到底还只是描述这些“测得的”数据,代表着只是大象的一部分信息。需要有更多的时空数据,或期待有更多的新仪器设备可以测到的新数据,来进行整合拼图。
在大数据时代,数据的重要性和价值已经毋庸置疑。信息是资源,数据也是生产力。数据正在改变竞争格局,认识数据的力量,并能够将数据转化为数据生产力,如为重大的生物医学问题找到结构性的解决方案,推动在癌症免疫治疗以及精准治疗领域的创新。
就像农业时代的土地、工业时代的能源,如今,数据已成为核心资源,已成为国家战略的一部分。如何进一步发展生命科学数据规范化存储,建立自己的数据中心,甚至成立全球数据中心;同时借助不断强大的信息技术,从数据到信息到知识,通过各类计算平台(包括云平台),实现快速分析与应用。当然,大数据也是一把双刃剑。数据共享、数据保密、数据缺失、隐私保护等安全问题需要格外重视,同时要保证数据的安全。生物安全已经成为我国国家安全战略的重要部分,将不断加强生物数据信息安全治理能力。
未来需要发展各种整合信息学的新方法,如面对大数据的压缩、整合方法、各组学的融合分析方法、各场景各类深度学习方法的整合使用、人工智能+人脑混合模型(人机整合)、全方位高精度分析方法,以及这些整合生物信息学的研究是否还要考虑其研究结果的可重复性(Reproducibility)的问题等等。
| [1] |
陈铭. 后基因组时代的生物信息学[J]. 生物信息学, 2004, 2(2): 29-34. CHEN Ming. Bioinformatics in the post-genomic era[J]. China Journal of Bioinformatics, 2004, 2(2): 29-34. DOI:10.3969/j.issn.1672-5565.2004.02.007 (  0) 0) |
| [2] |
陈铭. 整合生物信息学[J]. 计算机教育, 2006, 9: 7-10. CHEN Ming. Integrated bioinformatics[J]. Computer Education, 2006, 9: 7-10. DOI:10.16512/j.cnki.jsjjy.2006.09.002 (  0) 0) |
| [3] |
WATANABE K, STRINGER S, FREI O, et al. A global overview of pleiotropy and genetic architecture in complex traits[J]. Nature Genetics, 2019, 51(9): 1339-1348. DOI:10.1038/s41588-019-0481-0 (  0) 0) |
| [4] |
KANEHISA M, GOTO S. KEGG: Kyoto encyclopedia of genes and genomes[J]. Nucleic Acids Research, 2000, 28(1): 27-30. DOI:10.1093/nar/28.1.27 (  0) 0) |
| [5] |
TOMCZAK K, CZERWINSKA P, WIZNEROWICZ M. The Cancer Genome Atlas (TCGA): An immeasurable source of knowledge[J]. Contemporary Oncology, 2015, 19(1A): A68. (  0) 0) |
| [6] |
ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome[J]. Nature, 2012, 489(7414): 57. DOI:10.1038/nature11247 (  0) 0) |
| [7] |
ZHAO L, WANG J, LI Y, et al. NONCODEV6: An updated database dedicated to long non-coding RNA annotation in both animals and plants[J]. Nucleic Acids Research, 2021, 49(D1): D165-D171. DOI:10.1093/nar/gkaa1046 (  0) 0) |
| [8] |
BENSON D A, CAVANAUGH M, CLARK K, et al. Gen Bank[J]. Nucleic Acids Research, 2012, 41(D1): D36-D42. DOI:10.1093/nar/gks1195 (  0) 0) |
| [9] |
HARRISON P W, AHAMED A, ASLAM R, et al. The european nucleotide archive in 2020[J]. Nucleic Acids Research, 2021, 49(D1): D82-D85. DOI:10.1093/nar/gkaa1028 (  0) 0) |
| [10] |
FUKUDA A, KODAMA Y, MASHIMA J, et al. DDBJ update: Streamlining submission and access of human data[J]. Nucleic Acids Research, 2021, 49(D1): D71-D75. DOI:10.1093/nar/gkaa982 (  0) 0) |
| [11] |
CHEN F Z, YOU L J, YANG F, et al. CNGBdb: China national genebank database[J]. Hereditas, 2020, 42(8): 799-809. (  0) 0) |
| [12] |
National Genomics Data Center Members and Partners. Database resources of the national genomics data center in 2020[J]. Nucleic Acids Research, 2020, 48(D1): D24-D33. (  0) 0) |
| [13] |
FILLINGER S, DE LA GARZA L, PELTZER A, et al. Challenges of big data integration in the life sciences[J]. Analytical and Bioanalytical Chemistry, 2019, 411(26): 6791-6800. DOI:10.1007/s00216-019-02074-9 (  0) 0) |
| [14] |
BRAZMA A, HINGAMP P, QUACKENBUSH J, et al. Minimum information about a microarray experiment (MIAME)—toward standards for microarray data[J]. Nature Genetics, 2001, 29(4): 365-371. DOI:10.1038/ng1201-365 (  0) 0) |
| [15] |
HUCKA M, FINNEY A, SAURO H M, et al. The systems biology markup language (SBML): A medium for representation and exchange of biochemical network models[J]. Bioinformatics, 2003, 19(4): 524-531. DOI:10.1093/bioinformatics/btg015 (  0) 0) |
| [16] |
GREENER J G, KANDATHIL S M, MOFFAT L, et al. A guide to machine learning for biologists[J]. Nature Reviews Molecular Cell Biology, 2021, 1-16. (  0) 0) |
| [17] |
GREENE C S, TAN J, UNG M, et al. Big data bioinformatics[J]. Journal of Cellular Physiology, 2014, 229(12): 1896-1900. DOI:10.1002/jcp.24662 (  0) 0) |
| [18] |
LE T D. Causal inference methods and applications in Bioinformatics[J/OL]. Thermo Fisher Scientific, 2018. https://www.researchgate.net/publication/307477690.
(  0) 0) |
| [19] |
HALSEY L. The reign of the p-value is over: What alternative analyses could we employ to fill the power vacuum?[J]. Biology Letters, 2019, 15: 20190174. DOI:10.1098/rsbl.2019.0174 (  0) 0) |
| [20] |
CHEN D, CHEN M, ALTMAN T, et al. Bridging genomics and phenomics[M]//CHEN M, HOFESTÄDT. Approaches in Integrative Bioinformatics. Berlin: Springer, 2014: 299-333.
(  0) 0) |
| [21] |
JINDAL A, GUPTA P, SENGUPTA D. Discovery of rare cells from voluminous single cell expression data[J]. Nature Communications, 2018, 9(1): 1-9. DOI:10.1038/s41467-017-02088-w (  0) 0) |
| [22] |
LIU L, ZHANG Z, MEI Q, et al. PSI: A comprehensive and integrative approach for accurate plant subcellular localization prediction[J]. PLoS ONE, 2013, 8(10): e75826. DOI:10.1371/journal.pone.0075826 (  0) 0) |
| [23] |
陈铭. 系统生物学(Systems Biology)的几大重要问题[J]. 生物信息学, 2007, 5(3): 129-136. CHEN Ming. Several key problems in Systems Biology[J]. China Journal of Bioinformatics, 2007, 5(3): 129-136. DOI:10.3969/j.issn.1672-5565.2007.03.009 (  0) 0) |
| [24] |
CHEN M, HOFESTAD T R, Eds. Integrative Bioinformatics: History and Future[M]. Springer, 2022.
(  0) 0) |
| [25] |
陈铭. 生物信息学[M]. 4版. 北京: 科学出版社, 2022. CHEN Ming. Bioinformatics[M]. 4th ed. Beijing: Science Publish, 2022. (  0) 0) |
| [26] |
BATUT B, HILTEMANN S, BAGNACANI A, et al. Community-driven data analysis training for biology[J]. Cell Systems, 2018, 6(6): 752-758. DOI:10.1016/j.cels.2018.05.012 (  0) 0) |
| [27] |
BAGHERI H, SEVERIN A J, RAJAN H. Detecting and correcting misclassified sequences in the large-scale public databases[J]. Bioinformatics, 2020, 36(18): 4699-4705. DOI:10.1093/bioinformatics/btaa586 (  0) 0) |
| [28] |
ABNIZOVA I, TE BOEKHORST R, ORLOV Y L. Computational errors and biases in short read next generation sequencing[J]. Journal of Proteomics & Bioinformatics, 2017, 10(1): 1-17. (  0) 0) |
| [29] |
HORBACH S P J M, HALFFMAN W. The ghosts of HeLa: How cell line misidentification contaminates the scientific literature[J]. PLoS ONE, 2017, 12(10): e0186281. DOI:10.1371/journal.pone.0186281 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20

