食管癌是最常见、最具侵袭性的恶性肿瘤之一。每年全世界大约有40万名新确诊患者并且约有30万患者死亡[1]。据美国国家癌症研究院报道的数据显示食管癌患者的五年生存率仅为19.9%。近年来食管癌的治疗效果得到了很大改善,但食管癌的预后仍然不能令人满意。化疗和基于放疗的治疗方法已应用于许多患者,根据目前医学研究的统计分析,食管癌手术前的新辅助化疗或联合放化疗的患者比单独手术的患者的生存期会有所增加[2]。但是,对患者治疗的方法是因人而异的,有些患者的化疗效果很好,有些患者可能效果较差。所以确定哪些人适合使用哪种治疗手段就显得十分重要,患者也能避免尝试不同无效的治疗手段时带来的副作用的伤害。所以,可以预测化疗反应的分子标记物需要被更多的研究,找到适当的分子标记物,更好的识别哪些药物对患者有效,哪些药物患者会产生耐药性,从而在克服潜在的化疗耐药性的情况下提供新的治疗手段。
微小核糖核酸(miRNA)是一种非编码RNA,和转录因子功能相似,可以调节信使RNA(mRNA)的表达,miRNA起到的是抑制基因表达的功能,从而实现对各种生物学功能的调控,目前很多研究已经发现miRNA和转录因子在癌症的发生发展中起到关键的作用[3-4]。miRNA和转录因子作为生物标记物的研究也很多,研究人员发现在食管癌患者和正常样本中,多个miRNA表达异常,这些表达的差异直接影响了肿瘤的生长、增值、侵袭,miR-485-5p就可以使乙酰氨基葡糖转移酶水平下降从而抑制食管癌的增殖和侵袭,miR-502通过促进AKT磷酸化介导食管癌细胞增殖[5-6]。此外还有研究发现miRNA和转录因子的异常表达还影响着化学疗法的疗效,miR-205纳米制剂可以使得前列腺癌对化疗敏感,miR-146a通过下调CHOP的表达诱导机体耐药[7-9]。在多种癌症的研究中有转录因子和miRNA作为标志物的相关报道众多,miR-590-3p通过抑制Wnt/beta-catenin通路内的WIF1和DKK1基因促进了结直肠癌的增殖,转录因子ZNF217是乳腺癌进展过程中的预后生物标志物和治疗靶点[10-12],但是到目前为止miRNA是如何影响着食管癌对药物的耐药性的研究还很少。
本研究中我们整合识别了对不同药物敏感及耐药的细胞系,找到了在敏感和耐药细胞系中起到关键作用的基因集合,之后我们整合了多个转录因子及miRNA对靶基因调控关系数据库,构建了包含转录因子及miRNA的转录调控网络,这些调控关系是实验证实的,之后为了确定这些调控关系在食管癌中是被真正的激活,我们使用了食管癌患者的数据对网络内每一对关系进行相关性计算,得到在食管癌中显著相关的关系对,筛选后的网络进一步进行分析。使用不同药物作用的下耐药和敏感细胞系进行差异分析得到的差异表达基因,筛选出药物特异的调控模块,从根本上找到那些影响细胞系耐药相关的基因是被哪些转录因子或者miRNA调控的,这些转录因子及miRNA可能成为新的药物耐药性预测的标记物,为后续的药物研发及患者的治疗提供帮助。
1 材料方法 1.1 敏感及耐药细胞系识别从癌症药物敏感性基因组学数据库获(GDSC)[11]得了全部的食管癌细胞系表达数据及药物作用后的IC50值数据。然后分别对每一种药物治疗的所有细胞系进行划分,分为敏感和耐药细胞系。一个药物作用于多个细胞系,每个细胞系的IC50值有所差异,通过计算同种药物作用下不同细胞系IC50的均值及标准差,确定了分类标准,细胞系的IC50值大于均值加上0.8倍的标准差为耐药细胞系,IC50值小于均值减去0.8倍的标准差为敏感细胞系。为了保证后续计算的统计学准确性,要求耐药和敏感细胞系的数量均不小于5个,根据两个测度识别了食管癌敏感及耐药细胞系。
1.2 耐药性显著相关基因集合通过识别得到的多组药物相关的敏感和耐药细胞系进行差异表达计算,使用的方法为t检验,选取显著性结果的阈值为P<0.01,得到每组药物的耐药性相关基因集合。对全部13种药物的分类完成的细胞系进行了相关计算,得到13组差异表达基因。
1.3 食管癌转录调控网络构建为了识别出哪些miRNA和转录因子调控药物耐药及功能相关的基因,从多个数据库整理了实验证实的转录调控关系,miRNA调控靶基因的关系对来自miRTarBase[13]、TarBase[14],转录因子对靶基因及miRNA的调控关系来自于TRANSFAC[15],还整合了单独的转录因子对miRNA调控数据库TransmiR[16],所有的调控关系都是经过实验证实的,初始的转录调控网络内一共得到5 265条调控关系对,2 772个节点。
从TCGA数据库下载了食管癌的mRNA和miRNA表达数据[17],首先对下载的数据的miRNA名称注释为成熟miRNA名称,基因的ENSG名称注释成标准的基因缩写名称。使用得到的食管癌数据对网络内的每一对关系进行相关性计算,计算方法选择的是皮尔森相关,相关阈值选取P<0.05。进行筛选后得到的网络称之为食管癌特异的转录调控网络。网络内剩余2 445对调控关系,1 612个节点。
1.4 耐药相关模块挖掘为了得到各种药物耐药相关的转录调控模块,将敏感和耐药细胞系计算得到的差异表达基因投入网络,根据关联有罪理论,以及尽可能的找到网络内调控种子基因的关键基因,本研究以差异基因作为种子基因,进行扩一步搜索其相关调控基因,获取了药物特异的调控网络。网络并非为全联通网络,由多个分离的组分构成,由于网络内节点度比较高的基因起到重要作用,这里选取模块内的最大组分,最大组分的模块内包含最核心的基因以及最相比其他组分最多的节点数量,包含更为丰富的信息,最后剔除其余散点,这一模块称为相关药物的耐药模块。
1.5 功能富集分析及药物作用相关通路检验分析功能富集分析使用的是在线的网站工具DAVID(Database for Annotation, Visualization, and Integrated Discovery)[5],将模块内的miRNA剔除,使用模块内全部的转录因子及靶基因进行富集分析,分析GO_BP功能及KEGG通路的富集结果。
2 结果分析 2.1 食管癌特异转录调控网络通过整合多个数据库内的miRNA及转录因子对靶基因的调控关系,构建了所有关系均为实验证实的转录调控网。因为很多基因在不同组织内是特异性表达的,所以想通过真实的食管癌数据对调控关系进行验证,下载了TCGA的173例食管癌患者肿瘤数据,对所有的调控关系对进行相关性计算,使用的方法为Pearson相关,经过计算选取P值显著的关系对作为食管癌中能行使特定调控功能的关系对,由这些关系所组成的网络称之为食管癌特异的转录调控网络。我们可以基于这一网络去探索网络内某个重要基因的调控关系,去进一步研究该基因之所以能行驶功能是不是调控了其他基因导致的,或者从另一角度找到该基因的表达值改变的原因,是否是由于受其它基因的调控。构建了这样的食管癌特异的网络对后续的关键调控子相关调控机制的研究提供了便利(见图 1)。

|
图 1 食管癌特异的转录调控网络 Figure 1 Transcriptional regulatory network of characteristic esophageal cancer 注:(a)中网络内橙色的点为miRNA,蓝色的点为转录因子,绿色点为靶基因. |
通过对药物作用的食管癌细胞系进行区分,将同一种药物作用下不同耐药能力的细胞系进行区分,计算同种药物作用下耐药和敏感细胞系的差异表达基因,这些基因在药物作用的时候使细胞系产生耐药反应,从而使得药效降低。共筛选了满足分析条件的食管癌药物13种(见表 1)。
| 表 1 13种药物中敏感耐药细胞系数量 Table 1 The number of sensitive resistant cell lines in 13 drugs |
其中包含Trametinib、Topotecan、AZD7762和Afatinib等在内的10种药物均显示已经应用于食管癌患者的治疗[18-26],其余3种药物Teniposide、Dasatinib和Dactolisib也有研究发现其潜在的食管癌治疗的价值,而且发现了不同患者使用该药物时敏感性有一定差异[27-29]。对每一种药物进行差异表达分析,获得了13组差异表达基因集合。我们发现在不同药物的作用下,部分基因在两组样本中差异表达频率很高,这些基因可能影响多种药物在食管癌中的耐药性。尤其是FANCD2OS、GUCY2F、HES7三个基因,分析得知其在不同的6种药物中均显著的差异表达,通过文献查找也发现在以往的研究及试验中证实,这些基因发生突变和多个癌症的发生发展显著相关,而且影响患者的预后[30-33]。继续对这些耐药相关的基因的研究,从而发现是否是因为这些耐药相关基因发生了突变的患者预后较差的原因是否为对治疗药物产生了耐药性导致的。
这些耐药相关的基因是否受其他基因调控,或者是否依靠调控其他基因的表达从而真正实现患者耐药性的产生,将差异表达基因映射到食管癌特异的调控网络内,从网络内提取出和投入的种子基因最临近的基因集合,将筛选后的最大联调网络提取出来,作为食管癌各种药物特异的调控模块,选择网络内的最大连通组分,既保留了网络内的关键调控因子,也尽可能的保留了食管癌耐药相关的差异表达基因,从而能继续分析模块的功能,以及提取每一步的调控关系,找到关键的调控因子。根据上文体积的方法,最终挖掘出13种食管癌耐药模块(见图 2)。
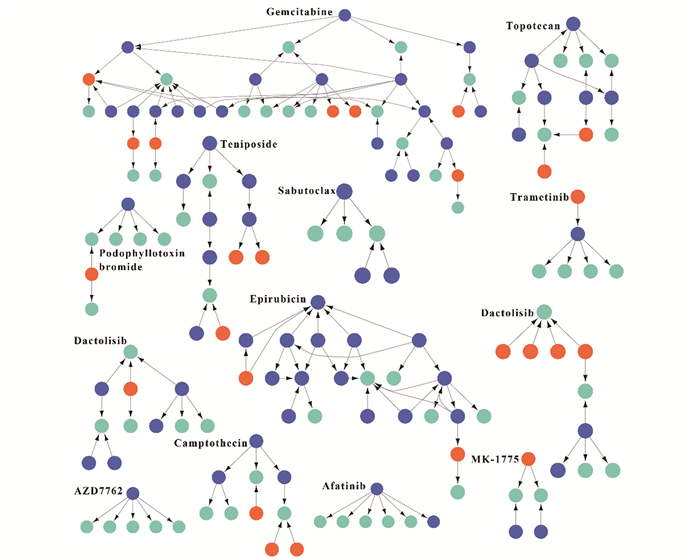
|
图 2 13种药物耐药特异模块 Figure 2 Resistance-related characteristic modules of 13 drugs |
因为每个药物耐药基因不同,模块的大小也有所差异。发现大多数模块内,耐药相关基因都位于模块的下游,也就是这些耐药相关基因的表达受到了一系列转录因子及miRNA的调控,上游的一个或几个关键的调控子发生改变,可能导致耐药相关基因的异常表达的出现。
挖掘后的模块进行功能分析,分析每个模块所行使的功能,我们使用是在线的功能注释工具DAVID,通过分析结果我们发现,很多模块的功能主要富集与细胞增殖及血管生成上,这些功能与肿瘤的发生发展相关,有趣的是我们也发现了吉西他滨模块内的CRHBP和TP53均是细胞对药物应答功能相关基因[34],该药物相关模块内基因的富集结果(见图 3)。
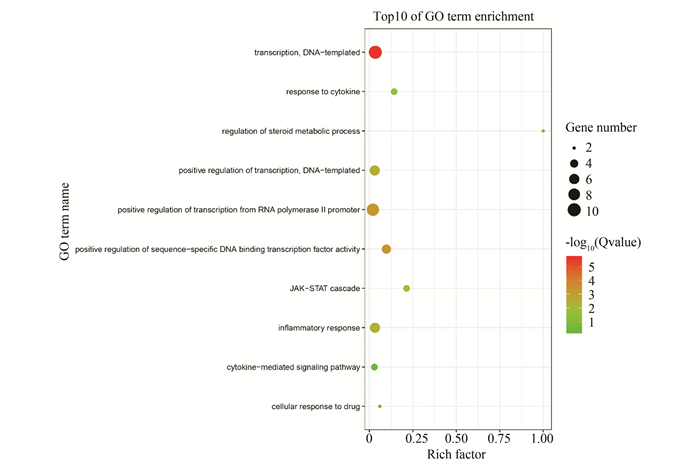
|
图 3 吉西他滨模块内基因富集分析结果 Figure 3 Results of gene enrichment analysis in Gemcitabine module 注:网络内橙色的点为miRNA,蓝色的点为转录因子,绿色点为靶基因. 注:节点越大表示该功能含有模块内基因数量越多,颜色越红表示结果越显著. |
本研究对13种药物进行了具体的分析,研究发现所有的耐药相关基因出现在模块的下游,针对相应的模块进行了详细的分析,以拓扑替康模块为例(见图 4)。

|
图 4 拓扑替康药物特异模块 Figure 4 Characteristic module of Topotecan 注:网络内橙色的点为miRNA,蓝色的点为转录因子,绿色点为靶基因. |
模块内黄色的点为差异表达的耐药相关基因,再对这些差异表达基因研究中我们发现,以CSF1基因为代表的模块内叶子节点上的基因,在很多研究中已经发现它和药物的耐药性显著相关[35],而且其行使功能也是由于miR-130 miRNA家族基因调控的[36],这与我们找到的模块内的调控关系是一致的。然后我们进一步分析了网络内最上游的转录因子及miRNA,这一模块内最上游的基因为SP1,其可以通过表达的改变调控相应的通路使得肿瘤患者产生耐药反应,这样的通路包括MAPK信号通路[37],MAPK通路内的RELA基因在模块内也是由SP1调控在进一步调控其他耐药相关基因。我们还发现网络内有两个miRNA位于叶子节点上,hsa-miR-9-5p和hsa-miR-125a-5p这样的位于叶子节点上的基因更可能直接行使功能,使机体产生耐药性。通过对最新的研究的检索,发下hsa-miR-9-5p再转录后调控中扮演者重要的角色,通过改变DNA拓扑异构酶Ⅱα致使相应的细胞系产生耐药性[38]。通过研究我们发现位于上游的基因可以通过一系列的调控关系,逐步的达到调控耐药相关基因的目的,位于根节点的基因能直接的反应出机体是否会产生耐药性,但往往单一的某个基因的异常不足以准确的确定机体是否产生耐药。所以能通过节点调控一个或者多个耐药相关的基因而且行使了重要功能的跟节点就显得尤为关键,本研究认为这些上游的基因可能成未来耐药研究的新的生物标记物。通过对模块的分析,本研究挖掘了不同药物模块内的关键调控基因,这些基因可能成为潜在的生物标记物(见表 2)。
| 表 2 13种药物模块内潜在关键调控子 Table 2 Potential key regulators in 13 drug modules |
药物耐药性是癌症治疗失败的一个重要原因,已经有研究发现了一些转录因子和miRNA可以影响药物的耐药性的产生,但这些研究大多是针对直接和耐药相关的基因,对这些基因为什么表达异常研究还有所欠缺,本文通过构建了一个食管癌特异的转录调控网络,基于这一网络将可以调控耐药基因的模块都挖掘出来,得到13种作用于食管癌细胞系的耐药模块。而且随着后续研究发现了包括SP1及hsa-miR-21-5p、hsa-miR-1-5p在内的关键的转录调控通路上游关键调控子,这些调控子可能经过一步或是几步的调控最终改变耐药基因的表达,从而导致患者耐药性的产生。各模块内的关键调控子可以作为潜在的生物标志物,用于检测病人是否适用相应药物。目前耐药及敏感样本的差异分析是根据不同的食管癌细胞系完成的,未来如果数据足够充足,使用表达模式更复杂的不同癌症亚型的样本人群数据进行分析可以进一步提升分析结果的准确性。目前的研究分析的调控关系完全是来源于整理了实验证实关系的数据库,随着研究的不断进展,以及潜在的调控关系可能还未被研究人员发现,现在使用的这些调控关系可能不够完整,会遗漏部分真实存在的调控关系对,导致网络的连通程度降低,选取网络最大连通组分的时候会有部分真实的调控关系的遗漏,如果能很好的解决假阳性率的问题,后续的研究中可以使用软件预测的调控关系对来代替真实调控关系,将会大大增加转录调控网络的大小以及最终挖掘的知识更加丰富。
| [1] |
JEMAL A, BRAY F, CENTER M M, et al. Global cancer statistics[J]. CA Cancer Journal for Clinicans, 2011, 61(2): 69-90. DOI:10.3322/caac.20107 (  0) 0) |
| [2] |
SJOQUIST K M, BURMEISTER B H, SMITHERS B M, et al. Survival after neoadjuvant chemotherapy or chemoradiotherapy for resectable oesophageal carcinoma: an updated meta-analysis[J]. Lancet Oncology, 2011, 12(7): 681-692. DOI:10.1016/S1470-2045(11)70142-5 (  0) 0) |
| [3] |
GIBBINGS D J, CIAUDO C, ERHARDT M, et al. Multivesicular bodies associate with components of miRNA effector complexes and modulate miRNA activity[J]. Nature Cell Biology, 2009, 11(9): 1143-1149. DOI:10.1038/ncb1929 (  0) 0) |
| [4] |
VOORHOEVE P M, SAGE C L, SCHRIER M, et al. A genetic screen implicates miRNA-372 and miRNA-373 as oncogenes in testicular germ cell tumors[J]. Cell, 2006, 124(6): 1169-1181. DOI:10.1016/j.cell.2006.02.037 (  0) 0) |
| [5] |
HUANG D W, SHERMAN B T, LEMPICKI R A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nature Protocols, 2009, 4(1): 44-57. DOI:10.1038/nprot.2008.211 (  0) 0) |
| [6] |
JIAO W P, ZHANG J Y, WEI Y Y, et al. MiR-139-5p regulates VEGFR and downstream signaling pathways to inhibit the development of esophageal cancer[J]. Digestive and Liver Disease, 2019, 51(1): 149-156. DOI:10.1016/j.dld.2018.07.017 (  0) 0) |
| [7] |
HUMMEL R, HUSSEY D J, HAIER J. MicroRNAs: Predictors and modifiers of chemo- and radiotherapy in different tumour types[J]. Europe Journal of Cancer, 2010, 46(2): 298-311. DOI:10.1016/j.ejca.2009.10.027 (  0) 0) |
| [8] |
NAGESH P K B, CHOWDHURY P, HATAMI E, et al. miRNA-205 nanoformulation sensitizes prostate cancer cells to chemotherapy[J]. Cancers (Basel), 2018, 10(9): 289. DOI:10.3390/cancers10090289 (  0) 0) |
| [9] |
TAN W F, LIAO Y, QIU Y, et al. miRNA 146a promotes chemotherapy resistance in lung cancer cells by targeting DNA damage inducible transcript 3 (CHOP)[J]. Cancer Letter, 2018, 428: 55-68. DOI:10.1016/j.canlet.2018.04.028 (  0) 0) |
| [10] |
ZHENG Yufeng, XU Huaxi, CEN D Z, et al. miR-590-3p promotes colon cancer cell proliferation via Wnt/beta-catenin signaling pathway by inhibiting WIF1 and DKK1[J]. European Review For Medical and Pharmacological Sciences, 2017, 21(21): 4844-4852. (  0) 0) |
| [11] |
YANG W J, SOARES J, GRENINGER P, et al. Genomics of drug sensitivity in cancer (GDSC): A resource for therapeutic biomarker discovery in cancer cells[J]. Nucleic Acids Research, 2013, 41(Database issue): D955-D961. DOI:10.1093/nar/gks1111 (  0) 0) |
| [12] |
LITTLEPAGE L E, ADLER A S, KOUROS-MEHR H, et al. The transcription factor ZNF217 is a prognostic biomarker and therapeutic target during breast cancer progression[J]. Cancer Discovery, 2012, 2(7): 638-651. DOI:10.1158/2159-8290.CD-12-0093 (  0) 0) |
| [13] |
HSU S D, LIN F M, WU W Y, et al. miRTarBase: a database curates experimentally validated microRNA-target interactions[J]. Nucleic Acids Research, 2011, 39(Database issue): D163-D169. DOI:10.1093/nar/gkq1107 (  0) 0) |
| [14] |
SETHUPATHY P, CORDA B, HATZIGEORGIOU A G. TarBase: A comprehensive database of experimentally supported animal microRNA targets[J]. RNA, 2006, 12(2): 192-197. DOI:10.1261/rna.2239606 (  0) 0) |
| [15] |
WINGENDER E, CHEN X, HEHL R, et al. TRANSFAC: an integrated system for gene expression regulation[J]. Nucleic Acids Research, 2000, 28(1): 316-319. DOI:10.1093/nar/28.1.316 (  0) 0) |
| [16] |
WANG J, LU M, QIU C X, et al. TransmiR: A transcription factor-microRNA regulation database[J]. Nucleic Acids Research, 2010, 38(Database issue): D119-D122. DOI:10.1093/nar/gkp803 (  0) 0) |
| [17] |
GROSSMAN R L, HEATH A P, FERRETTI V, et al. Toward a shared vision for cancer genomic data[J]. New England Journal of Medicine, 2016, 375(12): 1109-1112. DOI:10.1056/NEJMp1607591 (  0) 0) |
| [18] |
TONG Lian, LI Changying, WANG Haitao. Trametinib in the treatment of multiple malignancies harboring MEK1 mutations[J]. Cancer Treatment Reviews, 2019, 81: 101907. DOI:10.1016/j.ctrv.2019.101907 (  0) 0) |
| [19] |
ASBURY R F, LIPSITZ S, GRAHAM D, et al. Treatment of squamous cell esophageal cancer with topotecan: An eastern cooperative oncology group study (E2293)[J]. American Journal Of Clinical Oncology-Cancer Clinical Trials, 2000, 23(1): 45-46. DOI:10.1097/00000421-200002000-00013 (  0) 0) |
| [20] |
KWAK A W, YOON G, LEE M H, et al. Picropodophyllotoxin, an epimer of podophyllotoxin, causes apoptosis of human esophageal squamous cell carcinoma cells through ROS-Mediated JNK/P38 mapk pathways[J]. International Journal Of Molecular Sciences, 2020, 21(13). DOI:10.3390/ijms21134640 (  0) 0) |
| [21] |
BI Shuning, WEI Qiuren, ZHAO Zhijun, et al. Wee1 Inhibitor AZD1775 effectively inhibits the malignant phenotypes of esophageal squamous cell carcinoma In Vitro and In Vivo[J]. Frontiers In Pharmacology, 2019, 10: 864. DOI:10.3389/fphar.2019.00864 (  0) 0) |
| [22] |
WANG Yueshen, TIAN Jing, HAN Yong, et al. Gemcitabine plus vinorelbine as second-line therapy in patients with metastatic esophageal cancer previously treated with platinum-based chemotherapy[J]. Oncology Research, 2016, 24(2): 129-135. DOI:10.3727/096504016X14618564639213 (  0) 0) |
| [23] |
HONG J, MAACHA S, BELKHIRI A. Transcriptional upregulation of c-MYC by AXL confers epirubicin resistance in esophageal adenocarcinoma[J]. Molecular Oncology, 2018, 12(12): 2191-2208. DOI:10.1002/1878-0261.12395 (  0) 0) |
| [24] |
ZOU Jianling, LI Shuang, CHEN Zuha, et al. A novel oral camptothecin analog, gimatecan, exhibits superior antitumor efficacy than irinotecan toward esophageal squamous cell carcinoma in vitro and in vivo[J]. Cell Death & Disease, 2018, 9(6): 661. DOI:10.1038/s41419-018-0700-0 (  0) 0) |
| [25] |
WANG Jiannan, CHE Yun, YUAN Zuyang, et al. Acetyl-macrocalin B suppresses tumor growth in esophageal squamous cell carcinoma and exhibits synergistic anti-cancer effects with the Chk1/2 inhibitor AZD7762[J]. Toxicology Applied Pharmacology, 2019, 365: 71-83. DOI:10.1016/j.taap.2019.01.005 (  0) 0) |
| [26] |
HONG M H, HEO S G, LEE Y G, et al. Phase 2 study of afatinib among patients with recurrent and/or metastatic esophageal squamous cell carcinoma[J]. Cancer, 2020, 126(20): 4521-4531. DOI:10.1002/cncr.33123 (  0) 0) |
| [27] |
YANG Wanli, ZHAO Xinhui, HAN Yu, et al. Identification of hub genes and therapeutic drugs in esophageal squamous cell carcinoma based on integrated bioinformatics strategy[J]. Cancer Cell International, 2019, 19: 142. DOI:10.1186/s12935-019-0854-6 (  0) 0) |
| [28] |
TABATA M, TSUBAKI M, TAKEDA T, et al. Dasatinib reverses drug resistance by downregulating MDR1 and Survivin in Burkitt lymphoma cells[J]. BMC Complementary Medicine and Therapies, 2020, 20(1): 84. DOI:10.1186/s12906-020-2879-8 (  0) 0) |
| [29] |
WISE-DRAPER T M, MOORTHY G, SALKENI M A, et al. A phase Ib study of the dual PI3K/mTOR inhibitor dactolisib (BEZ235) combined with everolimus in patients with advanced solid malignancies[J]. Target Oncology, 2017, 12(3): 323-332. DOI:10.1007/s11523-017-0482-9 (  0) 0) |
| [30] |
CHEN Xia, LU Bin, MA Qian, et al. EphA3 inhibits migration and invasion of esophageal cancer cells by activating the mesenchymalepithelial transition process[J]. International Journal of Oncology, 2019, 54(2): 722-732. DOI:10.3892/ijo.2018.4639 (  0) 0) |
| [31] |
KATOH M, KATOH M. Integrative genomic analyses on HES/HEY family: Notch-independent HES1, HES3 transcription in undifferentiated ES cells, and Notch-dependent HES1, HES5, HEY1, HEY2, HEYL transcription in fetal tissues, adult tissues, or cancer[J]. International Journal of Oncology, 2018, 54(2): 722-732. DOI:10.3892/ijo.2018.4639 (  0) 0) |
| [32] |
SILVA P, NEUMANN M, SCHROEDER M P, et al. Acute myeloid leukemia in the elderly is characterized by a distinct genetic and epigenetic landscape[J]. Leukemia, 2017, 31(7): 1640-1644. DOI:10.1038/leu.2017.109 (  0) 0) |
| [33] |
WOOD L D, CALHOUN E S, SILLIMAN N, et al. Somatic mutations of GUCY2F, EPHA3, and NTRK3 in human cancers[J]. Humam Mutation, 2006, 27(10): 1060-1061. DOI:10.1002/humu.9452 (  0) 0) |
| [34] |
CHAPPELL W H, LEHMANN B D, TERRIAN D M, et al. p53 expression controls prostate cancer sensitivity to chemotherapy and the MDM2 inhibitor Nutlin-3[J]. Cell Cycle, 2012, 11(24): 4579-4588. DOI:10.4161/cc.22852 (  0) 0) |
| [35] |
ESCAMILLA J, SCHOKRPUR S, LIU C, et al. CSF1 receptor targeting in prostate cancer reverses macrophage-mediated resistance to androgen blockade therapy[J]. Cancer Research, 2015, 75(6): 950-962. DOI:10.1158/0008-5472.CAN-14-0992 (  0) 0) |
| [36] |
GU Jianjun, ZHANG Jianhe, CHEN Hongjie, et al. MicroRNA-130b promotes cell proliferation and invasion by inhibiting peroxisome proliferator-activated receptor-gamma in human glioma cells[J]. International Journal of Molecular Medicine, 2016, 37(6): 1587-1593. DOI:10.3892/ijmm.2016.2580 (  0) 0) |
| [37] |
ZHANG Yi, CHEN Haixuan, ZHOU Shuyan, et al. Sp1 and c-Myc modulate drug resistance of leukemia stem cells by regulating survivin expression through the ERK-MSK MAPK signaling pathway[J]. Molecular Cancer, 2015, 4: 56. DOI:10.1186/s12943-015-0326-0 (  0) 0) |
| [38] |
KANIA E E, CARVAJAL-MORENO J, HERNANDEZ V A, et al. hsa-miR-9-3p and hsa-miR-9-5p as post-transcriptional modulators of DNA Topoisomerase Ⅱαlpha in human leukemia K562 cells with acquired resistance to etoposide[J]. Molecular Pharmacology, 2020, 97(3): 159-170. DOI:10.1124/mol.119.118315 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


