2. 南开大学 生命科学学院,生物国家级实验教学示范中心,天津 300071
2. Biological Experimental Center, College of Life Science, Nankai University, Tianjin 300071, China
新型冠状病毒肺炎(COVID-19)自2019年底爆发以来相继在世界各国蔓延,先后被世界卫生组织宣布为国际关注的突发公共卫生事件(PHEIC)和全球性大流行病(Pandemic),对全球的公共安全构成了严重威胁[1-2]。据WHO新冠疫情数据显示,截至2020年10月1日,全球新冠确诊病例累计33 842 281例,其中死亡1 010 634例[3]。另据美国约翰斯·霍普金斯大学统计数据,截至同年10月2日,全球累计确诊超3 420万例,其中死亡超102万例[4]。
导致新型冠状病毒肺炎的病原是一种冠状病毒。冠状病毒在系统分类上属套式病毒目(Nidovirales)冠状病毒科(Coronaviridae)冠状病毒属(Coronavirus),是具有囊膜的ssRNA正链病毒,在自然界中广泛存在[5]。2020年2月11日,世界卫生组织将引起此次新冠肺炎的病毒正式命名为SARS-CoV-2[6]。它是目前已知的第七种能够感染人类的冠状病毒,具有高传染性和高隐蔽性[7],且临床上针对病毒感染性疾病的治疗较为困难。在我国第七版《新型冠状病毒肺炎诊疗方案》中,采用的抗病毒药物治疗推荐了三种方案:第一种是干扰素、洛匹那韦/利托那韦和利巴韦林的联合使用;第二种是抗疟疾药物磷酸氯喹的应用;第三种是阿比多尔的运用[8]。由于这三种都不是针对新冠肺炎的特效药,因此在诊疗方案中明确要求要注意上述药物的禁忌症、副作用以及药物间相互作用等[9]。当前对于新型冠状病毒的蛋白靶点的研究主要集中在特定蛋白靶点与已有药物的分子对接、分子动力学模拟等,探索相应蛋白靶点的结合药物及其作用机制;另外新冠肺炎的药物研究也有临床试验在进行中,据中国临床试验注册中心(Chinese Clinical Trial Registry,ChiCTR) 网站数据显示,截至2020年7月1日,我国共有666项(因各种原因撤销的45项未计算在内) 相关临床试验(包括药物、诊断试剂、心理干预等) 正在实施过程中[10]。但目前来看,老药新用的临床效果并不理想,且尚没有出现治疗新型冠状病毒肺炎的特效药,从长远角度考虑,进行疫苗和创新药物研发仍是当前重要研究任务[11]。
美国化学文摘社(Chemical Abstracts Service, CAS,美国化学会的分支机构,是全球化学信息最权威的机构,提供世界上最大的公开披露的化学信息的数据库)发布了SARS-CoV-2的蛋白靶点词库[12]。本研究对其发布的蛋白靶点进行生信分析,了解病毒的结构、侵袭过程以及基因组和蛋白质信息,借助分析工具和数据库资料分析与病毒复制等过程相关的蛋白通路和蛋白-蛋白相互作用关系,以获得新冠病毒和宿主细胞蛋白靶点组成的蛋白通路和互作网络,探索对于预防和治疗新冠肺炎有效的关键蛋白靶点和生物过程,希望为新冠肺炎的药物和疫苗研究提供可能的思路。
1 材料与方法 1.1 研究对象依据美国CAS提供的新型冠状病毒蛋白靶标词库《COVID-19 Protein Target Thesaurus》,一共可以得到63种与新冠病毒相关的蛋白靶点作为本次研究的基本材料。在这63种蛋白靶点中,可根据蛋白来源不同,分为宿主蛋白和病毒蛋白两大类。对于宿主蛋白,若一种蛋白具有发挥作用的不同亚型,则将其作为一类,对亚型进行细分,查找每个亚型靶点的Ensembl Gene ID。对于宿主蛋白靶点,白介素-1受体包括白介素-1受体1(IL-1R1)、白介素-1受体2(IL-1R2);血管紧张素Ⅱ受体包括血管紧张素Ⅱ-1型受体(AGTR1)、血管紧张素Ⅱ-2型受体(AGTR2);免疫球蛋白受体包括IgG受体Ⅱa的Fc片段(FCGR2A)、IgG受体Ⅱb的Fc片段(FCGR2B)、IgG受体Ⅲa的Fc片段(FCGR3A);核周蛋白α类包括核周蛋白α亚基1(KPNA1)、核周蛋白α亚基2(KPNA2)、核周蛋白α亚基3(KPNA3)、核周蛋白α亚基4(KPNA4)、核周蛋白α亚基5(KPNA5)、核周蛋白α亚基6(KPNA6)、核周蛋白α亚基7(KPNA7);核周蛋白β类包括皮质素传递蛋白1(TNPO1)、皮质素传递蛋白2(TNPO2)、入核转运受体5(IPO5);吞蛋白类包括吞蛋白A1(SH3GL2)、吞蛋白A2(SH3GL2)、吞蛋白A3(SH3GL3)、吞蛋白B1(SH3GLB1)、吞蛋白B2(SH3GLB2)。对于病毒蛋白靶点,可将具有相同功能的蛋白划分为一类。将3种同为病毒RNA依赖型RNA聚合酶的蛋白靶点归为一类,将2种核衣壳蛋白归为一类,并且将具有相同结构但有不同别名的蛋白合并为一类。由此分类后,可得到34种来自宿主细胞的蛋白靶点和19种来自病毒自身的蛋白靶点。宿主蛋白靶点种类多且复杂,病毒蛋白靶点则可分为参与病毒体构成的结构蛋白和不参与病毒体构成但在病毒增殖过程中发挥作用的非结构蛋白。本研究将分别对宿主蛋白靶位和病毒蛋白靶位的生物学特征及相互作用关系进行生信分析。
1.2 分析工具病毒和宿主的蛋白靶点序列数据来源于数据库NCBI(https://www.ncbi.nlm.nih.gov/)、Ensembl(http://asia.ensembl.org/index.html);数据分析工具为蛋白富集分析工具Matescape[13](https://metascape.org/gp/index.html)、蛋白网络分析工具STRING[14](https://string-db.org/);病毒蛋白靶点的图文描述来源于ViralZone网站[15-16](https://viralzone.expasy.org/;https://viralzone.expasy.org/9056);蛋白相互作用资源来源于ViralZone网站SARS-CoV-2蛋白组信息网站(https://viralzone.expasy.org/8996),ViralZone网站SARS-CoV-2互作组信息网站(https://viralzone.expasy.org/9077),UniProt数据库(https://covid-19.uniprot.org/uniprotkb?query=*)。
1.3 方法将CAS提供的新型冠状病毒的细胞蛋白靶点利用NCBI数据库工具转换成基因序列信息,并在Ensembl数据库检索基因的详细信息得到基因名称和ID,整理成表格信息,表中共有32种、48个蛋白靶点,同种类的蛋白靶点归纳在表格的最后部分,表中蛋白AL079342.2由于尚没有明确详细的研究记录,不列入分析网络内,故表中实有47个有效的Ensembl Gene ID。
1.3.1 蛋白质富集分析将47个不同的蛋白靶点以列表的形式输入到工具Metascape中,利用KEGG Pathway, GO Biological Processes, Reactome Gene Sets, Canonical Pathways, CORUM, TRRUST, DisGeNET和PaGenBase等资源进行路径和过程的富集分析,基因组中的所有基因都被用作富集背景,其中筛选条件p值<0.01、最小计数为3、富集因子>1.5的富集项,根据相似性进行层次聚类,生成条形图;为了进一步探索富集项之间的关系,我们将其呈现为网络图,并用Cytoscape生成可视化网络[17];最后利用BioGrid6、InWeb_IM7、OmniPath8等数据库和MCODE算法进行蛋白质相互作用的富集分析,得到所有基因相关蛋白质的全连接互作网络。
1.3.2 蛋白网络运用STRING数据库对基因的序列信息进行分析,做出包含已知相互作用、预测相互作用和根据蛋白质同源性得到的相互作用等的蛋白质互相作用网络,以及对各蛋白节点的描述、蛋白名称、注释等信息,并对结果进行整理分析。
分析蛋白质互作网络,得到相互作用强的蛋白网络,根据相互作用的强弱和子网络的分布查询对应的蛋白通路信息,并利用DAVID工具对相关作用强的基因做GO和KEGG分析[18],建立蛋白通路和新冠肺炎治疗的联系。
1.3.3 病毒运用NCBI数据库公布的SARS-CoV-2病毒组学信息[19],对CAS提供的已知的19种病毒蛋白靶位进行资料搜集,将靶点蛋白分类为结构蛋白和非结构蛋白两大类,整合组学信息后列出表格。然后,利用ViralZone网站中提供的图文资料得到有关病毒的结构、病毒的基因组蛋白组、病毒的复制周期、蛋白互作的描述及图表[15-16]。最后,整合CAS提供的靶点描述信息、NCBI提供的靶点所在开放阅读框的组学信息,并将其对应至ViralZone图片中结构蛋白靶点在病毒体上的具体位置,非结构蛋白靶点在病毒基因组中的相应位置,整理在UniProt数据库中通过相似性对比或已经由文献报道的蛋白质相互信息,梳理互作关系,分析靶点功能,得出结果。
2 结果分析 2.1 蛋白质靶位汇总表细胞蛋白靶位情况(见表 1);病毒蛋白靶位情况(见表 2)。
| 表 1 细胞蛋白靶位 Table 1 Cellular protein targets |
| 表 2 病毒蛋白靶位 Table 2 Viral protein targets |
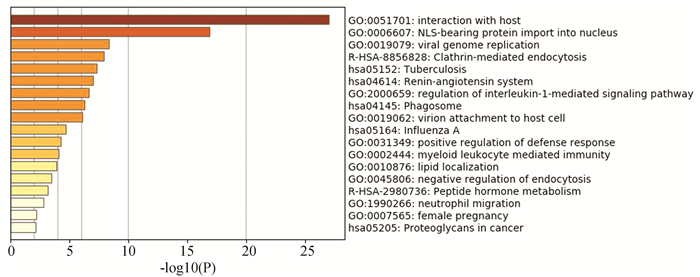
通过GO和KEGG获得富集分层聚类树(见图 1)。图中展示了前18个具有统计意义的通路,分别为与宿主的相互作用、NLS介导蛋白入核、病毒基因组复制、网格蛋白介导型内吞、肺结核病、肾素-血管紧张素系统、白细胞介素-1介导的信号通路的调控、吞噬体、病毒附着于宿主细胞、甲型流感、破骨细胞分化、髓系白细胞介导免疫、脂质定位、负向调节内吞作用、细胞对激素刺激的反应、肽类激素代谢、嗜中性粒细胞迁移等,这些人体内的生物过程均与新冠病毒的作用有关,因此,我们可以考虑调控这些关键的通路,以控制新冠病毒在人体内的复制与繁殖。其中,“与宿主的相互作用”和“NLS介导蛋白入核”的p值最小,具有最显著的统计学意义,这是由新冠病毒的致病机制决定的。新冠病毒通过呼吸道飞沫、接触等传播途径入侵人体,在人体内大量复制、繁殖,人体免疫系统调动防御机制杀死病毒,与大部分病毒相似,新冠病毒以相互作用的形式在宿主体内生存。核定位信号(NLS)是一段存在于核蛋白中,引导核蛋白入核的氨基酸序列,病毒在侵染过程中存在病毒蛋白的核质穿梭,尽管大部分RNA病毒能在细胞质中完成复制,但一些关键蛋白仍需进入核内进行复制,不仅如此,许多研究表明,部分病毒蛋白的核定位可以抑制核进口从而抑制宿主基因的转录[20-21],因此NLS介导蛋白入核是影响病毒致病力和宿主细胞重要的一环。
|
图 1 基因列表中富集项的条形图,用p值表示 Figure 1 Bar chart of enriched items from the gene list, coloured with p-values |
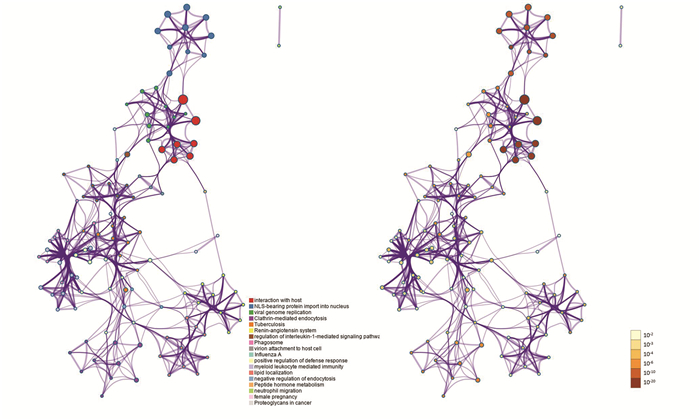
在层次聚类的基础上,相似度大于0.3的子树相连为一个簇,生成网络图(见图 2),p值最小的两个簇分别代表“与宿主的相互作用”和“NLS介导蛋白入核”,与上一结果对应一致。
|
图 2 富集项的网络 Figure 2 Network of enriched items 注:左图按聚类类型着色,拥有相似类型的节点通常相互接近;右图按p值着色,拥有更多基因的富集项往往拥有更显著的p值. |
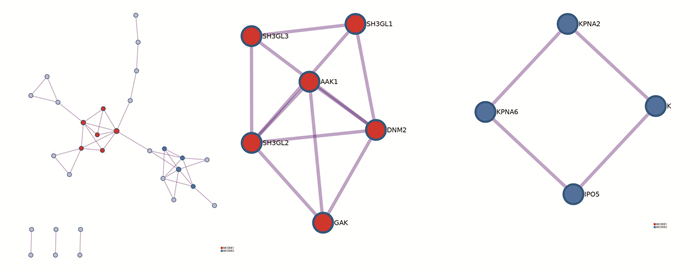
此外,对该基因列表还进行了蛋白质相互作用的富集分析(见图 3)。蛋白质互作网络包含与列表中至少一个其他成员形成物理相互作用的蛋白质子集,其中收集了两个为单个基因列表识别的MCODE网络。第一个网络由基因SH3GL1、SH3GL2、SH3GL3、AAK1、DNM2、GAK组成,其中发挥主要作用的生物过程为网格蛋白介导型内吞、网格蛋白依赖型内吞作用及其调节,网格蛋白介导型内吞是受体介导内吞的主要方式之一,在质膜蛋白内吞及胞内外信号传导过程中起着重要的调控作用,网格蛋白介导型内吞和依赖型内吞是许多病毒进入宿主细胞的重要途径,在研究抗病毒药物时,可通过抑制此途径阻断病毒感染[22];第二个网络由基因KPNA2、KPNA4、KPNA6、IPO5组成,其中发挥主要作用的生物过程为核定位信号(NLS)介导蛋白入核,此结果与上述聚类结果相对应,同时启发我们,对于新冠病毒入核机制的研究有利于发现潜在的抗病毒点,为靶向药物的研发提供新方向,例如,我们可以考虑通过改变病毒蛋白的细胞核定位信号来影响病毒的毒力。
|
图 3 基因列表中确定的蛋白质-蛋白质相互作用网络及MCODE成分 Figure 3 Protein-protein interaction network and MCODE components identified in the gene list |
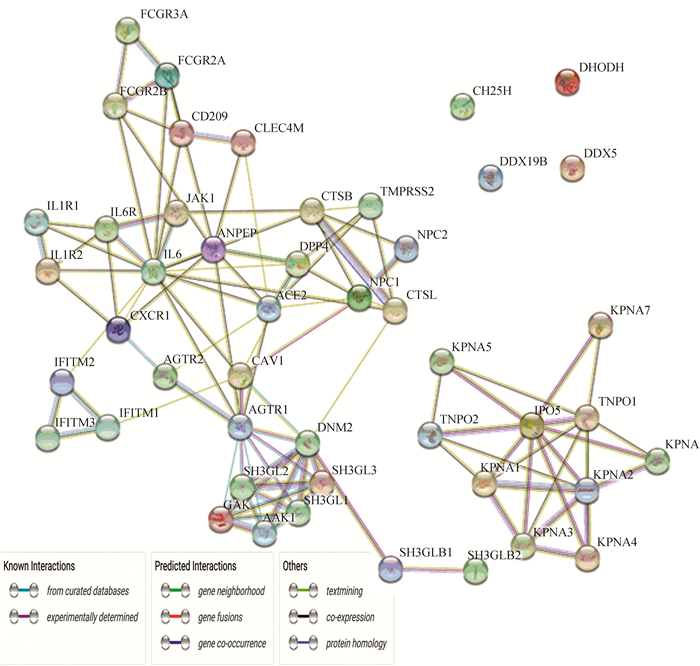
利用STRING数据库得到用基因表示的47种蛋白靶点相互作用网络(见图 4),其中有四种蛋白基因DHODH、DDX5、CH25H、DDX19B与其他蛋白在STRING数据库中没有得到任何已知或预测的相互作用,其与新冠肺炎治疗的关系仍需要探索。主要的数据来源为精选数据库KEGG、GO、Biocarta等得到的相互作用,MINT、HPRD等实验确定的相互作用和预测的基因邻接、基因融合、基因共现相互作用,以及参考文本挖掘、共表达、蛋白质同源性得到的相互作用。此蛋白网络分析是继上述富集分析的延伸,图 3中的蛋白网络只涵盖了全部研究基因中的一部分,而此蛋白网络是全部基因的整体蛋白互作网络,分析的蛋白质相互作用更加全面,并且对于子网络的分析更具体。其中有一个相互作用强的的子网络和两个相互作用较弱的、较大的子网络,每个子网络中的蛋白以及整个网络的蛋白存在较大联系或处于一个蛋白通路,包括肺结核、肾素-血管紧张素系统、细胞内吞作用、病毒在宿主细胞内的定向运动等蛋白通路和生物过程。
|
图 4 蛋白靶点相互作用网络 Figure 4 Interaction network of protein targets |
相互作用最强的子网络由基因SH3GL1、SH3GL2、SH3GL3、GAK、AAK1、DNM2、AGTR1组成,包括通过精选数据库、实验测定、文本挖掘得到的相互作用和基因共表达、蛋白质同源性的联系。在转换成基因ID后运用DAVID的Functional Annotation Tool做通路分析,基因占比在50%左右的生物过程有细胞内吞作用、中枢神经系统发育和信号传导,根据Viral Zone网站查询得知SARS-CoV-2病毒蛋白是通过细胞的内吞作用进入宿主细胞的,并且病毒的ORF8蛋白可能与“拦截”负载的Ⅰ类MHC分子,并将其保留在内质网中或将其靶向降解以避免肽在细胞表面出现这一过程有关[23],由此推断其中涉及两个基因DNM2和SH3GL2的通路抗原呈递细胞在其细胞表面与MHC Ⅱ类蛋白复合物联合表达外源性肽抗原过程,以及涉及四个基因SH3GL1、SH3GL2、SH3GL3、DNM2的通路细胞内吞作用,都与SARS-CoV-2病毒在人体内的侵袭过程有关,对这些基因所表达蛋白的研究和相应的药物研究对新型冠状病毒的防御和新冠肺炎的治疗有很大帮助。
另外两个相互作用较弱的子网络内部不同基因也存在联系,相互作用强的基因有IFITM1、IFITM2与IFITM3,FCGR2A与FCGR3A,CTSB与CTSL,IL6、IL6R与JAK1等。利用STRING的Analysis分析工具发现与这些基因相关联的GO生物过程,IFITM1、IFITM2和IFIFM3主要参与了病毒进入宿主细胞的负调节,对干扰素-α的反应,以及对干扰素-β的反应;FCGR2A和FCGR3A主要参与了内吞作用和涉及吞噬作用的Fc-γ受体信号通路;CTSB和CTSL主要参与了细胞对甲状腺激素的反应和胶原分解代谢过程;IL6、IL6R和JAK1主要参与了肝免疫反应,胶原蛋白合成过程的负调节,以及白介素6介导的信号通路[24],这些生物过程与免疫反应有很大关联。运用DAVID的Functional Annotation Tool对47个基因做整体的通路分析和功能解释,发现同样存在与新冠肺炎有关的蛋白通路。涉及基因最多的蛋白通路为含NLS的蛋白导入细胞核的过程,包括基因IPO5、TNPO1、TNPO2、KPNA1、KPNA2、KPNA3、KPNA4、KPNA5、KPNA6和KPNA7,即为一个单独的子网络,在Viral Zone网站查询得知SARS1在病毒复制或释放过程中病毒的ORF6蛋白和KPNA2存在交互过程,通过阻止蛋白质的核输入来破坏干扰素信号传导[25],与这一通路有关联,以其中蛋白为药物靶点对SARS-CoV-2的防御有研究意义。其次同样是细胞内吞作用,涉及SH3GL1、SH3GL2、SH3GL3、SH3GLB1、SH3GLB2、CAV1、CXCR1和DNM2共八个基因。发现与病毒相关度很高的通路有甲型流感,涉及JAK1、IL6、KPNA1、KPNA2和TMPRSS2共五个基因,但在蛋白网络中并没有显示出明显的相互作用;以及病毒或病毒的一部分在宿主细胞内的定向运动这一过程,与病毒在细胞内的活动有很强的关联,包括CLEC4M、CD209、KPNA1、KPNA2、KPNA3、KPNA4、KPNA5和KPNA7共8个基因;网络中与病毒有直接关系的蛋白通路还包括病毒附着后病毒或其核酸突破细胞膜进入宿主细胞的过程,免疫应答,病毒对宿主细胞过程的影响或调控,Ⅰ型干扰素信号通路,涉及基因都比较多,为四到八个之间,这些通路都与病毒的侵袭、复制等活动过程有关,通过药物对通路中某一蛋白的调控可以影响疾病的发展。病毒附着后病毒或其核酸突破细胞膜进入宿主细胞的过程,涉及CLEC4M、CD209、NPC1、ANPEP、ACE2、CTSB、DPP4和KPNA3共八个基因,除基因KPNA3外,其他基因都在蛋白网络中有关联,且相互作用较强,这一过程涉及病毒对细胞的侵入,对相应蛋白以药物控制可以一定程度地阻止SARS-CoV-2病毒的侵入;免疫应答的过程涉及基因为FCGR3A、FCGR2B、IFITM2、IFITM3、IL1R1、IL1R2和IL6,这七个基因的相互作用在蛋白网络中相对较弱,IL1R1对于辅助性T细胞1因子的合成是正调节作用,IL6对于辅助性T细胞2因子的合成同样是正调节,两者积极调节T细胞因子的产生,且两者与IL1R2共同参与了白介素1介导的信号通路的调控,IFITM2和IFITM3对于病毒进入宿主细胞有负调控作用,FCGR3A和FCGR2B都是低亲和力免疫球蛋白γFc区受体,参与了吞噬作用的Fc-γ受体信号通路,这七个基因都涉及了免疫过程。由此,针对这一过程的蛋白设计药物对免疫系统对病毒的防御过程做出调节,加强保护机制,可以作为治疗新冠肺炎的一个可行途径;另外两个通路病毒对宿主细胞的影响和Ⅰ型干扰素信号通路同样和病毒在体内的过程有关,以及蛋白网络中涉及的其他通路肺结核、肾素-血管紧张素系统、破骨细胞分化、造血细胞通路、金黄色葡萄球菌感染、蛋白质从细胞质到细胞核的定向运动、癌症中的转录失调蛋白质水解等,与病毒在体内的活动过程都有关联,其中在Viral Zone网站查询得知SARS1在病毒复制或释放过程中病毒的ORF6蛋白和KPNA2存在交互过程,通过阻止蛋白质的核输入来破坏干扰素信号传导[25],并且在病毒释放的过程中病毒的核蛋白(N)和宿主蛋白SMAd3存在交互作用,可能引起骨髓抑制[26],前者与Ⅰ型干扰素信号通路和蛋白质从细胞质到细胞核的定向运动有很大的关联,后者与破骨细胞分化和造血细胞等通路有密切关系。
总之,利用STRING做出的含47种基因或蛋白的蛋白互作网络,除四个基因外都有相互作用或处在同一蛋白通路,并且关系错综复杂,形成一些子网络和相连的网络,其中相对主要的并且与新冠肺炎相关度较高的通路包括细胞内吞作用、Ⅰ型干扰素信号通路、蛋白质从细胞质到细胞核的定向运动、病毒附着后病毒或其核酸突破细胞膜进入宿主细胞的过程、免疫应答和病毒对宿主细胞过程的影响或调控,这些通路涉及的蛋白可以作为药物的靶点,研究其结构和功能设计药物,以缓解或治疗新冠肺炎,所以研究这些蛋白靶点设计药物对新冠肺炎的治疗有重要意义。
2.4 病毒靶位分析由NCBI公布的SARS-CoV-2基因组、蛋白组数据可知,美国CAS提供的19种SARS-CoV-2蛋白靶位中,有15种为病毒的非结构蛋白(Non-structural proteins),有4种为结构蛋白(Structural proteins)(见表 2)。其中,前15种为非结构蛋白,后4种为结构蛋白。
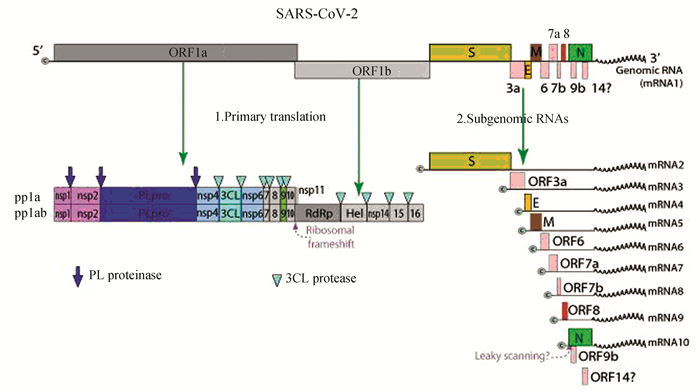
ViralZone网站公布了SARS-CoV-2基因组及表达信息(见图 5)。由图可知,SARS-CoV-2基因组编码13个开放阅读框(Open Reading Frame)。其中ORF1a/b由大约三分之二的全基因组长度组成,可通过初级翻译产生多肽pp1a和pp1ab,编码16种蛋白。其他ORFs占基因组的其余三分之一,通过基因组亚基因化表达过程,主要编码了4种蛋白——糖基化刺突蛋白(S)、包膜蛋白(E)、膜蛋白(M)、核衣壳蛋白(N),和其他辅助蛋白。
|
图 5 新型冠状病毒基因组及表达 Figure 5 SARS-CoV-2 genome and expression |
将病毒蛋白靶位(见表 2)对应至ViralZone图片(见图 5)蛋白靶点在病毒基因组中的相应位置可知,CAS提供的15种非结构蛋白靶点均由冠状病毒基因组编码的开放阅读框ORF1a/b翻译得到的两个多肽pp1a和pp1ab经过切割后形成。它们不仅可以参与构成病毒的复制-转录酶复合体(Replicase-transcriptase complex,RTC),对病毒的复制周期起关键作用,还可能具有抑制宿主基因表达、诱导细胞凋亡、抑制先天性免疫反应的作用,是病毒能够大量繁殖的优势所在。
CAS提供的4种结构蛋白靶点则是病毒基因组亚基因化表达的产物。在这4种相关的结构蛋白靶位中,有2种糖基化刺突蛋白(S),1种核衣壳蛋白(N)和1种膜蛋白(M)。刺突蛋白(S)位于病毒表面,可与宿主细胞的ACE受体结合介导病毒通过内吞作用进入细胞,还可与宿主蛋白TMPRESS2、Furin发生切割作用来获得融合能力,从而诱导病毒进入细胞。可见冠状病毒S蛋白与其宿主细胞表面受体的相互作用是决定宿主取向的关键因素,对其结构及功能的进一步研究可以指导疫苗的设计和研发[27-28]。核衣壳蛋白(N)参与病毒基因组的构成,使其保持“串珠状”结构,它也是一种高度免疫原性的磷蛋白,与病毒基因组复制和调节细胞信号通路有关。膜蛋白(M)主要位于病毒囊膜内表面,它在构成病毒内膜与核衣壳蛋白间连接支架的同时,也与子代病毒粒子的装配和释放有关。
SARS-CoV-2的结构蛋白和非结构蛋白靶位都在其感染细胞、大量增殖的过程中起关键作用,并同宿主细胞也具有一定的相互作用关系。因此,增加对这些蛋白靶点的生物学功能及互作机制的研究将对新冠肺炎的预防和治疗具有重要意义。
梳理ViralZone中给出的蛋白组描述、互作组描述信息,UniProt数据库中对病毒蛋白靶点功能注释和相互作用注释(见表 3)。
| 表 3 病毒蛋白靶点互作蛋白及功能 Table 3 Interacting proteins and functions of viral protein targets |
新冠肺炎已爆发许久,感染人群众多,但目前尚未研发出针对新冠病毒的特效药。上述分析启发我们,通过干扰宿主细胞的生物通路和病毒自身结构有可能为目前的新冠肺炎患者提供有效的治疗方案,为药物研发提供思路和方向。
在临床治疗上,可以通过干扰宿主细胞的生物通路抑制病毒感染。在蛋白富集分析的结果中我们发现网格蛋白介导型内吞是病毒进入细胞的主要途径,因此我们可以考虑在网格蛋白介导内吞过程中,靶向抑制发动蛋白或网格蛋白从而阻止病毒侵入细胞,在此领域较为常见的抑制剂有氯丙嗪等。在蛋白网络分析部分,我们得到了很多和宿主细胞免疫应答过程相关的生物过程,干扰与之相关的病毒蛋白或调节宿主细胞的关键通路,或可降低病毒毒力、恢复人体免疫力,例如干扰病毒ORF6蛋白或调控KPNA2使得干扰素信号传导过程不受影响,或补充所需干扰素以抑制细胞增殖、调节免疫;病毒的ORF8蛋白与避免肽在细胞表面表达有关,针对ORF8的抑制剂或使用IFN可能增强人体的免疫反应从而对抗新冠病毒[30]。
另外,经过我们的分析,在病毒入侵宿主细胞的过程中,NLS介导蛋白入核发挥了重要的作用,调控病毒蛋白NLS不仅可以减弱病毒的毒力,还可以恢复宿主细胞转录基因的表达,如研制靶向药物使NLS突变或缺失,从而起到有效的抗病毒作用。
病毒侵染宿主细胞时对其他蛋白通路的影响可能使人体感染相应疾病或出现相应疾病的类似临床表现,因此,旧药新用不失为一种治疗方案。例如治疗肺结核病常用的莫西沙星、左氧氟沙星和链霉素等、降压药氢氯噻嗪和氯沙坦等、减少免疫抑制的白细胞介素-1β抑制剂、用于治疗甲型流感的连花清瘟胶囊等,但这些用药方案只是根据蛋白通路的推断,同时这些药物不是针对新冠病毒的特效药,使用前应进行临床试验。
针对新冠病毒本身而言,与新冠肺炎有关的15种非结构蛋白对病毒的复制、调控、免疫起到了重要的作用,其中有些非结构蛋白可能影响病毒毒力,若能验证敲掉某非结构蛋白后病毒毒力大幅下降,则可为病毒疫苗的研发提供新思路。作为新冠肺炎靶位的结构蛋白有4种,2种S蛋白介导病毒附着并侵入宿主细胞,N蛋白与病毒基因组和宿主蛋白都存在相互作用并影响宿主造血细胞通路,M蛋白影响病毒的组装与免疫原性,针对上述结构蛋白可研发相应的病毒检测试剂和疫苗。
新冠肺炎的流行给医疗行业带来了重大压力,疫情当前,如何进行有效的预防和治疗成为了亟待解决的问题。根据蛋白靶位进行药物研发,使疾病的治疗有了更多的针对性;同时,旧药新用可以在原有的经验上节省时间。总之,对新冠病毒的蛋白靶位进行生信分析,让我们对病毒自身结构和侵染过程有了更多的了解,也为抗病毒药物和疫苗的研发提供了更多的参考。
4 结论上述分析获得了新冠病毒和宿主细胞蛋白靶点组成的蛋白通路和互作网络,由此我们进一步了解了新冠病毒的结构和侵染人体的过程:新冠病毒附着并通过内吞作用进入细胞,病毒基因组复制翻译,合成自身所需结构蛋白和非结构蛋白,抑制免疫反应,并胞吐出细胞。在此过程中,各种生物通路和组成病毒自身的蛋白质都起到了非常重要的作用。其中包括(但不限于):
1) 病毒附着后,通过网格蛋白介导的内吞作用和自身的S蛋白作用进入宿主细胞;
2) 病毒入侵细胞后,需要借助NLS进入细胞核完成复制。
3) 病毒进入细胞后,病毒的ORF8蛋白下调细胞表面MHC-Ⅰ的表达从而逃避免疫细胞的杀伤;
4) 在病毒侵染宿主细胞的过程中,还会影响其他蛋白通路,比如肺结核病、肾素-血管紧张素系统、白细胞介素-1介导的信号通路的调控、甲型流感、破骨细胞分化、造血细胞通路等。
| [1] |
WHO. WHO director-general's statement on IHR emergency committee on novel coronavirus (2019-nCoV)[EB/OL]. [2020-01-30]. https://www.who.int/dg/speeches/detail/who-director-general-s-statement-on-ihr-emergency-committee-on-novel-coronavirus-(2019-ncov).
(  0) 0) |
| [2] |
WHO. WHO director-general's opening remarks at the media briefing on COVID-19 - 11 March 2020[EB/OL]. [2020-03-11]. https://www.who.int/dg/speeches/detail/who-director-general-s-opening-remarks-at-the-media-briefing-on-covid-19---11-march-2020.
(  0) 0) |
| [3] |
WHO. WHO coronavirus disease (COVID-19) dashboard[EB/OL]. [2020-10-01]. https://covid19.who.int/.
(  0) 0) |
| [4] |
Johns Hopkins University & Medicine. Coronavirus resource center[EB/OL]. [2020-10-02]. https://coronavirus.jhu.edu/map.html.
(  0) 0) |
| [5] |
ViralZone. Betacoronavirus[EB/OL]. [2020]. https://viralzone.expasy.org/764?outline=all_by_species.
(  0) 0) |
| [6] |
WHO. Timeline: WHO's COVID-19 response[EB/OL]. [2020-09-09]. https://www.who.int/emergencies/diseases/novel-coronavirus-2019/interactive-timeline#event-158.
(  0) 0) |
| [7] |
HAO X, CHENG S, WU D, et al. Reconstruction of the full transmission dynamics of COVID-19 in Wuhan[J]. Nature, 2020, 584: 420-424. DOI:10.1038/s41586-020-2554-8 (  0) 0) |
| [8] |
医政医管局. 关于印发新型冠状病毒肺炎诊疗方案(试行第七版)的通知[EB/OL]. [2020-03-04]. http://www.nhc.gov.cn/yzygj/s7653p/202003/46c9294a7dfe4cef80dc7f5912eb1989.shtml. Medical Service and Management Administration. Notice on the issuance of 2019-nCoV pneumonia treatment plan (trial version 7)[EB/OL]. [2020-03-04]. http://www.nhc.gov.cn/yzygj/s7653p/202003/46c9294a7dfe4cef80dc7f5912eb1989.shtml. (  0) 0) |
| [9] |
医政医管局. 《新型冠状病毒肺炎诊疗方案(试行第七版)》解读[EB/OL]. [2020-03-04]. http://www.nhc.gov.cn/yzygj/s7652m/202003/a31191442e29474b98bfed5579d5af95.shtml. Medical Service and Management Administration. Interpretation of the issuance of 2019-nCoV pneumonia treatment plan (trial version 7)[EB/OL]. [2020-03-04]. http://www.nhc.gov.cn/yzygj/s7652m/202003/a31191442e29474b98bfed5579d5af95.shtml. (  0) 0) |
| [10] |
穆鑫, 赵颖, 张兰, 等. 我国治疗新冠肺炎药物临床研究现状浅析[J]. 中国临床药理学与治疗学, 2020, 25(8): 869-877. MU Xin, ZHAO Ying, ZHANG Lan, et al. Analysis of clinical research novel coronavirus pneumonia in China[J]. Clinical Pharmacology and Therapeutics in China, 2020, 25(8): 869-877. DOI:10.12092/j.issn.1009-2501.2020.08.005 (  0) 0) |
| [11] |
PAN H, PETO R, KARIM Q, et al. Repurposed antiviral drugs for COVID-19-interim WHO Solidarity trial results[EB/OL]. [2020-10-15]. DOI: 10.1101/2020.10.15.20209817.
(  0) 0) |
| [12] |
CAS. CAS COVID-19 protein target thesaurus[EB/OL]. [2020]. https://www.cas.org/covid-19-protein-target-thesaurus.
(  0) 0) |
| [13] |
ZHOU Y, ZHOU B, PACHE L, et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets[J]. Nature Communications, 2019, 10(1): 1523. DOI:10.1038/s41467-019-09234-6 (  0) 0) |
| [14] |
COOK H V, DONCHEVA N T, SZKLARCZYK D, et al. STRING: A virus-host protein-protein interaction database[J]. Viruses, 2018, 10(10): 519. DOI:10.3390/v10100519 (  0) 0) |
| [15] |
MASSON P, HULO C, DE CASTROE D, et al. Viralzone: Recent updates to the virus knowledge resource[J]. Nucleic Acids Research, 2013, 41(D1): D579-D583. DOI:10.1093/NAR/GKS1220 (  0) 0) |
| [16] |
HULO C, DE CASTRO E, MASSON P, et al. ViralZone: A knowledge resource to understand virus diversity[J]. Nucleic Acids Research, 2011, 39(Database issue): D576-D582. DOI:10.1093/nar/gkq901 (  0) 0) |
| [17] |
CLINE M S, SMOOT M, CERAMI E, et al. Integration of biological networks and gene expression data using Cytoscape[J]. Nature Protocols, 2007, 2(10): 2366-2382. DOI:10.1038/nprot.2007.324 (  0) 0) |
| [18] |
HUANG D W, SHERMAN B T, LEMPICKI R A. Systematic and integrative analysis of large gene lists using DAVID Bioinformatics Resources[J]. Nature Protocols, 2009, 4(1): 44-57. DOI:10.1038/nprot.2008.211 (  0) 0) |
| [19] |
NCBI. Severe acute respiratory syndrome coronavirus 2 iso late Wuhan-Hu-1, complete genome[EB/OL]. [2020-06-18]. https://www.ncbi.nlm.nih.gov/nuccore/NC_045512.
(  0) 0) |
| [20] |
MORI Y, OKABAYASHI T, YAMASHITA T, et al. Nuclear localization of Japanese encephalitis virus core protein enhances viral replication[J]. Journal of Virology, 2005, 79(6): 3448-3458. DOI:10.1128/JVI.79.6.3448-3458.2005 (  0) 0) |
| [21] |
DUAN Zhiqing, DENG Shanshan, JI Xinqin, et al. Nuclear localization of Newcastle disease virus matrix protein promotes virus replication by affecting viral RNA synthesis and transcription and inhibiting host cell transcription[J]. Veterinary Research, 2019, 22(1): 1-9. DOI:10.1186/s13567-019-0640-4 (  0) 0) |
| [22] |
周丽, 杨晓虹, 徐利保, 等. 网格蛋白介导型内吞作用与广谱抗病毒药[J]. 国际药学研究杂志, 2013, 40(1): 43-47+99. ZHOU Li, YANG Xiaohong, XU Libao, et al. Grid protein-mediated endocytosis and broad-spectrum antivirals[J]. Journal of International Pharmaceutical Research, 2013, 40(1): 43-47+99. DOI:10.13220/j.cnki.jipr.2013.01.019 (  0) 0) |
| [23] |
ZHANG Yiwen, ZHANG Junsong, CHEN Yingshi, et al. The ORF8 protein of SARS-CoV-2 mediates immune evasion through potently downregulating MHC-I[J/OL]. https://doi.org/10.1101/2020.05.24.111823. DOI: 10.1101/2020.05.24.111823.
(  0) 0) |
| [24] |
CARBON S, MUNGALL C. Gene Ontology Data Archive[J/OL]. https://zenodo.org/record/4495804#. Zenodo, 2018 Jul 2. DOI: 10.5281/zenodo.4495804.
(  0) 0) |
| [25] |
FRIEMAN M, YOUNT B, HEISE M, et al. Severe acute respiratory syndrome coronavirus ORF6 antagonizes STAT1 function by sequestering nuclear import factors on the rough endoplasmic reticulum/golgi membrane[J]. Journal of Virology, 2007, 81(18): 9812-9824. DOI:10.1128/JVI.01012-07 (  0) 0) |
| [26] |
ZHAO Xinggang, NICHOLLS J, CHEN Yeguang. Severe acute respiratory syndrome-associated coronavirus nucleocapsid protein interacts with Smad3 and modulates transforming growth factor-β signaling[J]. Journal of Biological Chemistry, 2008, 283(6): 3272-3280. DOI:10.1074/jbc.M708033200 (  0) 0) |
| [27] |
DE WILDE A H, SNIJDER E J, KIKKERT M, et al. Host factors in coronavirus replication[J]. Current Topics Microbiolofy Immunology, 2018, 419: 1-42. DOI:10.1007/82_2017_25 (  0) 0) |
| [28] |
WRAPP D, WANG N, CORBETT K S, et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation[J/OL]. Science, 2020. https://www.biorxiv.org/content/10.1101/2020.02.11.944462v1. DOI: 10.1126/science.abb2507.
(  0) 0) |
| [29] |
LI Jingjiao, GUO Mingquan, TIAN Xiaoxu, et al. Virus-host interactome and proteomic survey reveal potential virulence factors influencing SARS-CoV-2 pathogenesis[J]. Med (N Y), 2021, 2(1): 99-112. DOI:10.1016/j.medj.2020.07.002 (  0) 0) |
| [30] |
REN Lili, WANG Yeming, WU Zhiqiang, et al. Identification of a novel coronavirus causing severe pneumonia in human: a descriptive study[J]. Chinese Medical Journal, 2020, 133(9): 1015-1024. DOI:10.1097/CM9.0000000000000722 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


