基于系统学、分类学等的研究,SARS-CoV-2被归为冠状病毒中的Beta属,遗传物质为单股正链RNA,颗粒多为球状,直径一般在60 ~140 nm,通常具有多形性[1],病毒衣壳外由刺突样结构包裹,显微结构呈皇冠样。2020年1月7日,该病毒被初次检出且已被证明是截至目前第7种可以侵染人的冠状病毒,该病毒入侵机体可引发COVID-19。2019年12月,SARS-CoV-2的首例感染患者出现在湖北省武汉市,之后疫情迅速传播到中国众多省份及其他169个国家和地区[2]。COVID-19的初期临床病征通常与普通感冒相似,以浑身发热、乏力为主,部分严重个体可发展为脓毒症、凝血功能障碍等[3],有时甚至会危及神经系统。
与其他侵染人的冠状病毒相比,SARS-CoV-2的基因特征存在显著差别。据研究显示,生活于云南省的一种蝙蝠(Rhinolophus affinis)可能为SARS-CoV-2的自然宿主,其体内检测到的冠状病毒(BatCovRaTG13)在基因水平上与SARS-CoV-2极其相似,两者相似率高达96.2 %[4]。
棘突蛋白(Spike protein,S)、核衣壳蛋白(Nucleocapsid phosphoprotein,Np)、小包膜蛋白(Envelope protein,E)、囊膜蛋白(Membrane protein,M)为SARS-CoV-2编码的四种结构蛋白[5]。其中,Np相较其它三种蛋白(S、E、M)表达量更高,其为多功能碱性磷酸化蛋白,具有RNA伴侣活性,于细胞质中与RNA结合组成核糖核蛋白颗粒,从而保护病毒自身的遗传物质[6]。同时,它在病毒自身的复制、转录、组装等方面也占据了重要地位[7-8]。因具有免疫原性强和保守性高的特征,Np不仅能够诊断冠状病毒,而且可以作为抗病毒治疗的理想靶标[9-10]。
本研究通过对Wuhan-Hu-1株Np的碱基序列进行进化分析,并通过对Np氨基酸序列的空间结构及其B细胞优势抗原表位的分析预测,结合多种因素综合剖析以筛选合适的B细胞优势抗原表位,为研制SARS-CoV-2表位疫苗、快速诊断试剂和制备其单克隆抗体奠定基础,从而进一步解决COVID-19迫切的防控及治疗问题。
1 材料与方法 1.1 材料从NCBI数据库(https://www.ncbi.nlm.nih.gov/)中获取Wuhan-Hu-1株Np氨基酸序列(Protein ID:YP_009724397.2)。
1.2 方法 1.2.1 不同地区SARS-CoV-2 N蛋白基因进化分析以NCBI数据库中不同国家地区多株SARS-CoV-2 N蛋白基因为研究对象,采用MEGA5.05软件中的Neighbor-joining法进行分析,构建SARS-CoV-2 N蛋白碱基序列的系统发生树,并应用MegAlign软件分析不同毒株间的遗传距离。
1.2.2 Wuhan-Hu-1株Np三维结构的预测以Wuhan-Hu-1株Np氨基酸序列为研究对象,应用Phyre2在线工具(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)预测其三维空间结构,并应用Chimera软件模拟其立体结构。
1.2.3 Wuhan-Hu-1株Np的二级结构和抗原表位分析以DNAStar软件中的Protean模块下的方法剖析Wuhan-Hu-1株Np,步骤如下:首先对Np的二级结构(Garnier-Robson法和Chou-Fasman法)、柔性区段(Karplus-Schulz法)、表面可能性(Emini法)进行预测;然后分析Np的亲水性(Kyte-Doolittle法),再预测其抗原指数和抗原表位指数(分别用Jameson-Wolf法和Kolaskar-Tongaonkar法(http://tools.immuneepitope.org/tools/bcell/iedb_input));最后,参考Np的三维结构,对Wuhan-Hu-1株Np的B细胞优势抗原表位进行综合预测。
2 结果与分析 2.1 SARS-CoV-2 N蛋白基因进化分析以Wuhan-Hu-1株(WHU01)N蛋白和其他不同国家地区的SARS-CoV-2毒株N蛋白的碱基序列为分析对象,采用Neighbor-joining法(步长1 000,boot-strap值>20 %)构建系统发生树(见图 1)。MegAlign软件分析结果显示:不同国家地区分离的SARS-CoV-2毒株N蛋白碱基序列存在极高相似性(99.6 %~100 %)。结果表明,不同国家地区间的SARS-CoV-2毒株N蛋白碱基序列差异性很小,指示SARS-CoV-2毒株N蛋白基因高度保守。
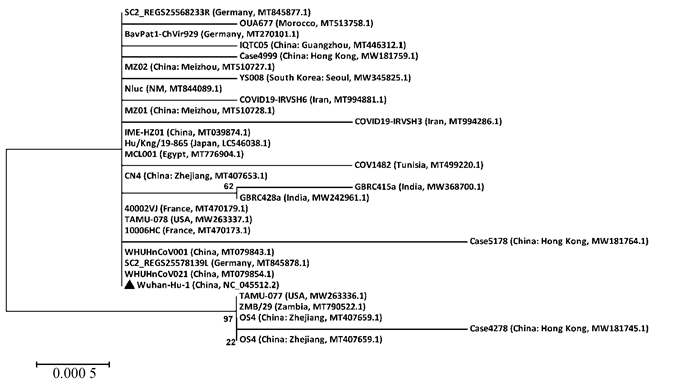
|
图 1 不同国家地区SARS-CoV-2 N蛋白碱基序列系统发生树 Figure 1 Phylogenetic tree of SARS-CoV-2 N protein base sequence isolated from different countries and regions |
应用Chimera软件模拟得到的Wuhan-Hu-1株Np的3D结构如图所示:蛋白中央存在一定数量的β-折叠,指示其内部可能含有大量疏水性残基;蛋白外围分布有一定数量的β-转角和无规则卷曲结构,且含少量的α-螺旋(见图 2)。
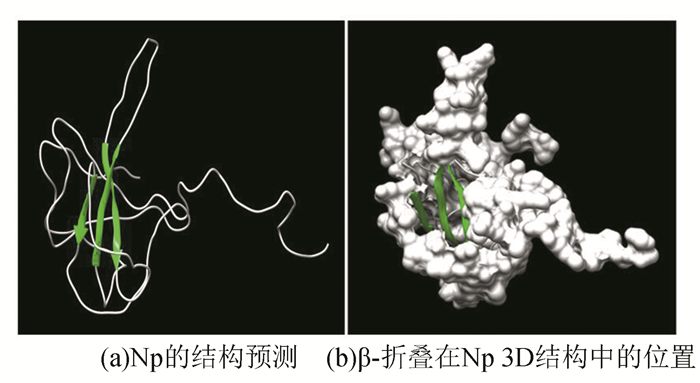
|
图 2 Wuhan-Hu-1株Np的结构预测 Figure 2 Protein structure prediction of Np in Wuhan-Hu-1 strain 注: (a)中亮绿色区域代表β-折叠结构,白色区域代表α-螺旋、β-转角及无规则卷曲结构. |
以两种不同的方法分别剖析Np的二级结构,最终结果可相互佐证。Garnier-Robson法预测的α-螺旋主要分布在氨基酸序列的中后段,且366~385区段跨区最大(20 aa),存在单个氨基酸组成的α-螺旋;有较多数量的β-折叠,其分布较均匀,最大的一个区段为262~274(13 aa);β-转角分布也较均匀,其中189~196区段最大(8 aa),存在单个氨基酸。Chou-Fasman法与Garnier-Robson法预测结果有部分区域重叠:α-螺旋呈分散分布,其中368~382区段存在跨最大区域的α-螺旋(15 aa),β-折叠呈分散分布,其预测数量相对较少;β-转角区域分布均匀,最大的一个区段为182~209(28 aa)(见图 3、表 1、表 2)。以Karplus-Schulz法预测出的Wuhan-Hu-1株Np柔性区段分布在5~15,17~50,57~83,89~106,114~119,125~128,136~153,161~169,174~208,211~216,225~250,254~266,275~299,309~310,325~329,339~348,361~392,401~416,可见Np存在多个柔性区段,有可能形成多个优势表位(见图 4)。
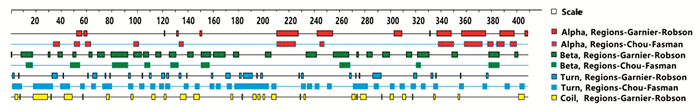
|
图 3 不同方法预测Wuhan-Hu-1株Np的二级结构 Figure 3 Secondary structure of Np in Wuhan-Hu-1 strain predicted by different methods |
| 表 1 Garnier-Robson法预测的Wuhan-Hu-1株Np的二级结构 Table 1 Secondary structure of Np in Wuhan-Hu-1 strain predicted by Garnier-Robson method |
| 表 2 Chou-Fasman法预测的Wuhan-Hu-1株Np的二级结构 Table 2 Secondary structure of Np in Wuhan-Hu-1 strain predicted by Chou-Fasman method |

|
图 4 Karplus-Schulz法预测Wuhan-Hu-1株Np的柔性区段 Figure 4 Flexible regions of Np in Wuhan-Hu-1 strain predicted by Karplus-Schulz method |
亲水性(Kyte-Doolittle法)分析显示(见图 5),Np亲水区段占比较高,几乎覆盖大多数区域,进一步证明该蛋白亲水性较强。表面可能性(Emini法)分析显示(见图 6),1~13,20~49,58~68,75~97,182~207,226~270,276~283,294~301,339~347,356~390,401~410,414~419区域的表面可能性较大。分析结果指示,两种方法预测的重复部分,极有可能位于Np表面,具备作为病毒抗原表位的特性。

|
图 5 Wuhan-Hu-1株Np的亲水性 Figure 5 Hydrophilicity of Np in Wuhan-Hu-1 strain |
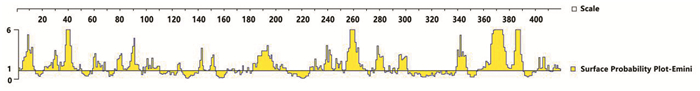
|
图 6 Wuhan-Hu-1株Np的表面可能性 Figure 6 Surface probability of Np in Wuhan-Hu-1 strain |
Jameson-Wolf法预测结果显示(见图 7),有多个抗原指数较高的区域分布于Wuhan-Hu-1株Np中,如2~12,17~48,58~71,75~85,88~108,113~130,137~153,171~216,226~267,274~300,337~349,355~390区域,其中171~216为抗原指数最高的区段,最有可能作为优势表位。
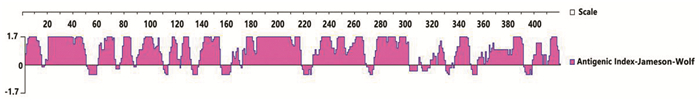
|
图 7 Wuhan-Hu-1株Np的抗原指数 Figure 7 Antigenic index of Np in Wuhan-Hu-1 strain |
Kolaskar-Tongaonkar法预测结果显示(见图 8),Wuhan-Hu-1株Np的抗原指数在0.874~1.197之间波动,均值为0.988。
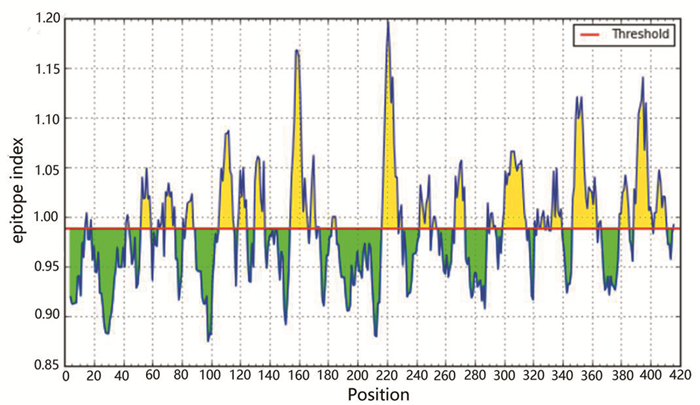
|
图 8 Wuhan-Hu-1株Np的抗原表位指数 Figure 8 Antigenic epitope index of Np in Wuhan-Hu-1 strain |
先对Wuhan-Hu-1株Np序列的柔性区段、亲水性指数、抗原指数、蛋白质表面可能性等抗原表位参数进行分析,再参考抗原表位指数结果,预测: 如果某一区段满足抗原指数≥ 0.988(均值)、表面可能性≥ 1、亲水性≥ 0,至少包含6个氨基酸残基,且内部或邻近区存在柔性区段,则推测该区段很大概率可以作为Np的B细胞抗原表位。结合空间预测结果,共筛选出符合上述条件的16个优势表位(见表 3)。
| 表 3 Wuhan-Hu-1株Np B细胞抗原表位的氨基酸序列 Table 3 Amino acid sequence of B-cell antigenic epitope of Np in Wuhan-Hu-1 strain |
抗原表位(Epitope)是指决定抗原特异性且位于分子表面的氨基酸序列及其组成的空间结构[11]。抗体或淋巴细胞表面与抗原相结合部位的形状、大小需与抗原表位相适合才能发挥其免疫学效应[12]。表位的分子量一般很小(5~7 aa),所在区段结构多数比较松散、具有可移动性。作为病毒抗原性的物质基础,表位预测可为疾病的诊断、药物的筛选与合成及表位疫苗的制备等奠定理论基础[13]。本研究从NCBI获取了Wuhan-Hu-1株Np氨基酸序列(419 aa),空间结构预测结果指示,Wuhan-Hu-1株Np空间构象形似锤状,中央的疏水核心聚集了一定数量的β-折叠且有较多的β-转角和无规则卷曲分布于蛋白表面,同时存在少量的α-螺旋。近期有报道称欧洲SARS-CoV-2基因发生突变并传入中国[14-16],鉴于该病毒中Np较强的抗原性和保守性,准确预测Wuhan-Hu-1株Np抗原表位可以为未来进一步的工作提供方向。
SARS-CoV-2粒子多呈皇冠状[17],Np是其主要编码的四种结构蛋白(Np、M、E、S)之一,在复制初期产生并与病毒遗传物质组成核壳体结构[6, 18]。Np通常大量表达于病毒侵染的初期[19-20],具有很强的抗原性。因此,常作为其他Beta冠状病毒如SARS-CoV、MERS-CoV等的快速诊断抗原和相关疫苗设计的靶标[21-24],具有特异、敏感、重复性好的优势。值得一提的是,广谱抗病毒药物和疫苗可对后续由病毒变异引起的新型传染病起到关键性作用,而Np在种属间的高度保守性[25],正使其占领了独特的优势。目前京天成生物、Bioss等科研机构已研制出针对Wuhan-Hu-1株Np的抗体,虽然还未进入临床使用,但为靶向Np抗体的设计开创了先例,而可识别Np的表位疫苗研制却存在较大空白。对于疫苗的制备,小分子蛋白更易做到快速、大量地表达,表位疫苗相对于减毒活疫苗和死疫苗又能达到更高的安全性[26]。所以,准确推断B细胞抗原表位的特定氨基酸序列,并通过表达相应基因以实现工厂化生产具有免疫原性的低毒多肽,可以成为制备诊断试剂、表位疫苗、单抗的新手段。为提高准确度,本研究采用新兴的多参数综合预测法。分析结果指示,Np抗原指数均值为0.988,抗原表位指数较高的区域分布均匀,最大值为1.197,其中52~59、69~75、83~89、106~115、119~124、130~136、154~166、217~227、243~249、267~273、299~315、333~339、347~363、379~385、389~401、403~411氨基酸区段覆盖了大部分抗原指数较高的区域,指示这些区段可能具有作为表位的潜能。本研究基于同源建模(TBM)参数预测结果,通过与同一毒株的小角X射线散射(SAXS)获得的低分辨率三维结构(pdb id: 6yun)[27]进行比较,发现二者内部均由若干近乎平行的β-折叠形成的领结形状偏离了中心的菱形瓦片结构组成,这与已报道的基于saxs的两个浮动NTD模型是相容的[28-29],但二者结果仍然存在一定的结构差异。这也许是由不同预测方法造成的,同时也体现了多参数预测以及后期验证的重要性。
Hopp-Woods开创的亲水性参数预测法[30-31]为抗原表位的预测奠定了基础,但与单一参数分析法相比,多参数预测的准确率明显更高[26, 32]。可见,由于抗原表位的多态性,表位抗原性取决于多种因素的互作而非任意单一条件决定。B细胞抗原表位应位于蛋白质表面且具有一定程度的灵活性,为更准确地与抗体或淋巴细胞表面配体相结合,抗原构象通常需要产生一定程度的改变,即其多具有柔韧性;此外,抗原表位生物学特性与其二级结构和三级结构也有较高相关性。准确剖析Np的抗原表位不但有助于进一步认识其表现出抗原性的结构基础,而且能够为指导药物的合成筛选、快速诊断试剂的研发制备和表位疫苗的设计生产等提供理论基础[33]。
4 结论通过对Wuhan-Hu-1株Np的碱基序列进行进化分析,并通过单独分析Np的二级结构、柔韧性、亲水性、表面可能性、抗原指数等多种参数,再利用三级结构预测结果筛选表位,最终综合以上所有参数多维筛选确定Np的B细胞优势抗原表位。研究结果为进一步阐明Np的结构特征及深入探究SARS-CoV-2入侵和致病机制提供了参考,以期助力解决SARS-CoV-2感染引发的肺炎疫情及迫切的防控问题。
| [1] |
詹菁, 刘倩, 张雨竹, 等. 新型冠状病毒2019-nCoV的一些初步认识[J]. 环境化学, 2020, 39(2): 283-291. ZHAN Jing, LIU Qian, ZHANG Yuzhu, et al. Preliminary understanding of the novel coronavirus 2019-nCoV[J]. Environmental Chemistry, 2020, 39(2): 283-291. DOI:10.7524/j.issn.0254-6108.2020021501 (  0) 0) |
| [2] |
LIU Yingxia, YANG Yang, ZHANG Cong, et al. Clinical and biochemical indexes from 2019-nCoV infected patients linked to viral loads and lung injury[J]. Science China(Life Sciences), 2020, 63(3): 364-374. DOI:10.1007/s11427-020-1643-8 (  0) 0) |
| [3] |
HUANG Chaolin, WANG Yeming, LI Xingwang, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China[J]. Lancet, 2020, 395(10223): 497-506. DOI:10.1016/S0140-6736(20)30183-5 (  0) 0) |
| [4] |
ZHOU Ping, YANG Xingluo, WANG Xianguang, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin[J]. Nature, 2020, 579: 270-273. DOI:10.1038/s41586-020-2012-7 (  0) 0) |
| [5] |
边葶苈, 周继勇, 廖敏. 冠状病毒非结构蛋白的研究进展[J]. 中国动物传染病学报, 2013, 21(4): 67-74. BIAN Tingli, ZHOU Jiyong, LIAO Min. Research advance on nonstructural proteins of coronavirus[J]. Chinese Journal of Veterinary Parasitology, 2013, 21(4): 67-74. DOI:10.3969/j.issn.1674-6422.2013.04.013 (  0) 0) |
| [6] |
STURMAN L S, HOLMES K V, BEHNKE J. Isolation of coronavirus envelope glycoproteins and interaction with the viral nucleocapsid[J]. Journal of Virology, 1980, 33(1): 449-462. DOI:10.1128/JVI.33.1.449-462.1980 (  0) 0) |
| [7] |
CHAN J F W, LAU S K P, TO K K W, et al. Middle east respiratory syndrome coronavirus: another zoonotic betacoronavirus causing SARS-Like disease[J]. Clinical Microbiology Reviews, 2015, 28(2): 465-522. DOI:10.1128/CMR.00102-14 (  0) 0) |
| [8] |
ZUMLA A, CHAN J F W, AZHAR E I, et al. Coronaviruses—drug discovery and therapeutic options[J]. Nature Reviews Drug Discovery, 2016, 15(5): 327-347. DOI:10.1038/nrd.2015.37 (  0) 0) |
| [9] |
杨海燕. 利用噬菌体肽库技术筛选SARS冠状病毒N蛋白B细胞表位的研究[D]. 广州: 第一军医大学, 2006. YANG Haiyan. Screening of B cell epitopes of SARS-cov N protein by phage display peptide library[D]. Guangzhou: Southern Medical University, 2006. (  0) 0) |
| [10] |
魏姗. 猪δ冠状病毒N蛋白单克隆抗体制备及抗原表位的初步鉴定[D]. 大庆: 黑龙江八一农垦大学, 2018. WEI Shan. Preparation of N protein monoclonal antibody of porcine delta coronavirus and identification of its antigenic epitope[D]. Daqing: Heilongjiang Bayi Agricultural University, 2018. (  0) 0) |
| [11] |
朱锡华, 吴玉章. 对表位生物学研究的认识和体会[J]. 上海免疫学杂志, 1998(1): 1-2. ZHU Xihua, WU Yuzhang. Understanding and understanding of epitope biology[J]. Shanghai Journal of Immunology, 1998(1): 1-2. (  0) 0) |
| [12] |
RUF J, CARAYON P, SARLES-PHILIP N, et al. Specificity of monoclonal antibodies against human thyroglobulin; comparison with autoimmune antibodies[J]. EMBO Journal, 1983, 2(10): 1821-1826. DOI:10.1002/j.1460-2075.1983.tb01664 (  0) 0) |
| [13] |
李海侠, 毛旭虎. 蛋白质抗原表位研究进展[J]. 微生物学免疫学进展, 2007(1): 54-58. LI Haixia, MAO Xuhu. Progress in the study of protein antigenic epitopes[J]. Progress in Microbiology and Immunology, 2007(1): 54-58. DOI:10.13309/j.cnki.pmi.2007.01.015 (  0) 0) |
| [14] |
PACHETTI M, MARINI B, BENEDETTI F, et al. Emerging SARS-CoV-2 mutation hot spots include a novel RNA-dependent-RNA polymerase variant[J]. Journal of Translational Medicine, 2020, 18(1): 179. DOI:10.1186/s12967-020-02344-6 (  0) 0) |
| [15] |
European Centre for Disease Prevention and Control. Rapid increase of a SARS-CoV-2 variant with multiple spike protein mutations observed in the United Kingdom, December 2020[N]. ECDC: Stockholm, 2020.
(  0) 0) |
| [16] |
CHEN H, HUANG X, ZHAO X, et al. The first case of new variant COVID-19 originating in the united kingdom detected in a returning student — Shanghai Municipality[N]. China CDC Weekly, 2021, 3(1): 1-3.
(  0) 0) |
| [17] |
ZHU N, ZHANG D, WANG W, et al. A novel coronavirus from patients with pneumonia in China, 2019[J]. New England Journal of Medicine, 2020, 382(8): 727-733. DOI:10.1056/NEJMoa2001017 (  0) 0) |
| [18] |
ESCORS D, ORTEGO J, LAUDE H, et al. The membrane M protein carboxy terminus binds to transmissible gastroenteritis coronavirus core and contributes to core stability[J]. Journal of Virology, 2001, 75(3): 1312-1324. DOI:10.1128/JVI.75.3.1312-1324.2001 (  0) 0) |
| [19] |
KUO L, MASTERS P S. Genetic evidence for a structural interaction between the carboxy termini of the membrane and nucleocapsid proteins of mouse hepatitis virus[J]. Journal of Virology, 2002, 76(10): 4987-4999. DOI:10.1128/jvi.76.10.4987-4999.2002 (  0) 0) |
| [20] |
NARAYANAN K, CHEN C J, MAEDA J, et al. Nucleocapsid-independent specific viral RNA packaging via viral envelope protein and viral RNA signal[J]. Journal of Virology, 2003, 77(5): 2922-2927. DOI:10.1128/jvi.77.5.2922-2927.2003 (  0) 0) |
| [21] |
李秀华, 王佑春. 抗SARS病毒N蛋白单克隆抗体的制备和初步应用[J]. 中华实验和临床病毒学杂志, 2004, 18(3): 255-257. LI Xiuhua, WANG Youchun. Development and preliminary application of monoclonal antibodies against N protein of SARS virus[J]. Chinese Journal of Experimental and Clinical Virology, 2004, 18(3): 255-257. DOI:10.3760/cma.j.issn.1003-9279.2004.03.015 (  0) 0) |
| [22] |
王平, 黄庆生, 薛小平, 等. SARS冠状病毒核衣壳蛋白的重组表达及其单克隆抗体的制备[J]. 细胞与分子免疫学杂志, 2005, 3(1): 50-52. WANG Ping, HUANG Qingsheng, XUE Xiaoping, et al. Preparation of specific monoclonal antibody against nucleocapsid protein of SARS coronavirus[J]. Chinese Journal of Cellular and Molecular Immunology, 2005, 3(1): 50-52. DOI:10.3321/j.issn:1007-8738.2005.01.014 (  0) 0) |
| [23] |
HU D, ZHU C, AI L, et al. Genomic characterization and infectivity of a novel SARS-like coronavirus in Chinese bats[J]. Emerging Microbes & Infections, 2019, 7(1): 1-10. DOI:10.1038/s41426-018-0155-5 (  0) 0) |
| [24] |
SONG D, HA G, SERHAN W, et al. Development and validation of a rapid immunochromatographic assay for detection of Middle East respiratory syndrome coronavirus antigen in dromedary camels[J]. Journal of Clinical Microbiology, 2015, 53(4): 1178-1182. DOI:10.1128/JCM.03096-14 (  0) 0) |
| [25] |
ZHOU P, YANG X L, WANG X G, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin[J]. Nature, 2020, 579(7798): 270-273. DOI:10.1038/s41586-020-2012-7 (  0) 0) |
| [26] |
郭春艳, 赵向绒, 胡军. B细胞抗原表位的研究进展及其应用[J]. 生物技术通讯, 2013, 24(2): 266-270. GUO Chunyan, ZHAO Xiangrong, HU Jun. Research and application of B cell epitope[J]. Letters in Biotechnology, 2013, 24(2): 266-270. DOI:10.3969/j.issn.1009-0002.2013.02.029 (  0) 0) |
| [27] |
ZINZULA L, BASQUIN J, BOHN S, et al. High-resolution structure and biophysical characterization of the nucleocapsid phosphoprotein dimerization domain from the Covid-19 severe acute respiratory syndrome coronavirus 2[J]. Biochemical Biophysical Research Communications, 2021, 538: 54-62. DOI:10.1016/j.bbrc.2020.09.131 (  0) 0) |
| [28] |
ZENG Weihong, LIU Guangfeng, MA Huan, et al. Biochemical characterization of SARS-CoV-2 nucleocapsid protein[J]. Biochemical Biophysical Research Communications, 2020, 527(3): 618-623. DOI:10.1016/j.bbrc.2020.04.136 (  0) 0) |
| [29] |
CHANG C K, HSU Y L, CHANG Y H, et al. Multiple nucleic acid binding sites and intrinsic disorder of severe acute respiratory syndrome coronavirus nucleocapsid protein: implications for ribonucleocapsid protein packaging[J]. Journal of Virology, 2009, 83(5): 2255-2264. DOI:10.1128/JVI.02001-08 (  0) 0) |
| [30] |
HOPP T P, WOODS K R. Prediction of protein antigenic determinants from amino acid sequences[J]. Proceedings of the National Academy of Sciences of the United States of America, 1981, 78(6): 3824-3828. DOI:10.1073/pnas.78.6.3824 (  0) 0) |
| [31] |
KYTE J, DOOLITTLE R F. A simple method for displaying the hydropathic character of a protein[J]. Journal of Molecular Biology, 1982, 157(1): 105-132. DOI:10.1016/0022-2836(82)90515-0 (  0) 0) |
| [32] |
孙建宏, 曹殿军. 细胞的抗原表位研究方法[J]. 动物医学进展, 2004, 25(5): 18-21. SUN Jianhong, CAO Dianjun. New approach for antigen epitope study[J]. Progress in Veterinary Medicine, 2004, 25(5): 18-21. DOI:10.3969/j.issn.1007-5038.2004.05.006 (  0) 0) |
| [33] |
纪锋, 徐黎明, 赵景壮, 等. 鲤春病毒血症病毒Shlj1株糖蛋白空间结构及其B细胞抗原表位的预测[J]. 大连海洋大学学报, 2017, 32(4): 440-446. JI Feng, XU Liming, ZHAO Jingzhuang, et al. Prediction of spatial structure and B cell antigenic epitope of glycoprotein in Shlj 1 strain of Spring Viremia of Carp[J]. Journal of Dalian Ocean University, 2017, 32(4): 440-446. DOI:10.16535/j.cnki.dlhyxb.2017.04.011 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


