结直肠癌(Colorectal cancer, CRC)是一种发生率极高的恶性肿瘤[1]。结肠腺癌(Colon adenocarcinoma, COAD)是结直肠癌最常见的病理类型之一(约占95%)[2]。然而目前现有的结肠腺癌的治疗手段(包括手术和化疗等综合治疗)仍然不能达到令人满意的效果,结肠腺癌的五年生存率依然不容乐观[3]。大部分患者发现和初诊为结肠腺癌时已处于癌症晚期,因此,现阶段的医疗诊治过程中晚就诊、缺乏可靠的生物标志物和不精准的治疗靶点已成为治疗结肠腺癌的主要障碍[4],而早期诊断和治疗对于改善患者的预后和生活质量至关重要。最新的研究表明,除了遗传变异以外,扰乱的、不平衡的表观遗传基因组也是癌症发生发展的重要原因[5]。DNA甲基化是一种重要的调控基因表达的表观遗传修饰,与癌症的发生和发展紧密相关[6]。异常的DNA甲基化通过激活癌基因和/或使肿瘤抑制基因失活诱导癌症的发生发展[7]。通过因此本课题旨在通过结肠腺癌DNA甲基化谱,分析患者的DNA甲基化模式并识别出与预后相关的结肠腺癌亚型。
1 材料与方法 1.1 DNA甲基化数据的获取和预处理从癌症基因组图谱(The Cancer Genome Atlas,TCGA,https://www.cancer.gov/about-nci/organization/ccg/research/structural-genomics/tcga)数据库中下载了结肠腺癌样本DNA甲基化数据和这些样本对应病人的临床信息,甲基化数据全部由Illumina Infinium HumanMethylation450 BeadChip芯片平台产生。选取324个记录了患者生存状态的结肠腺肿瘤样本数据进行接下来的分析。研究表明,发生在DNA启动子区的异常甲基化对癌症的发生发展起着重要调控作用。在本研究中启动子区域定义为转录起始位点上游2 kb至下游0.5 kb。首先选取癌症样本数据中属于启动子区的位点,去除其中的SNP位点、对应于性染色体上的位点和一个位点对应多个基因的位点。接下来去除70%以上的样本中有缺失的位点,最后使用k近邻算法(knn)补全其余的缺失值,该算法使用“impute” R包中的impute.knn()函数实现,其中参数k取10、maxp取3 000。
1.2 提取分类特征为寻找在癌症和癌旁样本中甲基化水平有差异且差异具有显著性的CpG位点,计算每个位点在癌症和癌旁样本中的甲基化水平平均值,求出均值差并用t检验方法计算该差异的显著性。本研究将均值差绝对差异>0.1且经Benjamini-Hochberg多重检验校正方法校正后p < 0.05的CpG位点视作差异甲基化位点。将324个样本按7∶3的比例划分为训练集与测试集,分组原则为随机分组且死亡率相似。最后分为226个样本的训练集和98个样本的测试集。
为获得与预后相关的结肠腺癌分子亚型,将使用显著影响生存的CpG位点作为分类特征。首先,使用每个样本所对应患者的生存状态和生存时间为每个差异甲基化CpG位点和患者的性别、年龄、临床分期构建单变量COX比例风险回归模型,分析每个CpG位点的甲基化水平、年龄、阶段与预后的关系。再将单变量中显著影响预后的临床因素作为协变量引入多变量模型筛选得到独立影响预后的CpG位点。对于每个CpG位点i,单变量和多变量COX比例风险回归模型的公式定义如下:
| $ h(t, x)_{i}=h 0(t) exp \left(\beta_{{methy }} { methy }_{i}\right) $ | (1) |
| $ h(t, x)_{i}=h 0(t) exp \left(\beta_{{methy }} { methy }_{i}+\beta_{{stage }} { stage }\right) $ | (2) |
式中h0(t)是基准风险方程,可以是任意一个针对时间t的非负方程,methyi是代表CpGi甲基化水平的矢量,stage是代表患者临床分期的矢量,βmethy和βstage代表回归系数。该模型使用Benjamini-Hochberg多重检验校正方法对P值进行校正。
1.3 识别预后相关的DNA甲基化亚组在指定聚类算法和度量距离时,使用ConcensusClusterPlus包内嵌的多种聚类算法和距离进行尝试,以探寻最优的聚类结果。根据输出的结果文件,使用欧几里得距离为度量的K均值聚类算法被纳入考虑范围。
分组数k的确定是本研究要解决的一个重要问题,对从k=2到k=10的不同分组数,应用上一步的聚类方法,除了固有的输出结果外,对不同分组数下类别的平均一致性和一致性变异系数进行了计算。最终用于确定聚类方法和聚类数的标准是:在某一聚类数k下,组间的一致性数值相对较高、变异系数相对较低,并且CDF曲线下面积在相邻的两个类别数量间的改变趋势较缓。变异系数根据下式计算:
| $ C V=\left(\frac{S D}{M N}\right) \times 100 \% $ | (3) |
式(3)中SD是同一分类数下不同分组间一致性的标准差,MN是同一分类数下不同分组间的平均一致性。根据上述标准,最终选择使用K均值算法,以欧几里得距离作为相似度指标的聚类方法,确定聚类数目k为7。
1.4 不同甲基化亚组间的预后分析通过与预后相关的CpG位点区分出了七个亚组,自然应该考察这七个DNA甲基化亚组间的预后情况。对得到的甲基化亚组进行Kaplan-Meier生存分析。这一分析通过“survival”R包中的函数survfit( )和survdiff( )完成。并使用对数秩检验(log-rank test)分析生存差异的统计学显著性。
1.5 各甲基化亚组间的特征描述目前结肠腺癌还没有通过临床病理特征定义的细分亚型,在分析并确定了不同DNA甲基化亚型间的预后差异之后,进一步分析各个甲基化亚型与临床特征的相关性,所有检验方法若无特殊说明均采取双侧检验。
根据临床数据的情况,使用费希尔精确检验(Fisher’s exact test)分析各临床特征在不同甲基化亚型间的分布是否有显著差异。
在每个甲基化亚型内,对每个临床特征和总体之间进行超几何检验,以分析各临床特征在各甲基化亚型内的富集是否具有显著性。
1.6 筛选甲基化亚型的特异性标记使用一种基于香农熵模型可从全基因组甲基化谱中鉴定差异甲基化区域(DMR)的软件QDMR[12](Quantitative Differentially Methylated Regions),从用于识别七个甲基化亚组的137个CpG位点中筛选每个亚组的特异性甲基化标记。由于在前述亚组确定的过程中采用了一致性聚类的方法,这意味着各个亚组内的样本有着相似的甲基化模式。因此,本研究计算用于一致性聚类的137个特征位点在七个亚组中的甲基化均值,以该均值来表征各位点在不同亚组中的甲基化状态,将得到285×6维矩阵作为QDMR的输入。在衡量特异性的过程中,参数SD被设置为0.07。
1.7 构建和评估甲基化亚型预测模型本研究的DNA甲基化亚型由前述无监督的一致性聚类方法得到,为了验证DNA甲基化分型结果并建立更为便捷精确的结肠腺癌甲基化分型方法,使用有着7个甲基化亚型标签的训练集构建有监督的SMO分类模型。在98个样本的测试集中使用1.2中得到的预测模型,根据样本的甲基化数据将患者划分到7个DNA甲基化亚型中,也就是使用SMO分类模型将训练集得到的7个甲基化亚型标签分配给测试集中的样本。然后对测试集中7个甲基化亚型进行生存分析,验证模型的稳定性和准确性。
2 结果和分析 2.1 结肠腺癌DNA甲基化特征的筛选本研究将均值差绝对差异> 0.1且p < 0.05的CpG位点视作样本对之间显著差异的CpG位点。最终得到26 158个差异甲基化位点,这一结果可视化使用R包ggplot(见图 1)。

|
图 1 结肠腺癌差异甲基化位点火山图 Figure 1 Volcano plot of differential methylation sites of colon adenocarcinoma 注:横坐标是癌症癌旁样本间不同CpG位点的均值差,纵坐标是经过多重检验校正后的p值负对数。在均值差阈值和P值分别设置为0.1和0.05的情况下,红色区域为差异且显著的低甲基化位点,蓝色区域是差异且显著的高甲基化位点。 |
为获得与预后相关的结肠腺癌分子亚型,使用显著影响存活的CpG位点作为分类特征。首先将所有结肠腺癌样本按照7∶3的比例划分为训练集和测试集,分别包含226个样本和98个样本。接着,为训练集中的样本基于生存时间和存活情况构建COX比例风险回归模型。单变量COX模型获得的2 838个与预后显著相关位点被用于多变量COX模型构建,3个显著的临床因素“分期”(gender: p=0.017, age: 0.004, stage: 0.016)被作为协变量也引入其中,最终多变量的回归模型获得137个依旧显著的位点,部分位点所对应的基因(见表 1)。
| 表 1 特异性位点基因对应表 Table 1 Characteristic sites and corresponding gene symbols |
P值使用Benjamini-Hochberg方法进行了多重检验校正,Hazard.Ratio代表着COX模型的风险比,风险比是相对而言的,风险比大于1的位点被认为与不良预后相关,小于1的位点被认为与良好预后相关。137个CpG位点将被用作分组特征。
2.2 基于一致性聚类的结肠腺癌亚组识别进一步对所获得的137个CpG位点进行一致性聚类以获得与预后相关的结肠腺癌DNA甲基化亚型。本研究计算每个类别的平均聚类一致性和聚类之间的变异系数,结合ConcensusClusterPlus包的输出结果(见图 2),根据1.3中描述的方法来确定选择最优的聚类算法和类别数k。
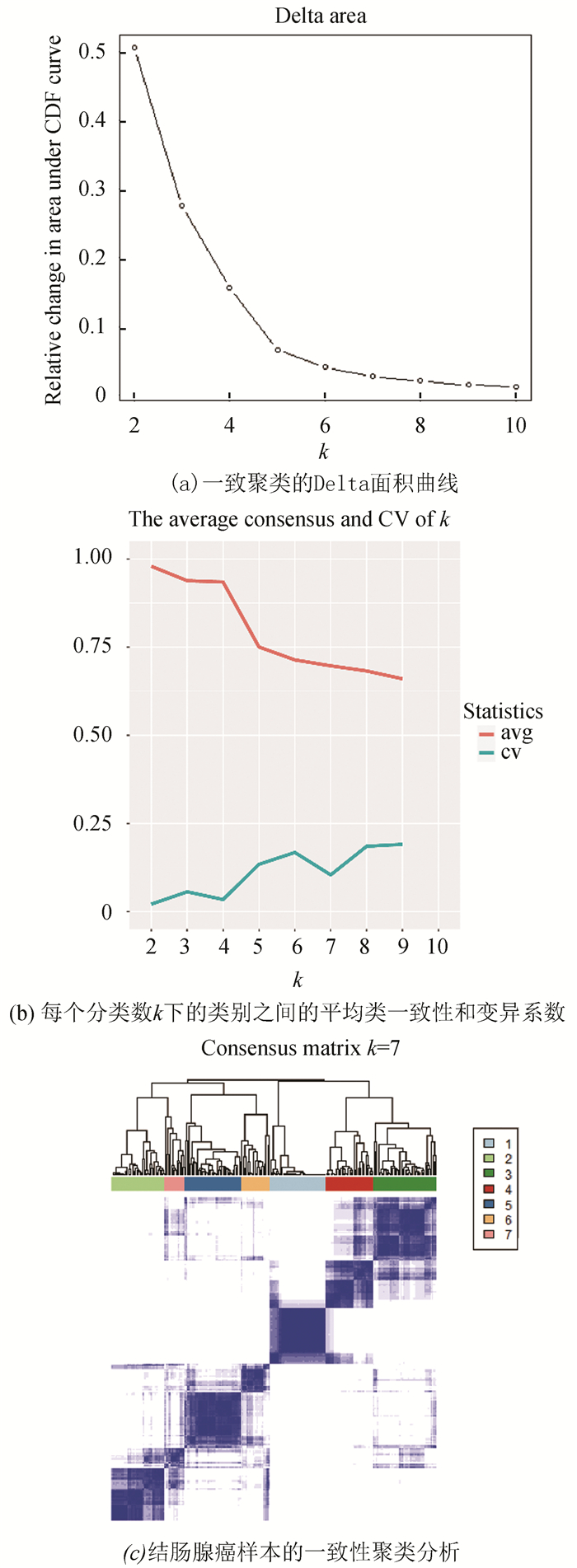
|
图 2 基于K均值算法的结肠腺癌亚型分类标准 Figure 2 Classification standards for colon adenocarcinoma subtypes based on k-means algorithm 注:(a)表示每个类别号k与k-1相比的累积分布函数(CDF)曲线下面积的相对变化。横轴表示类别编号k,纵轴表示CDF曲线下面积的相对变化。(b)中红线代表平均类一致性,蓝线代表类间的变异系数。 |
对于K均值算法,CDF曲线下面积改变趋势从聚类数目k=6开始明显趋缓且分类数为7时,其平均聚类一致性曲线和变异系数曲线有一个明显的拐点,拥有相对高的一致性系数和相对低的变异系数。因此,对于K均值算法,认为它在聚类数为7时具有较高的聚类稳定性,k=7是最适聚类数。最终确定使用聚类数k=7时K均值算法的聚类结果用于下一步的分析。
2.3 七个DNA甲基化亚组的预后分析因为使用的是与预后相关的CpG位点区分的亚组,进一步本研究对7个亚组进行Kaplan-Meier生存分析,并使用对数秩检验(log-rank test)研究生存差异的统计学显著性(见图 3)。与其他癌症类型相比较而言,结肠腺癌属于恶性程度高、预后状况差的一类肿瘤,目前还没有应用于临床的可以区分肿瘤预后的分型方法。在本文的DNA甲基化亚组中,生存分析显示这7个DNA甲基化亚组主要分为两大组,之间的预后差异存在显著性。三年(1 095 d)存活率分析结果表明,cluster1、cluster3、cluster4、cluster6生存时间明显优于其他亚型。
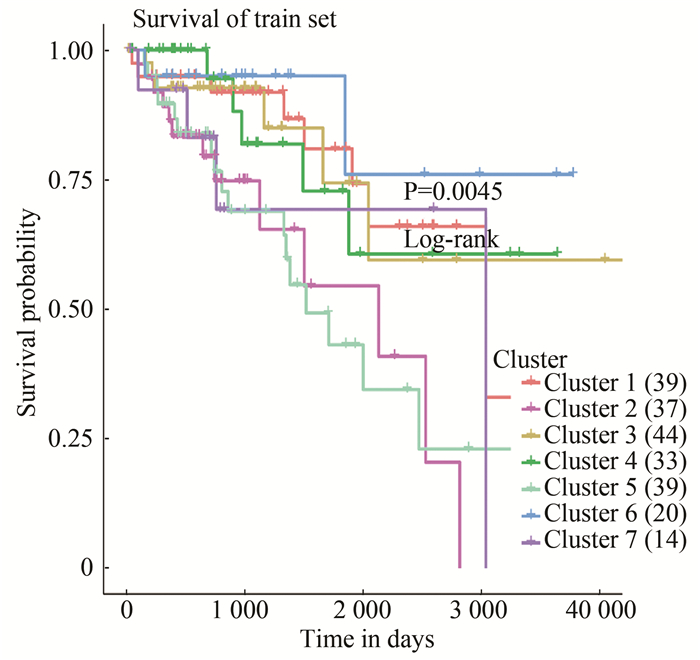
|
图 3 训练集中甲基化亚组的生存曲线 Figure 3 Survival of methylation subgroups in training set 注:横坐标是存活时间(d),纵坐标是存活概率. |
对各个亚型中样本的临床特征进行分析,临床特征年龄、肿瘤分期、T分期、N分期、M分期、组织学类型等较为经典的6个指标,同时,历年来有多篇NCS文章表明微卫星不稳定性与结肠腺癌的发生发展密切相关,故也将其选出作为结肠腺癌的重要临床特征。其中T分期是对原发肿瘤的评估,随着肿瘤体积的增加和邻近组织受累范围的增加,依次用T1~T4来表示;N分期是对区域淋巴结的评估,本研究中患者该项指标被纳入分析的分期有N0和N1,分别表示着没有区域淋巴结转移和1~3枚区域淋巴结转移;M分期是对肿瘤是否有远端转移的评估;肿瘤解剖学细分评估了肿瘤位置,不同位置的结肠腺癌肿瘤侵犯范围、术式选择等方面存在差异。
本研究在7个DNA甲基化亚型之间,使用Fisher’s exact test分析各临床特征在不同亚型之间的分布差异是否具有显著性(见表 2)。结果显示:年龄(p=0.017)、组织学类型((p=0.039)、微卫星不稳定性(p=0.003)、N分期((p=0.038)在不同亚型样本中的分布差异是显著的。这说明基于DNA甲基化谱分出的结肠腺癌亚型间的预后差异,一定程度上可由患者的年龄、发生部位、淋巴结受累情况、微卫星不稳定性去解释。而临床分期、肿瘤的大小和肿瘤转移情况,对于甲基化亚型的区分没有显著的指导意义。
| 表 2 甲基化亚型间Fisher's exact test结果 Table 2 Fisher's exact test results among methylation subtypes |
在确定了不同亚型间的临床特征有着差异性后,进一步分析临床特征在各亚型内的富集情况是否具有显著性。对每个临床特征在各亚型内的分布情况和在总体中的分布进行超几何检验,检验结果(见表 3)。
| 表 3 超几何检验各特征在亚型内富集的显著性 Table 3 Hypergeometric test for significance of feature enrichment in subtypes |
根据上一步对亚型间进行Fisher’s exact test的结果,将7个甲基化亚型和基于这两个临床特征下的样本分组进行比较(见图 4)。
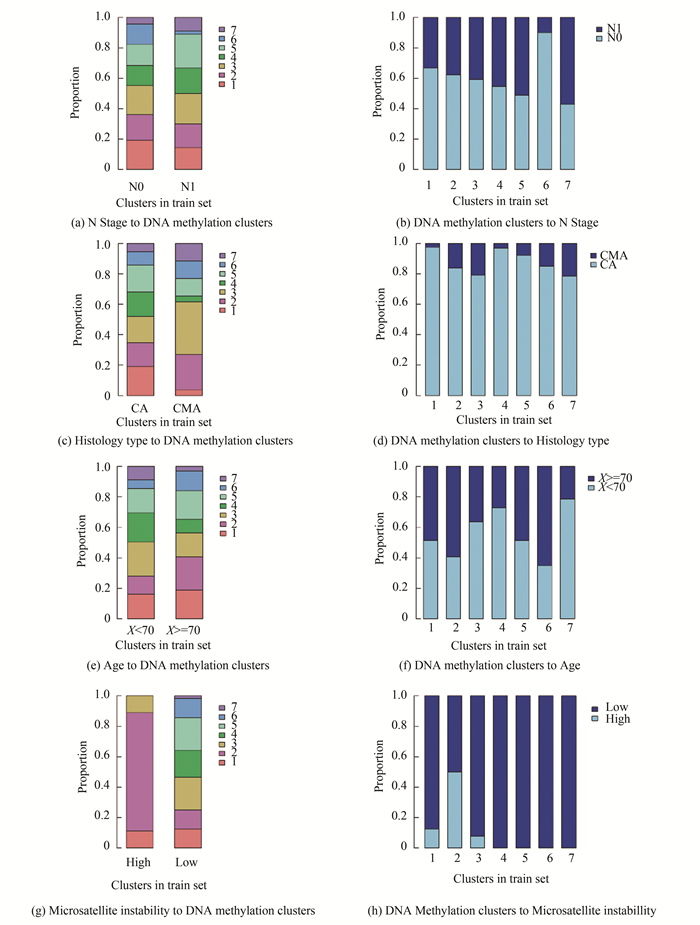
|
图 4 DNA甲基化亚型和N分期类型、组织学类型、年龄、微卫星不稳定性相互富集 Figure 4 Enrichment of DNA methylation clusters, N stage, histology type, age, and microsatellite instability 注:A. N分期类型对应于DNA甲基化亚型。B. DNA甲基化亚型对应N分期。C. 组织学类型对应于DNA甲基化亚型。D. DNA甲基化亚型对应组织学类型。E.年龄对应于DNA甲基化亚型。F. DNA甲基化亚型对应年龄。G. DNA甲基化亚型对应微卫星不稳定性。H. 微卫星不稳定性对应于DNA甲基化亚型。 |
提取训练集样本中通过QDMR得到的79个特征CpG位点的甲基化数据,部分位点对应的基因(见表 4)。生成特征位点甲基化矩阵。使用R包pheatmap为该矩阵绘制热图,并为它从0-1的甲基化值关联上蓝色到红色。每个类别中特性低甲基化位点在热图中显示出蓝色,特异高甲基化位点显示出红色(见图 5)。这79个特征位点可以作为结肠腺癌中不同DNA甲基化亚型的特异性DNA甲基化标记,代表着每个亚型独特的DNA甲基化模式(见图 5)。class1具有最大数量的特异性低甲基化位点,class2具有最大数量的特异性高甲基化CpG位点。
| 表 4 特异性位点基因对应表 Table 4 Characteristic sites and corresponding gene symbols |
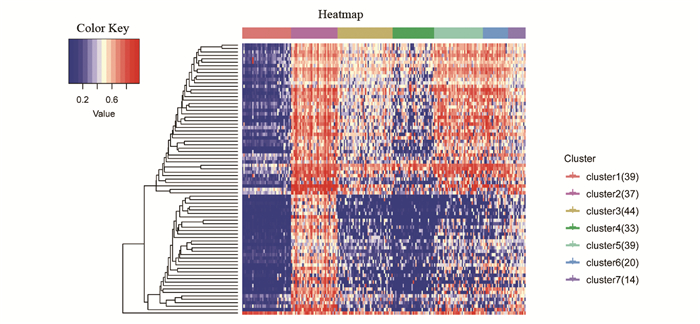
|
图 5 七个甲基化亚型中特征CpG位点甲基化水平 Figure 5 CpG site methylation level among seven subtypes |
为了验证本研究获得的甲基化亚型及更为准确高效地对样本进行分类,使用训练集样本构建基于决策树学习有监督的SMO分类模型(见表 5),所得到的模型有着87.61%的分类准确性,每一行代表模型预测的样本类别,每列代表样本真实的类别。C1-C7对应着class1-class7,表中对角线上的数字表示每个class中预测类别与实际类别相符的样本数量。
| 表 5 SMO分类器的混淆矩阵 Table 5 Confusion matrix of SMO classifier |
本研究的分类数量为7,而ROC曲线通常针对二分类的研究,故将7个亚型的真实值和模型的预测值转化为7个二分类对象以适应ROC曲线特征(见图 6),使用训练集样本训练出的该模型对6个亚型的分类都具有较高的AUC值(ROC曲线下面积), 而模型对class6的特异性和敏感性则相对较低。
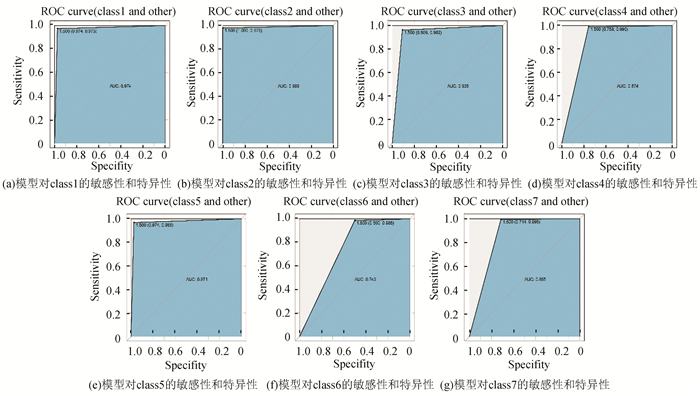
|
图 6 预测模型的敏感性和特异性 Figure 6 Sensitivity and specifity of prediction model |
将训练集中得到的模型应用到预留的检验集中,对检验集中未知类别标签的患者进行DNA甲基化亚型的分类。检验集中98个样本根据特征CpG位点的甲基化模式被分配到了7个不同的组内。对不同分组的3年(109 5 d)存活率进行分析,并使用对数秩检验(Log-rank test)分析发现生存差异具有较高的显著性(p=0.052)。生存曲线(见图 7)。
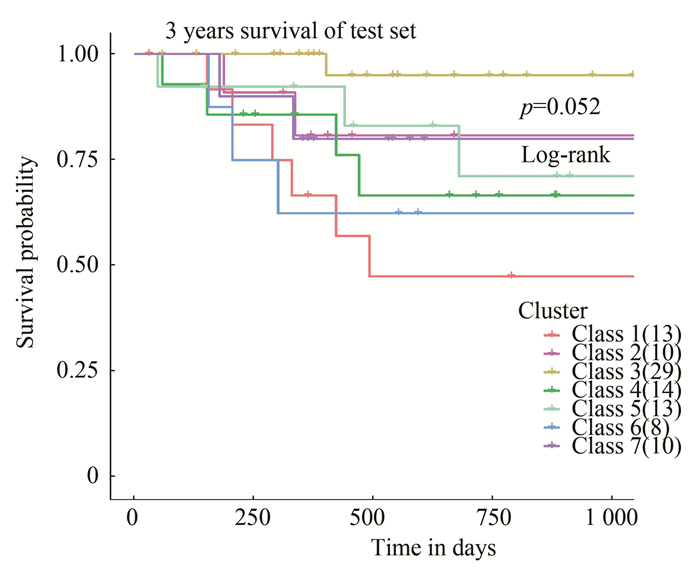
|
图 7 检验集生存分析结果 Figure 7 Survival analysis results of test set |
诱发癌症的因素复杂而多样,众所周知,目前的主流观点认为癌症由于基因突变引起[10],是一个累积渐进的过程,遗传改变的积累导致了恶性肿瘤的发生和发展。但是最新的研究表明,除了遗传变异以外,异常的DNA甲基化也在癌症的发生和发展中起到重要作用[11]。目前,异常的DNA甲基化在胃癌中是被广泛研究的一种失调的表观遗传机制。例如,有研究证明胃癌中一些肿瘤抑制基因或者肿瘤相关基因(如p16、RUNX3、MLH1和CDH1等)会被启动子的甲基化沉默[12]。这些研究都验证了DNA甲基化作为肿瘤标志物的重要价值。在确定了DNA甲基化与癌症的发生和进展相关联后,甲基化模式的动态性可能有助于癌症的早期诊断和评估:异常甲基化位点增加或减少的时间趋势可以帮助预测恶性肿瘤转化的速率和概率。因此,异常DNA甲基化被认为是可用来评估癌症前期进展的潜在早期诊断生物标志物[13],是临床实践中癌症诊断的理想靶标。此外,识别癌症特异或亚型特异的生物标志物也具有预测价值。
本研究中包含了大量的结肠腺癌样本的Infinium HumanMethylation450 BeadChip阵列数据集从TCGA数据库下载,这些数据可以用于本文的结肠腺癌异质性分析。庞大的样本量使得本研究能够更全面地探索结肠腺癌的分子亚型和分子异质性。
许多研究表明,表观遗传修饰(DNA甲基化)在结肠腺癌中的早期检测,以及改善分子分类,预后和辅助治疗中发挥关键作用,体现了基因组范围的分子水平分析在精准医学时代具有重要的生物学和临床意义[14]。利用基因启动子区域内筛选的预后相关的CpG位点进行无监督的聚类分析,通过一致性聚类获得七个预后不同的分子亚型,深入分析发现不同亚型之间有着分子或临床特征上的差异,这证实了结肠腺肿瘤的异质性及对结肠腺癌详细分类的必要性。
一致性聚类与其他无监督聚类方法(如层次聚类)相比,提供了聚类的类别数量的选取参考依据[15]。本研究首次提出建议根据DNA甲基化水平将结肠腺肿分为七个预后分子亚型。这种详细程度带来了较高的类内一致性,可更好地指导个性化医疗。在本研究中,SMO模型的甲基化亚型分类能力有着较高的稳定性。但是,第六亚型的AUC曲线太直,分析可能是由于样本量过少,过拟合导致。同时,在模型运用到检验集时,在训练集和检验集中,class6,class5,class3,class2的生存曲线差异较大,其可能是由于分类器对class6的灵敏性和特异性相对较低,导致预测类别有误所致。观察混淆矩阵可注意到实际类别为class6的不少样本被错误划分为class3,class5和class2,从而可以解释检验集中class3,class5和class2生存率提升的现象,这说明在一定程度上本研究建立的分类模型对于结肠腺癌DNA甲基化亚型的预测与预后情况相关联。值得注意的是,本研究建立的模型为各个甲基化亚型分配的样本数量占总样本数的相对数目多少在训练集和检验集中是相似的,例如:分配到class3的样本数目在训练集和检验集样本数均是最多。这也从一定程度上说明本研究建立的模型对甲基化亚型分类能力的稳定性。
在使用QDMR的分析中,发现了79个在亚型中特异性高/低甲基化的CpG位点,对应于76个基因,它们定义了结肠腺癌的特定DNA甲基化亚组。这些位点可以被视为诊断结肠腺癌的精准医疗的靶标和生物标志物。在这些特异性的CpG位点对应的基因中,需多先前已报道与结肠腺癌有关。其中,Sfrp5基因的甲基化被认为与结肠腺癌中致癌物诱导过程WNT基因的响应有关[16],Thbs4基因是大肠癌中一个与年龄相关的甲基化肿瘤抑制基因[17],GDF10基因与结肠癌发生密切相关[18]。
总之,使用TCGA数据库中的结肠腺肿瘤数据识别出七种不同的DNA甲基化预后分子亚型,这些亚型在分子水平或临床特征上也存在着显著差异。这可以对单一肿瘤内部的异质性更详细地解释。同时,该研究方法也可以推广到其他肿瘤的分型中。
4 结论使用TCGA中结肠腺癌的DNA甲基化数据,筛选到137个与预后相关的CpG位点。基于一致性聚类方法识别出7个与预后相关的亚型,这些亚型的预后差异显著且亚型特征可由年龄、N分期、微卫星不稳定性、解剖学发生部位反映。在不同的结肠腺癌亚型中筛选出79个特异性高/低甲基化位点,这些位点代表着每个亚型独特的甲基化模式。利用结肠腺癌亚型特异性DNA甲基化特征构建了基于序列最小最优化的分类模型,该模型对结肠腺癌亚型的分类准确性达到87.61%,提供了用于结肠腺癌分型的精确的DNA甲基化标记。
| [1] |
JATHAR S, KUMAR V, SRIVASTAVA J, et al. Technological Developments in lncRNA Biology[J]. Advances in Experimental Medicine and Biology, 2017, 1008: 283-323. DOI:10.1007/978-981-10-5203-3_10 (  0) 0) |
| [2] |
LIU H, ZHE Z, NAN W, et al. Integrative analysis of dysregulated lncRNA-associated ceRNA network reveals functional lncRNAs in gastric cancer[J]. Genes, 2018, 9(6): 303. DOI:10.3390/genes9060303 (  0) 0) |
| [3] |
YANG Yong, LI Xiaojia, LI Peng, et al. MicroRNA-145 regulates the proliferation, migration and invasion of human primary colon adenocarcinoma cells by targeting MAPK1[J]. International Journal of Molecular Medicine, 2018, 42(6): 3171-3180. DOI:10.3892/ijmm.2018.3904 (  0) 0) |
| [4] |
TSUKUDA K, TANINO M, SOGA H, et al. A novel activating mutation of the k-RAS gene in human primary colon adenocarcinoma[J]. Biochemical & Biophysical Research Communications, 2000, 278(3): 658. DOI:10.1006/bbrc.2000.3839 (  0) 0) |
| [5] |
LONGO D L, FEINBERG A P. The key role of epigenetics in human disease prevention and mitigation[J]. New England Journal of Medicine, 2018, 378(14): 1323-1334. DOI:10.1056/NEJMra1402513 (  0) 0) |
| [6] |
VANAJA K G, TIMP W, FEINBERG A P, et al. A loss of epigenetic control can promote cell death through reversing the balance of pathways in a signaling network[J]. Molecular Cell, 2018, 72(1): 60-70. DOI:10.1016/j.molcel.2018.08.025 (  0) 0) |
| [7] |
KLUTSTEIN M, NEJMAN D, GREENFIELD R, et al. DNA methylation in cancer and aging[J]. Cancer Research, 2016, 76(12): 3446-3450. DOI:10.1158/0008-5472.CAN-15-3278 (  0) 0) |
| [8] |
KOBOLDT D C, FULTON R S, MCLELLAN M D, et al. Comprehensive molecular portraits of human breast tumours[J]. Nature, 2012, 490(7418): 61-70. DOI:10.1038/nature11412 (  0) 0) |
| [9] |
STRANSKY N, EGLOFF A M, TWARD A D, et al. The mutational landscape of head and neck squamous cell carcinoma[J]. Science, 2011, 333(6046): 1157-1160. (  0) 0) |
| [10] |
GAN K A, SEBASTIAN C P, SEWELL J A, et al. Identification of single nucleotide non-coding driver mutations in cancer[J/OL]. Frontiers in Genetics, 2018. https://doi.org/10.3389/fgene.2018.00016, 2018-02-02.DOI: 10.1126/science.1208130.
(  0) 0) |
| [11] |
FEINBERG A P, KOLDOBSKIY M A, GÖNDÖR A, et al. Epigenetic modulators, modifiers and mediators in cancer aetiology and progression[J]. Nature Reviews Genetics, 2016, 17(5): 284-299. DOI:10.1038/nrg.2016.13 (  0) 0) |
| [12] |
LEE J H, PARK S J, ABRAHAM S C, et al. Frequent CpG island methylation in precursor lesions and early gastric adenocarcinomas[J]. Oncogene, 2004, 23(26): 4646-4654. DOI:10.1038/sj.onc.1207588 (  0) 0) |
| [13] |
MENDIZABAL I, YI S V. Whole-genome bisulfite sequencing maps from multiple human tissues reveal novel CpG islands associated with tissue-specific regulation[J]. Human Molecular Genetics, 2016, 25(1): 69-82. DOI:10.1093/hmg/ddv449 (  0) 0) |
| [14] |
PASCULLI B, BARBANO R, PARRELLA P. Epigenetics of breast cancer: Biology and clinical implication in the era of precision medicine[J]. Seminars in Cancer Biology, 2018, 51: 22-35. DOI:10.1016/j.semcancer.2018.01.007 (  0) 0) |
| [15] |
LANCICHINETTI A, FORTUNATO S. Consensus clustering in complex networks[J]. Scientific Reports, 2012, 336(2): 1-7. DOI:10.1038/srep00336 (  0) 0) |
| [16] |
ZHANG Y, LI Q, CHEN H. DNA methylation and histone modifications of Wnt genes by genistein during colon cancer development[J]. Carcinogenesis, 2013, 34(8): 1756-1763. DOI:10.1093/carcin/bgt129 (  0) 0) |
| [17] |
GRECO S A, CHIA J, INGLIS K J, et al. Thrombospondin-4 is a putative tumour-suppressor gene in colorectal cancer that exhibits age-related methylation[J]. BMC Cancer, 2010, 494: 1-10. DOI:10.1186/1471-2407-10-494 (  0) 0) |
| [18] |
SLATTERY M L, LUNDGREEN A, HERRICK J S, et al. Genetic variation in bone morphogenetic protein and colon and rectal cancer[J]. International Journal of Cancer, 2012, 130(3): 653-664. DOI:10.1002/ijc.26047 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


