星形细胞瘤(Astrocytoma)起源于星形胶质细胞,是颅内最常见的肿瘤,约占神经上皮组织肿瘤的70%以上。WHO分级为Ⅰ~Ⅱ级(低级别胶质瘤,LGG)与Ⅲ~Ⅳ级(胶质母细胞瘤,GBM)。该类肿瘤恶性程度高,预后较差,具有侵袭性生长的特点,手术难以全切且易复发,治疗非常困难,有效抑制星形细胞瘤的发生发展成为肿瘤生物治疗的难点之一[1-2]。微小RNA(microRNA,miRNA)是一类广泛存在于动植物体内的短链非编码RNA,仅有21~23个核苷酸序列,通过抑制翻译或促进细胞质中mRNA降解来控制转录后的基因表达[3]。研究表明,miRNA在肿瘤的生长发育、信号转导、细胞分化、增殖和能量代谢等进程中都发挥重要作用[4]。但有关miRNA与星形细胞瘤治疗的研究报道较少,本研究利用GEO数据库中有关星形细胞瘤的miRNA与mRNA两个表达数据集,通过生物信息学手段筛选与星形细胞瘤患者生存预后相关的miRNA-mRNA调控关系对,为该类肿瘤的基础研究与临床治疗提供重要靶点和参考。
1 材料与方法 1.1 检索策略与芯片数据以“astrocytoma”或“glioma”与“miRNA”或“mRNA”为检索词在美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)的基因表达综合数据库(Gene Expression Omnibus,GEO)检索芯片数据,共检索到5个符合要求的miRNA数据集(分别是GSE103228、GSE90603、GSE25631、GSE65626和GSE138764)与15个mRNA数据集(分别是GSE1993、GSE4271、GSE4412、GSE7696、GSE8692、GSE16011、GSE19728、GSE21354、GSE24072、GSE45921、GSE103227、GSE90598、GSE25630、GSE65626和GSE138999)。综合考虑样本数量、信息完整度与可靠性,下载miRNA表达数据集GSE138764,包含有9例正常脑组织样本和33例星形细胞瘤样本的miRNA测序信息,检测平台为GPL18402 Agilent-046064 Unrestricted_Human_miRNA_V19.0_Microarray (miRNA ID version)。下载mRNA表达数据集GSE19728,包含有4例正常脑组织样本和17例星形细胞瘤样本的mRNA测序信息,检测平台为GPL570 [HG-U133_Plus_2] Affymetrix Human Genome U133 Plus 2.0 Array。
1.2 数据处理与差异分析利用GPL注释文件将miRNA和mRNA的ID转换为基因名字,利用perl(perl5.30.2)语言将测序数据分为正常组和肿瘤组。下载R(R 3.6.3)语言中的limma包,其中的avereps函数进行数据矫正,normalizeBetweenArrays函数用于标准化处理,得到可进行差异分析的表达矩阵。设置FDR(BH)法矫正后的阈值P.adj<0.05,log2 FC|>2,进行差异分析,下载pheatmap包进行层次聚类分析并绘制差异基因热图和火山图。
1.3 靶基因预测与调控网络构建利用miRWalk3.0网页工具(http://mirwalk.umm.uni-heidelberg.de/)整合2个miRNA靶基因预测数据库TargetScan(http://www.targetscan,org/vert_72/)和miRDB(http://www.mirdb.org/cgi-bin/search.cgi)对GSE138764数据集中差异表达的miRNA进行靶基因预测。将预测到的靶基因与GSE19728差异表达的mRNA取交集,根据miRNA靶基因预测结果,明确取交集后的mRNA与miRNA之间的调控关系,整理出miRNA-mRNA关系对。将数据导入到Cytoscape 3.7.2软件中,构建miRNA-mRNA调控网络图。
1.4 mRNA数据验证与生存分析利用GEPIA2.0网页工具(http://gepia2.cancer-pku.cn/)输出网络中mRNA在LGG与GBM两类肿瘤及其正常组织的表达量统计图(设置P.adj<0.01,log2 FC|>1,Match TCGA normal and GTEx data),进而验证其在正常组织和肿瘤组织中的差异表达情况。筛选与网络中表达情况一致的mRNA,分别在LGG与GBM中进行生存分析,判断mRNA高低表达与生存率之间的关系(设置Log rank P < 0.05),分为“高表达,生存率高”、“低表达,生存率高”与“无明显差异”三种情况。
1.5 miRNA-mRNA调控关系对筛选筛选LGG与GBM中“高表达,生存率高”与“低表达,生存率高”的mRNA,利用miRNA-mRNA调控网络图确定相应的miRNA,使用OncoLnc工具(http://www.oncolnc.org/)判断miRNA高低表达与LGG或GBM患者生存率之间的关系(设置Log rank P < 0.05,截断值:25%)。根据miRNA对mRNA的功能发挥抑制作用这一生物学基础,我们制定关键miRNA-mRNA调控关系对筛选标准:即“高表达,生存率高”的mRNA对应的miRNA在同类型肿瘤中,应符合“低表达,生存率高”;“低表达,生存率高”的mRNA对应的miRNA在同类型肿瘤中,应符合“高表达,生存率高”。这些miRNA-mRNA调控关系对具有重要生物学意义,可为星形细胞瘤的研究与治疗提供重要靶点。
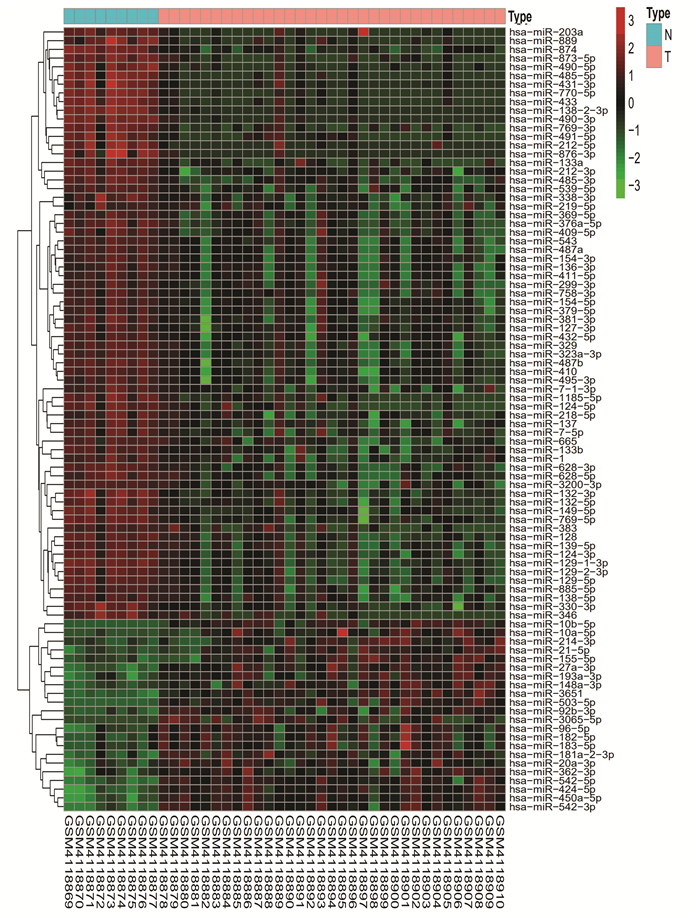
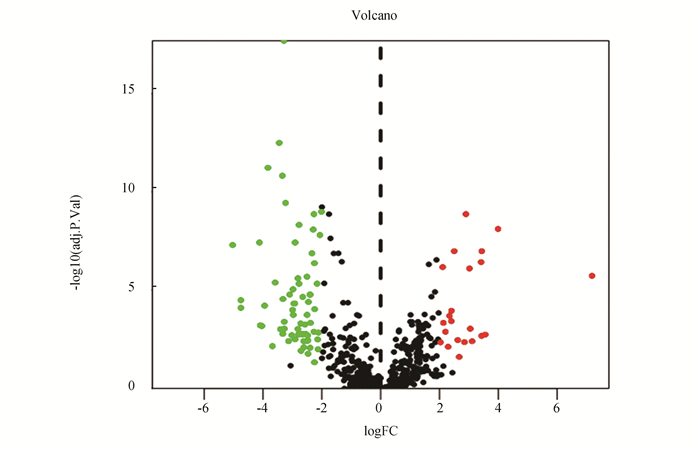
2 结果分析 2.1 差异表达miRNA和mRNAGSE138764数据集中星形细胞瘤与正常组织样本相比,差异表达的miRNA有90个(表达上调22个,下调68个),差异基因热图(见图 1),火山图(见图 2)(图中红色代表表达上调,绿色代表下调,黑色代表无明显差异)。GSE19728数据集中星形细胞瘤与正常组织样本相比,差异表达的mRNA有644个(表达下调476个,上调168个)。
|
图 1 差异表达的miRNA基因热图 Figure 1 Heat map of differentially expressed miRNA |
|
图 2 差异表达的miRNA火山图 Figure 2 Volcano plot of differentially expressed miRNA |
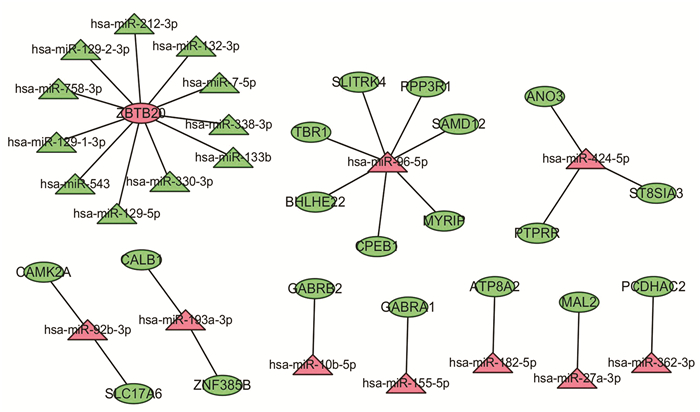
利用miRWalk3.0网页工具对90个差异表达的miRNA进行靶基因预测,TargetScan与miRDB数据库共预测到1 668个靶基因,与GSE19728数据集中644个差异表达的mRNA取交集得到20个mRNA,根据miRNA靶基因预测结果,明确20个mRNA与miRNA之间的调控关系,整理出miRNA-mRNA关系对30个(见表 1),绘制调控网络图(见图 3)(网络中红色代表表达上调,绿色代表下调;三角形代表miRNA,椭圆形代表mRNA)。
| 表 1 miRNA-mRNA调控关系 Table 1 miRNA-mRNA regulatory pairs |
|
图 3 miRNA-mRNA调控网络图 Figure 3 miRNA-mRNA regulatory network diagram |
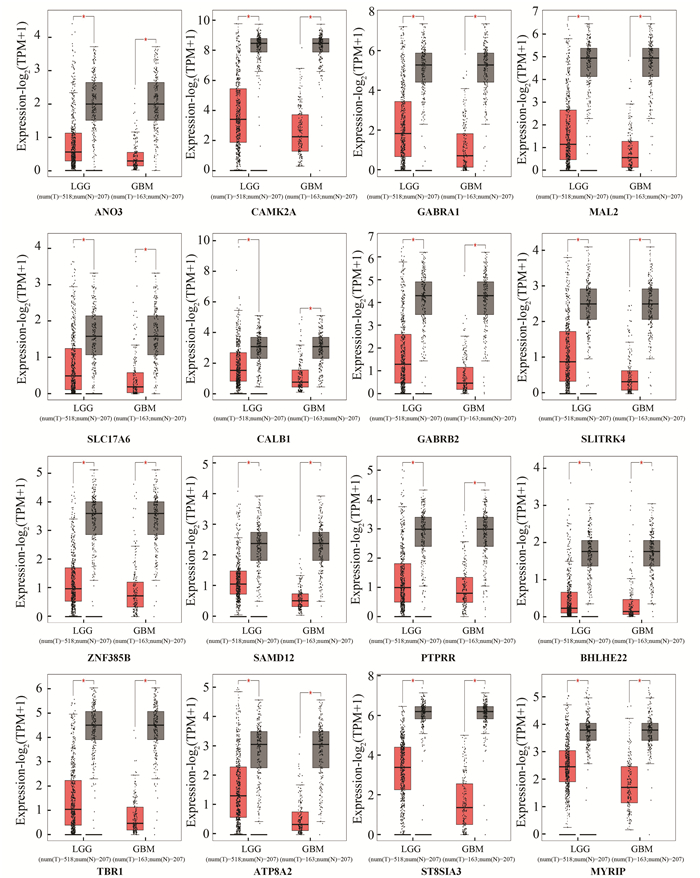
为了验证网络中20个mRNA在正常和肿瘤组织中的差异表达情况,利用GEPIA2.0网页工具分析其在癌症基因组图谱数据库(The Cancer Genome Atlas, TCGA, https://portal.gdc.cancer.gov/)中LGG与GBM两类肿瘤的表达情况,符合统计学差异的mRNA有16个,分别是ANO3、CAMK2A、GABRA1、MAL2、SLC17A6、CALB1、GABRB2、SLITRK4、ZNF385B、SAMD12、PTPRR、BHLHE22、TBR1、ATP8A2、ST8SIA3和MYRIP,它们在肿瘤组织中均表达下调,且与调控网络中的表达情况一致(见图 4)图中红色代表肿瘤组织,黑色代表正常组织)。
|
图 4 mRNA差异表达情况 Figure 4 mRNA differential expressions |
分别在LGG与GBM中对16个mRNA进行生存分析,其中9个mRNA在LGG中符合“高表达,生存率高”, 分别是ANO3、GABRA1、MAL2、SLC17A6、CALB1、GABRB2、PTPRR、ATP8A2和ST8SIA3(见图 5),7个mRNA在LGG中符合“无明显差异”;而16个mRNA在GBM中均“无明显差异”。
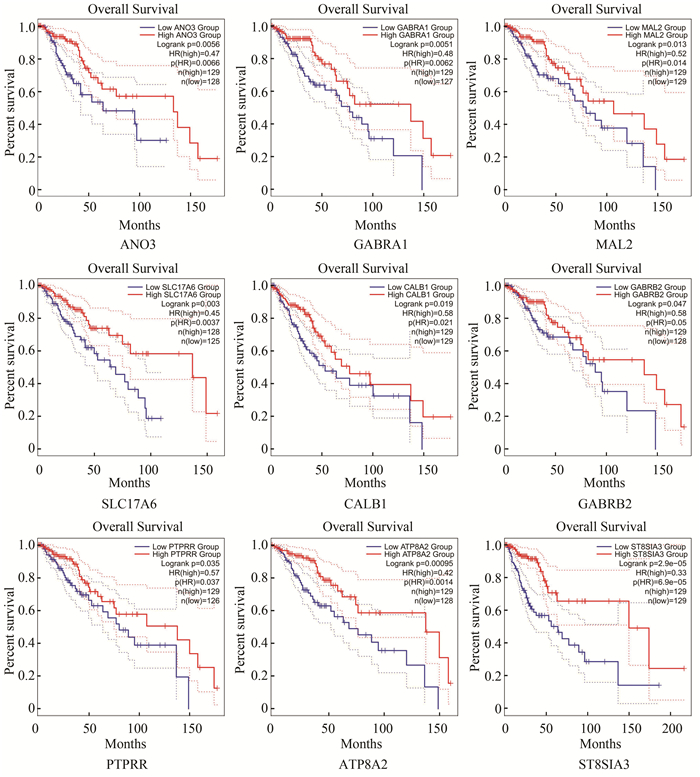
|
图 5 mRNA生存分析曲线 Figure 5 mRNA survival analysis curves |
结果2.3中得到的9个有统计学意义的mRNA均在LGG中符合“高表达,生存率高”这一情况,根据结果2.2中的调控网络确定其对应的miRNA,利用OncoLnc工具判断对应miRNA高低表达与LGG患者生存率之间的关系。依据miRNA-mRNA调控关系对筛选标准,应筛选在LGG中符合“低表达,生存率高”的miRNA,共得到5个miRNA,分别是hsa-miR-424-5p、hsa-miR-155-5p、hsa-miR-27a-3p、hsa-miR-193a-3p和hsa-miR-10b-5p,生存曲线(见图 6)。而其余miRNA的高低表达均与患者的生存率之间无明显差异,据此共筛选到miRNA-mRNA调控关系对7个,为后续研究提供参考(见表 2)。
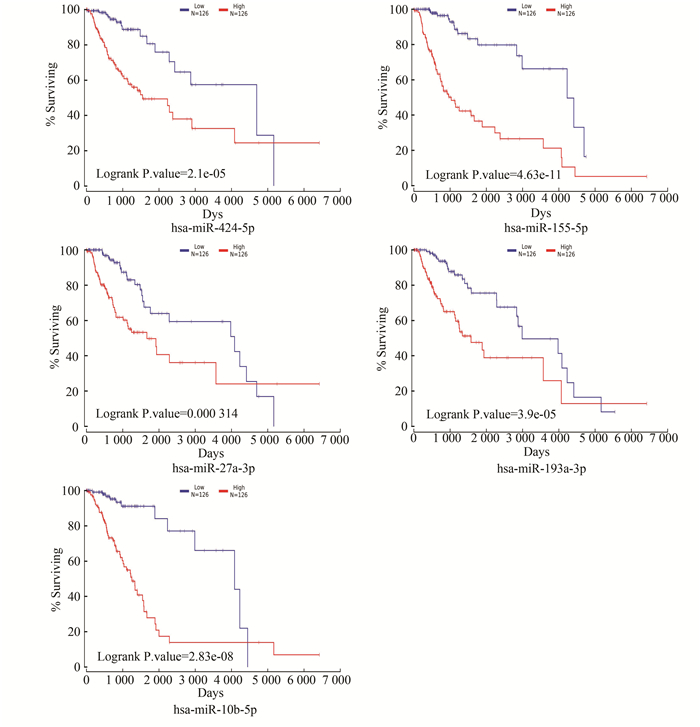
|
图 6 miRNA生存分析曲线 Figure 6 miRNA survival analysis curves |
| 表 2 生存预后相关miRNA-mRNA调控关系对 Table 2 miRNA-mRNA regulatory pairs associated with survival and prognosis |
成熟的miRNA通过碱基互补配对方式识别靶基因mRNA,发挥降解作用或直接阻断翻译过程[5]。目前已经有2 000多个miRNA被证实可在人体表达并参与调控30%的基因[6]。更重要的是,大量研究表明miRNA几乎参与肿瘤发生进展的全过程,包括持续增殖、无限复制、回避生长抑制、抵抗凋亡、血管生成以及肿瘤微环境构建等,在不同类型肿瘤中通过作用于特定的靶基因mRNA,发挥抑癌或促癌作用[7]。而随着细胞外泌体技术的发展,人为调控细胞内miRNA表达成为一种可能,这为精准治疗疾病提供了广阔的应用前景。细胞外泌体是几乎所有类型的细胞以胞吐方式分泌到细胞外的胞外囊泡,由40~100 nm的脂质双分子层构成,内含核酸或蛋白质分子,发挥细胞间信息交流作用[8]。将肿瘤细胞分泌的外泌体改造成高效的运载体,携带目标miRNA的类似物或抑制剂,回输后利用人体精准的通讯系统,可实现miRNA的精确调控,有望成为肿瘤生物治疗的重要工具[9]。本研究所筛选到的7个mRNA在LGG中均低表达,相应miRNA在LGG中高表达;生存分析则表明上述mRNA高表达,相应miRNA低表达,患者具有更高的生存率。因此利用外泌体技术特异性抑制这些miRNA表达,进而提高mRNA的翻译水平,有望成为该类肿瘤生物治疗的有效手段,但具体机制及可行性有待进一步研究。
本研究筛选到的7个mRNA有:ANO3、GABRA1、GABRB2、MAL2、CALB1、PTPRR和ST8SIA3。ANO3(Anoctamin 3)是钙离子激活氯离子通道蛋白家族成员之一,对该基因的功能知之甚少,但该基因的突变与一些常染色体显性颅颈肌张力障碍有关[10]。而该基因突变个体的细胞表现出内质网依赖性钙信号的异常[11],提示该基因可通过调节钙信号相关通路影响LGG的发生发展。GABRA1(Gamma-aminobutyric acid type A receptor subunit alpha 1)和GABRB2(Gamma-aminobutyric acid type A receptor subunit beta2)分别是γ-氨基丁酸A型受体α1亚基和γ-氨基丁酸A型受体β2亚基,它们均参与γ-氨基丁酸受体的形成。γ-氨基丁酸是哺乳动物大脑中主要的抑制性神经递质,作用于γ-氨基丁酸受体,该受体是配体门控氯离子通道,与癫痫的发生发展密切相关[12]。研究表明,γ-氨基丁酸相关受体的低表达与胶质瘤患者继发性癫痫呈正相关关系[13],因此提高该类受体表达可能减轻患者癫痫的发作,改善其生活质量与生存情况。MAL2(T cell differentiation protein 2)即T细胞分化蛋白2,该基因编码属于MAL蛋白脂类家族的多跨膜蛋白。该蛋白是脂质筏的组成部分,在极化细胞中,它主要定位于顶端膜下的内体结构,用于将膜结合蛋白和外源性物质从基底外侧转运至根尖表面[14]。研究表明,MAL2表达升高,可通过调节乳腺癌细胞系上皮-间充质转化促进其增殖、迁移和侵袭[15];而在结直肠癌中MAL2的高表达与患者的不良生存预后密切相关[16];miR-129通过靶向MAL2,下调其表达水平,进而调控甲状腺乳头状癌的生长和侵袭[17]。上述研究表明,MAL2参与多种肿瘤的进程,且该基因的低表达,会导致患者更高的生存率,这与我们在LGG中得到的结论相左,因此针对MAL2与LGG发生进展之间的关系,仍有待进一步的研究。CALB1(Calbindin 1)是钙结合蛋白超家族的成员,它被认为是缓冲钙离子进入刺激谷氨酸受体,在亨廷顿舞蹈病患者中这种蛋白常缺失[18]。研究表明,过表达CALB1通过调节p21和p27的表达水平,促进卵巢癌细胞增殖和集落形成,抑制细胞衰老[19];在非小细胞肺癌中miR-454-3p通过下调CALB1水平可抑制细胞增殖[20]。这提示我们,CALB1在多种肿瘤中可能发挥癌基因的作用,但由于谷氨酸受体主要存在于脑内,该受体与胶质瘤之间存在密切关系,研究表明,加入该受体的拮抗剂后,可抑制胶质瘤细胞的侵袭生长[21],而CALB1同样可以延缓谷氨酸受体的激活,因此我们猜想该基因在LGG中可能发挥抑癌作用,但有待进一步考证。PTPRR(Protein tyrosine phosphatase receptor type R)即蛋白质酪氨酸磷酸酶R型受体,是蛋白质酪氨酸磷酸酶(PTP)家族的一员。PTPs被认为是调控多种细胞过程的信号分子,包括细胞生长、分化、有丝分裂周期和致癌转化,在结直肠癌发生的早期该基因即被沉默[22],过表达PTPRR能否抑制癌细胞的转化,值得进一步研究。ST8SIA3(ST8 alpha-N-acetyl-neuraminide alpha-2, 8-sialyltransferase 3)是唾液酸转移酶家族成员,该基因在脑组织中高表达,通过唾液酸化(蛋白质糖基化的一种)可改变细胞膜糖蛋白和糖脂的唾液酸化糖基化的结构和功能,进而影响细胞的生物学行为[23]。随着蛋白质组学的发展,对于蛋白质翻译后修饰的调控逐渐成为关注的热点,唾液酸转移酶家族成员的高表达,广泛参与结直肠癌与非小细胞肺癌等的生长与侵袭过程[24-25]。但由于肿瘤细胞之间的异质性较强,同一修饰过程在不同肿瘤之间往往发挥不同作用,ST8SIA3在脑内的高表达,提示我们该基因以及唾液酸糖基化修饰与LGG之间的关系具有较高研究价值。
综上所述,本研究共筛选到7个与LGG患者生存预后显著相关的miRNA-mRNA调控关系对,为低级别胶质瘤的研究与治疗提供靶点和参考方向。不足之处在于,未对7个miRNA-mRNA的调控关系进行验证,它们之间是否存在靶关系需利用双荧光素酶报告实验等进行确定;GSE19728数据集中包含的正常组织样品较少,但所得结果在TCGA数据库中进一步得到了验证,以保证数据的准确性;所使用的miRNA与mRNA数据集中肿瘤样本并非来自同一组病人,导致说服力降低,如利用本研究思路对其他癌种进行研究,应注意样本的选择;所筛选的miRNA-mRNA调控关系对均与LGG患者生存预后相关,未在GBM中找到合适的关系对,提示后续研究应选择GBM来源的肿瘤样本进行分析。
4 结论1) 利用GEO数据库中GSE138764筛选到星形细胞瘤组织相较正常组织差异表达的miRNA 90个(表达上调22个,下调68个);利用GSE19728筛选到差异表达的mRNA 644个(表达下调476个,上调168个);通过miRNA靶基因预测,成功构建星形细胞瘤中miRNA-mRNA调控网络。
2) 对网络中的mRNA进行生存分析,筛选到9个mRNA在LGG中符合“高表达,生存率高”;对相应的miRNA进行生存分析,筛选到5个miRNA在LGG中符合“低表达,生存率高”;据此共筛选到miRNA-mRNA调控关系对7个,为后续研究提供参考。
致谢 感谢中南大学临床药理研究所刘昭前老师课题组提供的星形细胞瘤miRNA芯片数据集GSE138764对本研究的帮助;感谢中国人民解放军第150中心医院姚志强老师课题组提供的星形细胞瘤mRNA芯片数据集GSE19728对本研究的帮助;感谢山西省2019年大学生创新创业训练计划项目(No.2019189)对本课题的资助。
| [1] |
COLQUHOUN A. Cell biology-metabolic crosstalk in glioma[J]. International Journal of Biochemistry & Cell Biology, 2017, 89: 171-181. DOI:10.1016/j.biocel.2017.05.022 (  0) 0) |
| [2] |
STOYANOV G S, DZHENKOV D, GHENEV P, et al. Cell biology of glioblastoma multiforme: from basic science to diagnosis and treatment[J]. Medical Oncology, 2018, 35(3): 27. DOI:10.1007/s12032-018-1083-x (  0) 0) |
| [3] |
LU T X, ROTHENBERG M E. MicroRNA[J]. Journal of Allergy & Clinical Immunology, 2018, 141(4): 1202-1207. DOI:10.1016/j.jaci.2017.08.034 (  0) 0) |
| [4] |
TUTAR Y. miRNA and cancer; computational and experimental approaches[J]. Current Pharmaceutical Biotechnology, 2014, 15(5): 429. DOI:10.2174/138920101505140828161335 (  0) 0) |
| [5] |
MOHR A M, MOTT J L. Overview of microRNA biology[J]. Seminars in Liver Disease, 2015, 35(1): 3-11. DOI:10.1055/s-0034-1397344 (  0) 0) |
| [6] |
HAMMOND S M. An overview of microRNAs[J]. Advanced Drug Delivery Reviews, 2015, 87: 3-14. DOI:10.1016/j.addr.2015.05.001 (  0) 0) |
| [7] |
IORIO M V, CROCE C M. MicroRNA dysregulation in cancer: Diagnostics, monitoring and therapeutics. A comprehensive review[J]. EMBO Moloecular Medicine, 2012, 4(3): 143-159. DOI:10.1002/emmm.201100209 (  0) 0) |
| [8] |
BASU B, GHOSH M K. Extracellular vesicles in glioma: from diagnosis to therapy[J]. Bioessays, 2019, 41(7): e1800245. DOI:10.1002/bies.201800245 (  0) 0) |
| [9] |
OHNO S, TAKANASHI M, SUDO K, et al. Systemically injected exosomes targeted to EGFR deliver antitumor microRNA to breast cancer cells[J]. Moloecular Therapy, 2013, 21(1): 185-191. DOI:10.1038/mt.2012.180 (  0) 0) |
| [10] |
OLSCHEWSKI L, JESUS S, KIM H J. Role of ANO3 mutations in dystonia: A large-scale mutational screening study[J]. Parkinsonism Relat Disord, 2019, 62: 196-200. DOI:10.1016/j.parkreldis.2018.12.030 (  0) 0) |
| [11] |
PICOLLO A, MALVEZZI M, ACCARDI A. TMEM16 proteins: unknown structure and confusing functions[J]. Journal of Molecular Biology, 2015, 427(1): 94-105. DOI:10.1016/j.jmb.2014.09.028 (  0) 0) |
| [12] |
BHAT M A, GURU S A, MIR R. Association of GABAA receptor gene with epilepsy syndromes[J]. Journal of Molecular Neuroence, 2018, 65(2): 141-153. DOI:10.1007/s12031-018-1081-7 (  0) 0) |
| [13] |
云德波. 胶质瘤瘤周兴奋性氨基酸、抑制性氨基酸与胶质瘤继发性癫痫的相关性研究[D]. 桂林: 桂林医学院, 2014. YUN Debo. Study on relationship between excitatory amino acids, inhibitory amino acid in human glioma's surrounding and the presence of tumor-associated seizures[D]. Guilin: Medical College of Guilin, 2014. (  0) 0) |
| [14] |
MARZAUELA M, ALONSO M A. Expression of MAL and MAL2, two elements of the protein machinery for raft-mediated transport, in normal and neoplastic human tissue[J]. Histol Histopathol, 2004, 19(3): 925-933. DOI:10.14670/HH-19.925 (  0) 0) |
| [15] |
BHANDARI A, SHEN Y, SINDAN N, et al. MAL2 promotes proliferation, migration, and invasion through regulating epithelial-mesenchymal transition in breast cancer cell lines[J]. Biochemical & Biophysical Research Communications, 2018, 504(2): 434-439. DOI:10.1016/j.bbrc.2018.08.187 (  0) 0) |
| [16] |
LI Jingwen, LI Yongmin, LIU He, et al. The four-transmembrane protein MAL2 and tumor protein D52 (TPD52) are highly expressed in colorectal cancer and correlated with poor prognosis[J]. PLoS One, 2017, 12(5): e0178515. DOI:10.1371/journal.pone.0178515 (  0) 0) |
| [17] |
GAO Xuejun, CHEN Zhenyu, LI Aiqin, et al. MiR-129 regulates growth and invasion by targeting MAL2 in papillary thyroid carcinoma[J]. Biomedicine Pharmacotherapy, 2018, 105: 1072-1078. DOI:10.1016/j.biopha.2018.06.050 (  0) 0) |
| [18] |
CHRISTAKOS S, BARLETTA F, HUENING M, et al. Vitamin D target proteins: function and regulation[J]. Journal of Cellular Biochemistry, 2003, 88(2): 238-244. DOI:10.1002/jcb.10349 (  0) 0) |
| [19] |
CAO Longqiao, WANG Yanan, LIANG Ming, et al. CALB1 enhances the interaction between p53 and MDM2, and inhibits the senescence of ovarian cancer cells[J]. Molecular Medicine Reports, 2019, 19(6): 5097-5104. DOI:10.3892/mmr.2019.10212 (  0) 0) |
| [20] |
JIN Caijin, LIN Tao, SHAN Liqun. Downregulation of calbindin 1 by miR-454-3p suppresses cell proliferation in nonsmall cell lung cancer in vitro[J]. Cancer Biotherapy & Radiopharmaceuticals, 2019, 34(2): 119-127. DOI:10.3892/mmr.2019.10212 (  0) 0) |
| [21] |
王涛. 谷氨酸受体对胶质瘤U251细胞分泌侵袭因子MMP-2的调控作用[D]. 广州: 广州医科大学, 2012. WANG Tao. The regulation of glutamate receptors on invasion factor MMP-2 secreted by glioma U251 cells[D]. Guangzhou: Guangzhou Medical University, 2012. (  0) 0) |
| [22] |
MENIGATTI M, CATTANEO E, SABATES-BELLVER J, et al. The protein tyrosine phosphatase receptor type R gene is an early and frequent target of silencing in human colorectal tumorigenesis[J]. Moloecular Cancer, 2009, 8: 124. DOI:10.1186/1476-4598-8-124 (  0) 0) |
| [23] |
ANGATA K, SUZUKI M, MCAULIFFE J, et al. Differential biosynthesis of polysialic acid on neural cell adhesion molecule (NCAM) and oligosaccharide acceptors by three distinct alpha 2, 8-sialyltransferases, ST8Sia IV (PST), ST8Sia Ⅱ (STX), and ST8Sia Ⅲ[J]. Journal of Biological Chemistry, 2000, 275(24): 18594-18601. DOI:10.1074/jbc.M910204199 (  0) 0) |
| [24] |
BROWN J R, FUSTER M M, WHISENANT T, et al. Expression patterns of alpha 2, 3-sialyltransferases and alpha 1, 3-fucosyltransferases determine the mode of sialyl Lewis X inhibition by disaccharide decoys[J]. Journal of Biological Chemistry, 2003, 278(26): 23352-23359. DOI:10.1074/jbc.M303093200 (  0) 0) |
| [25] |
TANAKA F, OTAKE Y, NAKAGAWA T, et al. Expression of polysialic acid and STX, a human polysialyltransferase, is correlated with tumor progression in non-small cell lung cancer[J]. Cancer Research, 2000, 60(11): 3072-3080. DOI:10.1097/00002820-200006000-00012 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


