2. 云南省第一人民医院 消化内科,昆明 650032;
3. 云南省第一人民医院 肿瘤内科,昆明 650032
2. Department of Digestive Medicine, The First People's Hospital of Yunnan Province, Kunming 650032, China;
3. Department of Oncology, The First People's Hospital of Yunnan Province, Kunming 650032, China
结直肠癌(Colorectal cancer,CRC)是最常见的消化道恶性肿瘤,也是癌症患者死亡的主要原因之一,严重危害着人类健康与生存。据报道,2018年全球结直肠癌新发病例180万例,但死亡病例高达88.1万例,仅次于肺癌[1-2]。而且,约15%~25%的结直肠癌患者在确诊时即合并多发肝转移,其中绝大多数患者的肝转移灶无法获得根治性切除。目前,手术和全身化疗仍是结直肠癌的主要治疗方法[3]。近年来,以抗表皮生长因子受体(Epidermal growth factor receptor EGFR,如西妥昔单抗、帕尼单抗)和抗血管内皮生长因子(Vascular endothelial growth factor VEGF,如贝伐单抗,其他小分子抑制剂如瑞戈非尼等)为代表的靶向药物以及程序性死亡受体1/程序性死亡-配体1(programmed cell death-1/ programmed cell death-Ligand 1, PD-1/PD-L1)免疫治疗的应用,为晚期结直肠癌患者带来了希望[4-5]。但上述精准治疗都存在优势人群的筛选、显著获益的人数比例低,而且都存在一定的耐药性。因此,寻找结直肠癌发生与发展过程的关键基因和通路,有助于我们认识结直肠癌潜在的发病机制,为其诊治和靶点治疗提供新的参考。
肿瘤精准治疗的前提在于获得肿瘤分子图谱和分类,从中筛选并获得临床治疗靶点;而后者得益于二代测序技术的进步和分子靶向药物的问世。基于快速发展的微阵列和高通量测序技术,生物信息学的发展也日益蓬勃。公共数据库如GEO和TCGA数据库存储大量、复杂的生物信息数据,通过对这些数据进行差异分析、聚类比对、生物分子网络和通路分析以及可视化作图等能够更好地理解肿瘤的发病机制。
利用生物信息学的初步挖掘,筛选出潜在的结直肠癌发生发展相关功能基因,希望以此获得更多与结直肠癌发生、进展相关的分子机制,从而为挖掘结直肠潜在的预后指标和肿瘤靶点提供一定的帮助。
1 材料和方法 1.1 材料在NCBI(National center for biotechnology)的公共GEO数据库(http://www.ncbi.nlm.nih.gov/geo/, Gene expression omnibus)中检索含有人源结直肠癌样本信息的数据芯片,下载数据集GSE106582[6]作为分析对象。芯片信息:Illumina HumanHT-12 V4.0 expression beadchip,平台是GPL10558,该芯片中包含77例结直肠癌组织和117例癌旁组织(距离癌组织5cm处的粘膜组织)的表达数据。
1.2 筛选差异表达基因通过R包对样本进行PCA主成分分析,将样本分为癌组织和癌旁组织,利用GEO数据库的分析工具GEO2R对GSE106582芯片进行数据下载及差异表达基因筛选。筛选的标准为调准后p < 0.05,且|log2FC|>1.5,并将探针名称转化为标准基因名称。上调差异基因和下调差异基因结果以癌组织VS癌旁组织体现。
1.3 差异表达基因的GO功能富集和KEGG通路富集分析使用在线分析工具DAVID 6.8(http://david.ncifcrf.gov/)对差异表达基因进行GO功能富集分析和KEGG通路富集分析。P < 0.05认为具有统计学意义。
1.4 差异表达基因的相互作用分析将差异表达基因导入在线数据库STRING 10.5(https://string-db.org/),构建结直肠癌差异基因蛋白质相互作用网络,并运用Cytoscape3.1.1软件进行可视化分析。使用软件中的插入式分子复合物检测(MCODE,使用默认参数)筛选出PPI网络中的枢纽模块和关键基因。
1.5 差异表达基因的预后价值分析及ROC诊断利用在线工具GEPIA2(http://gepia2.cancer-pku.cn/)分析关键基因表达水平与结直肠癌患者总生存期(Overall survival,OS)的相关性。根据结直肠癌患者差异表达基因的表达情况及中位值分为高表达组和低表达组,绘制生存曲线,若log-rank P<0.05,则该基因被视为具有预后价值的基因。利用R语言包(PROC)对具有预后价值的基因进行受试者工作特征曲线(Receiver operating characteristic curve,ROC)分析,AUC>0.9则认为该基因对预后具有较高的灵敏度和特异度。下载数据集GSE21510对实验结果进行验证。
2 结果分析 2.1 样本分组对数据集GSE106582的样本进行分组,其PCA结果显示癌组织与癌旁组织的两类样本是相互独立的,可将样本分为癌组织和癌旁组织进行后续分析(见图 1)。
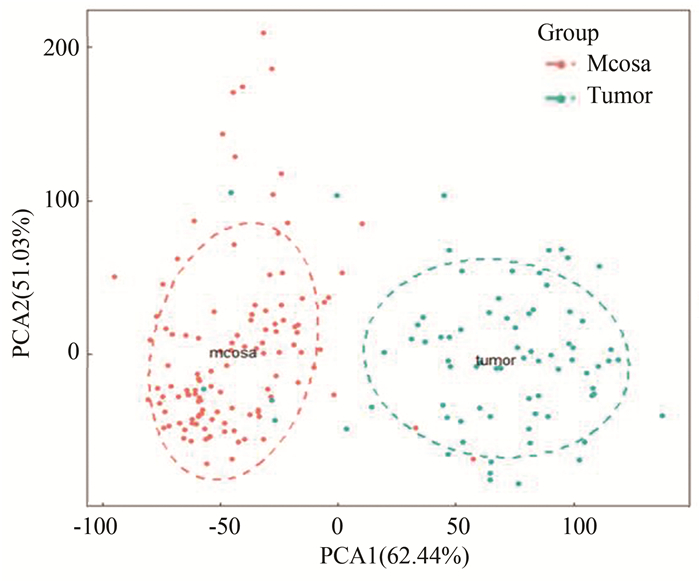
|
图 1 GSE106582 PCA分析图 Figure 1 PCA analysis diagram of GSE106582 |
在|log2FC|>1.5且调整后P < 0.05的条件下,从数据集GSE106582中共得到199个差异表达基因,其中56个是上调基因,143个是下调基因(见图 2)。
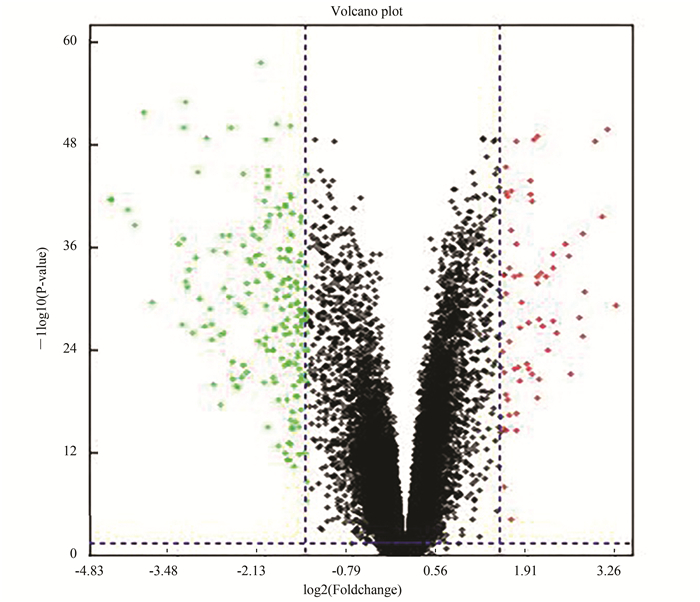
|
图 2 基因的表达水平及分布火山图 Figure 2 Volcano map of the expression level and distribution of genes |
运用DAVID在线工具对差异表达基因进行GO富集分析发现,其上调基因主要与胶原蛋白分解代谢过程(Collagen catabolic process),细胞外基质分解(Extracellular matrix disassembly),细胞外基质组织(Extracellular matrix organization)和胶原纤维组织(Collagen fibril organization)等生物学过程有关(见图 3a);而下调基因主要与碳酸氢盐运输(Bicarbonate transport),负生长调控(Negative regulation of growth),细胞对锌离子的反应(Cellular response to zinc ion)和一碳代谢过程(One-carbon metabolic process)等生物学过程有关(见图 3b)。
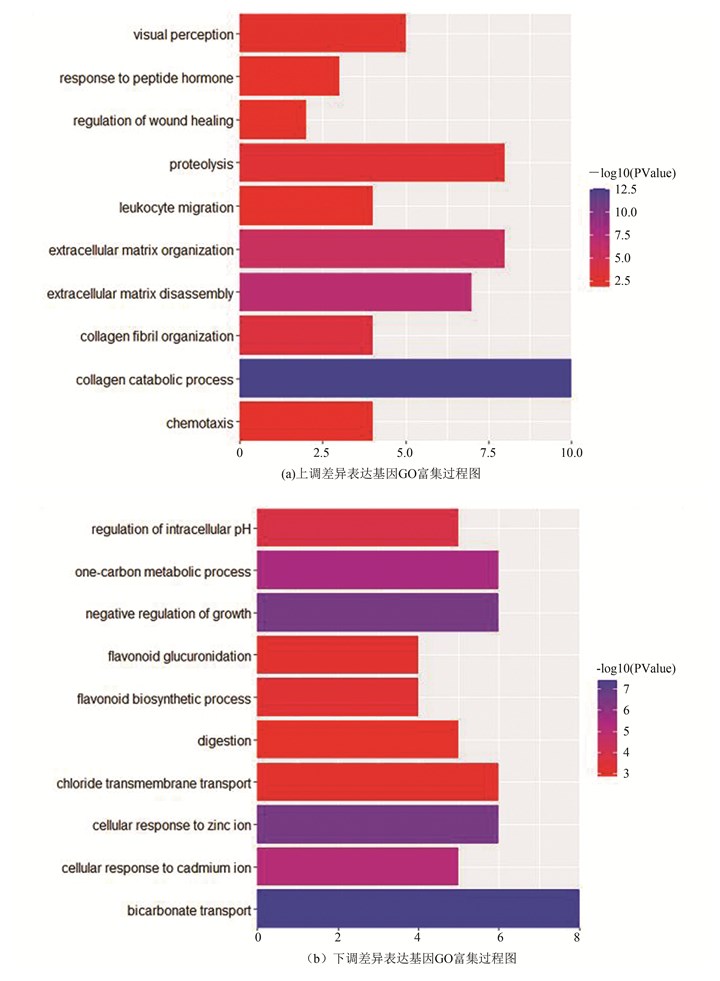
|
图 3 差异表达基因GO富集过程结果 Figure 3 Results of GO enrichment process of differentially expressed genes |
对差异表达基因进行KEGG通路富集分析发现,其上调基因主要参与细胞外基质-受体相互作用(ECM-receptor interaction),蛋白质消化与吸收(Protein digestion and absorption),PI3K/AKT信号通路(PI3K-Akt signaling pathway)和黏着斑(Focal adhesion)等信号通路(见图 4a);而下调基因主要参与矿物质吸收(Mineral absorption),药物代谢-细胞色素P450(Drug metabolism-cytochrome P450),化学致癌途径(Chemical carcinogenesis)和氮代谢(Nitrogen metabolism)等信号通路(见图 4b)。
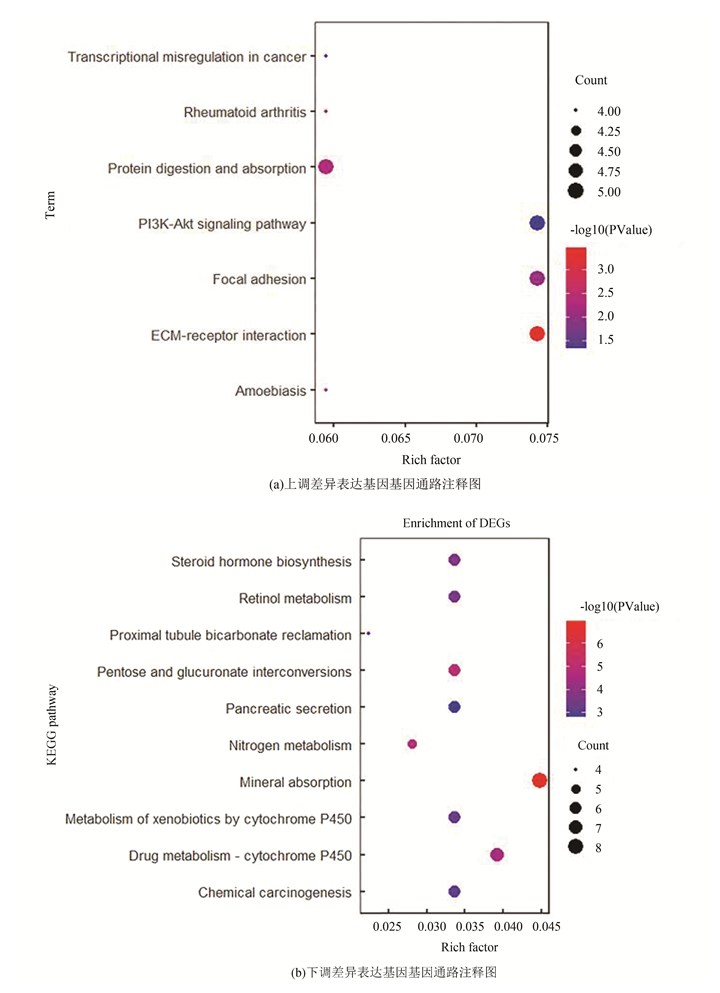
|
图 4 差异表达基因通路注释结果 Figure 4 Differentially expressed gene pathway annotation results |
将差异表达基因导入在线分析数据库STRING,并设定置信度0.4作为判断相互作用是否有意义的标准,去除网络中无连接的节点,构建PPI网络。将STRING构建的PPI网络导入Cytoscape3.1.1软件进行可视化分析,该网络有152个节点,377条边组成(图 5a)。使用MCODE插件分析出最显著的相互作用模块,该模块网络由13个节点,63条边组成(见图 5b)。上述13个节点/基因分别为BGN,COL1A1,COL1A2,MMP3,MMP1,TIMP1,CXCL12,PLAU,CXCL1,CXCL8,CXCL5,MMP7,SPP1。其中1个为下调基因(CXCL12),其余12个均为上调基因,后者有可能在结直肠的发生、发展过程中扮演癌基因角色,从而与结直肠癌的发生、发展与预后相关。
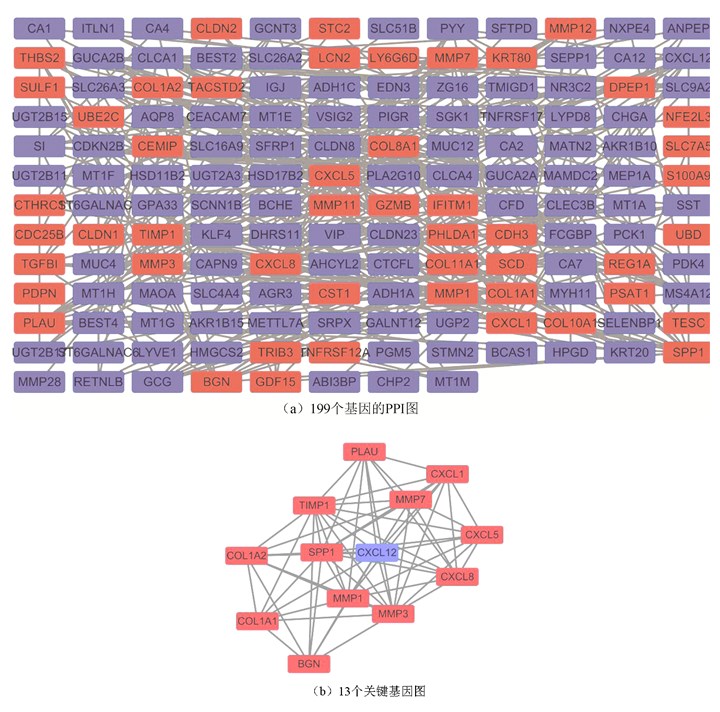
|
图 5 差异表达基因蛋白相互作用网络构建及可视化结果 Figure 5 Construction and visualization results of differentially expressed gene protein interaction network |
利用在线工具GEPIA2对显著模块中13个差异表达基因进行预后价值分析,结果显示:其中4个基因BGN,COL1A2,TIMP1,SPP1的高表达组的结直肠癌患者的OS显著低于相应的低表达组(见图 6a)。对上述的4个基因利用R语言包进行ROC诊断,结果显示,4个基因BGN,COL1A2,TIMP1,SPP1的ROC曲线下面积AUC分别为0.928,0.925,0.931和0.888,除了SPP1基因之外,另外3个基因的AUC均大于0.9,表明这3个基因BGN,COL1A2,TIMP1可作为灵敏度和特异度较高的结直肠癌不良预后指标(见图 6b)。通过GSE21510进行验证,BGN、COL1A2和TIMP1的诊断灵敏度要高于SPP1,与GSE106582实验结果是一致的。

|
图 6 预后价值分析结果 Figure 6 Prognostic value analysis results |
通过生物信息学分析方法对GEO数据库中的GSE106582芯片筛选结直肠癌与癌旁组织的差异表达基因,共获得199个差异基因,其中54个为上调基因,143个为下调基因基因。通过构建蛋白质相互作用网络图,得到13个核心差异表达基因,再应用STRING和GEPIA2工具对显著模块中13个差异表达基因进行预后价值分析,筛选出BGN、COL1A2和TIMP1可能是结直肠癌的不良预后指标。
双糖链蛋白聚糖(biglycan,BGN)是富含亮氨酸的蛋白多糖家族中的关键成员之一,通常在神经、骨、软骨、皮肤和肌肉中表达,发挥调节上皮细胞的形态、生长、粘附、骨矿化、炎症、迁移和分化功能[7]。BGN在膀胱癌、结直肠癌、胃癌等多种癌症中表达水平较高[8-10],参与多个肿瘤生物学过程包括细胞增殖、侵袭、上皮-间质转化、血管生成和化疗抵抗[9, 11]。其表达水平受多个因素多个信号通路调控,包括p38[12]、HIF-1[13]和NF-κB[14]等。在结直肠癌中,BGN/TLR4/NF-κB轴在炎症刺激下,能够以正反馈方式最终增强NF-κB信号通路活性,从而促进癌症的发生和进展[14]。
组织金属蛋白酶抑制剂-1(Tissue inhibitor of metalloproteinases-1,TIMP1)是基质金属蛋白酶家族(MMPs)的内源性抑制剂。TIMP1的过表达与多种癌症的不良预后相关,包括结直肠癌[15]、胃癌[16]、肺癌[17]。Batra等人发现,TIMP1的过度表达能够增加增殖和凋亡相关基因的表达水平[18];另有报道指出,通过诱导TIMP1特异性调控FAK-PI3K/AKT和MAPK通路,能够抑制TIMP1的表达,从而降低肿瘤细胞的增殖和转移,同时促进细胞凋亡[19];高水平的癌胚抗原和TIMP1与较短的结直肠癌患者生存时间相关,尤其是血浆TIMP1表达水平可以作为结直肠癌的潜在不良预后指标[20]。
Ⅰ型胶原α2链(Collagen type1 alpha2,COL1A2) 属于胶原蛋白基因家族中的成员之一。作为细胞外基质的主要成分之一,COL1A2主要通过细胞外基质受体通路和局部黏附通路影响细胞的增殖、分化、黏附和转移[21]。现有研究发现,COL1A2在肝癌、卵巢癌等癌组织中的表达水平明显高于正常组织,且高表达组患者的总体生存期较短[22-23]。在结直肠癌中,COL1A2的过度表达促进了EMT的发生。Mori[24]等报道COL1A2通过CpG岛过甲基化引起膀胱癌细胞增殖和迁移。类似地,COL1A2过甲基化现象也存在于结直肠癌,且COL1A2的过表达能够通过抑制NF-κB信号通路从而调控结直肠癌细胞的增殖、迁移与转移[25]。以上报道均提示BGN、COL1A2和TIMP1是结直肠癌的不良预后指标,与本研究生物信息学预测分析结论一致。
通过GO和KEGG富集分析发现,上调的差异表达基因主要富集在与胶原蛋白分解代谢过程,细胞外基质分解,细胞外基质受体相互作用和PI3K/AKT信号通路。细胞外基质在调节细胞内稳态和细胞间相互作用中发挥一定的作用,同时胶原蛋白是ECM的主要组分,在恶性肿瘤的细胞粘附、形态改变、侵袭和迁移过程中扮演重要的角色[26]。PI3K/Akt信号通路是多种肿瘤发生发展和炎症反应的重要信号通路,亦在促进肿瘤细胞增殖、调节细胞代谢、肿瘤生长、迁移和细胞骨架重塑等方面发挥重要作用[27]。下调的差异表达基因主要富集在碳酸氢盐运输、一碳代谢代谢过程、矿物质吸收、药物代谢-细胞色素P450和氮代谢通路等生物过程。一方面,上述途径主要集中在营养物质吸收和外源物质代谢等;另一方面,肠上皮细胞是防止细菌内毒素等有害物质进入人体的重要屏障,其主要功能是吸收营养、毒素和药物[28]。最近的研究表明,肠上皮细胞在维持肠道免疫稳态方面起着重要的作用,而相关信号通路的异常表达已被报道与结直肠癌的发生有关[29]。分析结果为PI3K/Akt信号通路在结直肠癌中的重要作用提供了更多的理论依据。目前PI3K/Akt/mTOR通路抑制剂依维莫司已被批准临床应用于晚期乳腺癌,而在结直肠癌中尚无报道。
4 结论综上所述,本研究通过多种生物信息学分析方法确定了199个差异表达基因,并发现BGN、COL1A2和TIMP1在结直肠癌中表达上调,可能是结直肠癌独立的不良预后因素。PI3K/Akt信号通路参与了结直肠癌的发生和转移。然而,BGN、COL1A2和TIMP1的表达水平或其生物学功能机制是否与PI3K/Akt信号通路有关目前尚不明确。对进一步深入研究结直肠癌发生和进展的分子机制、预后指标及治疗靶点的筛选具有重要意义,但需要进一步的实验和临床大样本的验证。
| [1] |
BRAY F I, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 (  0) 0) |
| [2] |
RAWLA P, SUNKARA T, BARSOUK A, et al. Epidemiology of colorectal cancer: incidence, mortality, survival, and risk factors[J]. Przeglad Gastroenterologiczny, 2019, 14(2): 89-103. DOI:10.5114/pg.2018.81072 (  0) 0) |
| [3] |
SIEGEL R L, MILLER K D, JEMAL A, et al. Cancer statistics, 2019[J]. CA: A Cancer Journal for Clinicians, 2019, 69(1): 7-34. DOI:10.3322/caac.21551 (  0) 0) |
| [4] |
WANG Y, ZHANG Y, ZHANG G, et al. Regorafenib overcomes chemotherapeutic multidrug resistance mediated by ABCB1 transporter in colorectal cancer: in vitro and in vivo study[J]. Cancer Letters, 2017, 396(1): 145-154. DOI:10.1016/j.canlet.2017.03.011 (  0) 0) |
| [5] |
SARTOREBIANCHI A, LOUPAKIS F, ARGILES G, et al. Challenging chemoresistant metastatic colorectal cancer: therapeutic strategies from the clinic and from the laboratory[J]. Annals of Oncology, 2016, 27(8): 1456-1466. DOI:10.1093/annonc/mdw191 (  0) 0) |
| [6] |
TAN X, TANG H, GONG L, et al. Integrating genome-wide association studies and gene expression profiles with chemical-genes interaction networks to identify chemicals associated with colorectal cancer[J]. Frontiers in Genetics, 2020, 11(385): 1-9. DOI:10.3389/fgene.2020.00385 (  0) 0) |
| [7] |
FALLON J R, MCNALLY E M. Non-Glycanated Biglycan and LTBP4: Leveraging the extracellular matrix for Duchenne Muscular Dystrophy therapeutics[J]. Matrix Biology, 2018, 68(1): 616-627. DOI:10.1016/j.matbio.2018.02.016 (  0) 0) |
| [8] |
SCHULZ G, GRIMM T, SERS C, et al. Prognostic value and association with epithelial-mesenchymal transition and molecular subtypes of the proteoglycan biglycan in advanced bladder cancer[J]. Urologic Oncology-Seminars and Original Investigations, 2019, 37(8): 57-64. DOI:10.1016/j.urolonc.2019.05.011 (  0) 0) |
| [9] |
GU X, MA Y, XIAO J, et al. Up-regulated biglycan expression correlates with the malignancy in human colorectal cancers[J]. Clinical and Experimental Medicine, 2012, 12(3): 195-199. DOI:10.1007/s10238-011-0155-4 (  0) 0) |
| [10] |
HU L, DUAN Y T, LI J F, et al. Biglycan enhances gastric cancer invasion by activating FAK signaling pathway[J]. Oncotarget, 2014, 5(7): 1885-1896. DOI:10.18632/oncotarget.1871 (  0) 0) |
| [11] |
XING X, GU X, MA T, et al. Knockdown of biglycan expression by RNA interference inhibits the proliferation and invasion of, and induces apoptosis in, the HCT116 colon cancer cell line[J]. Molecular Medicine Reports, 2015, 12(5): 7538-7544. DOI:10.3892/mmr.2015.4383 (  0) 0) |
| [12] |
UNGEFROREN H, LENSCHOW W, CHEN W B, et al. Regulation of biglycan gene expression by transforming growth factor-β requires MKK6-p38 mitogen-activated protein kinase signaling downstream of smad signaling[J]. Journal of Biological Chemistry, 2003, 278(13): 11041-11049. DOI:10.1074/jbc.M300035200 (  0) 0) |
| [13] |
DOE M R, ASCANO J M, KAUR M, et al. Myc posttranscriptionally induces HIF1 protein and target gene expression in normal and cancer cells[J]. Cancer Research, 2012, 72(4): 949-957. DOI:10.1158/0008-5472.CAN-11-2371 (  0) 0) |
| [14] |
HUANG H C, CAI B H, SUEN C S, et al. BGN/TLR4/NF-B mediates epigenetic silencing of immunosuppressive siglec ligands in colon cancer cells[J]. Cells, 2020, 9(2): 397-411. DOI:10.3390/cells9020397 (  0) 0) |
| [15] |
ISHIDA H, MURATA N, HAYASHI Y, et al. Serum levels of tissue inhibitor of metalloproteinases-1(TIMP-1) in colorectal cancer patients[J]. Surgery Today, 2003, 33(12): 885-892. DOI:10.1007/s00595-003-2628-x (  0) 0) |
| [16] |
GRUNNET M, MAUSORENSEN M, BRUNNER N, et al. Tissue inhibitor of metalloproteinase 1 (TIMP-1) as a biomarker in gastric cancer: A review[J]. Scandinavian Journal of Gastroenterology, 2013, 48(8): 899-905. DOI:10.3109/00365521.2013.812235 (  0) 0) |
| [17] |
CHANG Y H, CHIU Y J, CHENG H, et al. Down-regulation of TIMP-1 inhibits cell migration, invasion, and metastatic colonization in lung adenocarcinoma[J]. Tumor Biology, 2015, 36(5): 3957-3967. DOI:10.3109/00365521.2013.812235 (  0) 0) |
| [18] |
BATRA J, ROBINSON J, SOARES A S, et al. Matrix metalloproteinase-10 (MMP-10) interaction with tissue inhibitors of metalloproteinases TIMP-1 and TIMP-2: Binding studies and crystal structure[J]. Journal of Biological Chemistry, 2012, 287(19): 15935-15946. DOI:10.1074/jbc.M112.341156 (  0) 0) |
| [19] |
SONG G, XU S, ZHANG H, et al. TIMP1 is a prognostic marker for the progression and metastasis of colon cancer through FAK-PI3K/AKT and MAPK pathway[J]. Journal of Experimental & Clinical Cancer Research, 2016, 35(1): 148-148. DOI:10.1186/s13046-016-0427-7 (  0) 0) |
| [20] |
BIRGISSON H, NIELSEN H J, CHRISTENSEN I J, et al. Preoperative plasma TIMP-1 is an independent prognostic indicator in patients with primary colorectal cancer: A prospective validation study[J]. European Journal of Cancer, 2010, 46(18): 3323-3331. DOI:10.1016/j.ejca.2010.06.009 (  0) 0) |
| [21] |
HAQ F, AHMED N, QASIM M. Comparative genomic analysis of collagen gene diversity[J]. Biotech, 2019, 9(3): 83-91. DOI:10.1007/s13205-019-1616-9 (  0) 0) |
| [22] |
JI J, ZHAO L, BUDHU A, et al. Let-7g targets collagen type I α2 and inhibits cell migration in hepatocellular carcinoma[J]. Journal of Hepatology, 2010, 52(5): 690-697. DOI:10.1016/j.jhep.2009.12.025 (  0) 0) |
| [23] |
WU Y H, CHANG T H, HUANG Y, et al. COL11A1 promotes tumor progression and predicts poor clinical outcome in ovarian cancer[J]. Oncogene, 2014, 33(26): 3432-3440. DOI:10.1038/onc.2013.307 (  0) 0) |
| [24] |
MORI K, ENOKIDA H, KAGARA I, et al. CpG hypermethylation of collagen type I α2 contributes to proliferation and migration activity of human bladder cancer[J]. International Journal of Oncology, 2009, 34(6): 1593-1602. DOI:10.3892/ijo_00000289 (  0) 0) |
| [25] |
YU Y, LIU D, LIU Z, et al. The inhibitory effects of COL1A2 on colorectal cancer cell proliferation, migration, and invasion[J]. Journal of Cancer, 2018, 9(16): 2953-2962. DOI:10.7150/jca.25542 (  0) 0) |
| [26] |
AITKEN K J, BAGLI D J. The bladder extracellular matrix. Part I: architecture, development and disease[J]. Nature Reviews Urology, 2009, 6(11): 596-611. DOI:10.1038/nrurol.2009.201 (  0) 0) |
| [27] |
CHANG L, GRAHAM P H, HAO J, et al. PI3K/Akt/mTOR pathway inhibitors enhance radiosensitivity in radioresistant prostate cancer cells through inducing apoptosis, reducing autophagy, suppressing NHEJ and HR repair pathways[J]. Cell Death and Disease, 2014, 5(10): e1437-e1446. DOI:10.1038/cddis.2014.415 (  0) 0) |
| [28] |
GERBE F, LEGRAVEREND C, JAY P, et al. The intestinal epithelium tuft cells: specification and function[J]. Cellular and Molecular Life Sciences, 2012, 69(17): 2907-2917. DOI:10.1007/s00018-012-0984-7 (  0) 0) |
| [29] |
FERREIRA A R, FELGUEIRAS J, FARDILHA M, et al. Signaling pathways in anchoring junctions of epithelial cells: cell-to-cell and cell-to-extracellular matrix interactions[J]. Journal of Receptors and Signal Transduction, 2015, 35(1): 67-75. DOI:10.3109/10799893.2014.931426 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


