浸润性乳腺癌(Breast invasive carcinoma, BRCA)是全世界女性癌症死亡的第二大原因[1]。BRCA被认为是一种复杂的异质性疾病,基于组织病理学特征和治疗反应,其表现出显著的多样性,传统疗法(如乳房X线照相术)无法更好应对乳腺癌的挑战[2]。与肿瘤细胞增殖相关的基因标记在预测乳腺癌的预后方面具有局限性[3],需要不断完善乳腺癌的治疗策略,以达到更好的患者预后[1, 4]。免疫细胞浸润在乳腺癌发展进程中与预后相关联,关注免疫细胞浸润可以帮助患者寻找新的治疗方法,借助公共数据库进行数据分析,有助于更好地寻找乳腺癌预后或预测生物标志物[3, 5]。研究表明,BRCA1相关环指结构域-1 (BRCA1-associated RING domain, BARD1)在乳腺癌中上调,其高表达与乳腺癌的无病生存(Disease-free survival, DFS)和远处无转移生存(Distant metastasis-free survival, DMFS)差有关[6];血清脂质运载蛋白1(Lipocalin-1, LCN1)在BRCA中高表达,低表达LCN1与BRCA中无复发生存(Relapse-free survival, RFS)的不良预后相关[2];Janus激酶1 (Janus kinase-1, JAK1) 在乳腺癌中低表达,且与较差的总体生存(Overall survival, OS)和无复发生存(RFS)有关[7];脂滴包被蛋白(Perilipin-1, PLIN1)在乳腺癌中明显下调,与较差的总体生存(OS)相关[8],这些数据揭示了乳腺癌中具有预后意义的潜在基因治疗靶标。
细胞色素b561(Cytochromes b561, CYB561)又称Uniprot P49447,因在561 nm处具有吸光性而得名。CYB561氨基酸序列高度保守,是具有6个跨膜结构域的单体蛋白质[9]。该蛋白质附着在神经内分泌组织中的儿茶酚胺和神经肽分泌小泡的膜上,对儿茶酚胺的生物合成至关重要[10]。肿瘤抑制因子CYB561蛋白是抗坏血酸(ASC)可还原的跨膜二血红素蛋白,维持胞质和囊内抗坏血酸之间的氧化还原状态[11]。它在人体组织中广泛表达。在大脑中,它在皮质和海马中强烈表达,而在其他部位则表达程度较低,在各种神经内分泌组织中都证实了它的存在[12]。CYB561与细胞代谢和线粒体激活相关[13],研究表明其可能是卵巢癌的单基因预后生物标志物[14]。本研究发现CYB561在BRCA中失调,提示其可能在BRCA的发生发展中具有潜在作用。
1 材料和方法 1.1 Oncomine数据库分析在Oncomine数据库(https://www.oncomine.org/resource/login.html)中确定了CYB561基因在各种类型癌症中的表达水平[15]。根据以下值确定阈值:P值为0.001,倍数变化为2,所有基因的基因排名。
1.2 TIMER数据库分析在TIMER数据库(https://cistrome.shinyapps.io/timer/)中使用Diff Exp模块来探索肿瘤与相邻正常组织之间靶基因的差异表达;使用Gene模块分析了CYB561表达与免疫浸润的相关性,包括B细胞,CD4+ T细胞,CD8 + T细胞,中性粒细胞和树突状细胞[16-18]。
1.3 UALCAN数据库在UALCAN数据库(http://ualcan.path.uab.edu/index.html)中根据性别,年龄,种族,疾病阶段和肿瘤等级等进行多亚组分类,分析患者CYB561表达[19]。
1.4 cBioPortal数据库使用cBioPortal数据库(https://www.cbioportal.org/)分析CYB561基因组改变类型,不同表型的乳腺癌改变频率以及氨基酸中的蛋白质变化[20-21]。
1.5 LinkedOmics数据库利用LinkedOmics数据库(http://www.linkedomics.org/login.php),研究TCGA BRCA队列(n = 1 093)中与CYB561相关的差异表达基因[22]。
1.6 WebGestalt数据库对获得的差异基因,使用WebGestalt数据库(http://www.webgestalt.org/)进行基因本体论(gene ontology, GO)和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)富集分析,研究CYB561在BRCA中的生物学作用[23]。
1.7 PrognoScan数据库通过PrognoScan数据库(http://dna00.bio.kyutech.ac.jp/PrognoScan/index.html)分析了CYB561表达与BRCA患者预后之间的相关性,例如总生存期(OS)和无病生存期(DFS)和无远处转移生存期(DMFS)[24]。将阈值调整为Cox P值< 0.05。
1.8 GEPIA数据库通过GEPIA数据库(http://gepia.cancer-pku.cn/index.html)分析了TCGA和GTEx项目的BRCA中CYB561表达和患者总生存期(OS)之间的相关性[25]。
2 结果分析 2.1 不同类型人类癌症中CYB561的mRNA表达水平使用Oncomine数据库分析多种癌症类型的不同肿瘤和正常组织中的CYB561 mRNA水平表明,与正常组织相比,CYB561在乳腺癌(Breast Cancer)中表达更高(见图 1a),进一步分析Ma Breast 4,Curtis Breast,TCGA Breast和Finak Breast 4种不同的BRCA数据集,发现CYB561在BRCA组织中mRNA表达均显著高于在正常组织(p<0.01,Fold Change>2)(见图 1c~f)。使用TIMER数据库对CYB561的肿瘤与相邻正常组织之间的差异表达进行对比,发现CYB561在多种癌症中失调(见图 1b),提示其在BRCA和多种癌症发生发展进程中可能具有潜在作用。
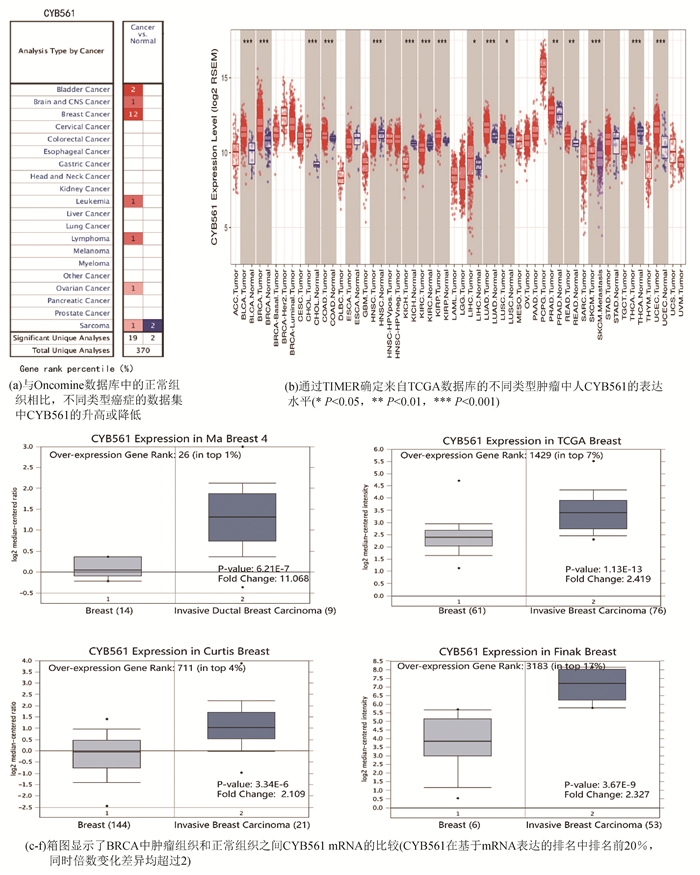
|
图 1 CYB561在不同类型的人类癌症中的表达水平(Oncomine和TIMER) Figure 1 CYB561 expression levels in different types of human cancer (Oncomine and TIMER) |
为探究CYB561表达量与临床特征之间的关系,对TCGA中1097例BRCA样品进行多亚组分析,结果显示BRCA患者中CYB561表达明显高于健康人(见图 2a~h)。在性别(见图 2b)、种族(见图 2d)、癌症等级(见图 2e)、更年期状态(见图 2f)和乳腺癌分型(见图 2g)亚组显示出组间差异,在年龄(见图 2c)和TP53突变状态(见图 2h)亚组间无显著差异。从第1阶段到第4阶段,CYB561表达在乳腺癌中呈逐渐增加,并且在第4阶段中发现最高的CYB561表达,表明CYB561表达与癌症各个阶段密切相关。CYB561在不同分型的BRCA中显示出差异,可用于辅助区分不同分型的乳腺癌。以上结果表明,高CYB561表达与浸润性乳腺癌多种临床特征相关,可将CYB561的表达作为BRCA中潜在的诊断指标。

|
图 2 BRCA患者不同亚组中CYB561的相对表达水平(UALCAN) Figure 2 Relative expression levels of CYB561 in different subgroups of patients with BRCA (UALCAN) 注:根据性别,年龄,性别,种族,肿瘤等级、更年期状态、乳腺癌分型和TP53突变状态进行分组(P<0.05) |
基因组突变与肿瘤发生密切相关[2]。浸润性乳腺癌中许多位点的乳腺浸润前病变均在染色体出现异常变化[26]。为此,使用cBioPortal数据库分析了BRCA中CYB561的基因组改变情况。数据显示,CYB561基因组遗传变异约为7%(见图 3a),包括拷贝数扩增、融合和意义不明的错义突变。而且,CYB561的遗传改变频率在各种浸润性乳腺癌中表现出差异(见图 3b), 且存在拷贝数变异和氨基酸点位突变(见图 3c~d)。以上结果提示,BRCA中发现CYB561的基因改变,可能在浸润性乳腺癌的发生发展中起重要作用。
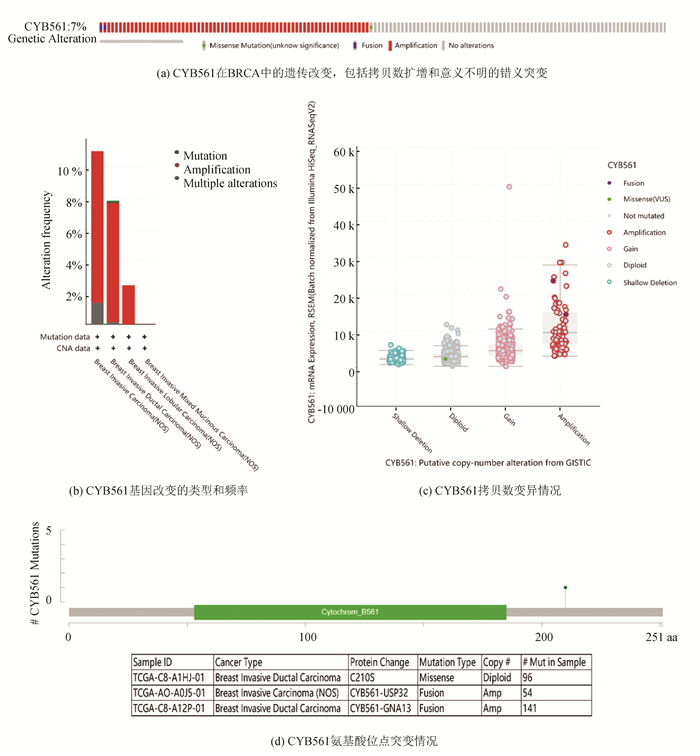
|
图 3 BRCA中CYB561的基因组突变(cBioPortal) Figure 3 Genomic mutations of CYB561 in BRCA (cBioPortal) |
CYB561在BRCA中潜在的功能,可能是由于CYB561基因组改变以及和多个基因共同作用的结果。为此,使用LinkedOmics分析出与CYB561正负相关50个关键基因(见图 4a~b)。并对其采用WebGestalt数据库进行GO的生物学功能(Biological Process, BP)和KEGG富集分析(见图 5a~b)。结果表明,其参与的生物学过程有线粒体基因表达、高尔基小泡运输、线粒体运输、前体代谢产物和能量的变化、细胞趋化性、白细胞分化、积极调节细胞因子的产生、细胞外结构组织、白细胞活化的调节;参与的信号通路有:蛋白酶体、内质网中的蛋白质加工、溶酶体、代谢途径、ECM受体相互作用、Wnt信号通路、细胞粘附分子、破骨细胞分化、细胞因子与细胞因子受体相互作用。富集分析结果,有助于了解CYB561及其相互作用基因在BRCA中的作用机理和过程。

|
图 4 BRCA中与CYB561相关的差异表达基因(LinkedOmics) Figure 4 Differentially expressed genes associated with CYB561 in BRCA (LinkedOmics) 注:(a, b)热图显示BRCA中与CYB561正相关和负相关的基因(前50名)。红色表示正相关的基因,蓝色表示负相关的基因。 |
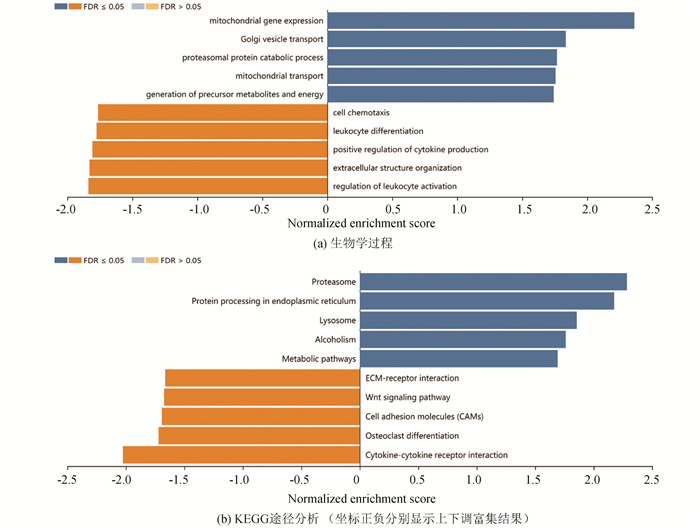
|
图 5 CYB561在BRCA中显著丰富的GO注释和KEGG途径(WebGestalt) Figure 5 Significantly enriched GO annotations and KEGG pathways of CYB561 in BRCA (WebGestalt) 注:使用GSEA分析了BRCA中CYB561共表达基因的GO注释和KEGG途径的显著富集。 |
了解CYB561在BRCA中参与的生物学过程后,我们使用PrognoScan和GEPIA评估了CYB561表达与BRCA癌症患者的预后关系。不同的数据集结果均显示,相较于低表达组,CYB561高表达组患者总体生存(Overall Survival, OS)、无病生存(Disease-Free Survival, DFS)、无远处转移生存(Distant Metastasis-Free Survival, DMFS)均较差(见图 6a~f),提示浸润性乳腺癌中高CYB561表达可能是一个独立的危险因素,并导致BRCA患者的不良预后。
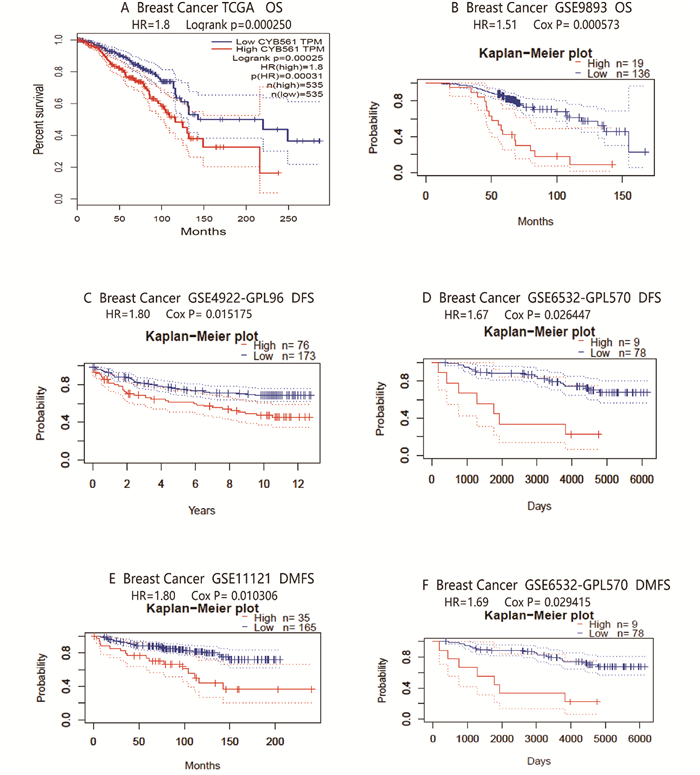
|
图 6 Kaplan-Meier生存曲线比较乳腺癌人群中CYB561的高表达和低表达(PrognoScan和GEPIA) Figure 6 Kaplan-Meier survival curves for comparing the higher and lower expressions of CYB561 in breast cancer cohorts (PrognoScan and GEPIA) |
浸润性淋巴细胞与部分癌症预后相关[27-28]。为此,我们探究了CYB561表达高导致BRCA不良预后与浸润性淋巴细胞的免疫浸润水平的相关性。结果表明,CYB561表达与BRCA的肿瘤纯度正显著相关,并与BRCA的B细胞、CD8+ T细胞和CD4+ T细胞浸润水平显著正相关(见图 7a, c, e),与树突状细胞和中性粒细胞浸润水平显著负相关(见图 7b, d),表明CYB561 mRNA水平在BRCA免疫浸润中具有潜在作用。累积存活表明,BRCA中CYB561的免疫浸润B细胞具有统计学意义(p < 0.05),表明B细胞显着影响预后(见图 7f),值得进一步探究。研究CYB561与免疫浸润的关系,有助于为未来BRCA免疫治疗提供新视野。
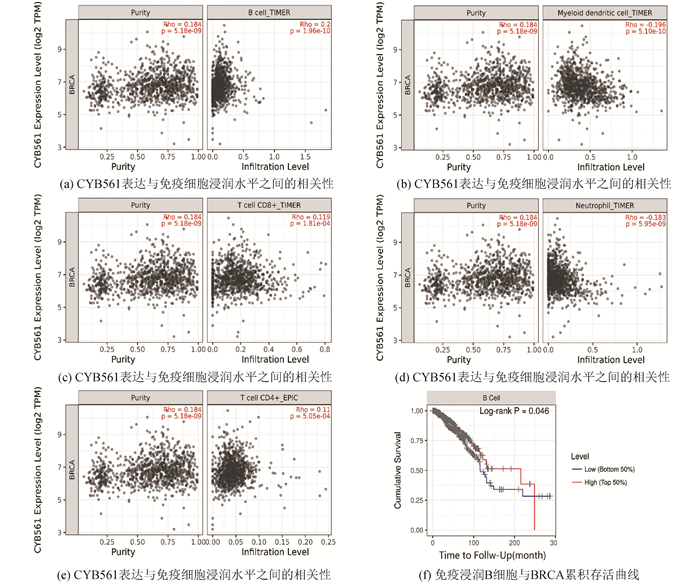
|
图 7 BRCA的B细胞,CD4 + T细胞,CD8 + T细胞,中性粒细胞和树突状细胞中与CYB561相关的免疫浸润(TIMER) Figure 7 Immune infiltrates in correlation with CYB561 in B cells, CD4+ T cells, CD8+ T cells, neutrophil, and dendritic cells of BRCA (TIMER) 注:(f)曲线正相关:Rho>0, p < 0.05; 负相关:Rho>0, p < 0.05. |
人类CYB561 mRNA的翻译控制通过肾上腺素途径的调节影响心率和血压[9, 30]。其他CYB561蛋白可还原铁化合物[31]。CYB561成员与生理过程有关,包括铁摄取神经内分泌组织中的ASC再生和肿瘤生长[32]。CYB561 3’-UTR中一个非常常见的遗传变异(MAF~45%-49%)会改变对特定微RNA(hsa-miR-1294)的反应,最终导致基础和应激的遗传变化[9]。正常人口腔角质形成细胞(NHOK)中的衰老发生与CYB561基因表达的改变有关[33]。编码CYB561基因中的致病性纯合突变,导致去甲肾上腺素的合成停止,引起的新型常染色体隐性体位性低血压综合征[34]。卵巢癌中CYB561 mRNA表达水平较低且与预后不良相关, 可能是已知拮抗剂或抑制剂的治疗靶标[14]。目前针对CYB561的研究较少,未见CYB561关于BRCA报道,其功能和作用机理有待进一步研究。
研究发现CYB561多种类型癌中失调,且在BRCA中高表达,在4种乳腺癌数据集中证实了我们的结论。基于临床病例特征进行亚组分析表明,CYB561对所有亚型和不同种族的乳腺癌诊断有用,可能是潜在的诊断和预后标志物,有待临床验证。基因组不稳定性更高的癌症具有更高的基因组多样性和更多的突变[35-36],部分位点存在基因组改变是BRCA的特点之一,CYB561基因组发生的遗传改变,提示其可能在BRCA中具有潜在作用。对CYB561相关的差异表达基因进行GO和KEGG富集分析,有助于了解CYB561在BRCA中参与的生物学过程和信号通路,为研究CYB561在BRCA中的作用提供方向。研究预后因素,有助于临床治疗的决策。研究表明,高表达CYB561与较差的OS、DFS和DMFS有关,可能是BRCA预后不良的预测指标,表明CYB561表达水平可能是BRCA生存需要考虑的重要因素。不同免疫细胞的浸润程度与BRCA预后存在相关性,基因组改变容易导致更多新抗原,进而产生更大的免疫浸润[35-36],分析发现CYB561表达与部分浸润性淋巴细胞的免疫浸润水平相关,且B细胞对BRCA预后有影响,值得进一步探究。我们的研究为理解CYB561在肿瘤免疫学中的潜在作用及其作为癌症生物标志物的应用提供了新视角。
4 结论本研究中的数据首次阐述了CYB561在BRCA中高表达,并与部分临床病理特征相关,高表达CYB561与不良预后相关,可作为BRCA潜在的预后指标,为了解CYB561 mRNA在肿瘤免疫学中的潜在作用及其预后价值提供了见识。尽管本研究提供了CYB561在BRCA中的一些有意义的数据,但也必须看到我们的研究存在一定的局限性: 一方面是我们的结果未在临床样本中得到验证,无法提供准确的临床数据支撑; 另一方面是BRCA的组织学类型和多个解剖部位的检验结果可能存在较大差异。后续我们将进行细胞生物学实验和人体组织标本测试以验证本研究结果,并评估CYB561抑制剂干扰免疫细胞的潜力。
| [1] |
YAGHOUBI A, KHAZAEI M, HASANIAN S M, et al. Bacteriotherapy in breast cancer[J]. International Journal of Molecular Sciences, 2019, 20(23): 5880. DOI:10.3390/ijms20235880 (  0) 0) |
| [2] |
YANG Y M, LI F, LUO X Y, et al. Identification of LCN1 as a potential biomarker for breast cancer by bioinformatic analysis[J]. DNA and Cell Biology, 2019, 38(10): 1088-1099. DOI:10.1089/dna.2019.4843 (  0) 0) |
| [3] |
KWON M J. Emerging immune gene signatures as prognostic or predictive biomarkers in breast cancer[J]. Archives of Pharmacal Research, 2019, 42(11): 947-961. DOI:10.1007/s12272-019-01189-y (  0) 0) |
| [4] |
MARILINA G A, MAXIMINO R. Immunotherapy: A challenge of breast cancer treatment[J]. Cancers, 2019, 11(12): 1822. DOI:10.3390/cancers11121822 (  0) 0) |
| [5] |
BADR N M, BERDITCHEVSKI F, SHAABAN A M. The immune microenvironment in breast carcinoma: predictive and prognostic role in the neoadjuvant setting[J]. Pathobiology, 2020, 87(2): 61-74. DOI:10.1159/000504055 (  0) 0) |
| [6] |
CHEN Y Z, ZUO D, REN H L, et al. Bioinformatics analysis of expression and alterations of BARD1 in breast cancer[J]. Technology in Cancer Research & Treatment, 2019, 18: 1533033819892260. DOI:10.1177/1533033819892260 (  0) 0) |
| [7] |
CHEN B, LAI J G, DAI D N, et al. JAK1 as a prognostic marker and its correlation with immune infiltrates in breast cancer[J]. Aging, 2019, 11(23): 11124-11135. DOI:10.18632/aging.102514 (  0) 0) |
| [8] |
ZHOU C, WANG M, ZHOU L, et al. Prognostic significance of PLIN1 expression in human breast cancer[J]. Oncotarget, 2016, 7(34): 54488-54502. DOI:10.18632/oncotarget.10239 (  0) 0) |
| [9] |
ZHANG K X, DEACON D C, RAO F W, et al. Human heart rate: Heritability of resting and stress values in twin pairs, and influence of genetic variation in the adrenergic pathway at a microribonucleic acid microRNA motif in the 3'-UTR of cytochrome b561[J]. Journal of the American College of Cardiology, 2014, 63(4): 358-368. DOI:10.1016/j.jacc.2013.09.025 (  0) 0) |
| [10] |
MCBRIDE O W, YI H F, SRIVASTAVA M. The human cytochrome b561 gene (CYB561) is located at 17q11-qter[J]. Genomics, 1994, 21(3): 662-663. DOI:10.1006/geno.1994.1332 (  0) 0) |
| [11] |
BÉRCZI B, ZIMÁNYI L, ASARD H. Dihydrolipoic acid reduces cytochrome b561 proteins[J]. European Biophysics Journal with Biophysics Letters, 2013, 42(2/3): 159-168. DOI:10.1007/s00249-012-0812-x (  0) 0) |
| [12] |
PRUSS R M, SHEPARD E A. Cytochrome b561 can be detected in many neuroendocrine tissues using a specific monoclonal antibody[J]. Neuroscience, 1987, 22(1): 149-157. DOI:10.1016/0306-4522(87)90205-3 (  0) 0) |
| [13] |
POLAK M E, THIRDBOROUGH S M, UNG C Y, et al. Distinct molecular signature of human skin langerhans cells denotes critical differences in cutaneous dendritic cell immune regulation[J]. Journal of Investigative Dermatology, 2014, 134(3): 695-703. DOI:10.1038/jid.2013.375 (  0) 0) |
| [14] |
WILLIS S, VILLALOBOS V M, GEVAERT O, et al. Single gene prognostic biomarkers in ovarian cancer: A meta-analysis[J]. PLoS ONE, 2016, 11(2): e0149183. DOI:10.1371/journal.pone.0149183 (  0) 0) |
| [15] |
RHODES D R, KALYANA-SUNDARAM S, MAHAVISNO V, et al. Oncomine 3.0: Genes, pathways, and networks in a collection of 18, 000 cancer gene expression profiles[J]. Neoplasia, 2007, 9(2): 166-180. DOI:10.1593/neo.07112 (  0) 0) |
| [16] |
LI T W, FAN J Y, WANG B B, et al. TIMER: A web server for comprehensive analysis of tumor-infiltrating immune cells[J]. Cancer Research, 2017, 77(21): e108-e110. DOI:10.1158/0008-5472.CAN-17-0307 (  0) 0) |
| [17] |
LI B, SEVERSON E, PIGNON J C, et al. Comprehensive analyses of tumor immunity: Implications for cancer immunotherapy[J]. Genome Biology, 2016, 17(1): 174. DOI:10.1186/s13059-016-1028-7 (  0) 0) |
| [18] |
LI T W, FU J X, ZENG Z X, et al. TIMER2.0 for analysis of tumor-infiltrating immune cells[J]. Nucleic Acids Research, 2020, 48(W1): W509-W514. DOI:10.1093/nar/gkaa407 (  0) 0) |
| [19] |
CHANDRASHEKAR D S, BASHEL B, BALASUBRAMANYA S A H, et al. UALCAN: A portal for facilitating tumor subgroup gene expression and survival analyses[J]. Neoplasia, 2017, 19(8): 649-658. DOI:10.1016/j.neo.2017.05.002 (  0) 0) |
| [20] |
GAO J J, AKSOY B A, DOGRUSOZ U, et al. Integrative analysis of complex cancer genomics and clinical profiles using the cBioPortal[J]. Science Signaling, 2013, 6(269): pl1. DOI:10.1126/scisignal.2004088 (  0) 0) |
| [21] |
CERAMI E, GAO J, DOGRUSOZ U, et al. The cBio cancer genomics portal: an open platform for exploring multidimensional cancer genomics data[J]. Cancer Discovery, 2012, 2(5): 401-404. DOI:10.1158/2159-8290.CD-12-0095 (  0) 0) |
| [22] |
VASAIKAR S V, STRAUB P, WANG J, et al. LinkedOmics: analyzing multi-omics data within and across 32 cancer types[J]. Nucleic Acids Research, 2018, 46(D1): D956-D963. DOI:10.1093/nar/gkx1090 (  0) 0) |
| [23] |
WANG J, DUNCAN D, SHI Z A, et al. WEB-based GEne set analysis toolkit (WebGestalt): update 2013[J]. Nucleic Acids Research, 2013, 41(Web Server issue): W77-W83. DOI:10.1093/nar/gkt439 (  0) 0) |
| [24] |
MIZUNO H, KITADA K, NAKAI K, et al. PrognoScan: A new database for meta-analysis of the prognostic value of genes[J]. BMC Medical Genomics, 2009, 2: 18. DOI:10.1186/1755-8794-2-18 (  0) 0) |
| [25] |
TANG Z F, LI C W, KANG B X, et al. GEPIA: A web server for cancer and normal gene expression profiling and interactive analyses[J]. Nucleic Acids Research, 2017, 45(W1): W98-W102. DOI:10.1093/nar/gkx247 (  0) 0) |
| [26] |
REIS-FILHO J S, LAKHANI S R. The diagnosis and management of pre-invasive breast disease: genetic alterations in pre-invasive lesions[J]. Breast Cancer Research: BCR, 2003, 5(6): 313-319. DOI:10.1186/bcr650 (  0) 0) |
| [27] |
OHTANI H. Focus on TILs: Prognostic significance of tumor infiltrating lymphocytes in human colorectal cancer[J]. Cancer immunity, 2007, 7(7): 4. (  0) 0) |
| [28] |
AZIMI F, SCOLYER R A, RUMCHEVA P, et al. Tumor-infiltrating lymphocyte grade is an independent predictor of sentinel lymph node status and survival in patients with cutaneous melanoma[J]. Journal of Clinical Oncology, 2012, 30(21): 2678-2683. DOI:10.1200/JCO.2011.37.8539 (  0) 0) |
| [29] |
GHIMIRE L V, KOHLI U, LI C, et al. Catecholamine pathway gene variation is associated with norepinephrine and epinephrine concentrations at rest and after exercise[J]. Pharmacogenetics and Genomics, 2012, 22(4): 254-260. DOI:10.1097/FPC.0b013e328350a274 (  0) 0) |
| [30] |
PICCO C, SCHOLZ-STARKE J, NASO A, et al. How are cytochrome b561 electron currents controlled by membrane voltage and substrate availability?[J]. Antioxidants & Redox Signaling, 2014, 21(3): 384-391. DOI:10.1089/ars.2013.5809 (  0) 0) |
| [31] |
ASARD H, BARBARO R, TROST P, et al. Cytochromes b561: Ascorbate-mediated trans-membrane electron transport[J]. Antioxid Redox Signal, 2013, 19(9): 1026-1035. DOI:10.1089/ars.2012.5065 (  0) 0) |
| [32] |
KANG M K, KAMETA A, SHIN K, et al. Senescence-associated genes in normal human oral keratinocytes[J]. Experimental Cell Research, 2003, 287(2): 272-281. DOI:10.1016/s0014-4827(03)00061-2 (  0) 0) |
| [33] |
VAN DEN BERG M P, ALMOMANI R, BIAGGIONI I, et al. Mutations in CYB561 causing a novel orthostatic hypotension syndrome[J]. Circulation Research, 2018, 122(6): 846-854. DOI:10.1161/CIRCRESAHA.117.311949 (  0) 0) |
| [34] |
PAN J H, ZHOU H, COOPER L, et al. LAYN is a Prognostic biomarker and correlated with immune infiltrates in gastric and colon cancers[J]. Frontiers in Immunology, 2019, 10: 6. DOI:10.3389/fimmu.2019.00006 (  0) 0) |
| [35] |
KARN T, JIANG T T, HATZIS C, et al. Association between genomic metrics and immune infiltration in triple-negative breast cancer[J]. JAMA Oncology, 2017, 3(12): 1707-1711. DOI:10.1001/jamaoncol.2017.2140 (  0) 0) |
| [36] |
SAFONOV A, JIANG T T, BIANCHINI G, et al. Immune gene expression is associated with genomic aberrations in breast cancer[J]. Cancer Research, 2017, 77(12): 3317-3324. DOI:10.1158/0008-5472.CAN-16-3478 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


