2. 西安交通大学 生命科学与技术学院,生物医学信息工程教育部重点实验室,西安 710049;
3. 西安交通大学 医学部, 西安 710061;
4. 河北燕达陆道培医院,河北 廊坊 065201
2. The Key Laboratory of Biomedical Information Engineering of Ministry of Education, School of Life Sciences and Technology, Xi'an Jiaotong University, Xi'an 710049, China;
3. Health Science Center, Xi'an Jiaotong University, Xi'an 710061, China;
4. Hebei Yanda Lu Daopei Hospital, Langfang 065201, Hebei China
染色体是人类遗传物质(DNA)的载体,在人类体细胞中共有46条染色体,22对为与性别无关的常染色体,剩余的两条为性染色体,其中女性性染色体成对出现表现为XX,男性为XY[1]。任何的染色体异常都会导致很严重的疾病,如子代流产、死胎、新生儿死亡、先天畸形、智力低下、发育迟缓以及血液肿瘤疾病等[2]。染色体的异常包括数目异常和结构异常,分别是指整条染色体的增加或减少和染色体部分片段的断裂重组,这些异常的判断需要由训练有素的专业人员基于细胞分裂至中期的显微镜观察图,先进行染色体核型分析[3],再从核型中判断染色体是否异常,整个流程费时、费力。随着遗传病诊断需求的增加,染色体自动分类和异常诊断日渐重要,因此发展染色体核型的自动分析方法和异常染色体的自动识别方法来辅助临床诊断是非常有必要的。
染色体核型分析最为关键的一步是染色体分类,该任务实质上属于图像分类问题。早期的染色体分类问题,通过提取染色体的视觉特征,构建分类器进行分类,MARKOU[4]等通过提取染色体特征,构建支持向量机(SVM)分类器,染色体分类达到93.5%的准确度。自卷积神经网络(CNN)提出以来,利用CNN进行图像分类的研究越来越多,CNN结构来自HUBEL和WIESEL对猫的视觉皮层的建模[5],有着强大的特征提取功能,基于早期CNN的改进模型如AlexNet[6]、VGG[7]、ResNet[8]等一系列网络不断刷新了图像分类的准确度。目前已有一些研究将CNN方法应用于染色体分类:HU[9]等人构建了一个6层卷积层,3层池化层和4层随机失活层以及2个全连接层对染色体进行了24分类;SWATI[10]等人提出一种自动矫直染色体的方法后输入孪生网络对染色体进行24分类,实验表明比常规的CNN分类更高;SHARMA[11]采用众包预处理得到单条染色体,然后输入CNN网络进行分类。
对于染色体异常识别问题,WANG[12]等人结合染色体大小、平均灰度、标准差、着丝粒指数、最暗带指数以及最暗带比来识别22对染色体,然后运用模板匹配方法进行正常/异常分类,达到了93.3%的准确度;SAEID[13]等人根据形态特征(长度、长短臂比值)对染色体的异常进行了识别。
对于易位重组位点的自动识别问题,LEGEAND[14]等结合不同核型指定参考染色体密度谱后,采用动态时间规划方法(DTW)对染色体密度谱进行了易位重组位点识别。
但是,目前染色体分类和异常染色体易位重组位点的自动识别方法还存在一些不足。首先,现有的染色体类别识别往往都基于一个具体的分类网络进行,特征提取能力有限,因此本文针对染色体24分类,利用基于纹理特征的多通道模型进行特征提取,并和ResNet18基本模型进行模型融合,构建了整合的染色体分类网络。其次,大多异常染色体判别方法都是基于染色体的多个形态特征进行识别,然而当染色体发生易位时密度谱特征是变化最为明显的特征,因此本文采用密度谱作为异常染色体判别的主要特征,集成支持向量机、随机森林和XGBoost模型进行异常染色体判别。最后,异常染色体中易位重组位点的自动识别方法目前也较少有人研究,本文借助于DTW算法在判别序列相似度方面的优势,用于染色体密度谱序列进行易位重组位点的判别,并在临床数据中进行了验证。此外,本文通过以上的研究能够构建一套比较完整的易位重组位点的自动识别流程,有较强的现实意义和应用价值。
1 数据集介绍 1.1 数据来源数据来源于河北燕达陆道培医院,其中包含准确分类的1-22号常染色体、X、Y性染色体,共24类;异常染色体t(9;22) (q34;q11.2),即9号、22号易位染色体(t(9;22) (q34;q11.2)表示9号染色体3区4带和22号染色体1区1带发生易位)。所有数据来自显微镜拍摄的样本,染色体类型由专业医师确认。
1.2 染色体分类数据集该数据集包含了专业医师手动分割出的24类染色体,每一类染色体含有179张左右的染色体图片,24号染色体为男性性染色体仅有28张图片,共计4 108张,数据具体情况(见表 1),下载地址http://gr.xjtu.edu.cn/web/jianzu/useful-links。
| 表 1 24类染色体分类数据信息汇总 Table 1 Summary of data information for 24 classes of chromosomes |
该数据集包含了正常的9号染色体图片和发生易位的9号染色体图片(异常),其中正常染色体图片有61张图片,异常染色体有53张图片,数据量描述如表 2所示,下载地址http://gr.xjtu.edu.cn/web/jianzu/useful-links。
| 表 2 正常和异常9号染色体数据信息汇总 Table 2 Summary of data information for normal and abnormal chromosomes 9 |
本研究旨在建立一套自动识别染色体易位重组位点的方法流程。总体流程图(见图 1),首先,结合带有标签的24类染色体数据,构建染色体类别识别网络,提出基于纹理特征的多通道网络模型进行染色体24分类,再与ResNet18基本模型进行模型融合,预测染色体的类别。然后,对每一类染色体,构建染色体易位异常识别模型,结合带有正常和异常标签的9号染色体,基于染色体密度谱序列利用投票的方法集成支持向量机、随机森林和XGBoost分类模型,构建了染色体异常判别器。最后,基于识别出的异常染色体的密度谱,利用动态时间规划(DTW)算法计算出了发生易位的重组位点。本文接下来的3节分别对该框架的染色体类别识别、异常染色体判别和重组位点计算进行详细介绍。
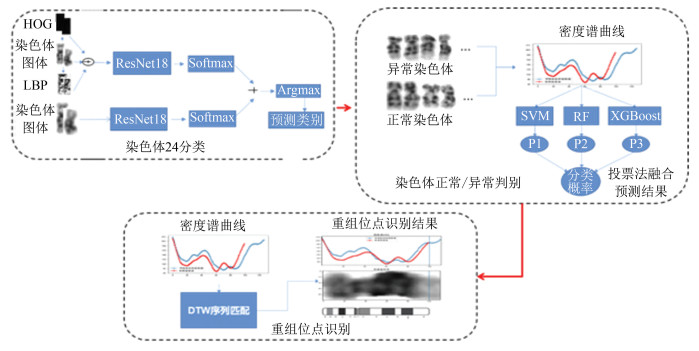
|
图 1 染色体重组位点自动识别总体流程图 Figure 1 Overall flow chart of automatic identification of chromosome recombination sites |
不同类别的染色体在长度、条带、长短臂比例等多方面有着较大的区别,在有限的数据集下训练卷积神经网络难以提取到深层的特征,如何充分利用现有数据成为关键。
一方面,通过随机旋转10度、随机中心裁剪、随机水平翻转进行数据增强,实验表明这有效地增强了分类的效果。另一方面,不仅仅直接使用统一维度(224×224)的原始图像数据训练了ResNet18基础网络模型,还通过对染色体图像进行预处理,提取了纹理特征,得到方向梯度直方图特征(HOG)和局部二值模式特征(LBP),并同时输入ResNet18网络模型,训练了多通道网络模型。相比ResNet18基础网络模型,基于纹理特征的多通道网络模型更注重染色体条带间的灰度变化信息,而ResNet18基础网络模型由于没有直接利用提取好的纹理特征,会更注重染色体的轮廓信息等。
最后,在现有数据上进行了24分类的测试。由于数据有限,我们对ResNet18基础网络模型和基于纹理特征的多通道网络模型都进行了4折交叉验证,最后把训练完成的两个模型进行融合,预测染色体的类别。本文设计的分类流程如(见图 2)。
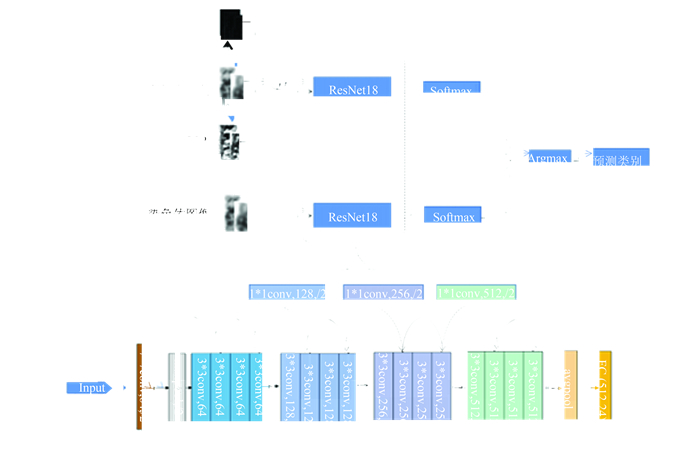
|
图 2 染色体24分类流程图 Figure 2 Flow chart of chromosome 24 classification |
首先,将原始图片转化为224×224,通过LBP映射、HOG映射预处理,得到LBP特征图、HOG特征图。然后,再把LBP特征图、HOG特征图和原始染色体图像组合为3×224×224的多通道输入。
具体的多通道输入模型为经过调整的ResNet18模型,将最后一层调整为一个(512,24)的全连接层以适应我们的分类任务。同时,训练了维度为224×224单通道原始图像作为输入的ResNet18基础网络模型。2个模型都采用了部分ImageNet预训练的参数进行初始化,并利用数据增强和4折交叉验证以增强分类的效果。
3.2 模型融合对ResNet18基础网络模型和基于纹理特征的多通道网络模型分别进行4折交叉验证,可以分别得到4种不同训练集、验证集划分,相当于2个子模型一共训练了8种情形。这8种情形在同一个测试集上可以得到共8个特征图,每个特征图的维度为训练批次数目×训练批次大小×24,最后一个维度对应了标签的独热编码。
模型融合的方式是把2个子模型得到的共8个特征图在最后一个维度相加(与取平均等价),得到的结果取最大值,得到输出的预测标签,与真实标签比对,最终得到分类准确率。
3.3 损失函数本文讨论的染色体类别识别问题是一个24分类问题,因此可以直接使用基本的多类别交叉熵损失。对于样本数据集(X, Y)={(xi, yi)}, yi为真实标签, 现在有24个类别标签,第i个样本第k个标签的预测概率记为pi, k,则该多分类网络的损失函数为:
| $ L\left( {X, Y, P} \right) = - \frac{1}{N}\sum\limits_{i = 1}^N {\sum\limits_{k = 1}^{24} {{y_{i, k}}} } \log \left( {{p_{i, k}}} \right). $ |
染色体密度谱是指染色体横截面处灰度值的均值构成的序列,其计算过程由算法1给出:
由于不同类别的染色体有着唯一的带状信息,因此密度谱序列能够直接反映染色体的带状信息。因此当染色体发生易位时,会直接反映在密度谱序列上,图 3展示了正常和异常染色体密度谱的序列图,红色曲线代表正常9号染色体的密度谱,蓝色曲线代表异常9号染色体密度谱。
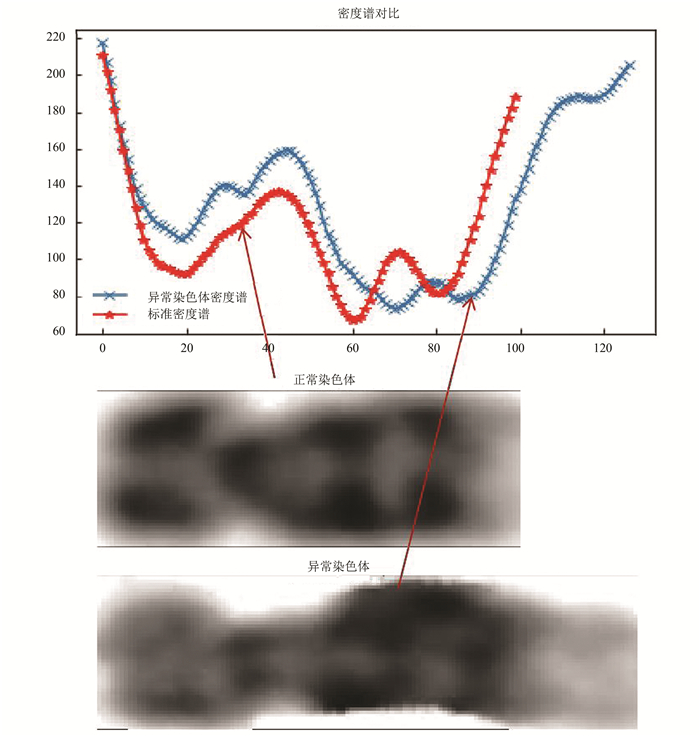
|
图 3 正常和异常9号染色体密度谱曲线图 Figure 3 Density profiles of normal and abnormal chromosomes 9 |
传统的染色体易位异常的判断是依靠专业人员进行判断,这种判别方式耗费人力和时间,因此本文基于带有标签的9号正常和异常染色体数据,构建分类器模型来自动判别正常和异常染色体。在训练集上分别训练支持向量机,随机森林和XGBoost分类器,在测试集上采用投票法对三种分类器的分类结果进行投票融合(见图 4),得到最终的正常/异常预测结果。
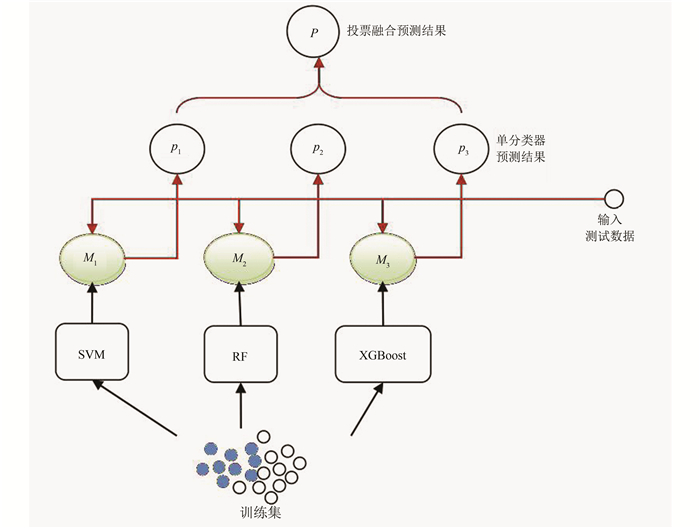
|
图 4 异常染色体集成识别器构建流程图 Figure 4 Flow chart of integrated identifier for abnormal chromosomes |
在识别出染色体类别并识别出异常染色体之后,进一步需要识别染色体发生易位后的重组位点。动态时间规划方法(DTW)是一种衡量不同长度序列之间相似度的方法,本文基于此方法进行重组位点的计算,基于动态时间规划方法(DTW)的易位重组位点识别算法由算法2给出:
通过第3,4,5节提出的方法,输入单张染色体图像即可识别染色体类别和判别其是否异常,并自动识别易位异常染色体的重组位点。
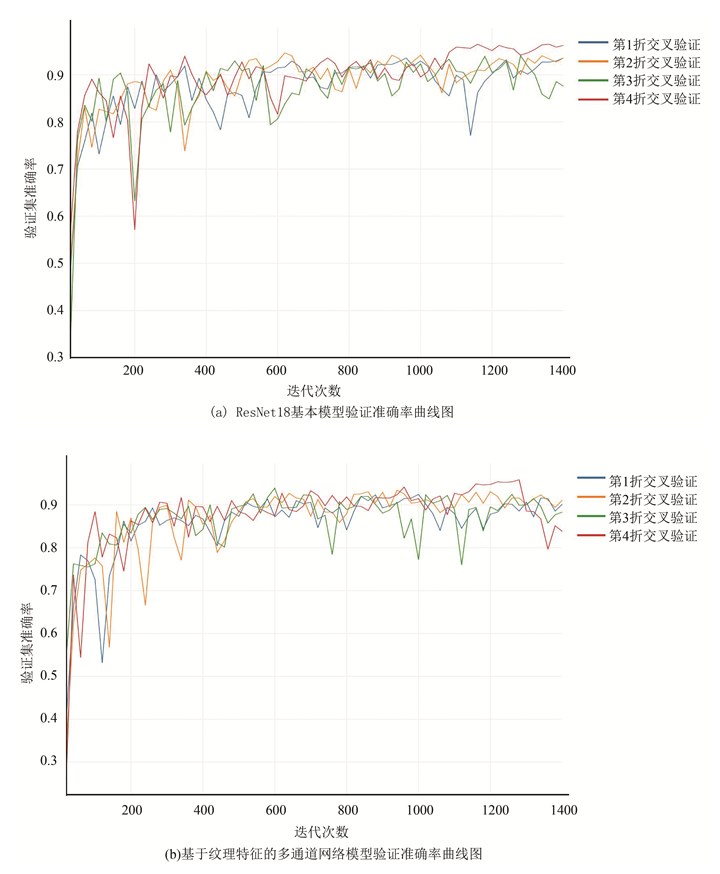
6 实验及结果 6.1 染色体24分类结果对收集到的4 108张染色体图像数据集按照8∶1∶1的比例进行训练集、验证集和测试集的划分。首先,通过3.1提出的染色体特征提取方法进行特征提取,网络参数由表 3给出,将得到的特征进行融合后进行全连接分类,得到准确率曲线(见图 5)。
| 表 3 染色体24分类网络参数说明 Table 3 Parameter description of chromosome 24 classification network |
|
图 5 染色体24分类模型准确率曲线图 Figure 5 Accuracy curves of chromosome 24 classification model |
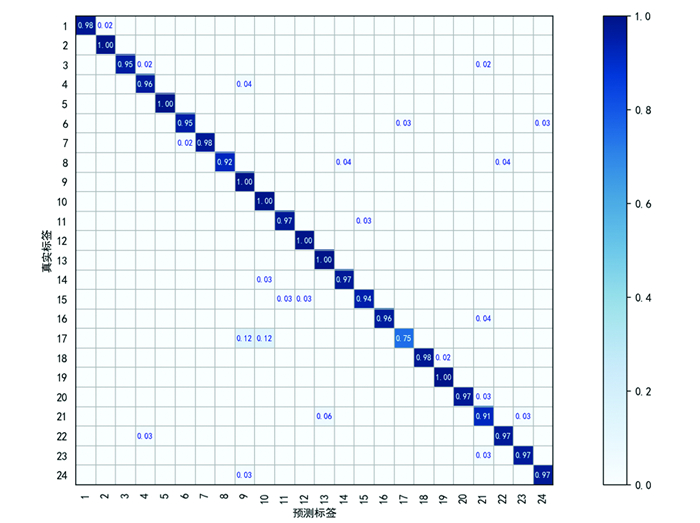
用测试集对ResNet18基本模型、基于纹理特征的多通道网络模型和两模型的融合模型进行测试,分类准确率分别达到了95.86%,95.99%和97.08%。换句话说,融合模型达到了更好的分类效果。为了分析融合分类模型对每一类染色体的分类情况,计算得到的混淆矩阵(见图 6),从混淆矩阵可以得到,有20类染色体的分类准确率均达到了95%及以上。只有第17类染色体的分类精度较低,说明对应的图像数据提取出的特征代表性还不够,会与9号和10号染色体混淆。特别地,对于23和24号染色体,即X、Y性染色体,尽管数据较少,但是用融合模型得到的分类准确率都达到了97%。
|
图 6 染色体24分类混淆矩阵 Figure 6 Confusion matrix of chromosome 24 classifications |
按照16∶4∶5的比例对115张正常和异常9号染色体数据集进行训练集、验证集和测试集的划分,采用公平投票的方式在测试集上对第4.2节得到的判别模型进行测试,正常和异常染色体判别准确率均达到了100%。这表明基于密度谱特征构建分类器对染色体易位异常进行判别是非常有效的。
6.3 重组位点识别结果首先,计算标准密度谱:使用专家确定的10条正常染色体,计算10条染色体的平均长度和宽度,为了统一标准,将10条染色体的长度和宽度规范化为平均长度100和宽度50,按照算法1计算密度谱,取该10条正常染色体密度谱的均值作为标准的参考密度谱A;
其次,计算异常染色体的密度谱:按照算法1计算待识别的异常染色体的密度谱序列B;
最后,计算易位位点: 按照算法2通过序列A和B计算易位重组位点。
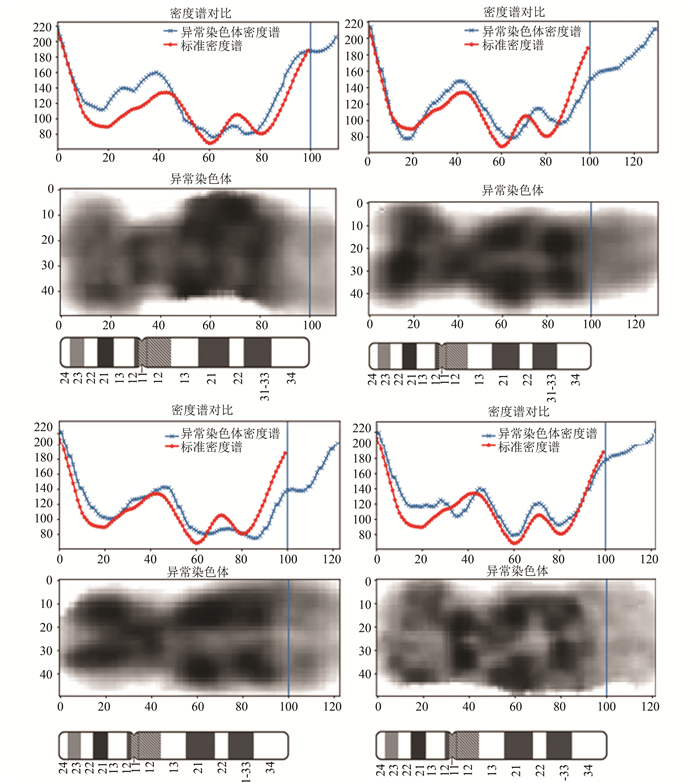
选择了27张异常的9号易位染色体进行易位重组位点识别测试,得到的部分识别结果(见图 7)。
|
图 7 易位重组位点识别结果图 Figure 7 Identification results of translocation recombination sites |
由图 7可以看出利用算法2可以准确定位出发生易位的位点。由于计算出的位点是基于图片像素位点,医学上将染色体的条带信息进行编号得到染色体G显带模式图,将计算的像素位点映射到染色体G显带模式图上,得到临床上的位点描述,可以得到发生易位的区域均在9号染色体的3区4带,这与临床医生根据原始数据的诊断结果一致,说明本算法可以较为准确的识别染色体易位的发生区域。
7 讨论染色体易位重组位点的自动精准识别对辅助临床诊断具有重要的应用价值。本文结合河北燕达陆道培医院采集到的24类染色体数据和9号正常与异常染色体数据,构建了一套自动识别染色体易位重组位点的模型和方法。对于任意给定的一对染色体图像,我们首先可以自动将其进行分类;如果是9号染色体,我们可以进一步判断它是正常染色体还是易位异常染色体;如果是易位异常染色体,我们还可以进一步自动判断其易位的重组位点,具有较高的准确率。本论文的主要创新点包括: 第一,通过将染色体灰度图像转换成密度谱曲线,将染色体密度谱曲线作为正常和易位异常染色体的分类特征,采用集成分类器的分类方法,使得正常和异常染色体的分类准确率达到了100%。第二,基于动态时间规划(DTW)的算法,我们设计了染色体易位重组位点的自动识别算法,在异常染色体的密度谱曲线上准确找到了重组位点,并映射至染色体G显带模式图上,得到的诊断结果与临床专家的诊断结果一致,相关方法也可以应用到其他类别异常染色体重组位点的识别。具体来讲,本文提出的模型和方法具有以下优点和可以进一步改进的地方:
首先,为了提取染色体更多的纹理特征,本文在ResNet18基本模型的基础上,提出了基于纹理特征的多通道网络模型和融合模型,进行了特定的特征提取和特征融合,同时辅以数据增强,有利于对染色体图像特征的提取,且分类准确度达到了较高的水平。从表 4可以看出,相比已有研究结果,我们提出的融合模型达到了较好的分类预测效果。但是由于临床数据不易获取,目前训练和测试数据的数量有限,数据在质量上也存在显著的类别间不平衡因素和图像质量的问题,随着不同类型染色体数据的不断收集和增强,以及更高级针对小样本数据的智能学习方法、稳健方法的应用,本文提出的分类模型将达到更高的准确率。
| 表 4 染色体分类结果比较 Table 4 Comparison of chromosome classification results |
其次,在染色体异常判别分析中,WANG[12]等人运用模板匹配方法进行正常/异常染色体分类,达到了93.3%的准确度。然而,我们基于密度谱曲线特征,采用集成的思想融合多个分类器,对9号正常和异常染色体的判别准确率达到了100%,取得了更好的效果。后续可以继续收集22号或者其他类别异常染色体的数据信息,可将此集成分类器应用于22号或其他类别染色体的异常识别。
最后,本文基于动态时间规划方法(DTW)对9号易位染色体进行了易位重组位点的识别,能准确的定位到易位发生的区域,并通过与临床专家诊断的结果进行比较,验证了算法的有效性和可信度。相关识别方法可推广至22号或者其他类别染色体易位点的识别。但是由于图像质量的影响,有少数的易位重组位点识别不准确,因此对于染色体图像质量的增强、评估是一个很好的改进方向。此外,构建一个正常染色体的标准密度谱曲线数据库,也将大大提高重组位点的识别准确率。
8 结论总的来讲,基于24类染色体灰度图像数据和9号正常与易位异常染色体数据,本文提出了一套自动识别易位染色体重组位点的方法流程。基于多网络提取特征和模型融合的染色体分类网络的设计,加强了染色体特征提取的能力,达到了较高的分类准确率。基于特定纹理特征融合的染色体分类方法在我们的测试数据上达到了97.08%的分类准确率;基于密度谱曲线的正常和异常染色体判别器在目前的数据集上达到了100%的分类准确率,说明利用密度谱这一特征进行染色体异常判别是非常有效的。对于异常染色体,基于动态时间规划方法(DTW)对重组位点进行了识别,计算出可能的重组位点并映射至染色体G显带模式图上,得出了临床直观的诊断结果,并由临床专家进行判断检验,提升了本文算法的可信度。本文提出的自动识别易位重组位点的方法流程具有较强的现实意义,有望完善和设计成一套软件系统,应用于临床实践。
| [1] |
TANVI A, RENU D. Geometric feature-based classification of segmented human chromosomes[J]. International Journal of Image and Graphics, 2020, 20(1): 2050006. DOI:10.1142/S0219467820500060 (  0) 0) |
| [2] |
张成成, 宋婕萍, 徐淑琴, 等. 基于深度卷积神经网络对中期染色体分类的应用于研究[J]. 中国临床新医学, 2020, 13(2): 123-124. Zhang Chengcheng, SONG Jieping, XU Shuqin, et al. Application of deep convolution neural network in metaphase chromosome classification[J]. New Clinical Medicine in China, 2020, 13(2): 123-124. DOI:10.3969/j.issn.1674-3806.2020.02.04 (  0) 0) |
| [3] |
NATARAJAN A T. Chromosome aberrations: Past, present and future[J]. Mutation Research, 2002, 504(1): 3-16. DOI:10.1016/S0027-5107(02)00075-1 (  0) 0) |
| [4] |
MARKOU C, MARAMIS C, DELOPOULOS A, et al. Automatic chromosome classi cation using support vector machines[R]. Thessaloniki: Aristotle University of Thessaloniki, 2012.
(  0) 0) |
| [5] |
HUBEL D H, WIESEL T N. Receptive fields of single neurones in the cat's striate cortex[J]. The Journal of Physiology, 1959, 148(3): 574-591. DOI:10.1113/jphysiol.1959.sp006308 (  0) 0) |
| [6] |
KRIZHEVSKY A, SUTSKEVER I, HINTON G. ImageNet classification with deep convolutional neural networks[J]. Neural Information Process Systems, 2012, 25: 1-9. DOI:10.1145/3065386 (  0) 0) |
| [7] |
SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition[C]//International Conference on Learning Representations (ICLR), 2015: 1-14. https://arxiv.org/abs/1409.1556.
(  0) 0) |
| [8] |
HE Kaiming, ZHANG Xiangyu, REN Shaoqing, et al. Deep residual learning for image recognition[C]//IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016: 770-774. DOI: 10.1109/CVPR.2016.90.
(  0) 0) |
| [9] |
HU Xi, YI Wenling, JIANG Ling, et al. Classification of metaphase chromosomes using deep convolutional neural network[J]. Journal of Computational Biology, 2019, 26(5): 473-480. DOI:10.1089/cmb.2018.0212 (  0) 0) |
| [10] |
SWAT I, GUPTA G, YADAV M, et al. Siamese networks for chromosome classification[C]//Proceedings of the IEEE International Conference on Computer Vision (ICCV). Venice: IEEE, 2017: 72-81. DOI: 10.1109/ICCVW.2017.17.
(  0) 0) |
| [11] |
SHARMA M, SAHA O, SRIRAMAN A, et al. Crowdsourcing for chromosome segmentation and deep classification[C]// Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition (CVPR) Workshops Honolulu: IEEE. 2017: 34-41. DOI: 10.1109/CVPRW.2017.109.
(  0) 0) |
| [12] |
WANG Xingwei, ZHENG Bin, LI Shibo, et al. Automated identification of abnormal metaphase chromosome cells for the detection of chronic myeloid leukemia using microscopic images[J]. Journal of Biomedical Optics, 2010, 15(4): 046026. DOI:10.1117/1.3476336 (  0) 0) |
| [13] |
SAEID A, HASSAN K, MAHLAGHA A, et al. Automatic identification of chromosomal abnormalities in metaphase karyotype using paired images in human chromosomes[C]//International Conference on Knowledge-Based Engineering and Innovation(KBEI), 2015: 762-769. DOI: 10.1109/KBEI.2015.7436140.
(  0) 0) |
| [14] |
LEGEAND B, CHANG C S, ONG S H, et al. Automated identification of chromosome segments involved in translocations by combining spectral karyotyping and banding analysis[J]. IEEE Transactions on Systems, 2008, 38(6): 1374-1384. DOI:10.1109/TSMCA.2008.2003963 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


