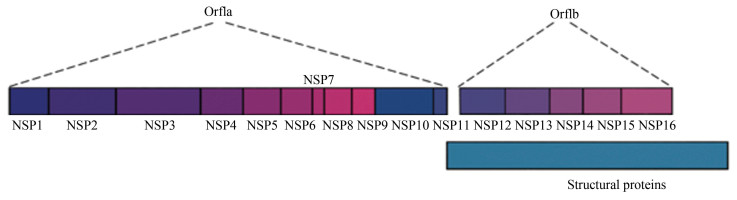
截至2020年9月15日,全球累计确诊新型冠状病毒肺炎病例29 455 791例,死亡932 961例,已感染全球229个国家或地区。SARS-CoV-2基因组3′末端的13个开放阅读框(Open reading frame, ORF)编码4种结构蛋白: 刺突蛋白(S,Spike Protein)、包膜蛋白(E,Envelope Protein)和膜蛋白(M,Membrane Protein)、核衣壳蛋白(N,Nucleocapsid Protein)。5′端的开放阅读框(ORF1a和ORF1b)编码多聚蛋白pp1a和pp1ab[1]。蛋白酶水解两种多聚蛋白后生成16种非结构蛋白(见图 1)[2-3]。NSP12是病毒RNA依赖的RNA聚合酶,(RNA-dependent RNA polymerases,RdRp)。NSP7和NSP8被证明是SARS-CoV-2 RNA转录和复制的核心元件[4]。
|
图 1 SARS-CoV-2的基因组特征 Figure 1 Genome structure of SARS-CoV-2 |
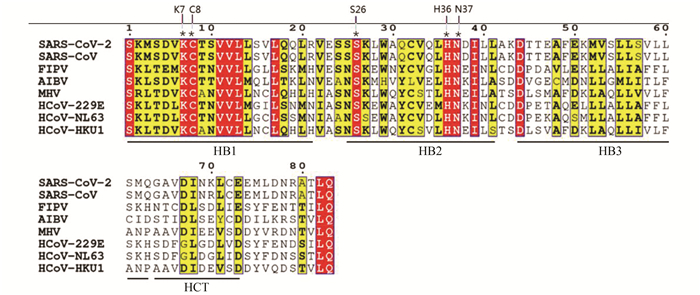
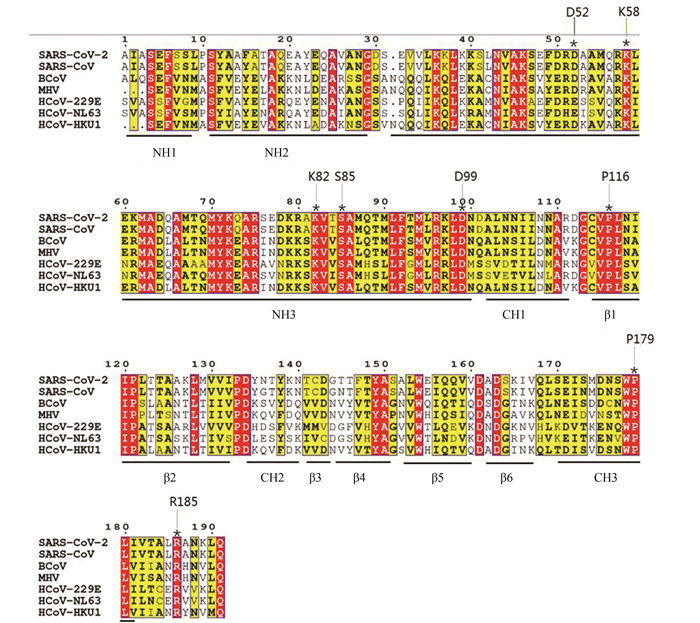
SARS-CoV-2基因组序列来自NCBI(参考序列号:NC_045512.2)。使用SARS-CoV-2的NSP7和NSP8氨基酸序列在冠状病毒家族中(SARS-CoV,猫传染性腹膜炎病毒(Feline Infectious Peritonitis Virus, FIPV),冠状病毒(Bovine Coronavirus,BCoV),禽传染性支气管炎病毒(Avian Infectious Bronchitis, IBV),鼠肝炎病毒(Mouse Hepatitis Virus, MHV),人冠状病毒229E(Human Coronavirus 229E, HCoV-229E),人冠状病毒NL63(Human Coronavirus NL63, HCoV-NL63),人冠状病毒HKU1(Human Coronavirus HKU1, HCoV-HKU1)进行比对。将比对结果提交至http://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi[5](见图 2、图 3)。
|
图 2 冠状病毒家族NSP7保守性分析 Figure 2 Conservative analysis of NSP7 in the coronavirus family 注:N端螺旋束(HB,N-terminal helical bundle),短螺旋(HCT)。RNA依赖的RNA聚合酶活性位点(K7, N37)。引物依赖性聚合酶活性位点(H36)。NSP7-NSP8-NSP12复合体结合RNA的活性位点(C8, S26)。 |
|
图 3 冠状病毒家族NSP8保守性分析 Figure 3 Conservative analysis of NSP8 in the coronavirus family 注:NH1-3(螺旋),CH1-3(α螺旋),β1-7(β链),RNA聚合酶活性位点(D99,P116,P179,R185),RNA结合位点(K58),体外NSP8正常功能活性位点(D52, K82, S85)。 |
新型冠状肺炎病毒的NSP7氨基酸序列与SARS-CoV的极为相似。仅70位精氨酸突变为赖氨酸。精氨酸和赖氨酸均为分子量相似带正电碱性氨基酸。该位点突变不会影响蛋白结构及功能。
NSP8的氨基酸序列比对结果见图 3。15位的氨基酸由酪氨酸突变为苯丙氨酸,理化性质相近。132位的氨基酸由缬氨酸突变为异亮氨酸,它们分子量和等电点相差不大且均为脂肪族疏水性氨基酸。173位氨基酸由天冬酰胺突变为丝氨酸,它们分子量和等电点相近,均为极性氨基酸。
1 SARS-CoV-2 NSP7和NSP8的结构特征NSP7带有N端螺旋束(N-terminal Helical Bundle, HB):HB1(1-21),HB2(25-42),HB3(44-63),HCT(65-73)(见图 2)。HB1,HB2,HB3通过疏水残基互作形成一个三链反平行螺旋束[6]。
NSP8含有N端的“轴”结构域(氨基酸1-100)和C端“头部”结构域(氨基酸102-181)[7](见图 3),且存在3个螺旋结构域(NH1,NH2,NH3),三个α螺旋(CH1,CH2,CH3),七个β链(β1-7)。SARS-CoV RNA依赖性RNA聚合酶的折叠结构与SARS-CoV NSP8的C端结构类似,这说明NSP8和NSP12有相似的功能。SARS-CoV的NSP8可识别冠状病毒单链RNA中的特定短序列[7],以低保真度催化最多6个核苷酸的合成。形成了NSP8-RNA-核苷酸的三元复合物后,就会在二核苷酸合成之前或合成时被“限速”。这与依赖DNA的RNA引发酶功能相似。NSP8使冠状病毒的复制更快、更准确,而不必依赖5’端的引物。
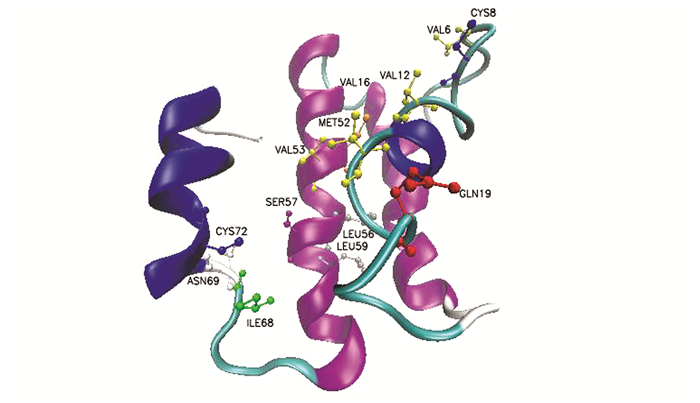
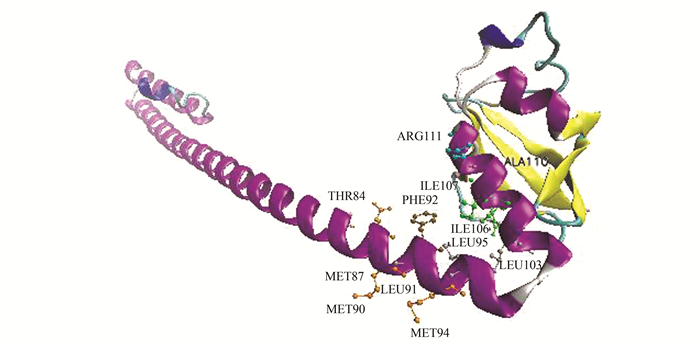
SARS-CoV和SARS-CoV-2 NSP7和NSP8的晶体结构高度相似[8]。在SWISS-MODEL以1ysy.1.A和6m5i.1作为模板分别对SARS-CoV的NSP7,NSP8氨基酸序列进行同源建模[9]。使用VMD分析结果[10](见图 4)。NSP8 NH螺旋的Met87,Met 90,Leu91,Met94,Leu103和NSP7 HB1螺旋的Val6,Cys8,Val12,Val16形成疏水核心[11]。NSP8的Thr84和NSP7的Gln19之间的侧链形成一个氢键。NSP7的HB3和HCT与NSP8的CH1相互作用, NSP8的Phe92,Leu95,Leu103,Ile106,IlE107和Ala110等残基与NSP7的Leu56,Leu59,Ile68形成侧键。NSP8 Arg111的侧链和主链分别与NSP7 Cys72的主链羰基和Asn69的侧链形成氢键。在NSP8 Ile119的主链酰胺基和NSP7 Ser57的侧链之间形成另一个氢键[12]。
|
图 4 SARS-CoV NSP7蛋白的三级结构 Figure 4 Tertiary structure prediction of SARS-CoV NSP7 protein |
SARS-CoV与SARS-CoV-2 NSP7和NSP8的氨基酸序列及蛋白空间构型高度相似,可推测两种非结构蛋白在SARS-CoV-2中可能具有相似的功能。NSP12是冠状病毒中RNA依赖性的RNA聚合酶,纯化的NSP12在体外拥有良好的催化活性[13]。NSP8能分别与NSP7和NSP12形成NSP7-NSP8和NSP8-NSP12复合物[14],但这两个复合物均无聚合酶活性。在相同温度下将NSP7和NSP8以1∶1混合,后向混合物中添加等量NSP12可检测到高RNA聚合酶活性[15]。这表明NSP7-NSP8复合物是激活RNA聚合酶活性的重要因素,可以协同激活NSP12的引物依赖性活性。即NSP7-NSP8-NSP12复合物具有完整的RNA聚合酶活性。
NSP7的K7和N37氨基酸位点改变使冠状病毒RNA依赖性RNA聚合酶完全失活,替换NSP7的H36使冠状病毒引物依赖性聚合酶活性减少62%[16]。K7,H36,N37突变使NSP7-NSP8-NSP12复合体无法结合RNA[17],表明NSP7在RNA结合中起至关重要的作用。NSP8的D99A,P116A,P183A,R190A突变后均会降低聚合酶活性[16]。
2.2 SARS-CoV的多种非结构蛋白可作为辅助因子结合NSP7-8-12复合物SARS-CoV的NSP14具有鸟嘌呤N7甲基转移酶活性,可以甲基化GTP,dGTP以及帽子类似物[18],这一过程对于病毒RNA的稳定性, 逃逸宿主先天免疫等过程都是至关重要的。N7甲基转移酶NSP14可结合NSP7-NSP8-NSP12复合物,且不影响复合物的活性[16]。NSP14还具有3’-5’的核酸外切酶活性[18]。NSP10能使NSP14的核糖核酸外切酶活性提高35倍[15]。NSP10-NSP14复合物在3’-5’方向水解双链RNA,并在3’方向切除单个错配的核苷酸。这表明NSP10和NSP14可能会作为辅蛋白增强SARS-CoV RNA聚合酶复合酶的稳定性和活性。
3 抗原表位与相关药物 3.1 SARS-CoV-2 NSP7和NSP8的抗原表位预测冠状病毒进入细胞后,pp1a和pp1ab蛋白水解,产生非结构蛋白。部分非结构蛋白诱导细胞膜重排,形成双膜囊泡或小泡结构。这也正是冠状病毒复制转录复合体的组装和锚定位点。在感染早期,NSP7、NSP8位于宿主核周区并形成病毒复制复合体,而在感染后期定位在膜蛋白聚集位点。
内源性抗原经过抗原提呈细胞(Antigen Presentation Cel1,APC)处理后,组织相容性复合体MHC I(Major Histocompatibility Complex Class I)将其递呈到细胞表面。人类的MHC通常称为HLA(Human Leucocyte Antigen,HLA)[19]。HLA复合体位于人第6对染色体的短臂上,共有HLA-A、HLA-B、HLA-C、HLA-DR、HLA-DQ、HLA-DP 6个位点。HLA-A2是HLA-A血清型组中的人白细胞抗原血清亚型。HLA-A2是最常见的HLA基因型,我国是HLA-A2的高阳性地区,约50%左右的人表达HLA-A2抗原[20]。表征引发适应性免疫应答的细胞表位是开发相关诊断和治疗工具的第一步。目前的软件算法如HLArestrictor[21]只是根据多肽与MHC的亲和力进行预测,但肽段与MHC结合的稳定性或抗原肽转运体(TAP)和内质网氨基肽酶基因(Endoplasmic Reticulum Aminopeptidase, ERAP)剪切同样会影响预测的肽段作为MHC分子配体的能力[22]。NetMHCpan-4.0能利用机器学习和IEDB数据库的MHC I数据进行筛选,使用亲和力(BA)和质谱洗脱配体的数据,通过人工神经网络的方法整合,整理出特定MHC分子结合肽段的亲和力值和肽段的长度[23]。NetMHCpan-4.0增加了T细胞免疫表位的预测准确性。
分子对接通过表位肽作为配体小分子和MHC I受体的相互作用预测其亲和力,能基于结构设计疫苗,整体考虑配体与受体的结合效果。本文使用NetMHCpan-4.0预测SARS-CoV-2所有的HLA-A2表位[24-25],利用筛选出的表位肽作为配体, PDB库中的MHC I作为受体,利用DINC进行分子对接和可视化[26]。计算预测得出的多肽与MHC I类分子的结合能,筛选出了高亲和力的表位。NSP7包含9个氨基酸的一条KLWAQCVQL配体,其结合亲和力13.6 nM(见表 1)。利用PDB模板2X4O(HLA-A2与HIV-1结合的晶体模型[27])作为受体进行分子对接后其结合能为-5.6 kcal/mol。这提示该多肽可与MHC I受体形成复合物并稳定存在。NSP8的多肽ALWEIQQV,ALWEIQQVV结合性强,分别以1HHI[28], 3TO2[29]为模板进行分子对接后结合能分别为-5.40 kcal/mol和-3.00 kcal/mol。这两条多肽均能与MHC I形成复合物并稳定存在。Grifoni Alba[30]发表的文章也提到这三条多肽可作为良好的CD8+抗原表位。白细胞抗原表位筛选出的多肽可为新冠肺炎疫苗设计提供帮助。
| 表 1 NSP7(上)和NSP8(下)的抗原多肽预测 Table 1 Antigenic peptide prediction of NSP7(up) and NSP8(down) |
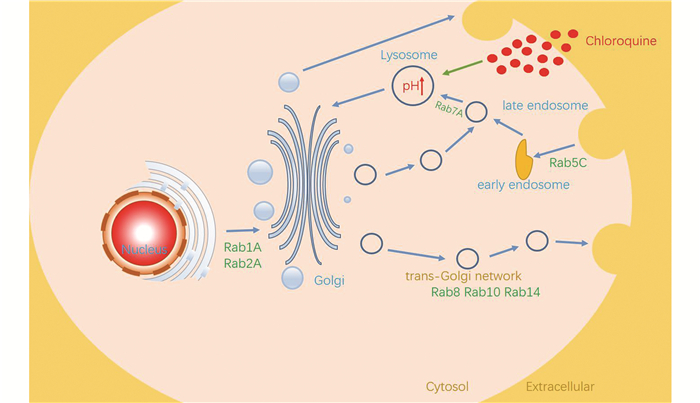
囊泡以芽生形成,囊泡内包裹被运输的蛋白质。当它到达靶细胞器时即与其融合,将蛋白质从一个细胞器运送到另一个细胞器。Rab是这一运输机制的关键调控因子,可作为“开关”调节囊泡的形成,运输以及融合[31]。Rab1A、Rab2A促进内质网(ER)迁移到高尔基体。Rab8A介导从反式高尔基体网络((Trans golgi network,TGN)到质膜的运输(见图 5)。Rab5C参与形成早期内体,是内体运输的必需分子。Rab7A介导晚期内体和溶酶体的融合。Rab8A、Rab10、Rab14调节反式高尔基体网络到细胞质膜的生物合成运输[32]。NSP7能与Rab家族的多个成员(Rab1A,Rab2A,Rab5C,Rab7A,Rab8A,Rab10,Rab14,Rab18)直接相互作用[33],调节病毒复制和病毒粒子的组装、转运。
|
图 5 SARS-CoV NSP8的蛋白三级结构 Figure 5 Tertiary structure prediction of SARS-CoV NSP8 protein |
刺突蛋白(S,Spike Protein)、包膜蛋白(E,Envelope Protein)和膜蛋白(M,Membrane protein)组成SARS-CoV-2的包膜结构。S蛋白在病毒包膜与宿主细胞膜融合的过程中起到关键性作用;M蛋白参与了病毒包膜的形成与出芽过程。抑制Rab7A可能会遏制病毒包膜蛋白的裂解,导致病毒颗粒感染能力降低[34],影响NSP7在冠状病毒的复制和出芽中的作用。
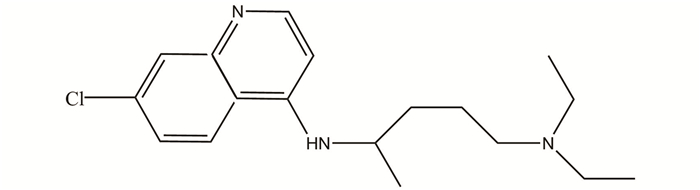
3.3 氯喹氯喹(见图 6,ChewDraw https://www.chemdraw.com.cn/)具有抗多种病毒的活性。可用于辅助治疗I型HIV,乙型肝炎病毒,HCoV-229E, SARS-CoV[35]。还具有免疫调节活性,能抑制肿瘤坏死因子和白介素6的释放[36]。
|
图 6 氯喹结构式 Figure 6 Chemical structure of chloroquine |
在感染前24小时添加药物,氯喹能有效预防SARS-CoV感染Vero E6细胞[37]。即使在病毒感染后3~5小时加入氯喹也有显著的抗病毒感染效果。一般认为氯喹通过升高反式高尔基体网络和内体的pH进而引起血管紧张素转化酶2末端糖基化受损,使SARS-CoV刺突蛋白与血管紧张素转化酶2结合能力降低[38]。
在细胞培养物中添加氯喹,氯喹会进入细胞发生质子化并聚集在低pH的细胞器(内体,高尔基囊泡和溶酶体)[35]。感染宿主后冠状病毒的出芽(Viral Budding)发生在pH=6.5的高尔基体中。pH=7.0的内质网(Endoplasmic Reticulum, ER)或内质网-高尔基体中间体会形成双膜囊泡(病毒的复制场所)[39]。SARS-CoV的NSP7和NSP7-NSP8复合物具有结构多态性,在不同pH环境(pH6.5与pH7.5)的构象不同[40]。而氯喹一般通过升高反式高尔基体网络以及溶酶体的pH发挥抗病毒作用(见图 7),这表明该药物可能升高pH改变NSP7和NSP7-NSP8复合物的空间构型,进而影响病毒RNA的复制和基因组装配过程。
|
图 7 氯喹和Rab家族在高尔基体网络中的作用 Figure 7 Function of chloroquine and Rab family in the Golgi network |
半最大效应浓度(Concentration For 50% Of Maximal Effect, EC50)指能引起50%最大效应的浓度,是评估药物安全的重要指标。氯喹/羟氯喹已在体外实验中显示出良好的抗病毒和预防病毒感染活性。一项研究指出,尽管细胞外氯喹/羟氯喹的EC50非常低,但由于这两种药物在细胞中具有富集效应。细胞内实际的氯喹/羟氯喹浓度远远高于细胞外的浓度,因此氯喹和羟氯喹真正的EC50非常高[41]。这意味着在患者可耐受范围内氯喹和羟氯喹的抗病毒作用不及体外作用。
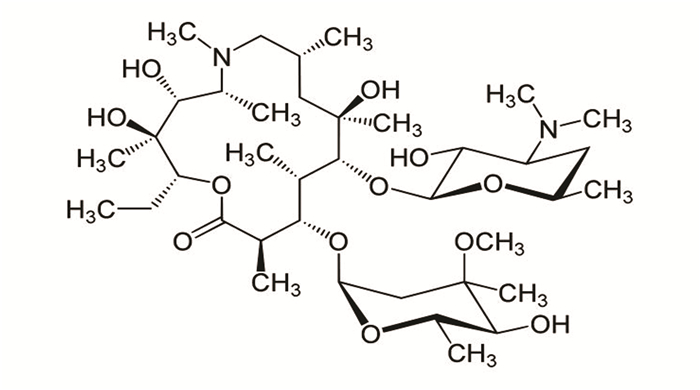
3.4 阿奇霉素线粒体28S核糖体蛋白负责将氨基酸残基添加到新生的多肽链。SARS-CoV-2的NSP8蛋白与宿主线粒体核糖体蛋白28S亚基(MRPS2,MRPS5,MRPS25,MRPS27)结合[42],这些亚基参与线粒体的翻译。阿奇霉素导致线粒体的空泡化和肿胀,破坏MCF-12A细胞和成纤维细胞线粒体內脊,使DNA氧化损伤产物8-羟基-2-脱氧鸟苷显著增加[43],引起线粒体毒性。阿奇霉素(见图 8,ChewDraw,https://www.chemdraw.com.cn/)还会与线粒体核糖体蛋白结合,抑制蛋白质转肽和核糖体亚基的组装阻止细胞的线粒体合成病毒蛋白[44]。
|
图 8 阿奇霉素结构式 Figure 8 Chemical structure of azithromycin |
已发现部分患者存在新冠肺炎和细菌型肺炎共感染的情况[45]。在新的治疗指南中已批准使用大环内酯类抗生素配合抗病毒药物进行治疗。美国胸科学会和传染病学会提出的治疗方案中建议阿奇霉素和克拉霉素联合用于治疗一线新冠肺炎患者[46]。阿奇霉素可能在新冠肺炎的辅助治疗中有效。
3.5 小分子抑制剂p97是最丰富的胞质ATP酶之一,在古细菌和真核生物中高度保守。p97是蛋白质内稳态的关键组成部分,影响膜融合与囊泡运输。p97的小分子抑制剂NMS-873能通过降低病毒蛋白的表达抑制甲型和乙型流感病毒的复制。Bojkova Denisa[47]测试了NMS-873对SARS-CoV-2复制的影响。NMS-873在低纳摩尔浓度下完全抑制SARS-CoV-2在细胞中的复制。NMS-873作用于囊泡运输过程可能抑制了NSP7对病毒复制、转运等功能的调节。
4 讨论冠状病毒家族成员中nsp7和nsp8序列高度保守,且氨基酸活性位点位点均未突变。NSP7和NSP8对于冠状病毒家族的RNA复制十分重要,其结构受到破坏会导致病毒RNA合成受严重影响。冠状病毒的非结构蛋白7和8可形成多种复合物调控多种功能。
阿奇霉素和氯喹有良好的体外抗病毒活性,宿主体内相互作用蛋白表明两种非结构蛋白可能与氯喹和阿奇霉素的药物机制有关。法国马赛医院[48]发表一项临床试验结果表明羟氯喹与阿奇霉素联合治疗可使COVID-19患者短时间转阴。Mandeep Mehra[49]于5月22日发表的一篇文章指出,氯喹或阿奇霉素会导致QTc间期的延长。QTc间期延长表示心脏复极延迟,反映了心电异常,通常与心律失常敏感性增高密切相关。氯喹和阿奇霉素具有钠通道阻滞特性,可能在COVID-19中存在心肌损伤和缺氧的情况下促成心律失常和心力衰竭[50]。由于研究人员无法排除未观察到的混杂因素对患者预后的影响,这个假设还有待检验。任何治疗COVID-19患者的药物都需要进一步证明其有效性和安全性。
| [1] |
PARK S E. Epidemiology, virology, and clinical features of severe acute respiratory syndrome-coronavirus-2 (SARS-CoV-2; Coronavirus Disease-19)[J]. Clinical and Experimental Pediatrics, 2020, 63(4): 119-124. DOI:10.3345/cep.2020.00493 (  0) 0) |
| [2] |
BRIERLEY I, DIGARD P. Characterization of an efficient coronavirus ribosomal frameshifting signal: Requirement for an RNA pseudoknot[J]. Cell, 1989, 57(4): 537-547. DOI:10.1016/0092-8674(89)90124-4 (  0) 0) |
| [3] |
ZHU N, ZHANG D, WANG W, et al. A novel coronavirus from patients with pneumonia in China, 2019[J]. New England Journal of Medicine, 2020, 382(8): 727-733. DOI:10.1056/NEJMoa2001017 (  0) 0) |
| [4] |
GAO Y, YAN L, HUANG Y, et al. Structure of the RNA-dependent RNA polymerase from COVID-19 virus[J]. Science, 2020, 368(6492): 779-782. DOI:10.1126/science.abb7498 (  0) 0) |
| [5] |
ROBERT X, GOUET P. Deciphering key features in protein structures with the new ENDscript server[J]. Nucleic Acids Research, 2014, 42: W320-W324. DOI:10.1093/nar/gku316 (  0) 0) |
| [6] |
LU S, WANG J, CHITSAZ F, et al. CDD/SPARCLE: the conserved domain database in 2020[J]. Nucleic Acids Research, 2020, 48(D1): D265-D268. DOI:10.1093/nar/gkz991 (  0) 0) |
| [7] |
IMBERT I, GUILLEMOT J C, BOURHIS J M, et al. A second, non-canonical RNA-dependent RNA polymerase in SARS coronavirus[J]. The EMBO Journal, 2006, 25(20): 4933-4942. DOI:10.1038/sj.emboj.7601368 (  0) 0) |
| [8] |
PENG Q, PENG R, YUAN B, et al. Structural and biochemical characterization of the nsp12-nsp7-nsp8 core polymerase complex from SARS-CoV-2[J]. Cell Reports, 2020, 31(11): 107774. DOI:10.1016/j.celrep.2020.107774 (  0) 0) |
| [9] |
SCHWEDE T, KOPP J, GUEX N, et al. SWISS-MODEL: An automated protein homology-modeling server[J]. Nucleic Acids Research, 2003, 31(13): 3381-3385. DOI:10.1093/nar/gkg520 (  0) 0) |
| [10] |
HUMPHREY W, DALKE A, SCHULTEN K. VMD: Visual molecular dynamics[J]. Journal of Molecular Graphics and Modelling, 1996, 14(1): 33-38, 27-38. DOI:10.1016/0263-7855(96)00018-5 (  0) 0) |
| [11] |
TE VELTHUIS A J, VAN DEN WORM S H, SNIJDER E J. The SARS-coronavirus nsp7+nsp8 complex is a unique multimeric RNA polymerase capable of both de novo initiation and primer extension[J]. Nucleic Acids Research, 2012, 40(4): 1737-1747. DOI:10.1093/nar/gkr893 (  0) 0) |
| [12] |
ZHAI Y, SUN F, LI X, et al. Insights into SARS-CoV transcription and replication from the structure of the NSP7-NSP8 hexadecamer[J]. Nature Structural & Molecular Biology, 2005, 12(11): 980-986. DOI:10.1038/nsmb999 (  0) 0) |
| [13] |
TE VELTHUIS A J, ARNOLD J J, CAMERON C E, et al. The RNA polymerase activity of SARS-coronavirus nsp12 is primer dependent[J]. Nucleic Acids Research, 2010, 38(1): 203-214. DOI:10.1093/nar/gkp904 (  0) 0) |
| [14] |
KRICHEL B, FALKE S, HILGENFELD R, et al. Processing of the SARS-CoV pp1a/ab nsp7-10 region[J]. The Biochemical Journal, 2020, 477(5): 1009-1019. DOI:10.1042/bcj20200029 (  0) 0) |
| [15] |
BOUVET M, IMBERT I, SUBISSI L, et al. RNA 3'-end mismatch excision by the severe acute respiratory syndrome coronavirus nonstructural protein nsp10/nsp14 exoribonuclease complex[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(24): 9372-9377. DOI:10.1073/pnas.1201130109 (  0) 0) |
| [16] |
SUBISSI L, POSTHUMA C C, COLLET A, et al. One severe acute respiratory syndrome coronavirus protein complex integrates processive RNA polymerase and exonuclease activities[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(37): E3900-E3909. DOI:10.1073/pnas.1323705111 (  0) 0) |
| [17] |
VAN DIJK A A, MAKEYEV E V, BAMFORD D H. Initiation of viral RNA-dependent RNA polymerization[J]. Journal of General Virology, 2004, 85(Pt 5): 1077-1093. DOI:10.1099/vir.0.19731-0 (  0) 0) |
| [18] |
金旭. 冠状病毒N7甲基转移酶甲基化核苷酸GTP的特性研究[D]. 武汉: 武汉大学, 2013. JIN Xu. Study on the characteristics of the methylated nucleotide GTP of coronavirus N7 methyltransferase[D]. Wuhan: Wuhan University, 2013. (  0) 0) |
| [19] |
TAYLOR C J, BOLTON E M, BRADLEY J A. Immunological considerations for embryonic and induced pluripotent stem cell banking[J]. Philosophical Transactions of the Royal Society of London B Biological Sciences, 2011, 366(1575): 2312-2322. DOI:10.1098/rstb.2011.0030 (  0) 0) |
| [20] |
侯亚非, 孙宗棠, 曲春枫, 等. 北方汉族人群HLA-A2亚型分布及p53的合成肽体外诱导CTL反应[J]. 中华微生物学和免疫学杂志, 1999, 19(1): 47-50. HOU Yafei, SUN Zongtang, QU Chunfeng, et al. Distribution of HLA-A2 subtypes in northern Han population and p53 synthetic peptides induce CTL response in vitro[J]. Chinese Journal of Microbiology and Immunology, 1999, 19(1): 47-50. DOI:10.3760/j:issn:0254-5101.1999.01.018 (  0) 0) |
| [21] |
ERUP LARSEN M, KLOVERPRIS H, A STRYHN, et al. HLArestrictor-a tool for patient-specific predictions of HLA restriction elements and optimal epitopes within peptides[J]. Immunogenetics, 2011, 63(1): 43-55. DOI:10.1007/s00251-010-0493-5 (  0) 0) |
| [22] |
TROLLE T, MCMURTREY C P, SIDNEY J, et al. The length distribution of class i-restricted t cell epitopes is determined by both peptide supply and mhc allele-specific binding preference[J]. Journal of Immunology (Baltimore, Md. : 1950), 2016, 196(4): 1480-1487. DOI:10.4049/jimmunol.1501721 (  0) 0) |
| [23] |
REYNISSON B, ALVAREZ B, PAUL S, et al. NetMHCpan-4.1 and NetMHCⅡpan-4.0: Improved predictions of MHC antigen presentation by concurrent motif deconvolution and integration of MS MHC eluted ligand data[J]. Nucleic Acids Research, 2020, 48: W449-W454. DOI:10.1093/nar/gkaa379 (  0) 0) |
| [24] |
NERLI S, SGOURAKIS N G. Structure-based modeling of SARS-CoV-2 peptide/HLA-A02 antigens[J/OL]. http://www.researchgate.net/publication/340244204.2020.3. DOI: 10.1101/2020.03.23.004176.
(  0) 0) |
| [25] |
JURTZ V, PAUL S, ANDREATTA M, et al. NetMHCpan-4.0: Improved Peptide-MHC Class I Interaction Predictions Integrating Eluted Ligand and Peptide Binding Affinity Data[J]. The Journal of Immunology, 2017, 199(9): 3360-3368. DOI:10.4049/jimmunol.1700893 (  0) 0) |
| [26] |
ANTUNES D A, MOLL M, DEVAURS D, et al. DINC 2.0: A new protein-peptide docking webserver using an incremental approach[J]. Cancer Research, 2017, 77(21): e55-e57. DOI:10.1158/0008-5472.can-17-0511 (  0) 0) |
| [27] |
CELIE P H, TOEBES M, RODENKO B, et al. UV-induced ligand exchange in MHC class I protein crystals[J]. Journal of the American Chemical Society, 2009, 131(34): 12298-12304. DOI:10.1021/ja9037559 (  0) 0) |
| [28] |
MADDEN D R, GARBOCZI D N, WILEY D C. The antigenic identity of peptide-MHC complexes: A comparison of the conformations of five viral peptides presented by HLA-A2[J]. Cell, 1993, 75(4): 693-708. DOI:10.1016/0092-8674(93)90490-h (  0) 0) |
| [29] |
LIU J, QI J, GAO F, et al. Functional and structural definition of a clustering region of HLA-a2-restricted cytotoxic t lymphocyte epitopes[J]. Science & Technology Review, 2011, 29: 19-26. DOI:10.2210/pdb3TO2/pdb (  0) 0) |
| [30] |
GRIFONI A, SIDNEY, ZHANG Y, et al. A sequence homology and bioinformatic approach can predict candidate targets for immune responses to SARS-CoV-2[J]. Cell Host & Microbe, 2020, 27(4): 671-680.e672. DOI:10.1016/j.chom.2020.03.002 (  0) 0) |
| [31] |
HUTAGALUNG A H, NOVICK P J. Role of Rab GTPases in membrane traffic and cell physiology[J]. Physiol Review, 2011, 91(1): 119-149. DOI:10.1152/physrev.00059.2009 (  0) 0) |
| [32] |
SHI M M, SHI C H, XU Y M. Rab GTPases: The key players in the molecular pathway of parkinson's disease[J]. Frontiers in Cellular Neuroscience, 2017, 11: 81. DOI:10.3389/fncel.2017.00081 (  0) 0) |
| [33] |
GORDON D E, JANG G M, BOUHADDOU M, et al. A SARS-CoV-2 protein interaction map reveals targets for drug repurposing[J]. Nature, 2020, 583(7816): 1-13. DOI:10.1038/s41586-020-2286-9 (  0) 0) |
| [34] |
CAILLET M, JANVIER K, PELCHEN-MATTHEWS A, et al. Rab7A is required for efficient production of infectious HIV-1[J]. PLoS Pathogens, 2011, 7(11): e1002347. DOI:10.1371/journal.ppat.1002347 (  0) 0) |
| [35] |
KEYAERTS E, VIJGEN L, MAES P, et al. In vitro inhibition of severe acute respiratory syndrome coronavirus by chloroquine[J]. Biochemical and Biophysical Research Communications, 2004, 323(1): 264-268. DOI:10.1016/j.bbrc.2004.08.085 (  0) 0) |
| [36] |
ZHAO M. Cytokine storm and immunomodulatory therapy in COVID-19: Role of chloroquine and anti-IL-6 monoclonal antibodies[J]. International Journal of Antimicrobial Agents, 2020, 55(6): 105982. DOI:10.1016/j.ijantimicag.2020.105982 (  0) 0) |
| [37] |
VINCENT M J, BERGERON E, BENJANNET S, et al. Chloroquine is a potent inhibitor of SARS coronavirus infection and spread[J]. Virology Journal, 2005, 2: 69. DOI:10.1186/1743-422x-2-69 (  0) 0) |
| [38] |
IMBERT I, SNIJDER E J, DIMITROVA M, et al. The SARS-Coronavirus PLnc domain of nsp3 as a replication/transcription scaffolding protein[J]. Virus Research, 2008, 133(2): 136-148. DOI:10.1016/j.virusres.2007.11.017 (  0) 0) |
| [39] |
STERTZ S, REICHELT M, SPIEGEL M, et al. The intracellular sites of early replication and budding of SARS-coronavirus[J]. Virology, 2007, 361(2): 304-315. DOI:10.1016/j.virol.2006.11.027 (  0) 0) |
| [40] |
LI S, ZHAO Q, ZHANG Y, et al. New nsp8 isoform suggests mechanism for tuning viral RNA synthesis[J]. Protein Cell, 2010, 1(2): 198-204. DOI:10.1007/s13238-010-0028-8 (  0) 0) |
| [41] |
FAN J, ZHANG X, LIU J, et al. Connecting hydroxychloroquine in vitro antiviral activity to in vivo concentration for prediction of antiviral effect: a critical step in treating COVID-19 patients[J]. Clinical Infectious Diseases: An Official Publication of the Infectious Diseases Society of America, 2020, 71(12): 3232-3236. DOI:10.1093/cid/ciaa623 (  0) 0) |
| [42] |
GORDON D E, JANG G M, BOUHADDOU M, et al. A SARS-CoV-2 protein interaction map reveals targets for drug repurposing[J]. Nature, 2020, 583(7816): 459-468. DOI:10.1038/s41586-020-2286-9 (  0) 0) |
| [43] |
JIANG X, BAUCOM C, ELLIOTT R L. Mitochondrial toxicity of azithromycin results in aerobic glycolysis and DNA damage of human mammary epithelia and fibroblasts[J]. Antibiotics (Basel), 2019, 8(3): 110-127. DOI:10.3390/antibiotics8030110 (  0) 0) |
| [44] |
PETERS D H, FRIEDEL H A, MCTAVISH D. Azithromycin: A review of its antimicrobial activity, pharmacokinetic properties and clinical efficacy[J]. Drugs, 1992, 44(5): 750-799. DOI:10.2165/00003495-199244050-00007 (  0) 0) |
| [45] |
ZHOU F, YU T, DU R, et al. Clinical course and risk factors for mortality of adult inpatients with COVID-19 in Wuhan, China: A retrospective cohort study[J]. Lancet, 2020, 395(10229): 1054-1062. DOI:10.1016/S0140-6736(20)30566-3 (  0) 0) |
| [46] |
METLAY J P, WATERER G W. Treatment of community-acquired pneumonia during the coronavirus disease 2019 (COVID-19) pandemic[J]. Annals of Internal Medicine, 2020, 173(4): 304-305. DOI:10.7326/M20-2189 (  0) 0) |
| [47] |
BOJKOVA D, KLANN K, KOCH B, et al. Proteomics of SARS-CoV-2-infected host cells reveals therapy targets[J]. Nature, 2020, 583: 469-472. DOI:10.1038/s41586-020-2332-7 (  0) 0) |
| [48] |
GAUTRET P, LAGIER J C, PAROLA P, et al. Hydroxychloroquine and azithromycin as a treatment of COVID-19: Results of an open-label non-randomized clinical trial[J]. International Journal of Antimicrobial Agents, 2020, 56(1): 105949. DOI:10.1016/j.ijantimicag.2020.105949 (  0) 0) |
| [49] |
MEHRA M R, DESAI S S, RUSCHITZKA F, et al.Hydroxychloroquine or chloroquine with or without a macrolide for treatment of COVID-19: a multinational registry analysis[J/OL]. http://doi.org/10.1016/s0140-6736(20)31180-6.2020.05.22. DOI: 10.1016/s0140-6736(20)31180-6.
(  0) 0) |
| [50] |
GUO T, FAN Y, CHEN M, et al. Cardiovascular implications of fatal outcomes of patients with coronavirus disease 2019 (COVID-19)[J]. JAMA Cardiology, 2020, 5(7): E1-E8. DOI:10.1001/jamacardio.2020.1017 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


