细胞自噬是存在于真核生物中进化上的一种保守的分解代谢过程,又被称做“II型细胞死亡”其过程主要是将受损的细胞器和蛋白质输送至溶酶体进行降解[1]。因此它对控制细胞内蛋白质和细胞器的质量和数量方面具有重要作用。自噬的失调会引起多种疾病的发生,包括神经退行性疾病、肿瘤、免疫性疾病及感染性疾病等[2-4]。在肿瘤疾病中,自噬通过清除受损的细胞器和异常表达的蛋白质从而减轻细胞的损伤,抑制肿瘤的早期发生发展[5]。然而也有研究表明,自噬使肿瘤细胞具有了有更加良好的应激耐受性,提高了肿瘤细胞的生存能力,并促进了其再生[6]。在化疗过程中抑制自噬可以促进肿瘤细胞的死亡,降低耐药性[7]。尽管有越来越多的文献报道了自噬与肿瘤的关系,但对于二者之间的具体的关系仍旧没有定论,进一步研究自噬与肿瘤的关系有其必要性。近年来,自噬相关基因预测乳腺癌、肝癌、膀胱癌患者的临床预后得到了证实[8-10],而对自噬与神经胶质母细胞瘤的相关性仅有少量文献报道。
胶质母细胞瘤(GBM)是成人恶性脑肿瘤中最常见的肿瘤。病理分级为IV级,是世界卫生组织(WHO)脑肿瘤分类的最高等级。即便GBM患者术后进行标准化疗和辅助放疗,其中位生存时间仍然只有12~15个月[11],针对其较低的生存时间,越来越多医师探讨GBM的治疗方法,比如通过识别在实体瘤的肿瘤细胞和肿瘤微环境的主要成分中广泛表达的CD155,使用重组脊髓灰质炎病毒治疗胶质母细胞瘤[12],利用嵌合抗原受体(CAR)改造的T细胞,靶向肿瘤相关抗原白细胞介素13受体α2(IL13Rα2)[13]。但是这些方法目前并不具备向临床推广的能力,其临床有效性也仅在少数患者身上证实。近期有文献报道了细胞自噬与胶质瘤之间的关系,提示自噬可能是胶质瘤发生发展的中必不可少的机制之一。一项III期临床试验显示,唯一经美国食品药品监督管理局(FDA)批准的且能够抑制自噬的抗疟疾药物氯喹在GBM中具有抗肿瘤作用,其患者在手术、化疗和放疗后,中位生存期均有所增加[14]。通过药理抑制剂抑制FH535抑制WNT-CTNNB1信号使得SQSTM1表达上调,可增加GBM中的自噬通量,从而诱导GBM细胞凋亡[15]。但大多数对于自噬在肿瘤发生作用的研究都是基于细胞系或者动物模型中的有限的自噬相关基因,自噬相关基因对于人体影响的预后价值尚未在GBM中得以体现。本研究基于多个数据库综合分析了自噬基因在GBM患者中的临床病理相关性及其预后价值。
1 资料与方法 1.1 资料来源从人类自噬数据库HADb(http://autophagy.lu/clustering/index.html.)获得了232个自噬相关基因。从癌症基因组图谱(TCGA)获取169个GBM样本,5个正常样本数据,包含了基因表达数据。在UCSC(https://xenabrowser.net)中下载了对应的临床数据。使用GEPIA网站(http://gepia.cancer-pku.cn/index.html)中的数据(207个GBM样本,163个正常样本)及CGGA数据库(http://www.cgga.org.cn/index.jsp)中的mRNA325数据集(Ⅱ级胶质瘤样本103个,Ⅲ级胶质瘤样本79个,GBM样本139个)进行验证和分析。
1.2 统计学方法差异基因的筛选方法为limma包(版本1.30.4)中的Wilcoxon秩和检验,GO、KEGG分析使用clusterProfiler包(版本3.10.1),KM分析及COX多因素分析均使用SPSS22.0(IBM Crop.)完成,COX分析使用的方法为Forward:LR, 使用GraphPad Prism 8(GraphPad Software,Inc.)生成图形。通过survivalROC(版本1.0.3)完成随时间依赖性的ROC分析。临床相关性分析使用beeswarm包(版本0.2.3)完成。GSEA分析使用软件GSEA4.03(http://software.broadinstitute.org/gsea/index.jsp),自噬基因相关图基因来源于ARN(http://arn.elte.hu/),数据分析使用的R软件版本为R3.53。
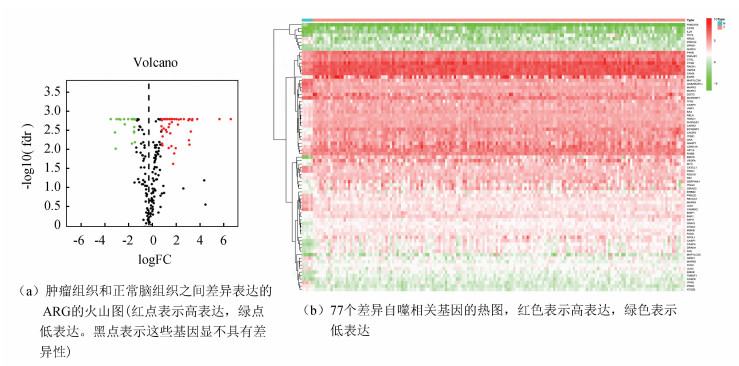
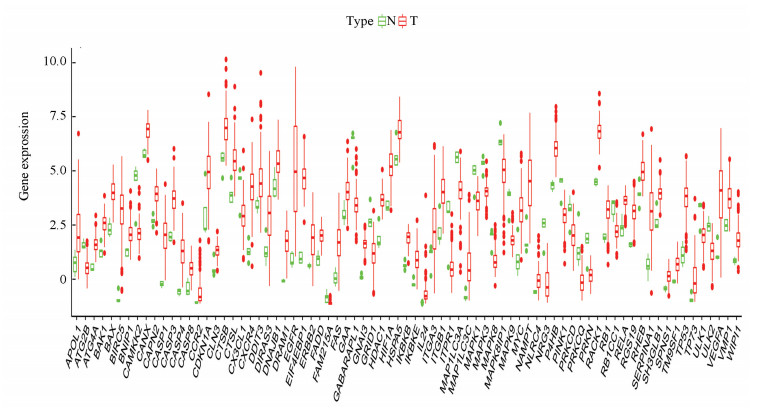
2 结果分析 2.1 GBM中差异表达的自噬相关基因通过在人类基因数据库HADb下载总共直接或间接参与自噬的232个基因。提取TCGA中这些自噬相关基因的表达数据后,使用Wilcoxon秩和检验做差异分析,筛选标准为FDR < 0.05和| log2(倍数变化)|> 1。得出56个基因表达显著高于正常脑组织,为上调基因。21个基因的表达明显低于正常脑组织。为下调基因(见图 1)。运用箱线图显示这些基因在正常脑组织样本及肿瘤样本中的表达(见图 2)。
|
图 1 差异自噬基因火山图和热图 Figure 1 Differential autophagy gene volcano map and heatmap |
|
图 2 差异自噬基因箱线图 Figure 2 Differential autophagy gene box plot 注:77个ARG在肿瘤组织和正常脑组织的表达。红点代表肿瘤组织样本,绿色代表正常脑组织样本。 |
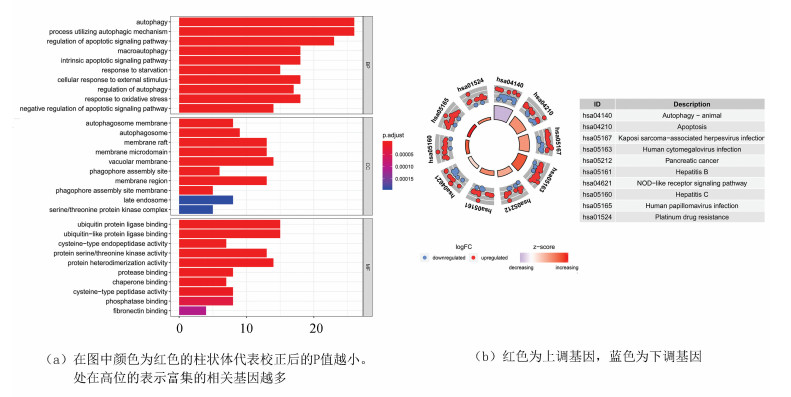
在GO富集分析中的具有显著分析的生物学过程(BP)主要包含:调节自噬、凋亡信号通路的调控及细胞对外界刺激反应方面;细胞组分(CC)主要包含:自噬体膜和自噬小体;分子功能(MF)主要包含:泛素样蛋白连接酶结合、半胱氨酸-类型肽链内切酶的活动及蛋白丝氨酸/苏氨酸激酶活性。分析结果见图 3(P < 0.001,FDR < 0.05)。在KEGG富集分析中显示,77个ARG与动物自噬、细胞凋亡、胰腺癌、类NOD受体信号通路、结直肠癌、膀胱癌、小细胞肺癌、ErbB信号通路、肿瘤坏死因子信号通路显著相关(P < 0.001, FDR < 0.05)。分析结果如图 4所示。

|
图 3 GO分析结果 Figure 3 Results of GO analysis |
|
图 4 KEGG分析结果 Figure 4 Results of KEGG analysis |
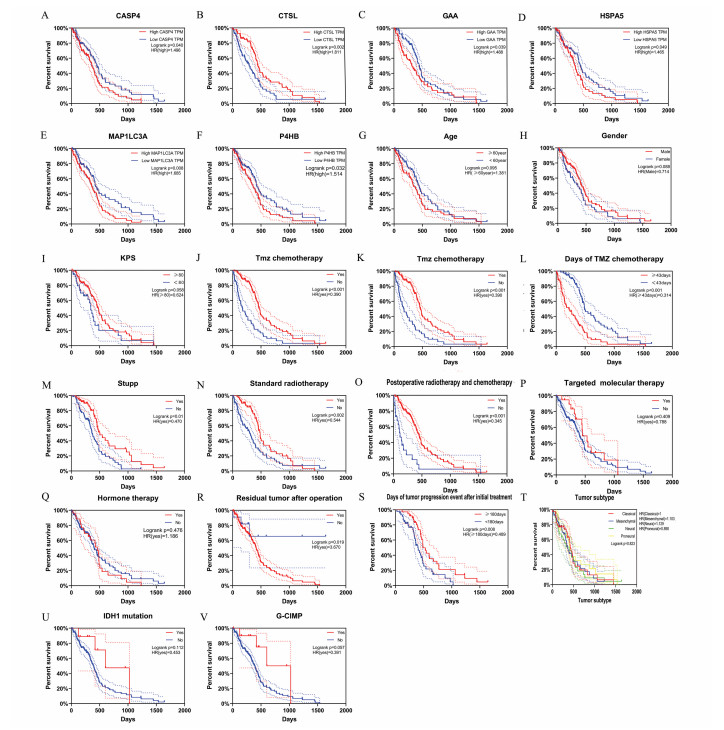
在剔除掉TCGA数据组中没有生存数据的患者后,对数据进行预后评估。KM分析显示,6个自噬基因(HSPA5、GAA、P4HB、CASP4、CTSL、MAP1LC3A)及Tmz化疗、Tmz长期化疗、Tmz化疗天数、Stupp方案、放疗、术后放化疗、术后肿瘤残余、首次治疗后肿瘤进展时间与GBM预后相关(P < 0.05),结果见表 1及图 5。其中6个自噬基因、术后肿瘤残余、肿瘤在6个月内有进展为高危因素;Tmz化疗、Tmz长期化疗、Tmz化疗天数、Stupp方案、放疗、术后放化疗、为保护因素。随后,通过纳入Kaplan-Meier分析有意义的及临床上认为与GBM患者预后显著相关的因素(KPS评分、Stupp治疗方案、放疗、术后肿瘤残余、首次治疗后肿瘤进展时间、肿瘤分型、IDH1状态、6个自噬基因)[16-18]进行COX分析,筛选出CTSL(P=0.007,HR=2.605,95%CI =1.303-5.211)和Stupp方案(P=0.032,HR=0.464,95%CI=0.230-0.936)作为GBM总体存活(overall survive, OS)的独立预后指标(见表 2)。
| 表 1 GBM患者关键自噬基因与临床病理参数的单因素分析 Table 1 Single factor analysis of key autophagy genes and clinicopathological parameters in GBM patients |
|
图 5 Kaplan-Meier分析的结果图(A-V) Figure 5 Results of Kaplan-Meier analysis (A-V) |
| 表 2 COX分析结果 Table 2 Results of COX analysis |
对CTSL预测生存的可靠性分析结果显示单个自噬基因CTSL在1年,3年,5年生存率为(AUC=0.665,AUC=0.628,AUC=0.549),其预测预后的可靠性仍需要探讨(见图 6)。
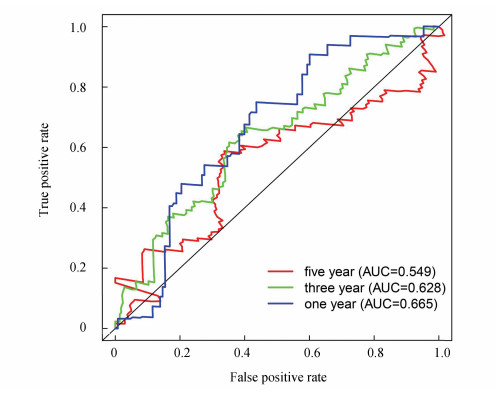
|
图 6 随时间依赖性的ROC分析 Figure 6 Results of time-dependent ROC analysis |
为了探索CTSL在GBM中的表达与临床病理的关系,从TCGA数据库获得并分析了mRNA表达谱。并列出有差异性的结果,如图 7(a-d)所示。CTSL表达的增加与非G-CIMP(P=0.015),TMZ长期化疗(P =0.012),TMZ化疗(P=0.012),肿瘤亚型(P < 0.001)相关.
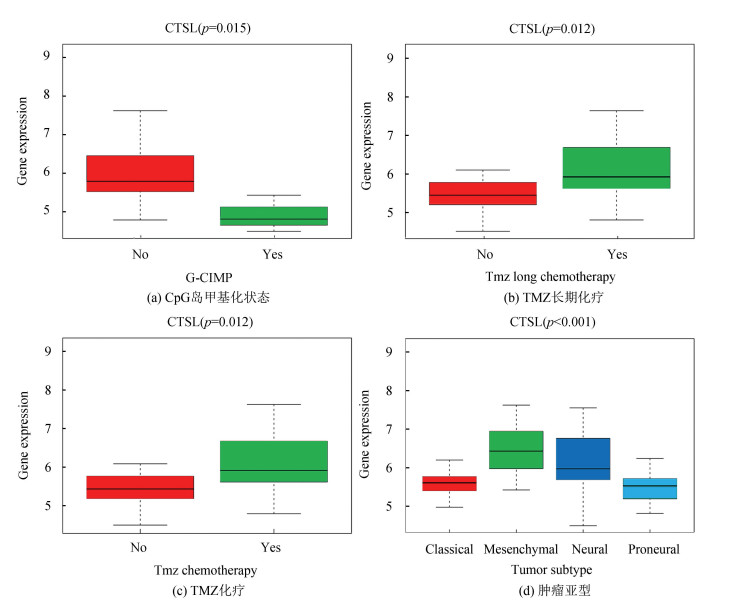
|
图 7 TCGA队列中CTSL表达与临床病理变量之间的关联 Figure 7 Correlation between CTSL expression and clinicopathological variables in TCGA cohort |
GEPIA的数据分析显示CTSL表达在正常人和GBM患者中具有差异性,单因素分析显示CSTL为高危因素, 见图 8(Lograngk P=0.007 9, HR=1.6)。CGGA数据库的mRNA325数据集显示:(1)GBM中CTSL的表达与Ⅱ级胶质瘤、Ⅲ级胶质瘤中CTSL的表达均具有差异性;(2)以CTSL中位值分组的Kaplan-Meier分析仅在GBM中有差异性(P=0.001 8);(3)IDH野生型GBM及1p / 19q无缺失型GBM中的CTSL表达量更高(见图 9)。
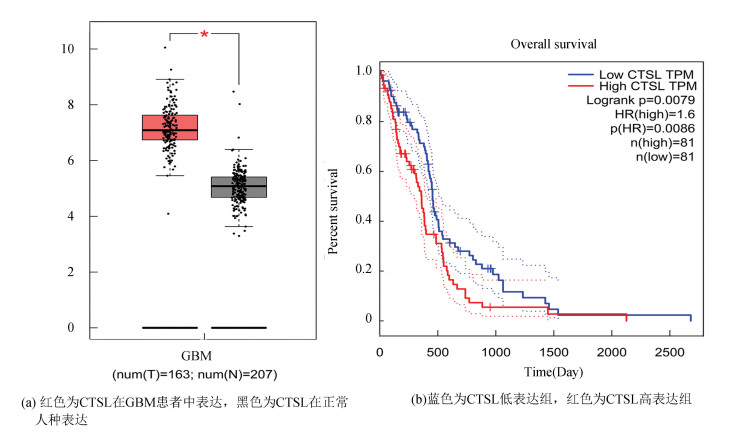
|
图 8 GEPIA分析结果 Figure 8 Results of GEPIA analysis |
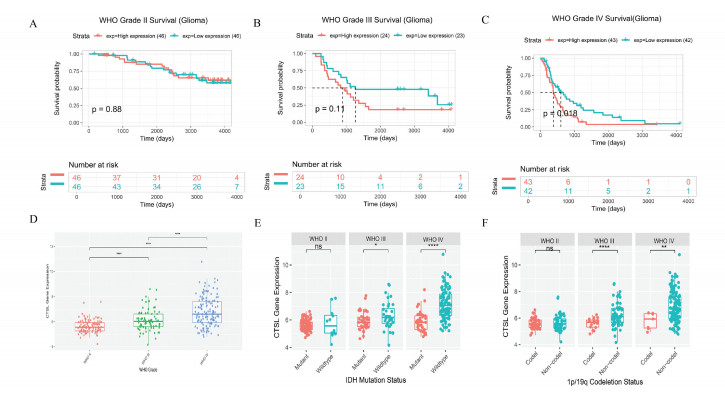
|
图 9 CGGA分析结果 Figure 9 Results of CGGA analysis 注:(A, B, C)CTSL的高低表达分组在Ⅱ级胶质瘤、Ⅲ级胶质瘤及GBM中KM分析(D)CTSL在Ⅱ级胶质瘤、Ⅱ级胶质瘤及GBM中的表达差异(E, F)CTSL在IDH突变数据及1p/19q编码数据中的表达。 |
根据CTSL表达的中位值将其分为高、低两类,运用GSEA进行分析,在MSigDB集合(kegg.v6.2.symbols.gmt, h.all.v7.0.symbols.gmt, c5.all.v7.0.symbols.gmt)的富集中观察到了显着差异(P < 0.05,FDR < 0.05)。结果见图 10及表 3,在溶酶体、类NOD受体信号通路、kras通路信号上调、活性氧通路、血管生成、低氧症、细胞化疗、巨噬细胞迁移、巨噬细胞迁移的监管等与自噬和肿瘤相关的途径中,CTSL有显著富集。
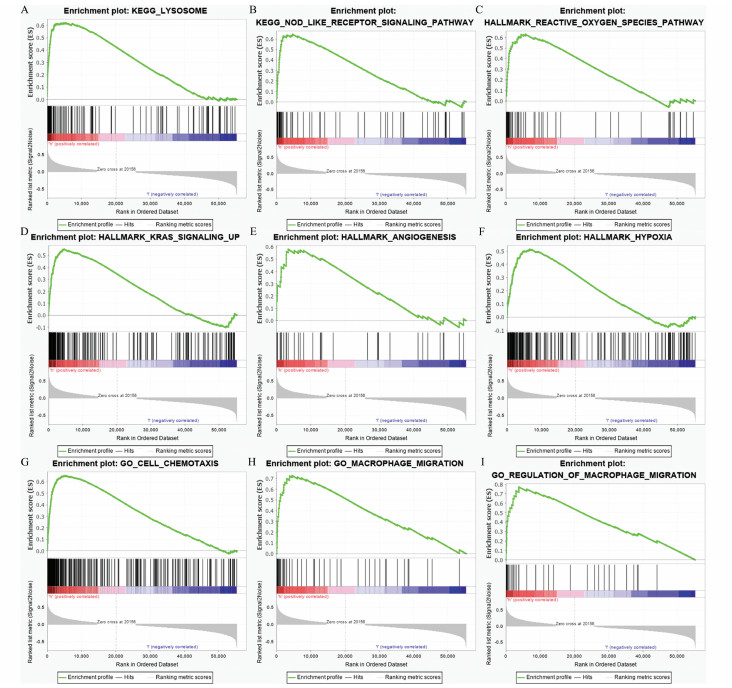
|
图 10 GSEA分析结果 Figure 10 Results of GSEA analysis |
| 表 3 GSEA分析结果 Table 3 Results of GSEA analysis |
为了研究自噬基因CTSL在自噬调节和胶质瘤起到的作用,我们从自噬调节网络(ARN)中提取了与它们直接相关的邻居。ARN是一个包含自噬基因相互作用及预测数据的数据库。[19]我们发现与CTSL直接相关的自噬基因为: COL18A1, CD74, IGFBP-3, C3.同时COL18A1和CD74还被证明与胶质瘤相关(见图 11)。

|
图 11 与CTSL相关基因图 Figure 11 Gene map related to CTSL |
研究中的筛选出的77个差异自噬基因,GO和KEGG富集分析结果显示了其在调节自噬和各种肿瘤方面具备重要作用。同类型研究显示通过自噬基因在GBM中的集中富集在细胞凋亡,坏死,免疫力和炎症反应中,并通过COX模型联合Kaplan-Meier分析有意义的自噬基因,以风险值为分组,对于GBM的预后预测更显著,其中自噬基因P4HB、HSPA5同样为高危因素,但由于采取统计学方法及数据库不同,最终筛选结果具有一定差异[20, 21]。Kaplan-Meier生存分析显示6个自噬基因(HSPA5、GAA、P4HB、CASP4、CTSL、MAP1LC3A)、术后肿瘤残余、肿瘤在6个月内有进展、为高危因素;Tmz化疗、Tmz长期化疗、Tmz化疗天数、Stupp方案、放疗、术后放化疗为保护因素。且临床广泛应用的STUPP方案在COX结果中被验证为是一种保护因素,结果与其他实验结果和临床认知基本相符[21, 22]。同时本研究显示43天的TMZ化疗(根据数据中位数分组)似乎更具有意义。但年龄分析并未显示显著的差异性,这与Brodbelt等人对胶质瘤的研究并不相符[23],这可能与本文样本较少有关。
CTSL是一种溶酶体酸性半胱氨酸蛋白酶,具有促进肿瘤转移的功能,高表达的CTSL患者生存时间更短,与正常组织、Ⅱ级胶质瘤组织、Ⅲ级胶质瘤组织相比,CTSL在GBM中具有差异性表达,且在COX分析中CTSL的高表达被确定为一种独立危险因素,有越来越多的研究显示CTSL的表达与肿瘤的临床及预后密切相关。在已有的研究中发现CTSL在人胃癌组织中具有显著的高表达,通过作用于人胃癌中的CDP / Cux / VEGF-D途径促进胃癌组织内的血管生成[24]。Dhivya在体内和体外实验均证实了使用KGP94抑制CTSL的表达能使肿瘤细胞诱导的血管生成显着减少,并提出与乳腺癌患者中CTSL的表达上调与复发和转移发生率增加以及整体生存率下降密切相关[25],其结论与本文GSEA结果显示CTSL在血管通路富集结果相符。
临床相关性分析显示CTSL在Tmz化疗后表达更高, 而肿瘤细胞对Tmz的不敏感性和耐药性是GBM患者治疗失败和预后不良的主要原因,自噬抑制剂在临床、体外与体内模型中均表现出与临床药物替莫唑胺的优异协同作用[26]。冯建波等人的研究发现LRRC4与DEPTOR / mTOR复合物的相互作用抑制了GBM细胞的自噬并增加了GBM的Tmz治疗反应[27]。而目前仅有动物实验表明抑制CTSL的表达可以使G2 / M细胞周期停滞从而增加人神经胶质瘤U251细胞的放射敏感性[28]。同时我们研究发现CTSL在非GCIMP型、间质型、IDH野生型及1p/19q无缺失型胶质母细胞瘤中为高表达,这几种类型的GBM均被证实为胶质母细胞瘤的高危分型[29-31],提示对通过抑制自噬基因CTSL的表达,对GBM的化疗耐药性及高危型GBM的预后可能具有改善作用,但这一结论仍需实验证实。与Ⅱ级胶质瘤、Ⅲ级胶质瘤相比,CTSL在GBM中具有差异性表达,这提示CTSL的表达可能可以作为胶质瘤的诊断标准。
与CTSL直接相关的自噬基因为: COL18A1, CD74, IGFBP-3, C3。单个COL18A1基因编码成人类胶原蛋白XVIII,而内皮抑素位于人类胶原蛋白XVIII的C端非胶原结构域的末端[32],其通过调节Bcl-2表达影响内皮细胞的自噬[33]。且有研究表明经胶质瘤组织内皮抑素水平与恶性程度呈正相关[34]。CD74通过AMPK-mTOR-Skp2介导的自噬调节作用来抑制乙醇引起的的心肌功能障碍,炎症和细胞凋亡[35]。同时Zeiner等人实验表明人类神经胶质瘤中CD74的表达与患者的生存呈正相关[36]。IGFBP-3的表达在缺氧环境中会上调,当其GRP78结合后,会促进自噬和应激存活,在受到治疗剂的挑战时,可以明显增强肿瘤的进展,也可以帮助肿瘤细胞存活[37]。与CTSL相关联的C3'通过和ATG16L1之间的直接相互作用,使得自噬系统可以有效地靶向细胞[38]。这些结果表明,CTSL在某种条件下与其他自噬相关成分相互作用从而影响自噬和胶质瘤的进展。前期自噬通过吞噬肿瘤细胞来抑制自噬,后期由于肿瘤细胞的迅速生长,造成缺氧环境,从而激活自噬,反而保护肿瘤细胞,促进其增殖与后期的复发,此结果在相关文章中也有提及错误!未指定书签[3, 34, 39]。
GBM作为一种恶性程度高,预后差的疾病,已经有学者通过生物信息学方法筛选GBM的核心基因,并提出基因KCNAB2可能为有效靶标[40]。本研究则进一步探讨了自噬基因与GBM的关系,发现了自噬基因CTSL的高表达是独立危险因素,并提出CTSL的高表达可能与化疗耐药性、高危型GBM的预后相关。不足之处在于自噬基因数据库未能全部覆盖,且由于胶质母细胞瘤恶性程度高,单一基因预测预后AUC值较低,文章的结论缺少实验证据的支持。
4 结论1) 自噬基因CTSL在胶质母细胞瘤患者中显著高表达,在高危型和接受化疗后胶质母细胞瘤患者表达量更高。
2) 自噬基因CTSL高表达与胶质母细胞瘤患者生存时间负相关,COX分析显示为独立危险因素,有作为有效靶标的潜质。
3) 与自噬基因CTSL相关的成分研究表明其在某种条件下CTSL与其他自噬相关成分相互作用从而影响自噬和胶质瘤的进展。
| [1] |
ROMERO M A, BAYRAKTAR E O, BAGCA B G, et al. Role of autophagy in breast cancer development and progression: opposite sides of the same coin[J]. Advances in Experimental Medicine & Biology, 2019, 1152: 65-73. DOI:10.1007/978-3-030-20301-6_5 (  0) 0) |
| [2] |
GUO Feng, LIU Xinyao, CAI Huaibin, et al. Autophagy in neurodegenerative diseases: pathogenesis and therapy[J]. Brain Pathology, 2018, 28(1): 3-13. DOI:10.1111/bpa.12545 (  0) 0) |
| [3] |
AMARAVADI R, KIMMELMAN A C, WHITE E. Recent insights into the function of autophagy in cancer[J]. Genes & Development, 2016, 30(17): 1913-1930. DOI:10.1101/gad.287524.116 (  0) 0) |
| [4] |
JIANG P, MIZUSHIMA N. Autophagy and human diseases[J]. Cell Research, 2014, 24(1): 69-79. DOI:10.1038/cr.2013.161 (  0) 0) |
| [5] |
WHITE E, KARP C, STROHECKER A M, et al. Role of autophagy in suppression of inflammation and cancer[J]. Current Opinion in Cell Biology, 2010, 22(2): 212-217. DOI:10.1016/j.ceb.2009.12.008 (  0) 0) |
| [6] |
WHITE E, DIPAOLA R S. The double-edged sword of autophagy modulation incancer[J]. Clinical Cancer Research, 2009, 15(17): 5308-5316. DOI:10.1158/1078-0432.CCR-07-5023 (  0) 0) |
| [7] |
SUI X, CHEN R, WANG Z, et al. Autophagy and chemotherapy resistance: a promising therapeutic target for cancer treatment[J]. Cell Death & Disease, 2013, 4(10): 838. DOI:10.1038/cddis.2013.350 (  0) 0) |
| [8] |
GU Yunyan, LI Pengfei, PENG Fuduan, et al. Autophagy-related prognostic signature for breast cancer[J]. Molecular Carcinogenesis, 2016, 55(3): 292-299. DOI:10.1002/mc.22278 (  0) 0) |
| [9] |
LIN Peng, HE Rongquan, DANG Yiwu, et al. An autophagy-related gene expression signature for survival prediction in multiple cohorts of hepatocellular carcinoma patients[J]. Oncotarget, 2018, 9(25): 17368-17395. DOI:10.18632/oncotarget.24089 (  0) 0) |
| [10] |
WANG Shishuo, CHEN Gang, LI Shenghua, et al. Identification and validation of an individualized autophagy-clinical prognostic indexin bladder cancer patients[J]. OncoTargets and Therapy, 2019, 12: 3695-3712. DOI:10.2147/OTT.S197676 (  0) 0) |
| [11] |
WEN P Y, KESARI S. Malignant gliomas in adults[J]. The New England Journal of Medicine, 2008, 359(5): 492-507. DOI:10.1056/NEJMra0708126 (  0) 0) |
| [12] |
DESJARDINS A, GROMEIER M, HERNDON J N, et al. Recurrent glioblastoma treated with recombinantpoliovirus[J]. The New England Journal of Medicine, 2018, 379(2): 150-161. DOI:10.1056/NEJMoa1716435 (  0) 0) |
| [13] |
BROWN C E, ALIZADEH D, STARR R, et al. Regression of glioblastoma after chimeric antigen receptor T-cell therapy[J]. The New England Journal of Medicine, 2016, 375(26): 2561-2569. DOI:10.1056/NEJMoa1610497 (  0) 0) |
| [14] |
JAWHARI S, RATINAUD M H, VERDIER M. Glioblastoma, hypoxia and autophagy: a survival-prone 'menage-a-trois'[J]. Cell Death & Disease, 2016, 7(10): e2434. DOI:10.1038/cddis.2016.318 (  0) 0) |
| [15] |
NAGER M, SALLAN M C, VISA A, et al. Inhibition of WNT-CTNNB1 signaling upregulates SQSTM1 and sensitizes glioblastoma cells to autophagy blockers[J]. Autophagy, 2018, 14(4): 619-636. DOI:10.1080/15548627.2017.1423439 (  0) 0) |
| [16] |
WU M, MISKA J, XIAO T, et al. Race influences survival in glioblastomapatients with KPS≥80 and associates with genetic markers of retinoic acid metabolism[J]. Journal of Neuro-Oncology, 2019, 142(2): 375-384. DOI:10.1007/s11060-019-03110-5 (  0) 0) |
| [17] |
STUPP R, DIETRICH P Y, OSTERMANN K S, et al. Promising survival for patients withnewly diagnosed glioblastoma multiforme treated with concomitant radiation plus temozolomide followed by adjuvant temozolomide[J]. Journal of Clinical Oncology, 2002, 20(5): 1375-1382. DOI:10.1200/JCO.2002.20.5.1375 (  0) 0) |
| [18] |
BROWN T J, BRENNAN M C, LI M, et al. Association of the extent of resection with survival in glioblastoma[J]. JAMA Oncology, 2016, 2(11): 1460. DOI:10.1001/jamaoncol.2016.1373 (  0) 0) |
| [19] |
TUREI D, FOLDVARI-NAGY L, FAZEKAS D, et al. Autophagy Regulatory Network - a systems-level bioinformatics resource for studying the mechanism and regulation of autophagy[J]. Autophagy, 2015, 11(1): 155-165. DOI:10.4161/15548627.2014.994346 (  0) 0) |
| [20] |
WANG Yunling, ZHAO Weijiang, XIAO Zhe, et al. A risk signature with four autophagy-related genes for predicting survival of glioblastoma multiforme[J]. Journal of Cellular and Molecular Medicine, 2020, 24(7): 3807-3821. DOI:10.1111/jcmm.14938 (  0) 0) |
| [21] |
WANG Qiangwei, LIU Hanjie, ZHAO Zheng, et al. Prognostic correlation of autophagy-related gene expression-based risk signature in patients with glioblastoma[J]. Onco Targets Therapy, 2020, 13: 95-107. DOI:10.2147/OTT.S238332 (  0) 0) |
| [22] |
GALANIS E, BUCKNER J. Chemotherapy for high-grade gliomas[J]. British Journal of Cancer, 2000, 82(8): 1371-1380. DOI:10.1054/bjoc.1999.1075 (  0) 0) |
| [23] |
BRODBELT A, GREENBERG D, WINTERS T, et al. Glioblastoma in England: 2007-2011[J]. European Journal of Cancer, 2015, 51(4): 533-542. DOI:10.1016/j.ejca.2014.12.014 (  0) 0) |
| [24] |
PAN Tao, JIN Zhijian, YU Zhenjia, et al. Cathepsin L promotes angiogenesis by regulating the CDP/Cux/VEGF-D pathway in human gastric cancer[J/OL]. Gastric Cancer, 2020. https://doi.org/10.1007/s10120-020-01080-6. DOI:10.1007/s10120-020-01080-6.
(  0) 0) |
| [25] |
SUDHAN D R, RABAGLINO M B, WOOD C E, et al. Cathepsin L in tumor angiogenesis and its therapeutic intervention by the small molecule inhibitor KGP94[J]. Clinical & Experimental Metastasis, 2016, 33(5): 461-473. DOI:10.1007/s10585-016-9790-1 (  0) 0) |
| [26] |
HWANG H Y, CHO Y S, KIM J Y, et al. Autophagic inhibition via lysosomal integrity dysfunction leads to antitu or activity in glioma treatment[J]. Cancers (Basel), 2020, 12(3): 543. DOI:10.3390/cancers12030543 (  0) 0) |
| [27] |
FENG J, ZHANG Y, REN X, et al. Leucine-rich repeat containing 4 act as an autophagy inhibitor that restores sensitivity of glioblastoma to temozolomide[J]. Oncogene, 2020, 39(23): 4551-4566. DOI:10.1038/s41388-020-1312-6 (  0) 0) |
| [28] |
ZHANG Qingqing, WANG Wenjuan, LI Jun, et al. Cathepsin L suppression increases the radiosensitivity of human glioma U251 cells via G2/M cell cycle arrest and DNA damage[J]. Acta Pharmacologica Sinica, 2015, 36(9): 1113-1125. DOI:10.1038/aps.2015.36 (  0) 0) |
| [29] |
卫力文, 姚冰, 孙佩欣, 等. 63例高级别胶质瘤中IDH1和TERT突变状态的预后价值[J]. 现代肿瘤医学, 2020, 28(14): 2412-2416. WEI Liwen, YAO Bing, SUN Peixin, et al. Prognostic significance of IDH1 and TERT promotor mutations in 63 patients with high-grade gliomas[J]. Journal of Modern Oncology, 2020, 28(14): 2412-2416. DOI:10.3969/j.issn.1672-4992.2020.14.010 (  0) 0) |
| [30] |
许学文, 宋廉, 杨晓晓, 等. CXCL12-CXCR4轴促进胶质母细胞瘤前神经间质转化[J]. 江苏大学学报(医学版), 2020, 30(1): 18-23. XU Xuewen, SONG Lian, YANG Xiaoxiao, et al. CXCL12-CXCR4 axis promotes proneural-mesenchymal transition in glioblastoma[J]. Journal of Jiangsu University(Medicine Edition), 2020, 30(1): 18-23. DOI:10.13312/j.issn.1671-7783.y190203 (  0) 0) |
| [31] |
LEBLANC V G, MARRA M A. DNA methylation in adult diffuse gliomas[J]. Briefings in Functional Genomics, 2016, 15(6): 491-500. DOI:10.1093/bfgp/elw019 (  0) 0) |
| [32] |
HELJASVAARA R, AIKIO M, RUOTSALAINEN H, et al. Collagen XVⅢ in tissue homeostasis and dysregulation-Lessons learned from model organisms and human patients[J]. Matrix Biology, 2017(57/58): 55-75. DOI:10.1016/j.matbio.2016.10.002 (  0) 0) |
| [33] |
NGUYEN T M, SUBRAMANIAN I V, XIAO X, et al. Endostatin induces autophagy in endothelial cells by modulating Beclin 1 and beta-catenin levels[J]. Journal of Cellular & Molecular Medicine, 2010, 13(9b): 3687-3698. DOI:10.1111/j.1582-4934.2009.00722.x (  0) 0) |
| [34] |
MORIMOTO T, AOYAGI M, TAMAKI M, et al. Increased levels of tissue endostatin in human malignant gliomas[J]. Clinical Cancer Research, 2002, 8(9): 2933-2938. (  0) 0) |
| [35] |
YANG Lifang, WANG Shuyi, MA Jipeng, et al. CD74 knockout attenuates alcohol intake-induced cardiac dysfunction through AMPK-Skp2-mediated regulation of autophagy[J]. Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease, 2019, 1865(9): 2368-2378. DOI:10.1016/j.bbadis.2019.05.020 (  0) 0) |
| [36] |
ZEINER P S, PREUSSE C, BLANK A E, et al. MIF Receptor CD74 is restricted to Microglia/Macrophages, associated with a M1-Polarized immune milieu and prolonged patient survival in gliomas[J]. Brain Pathology, 2015, 25(4): 491-504. DOI:10.1111/bpa.12194 (  0) 0) |
| [37] |
GRKOVIC S, O'REILLY V C, HAN S, et al. IGFBP-3 binds GRP78, stimulates autophagy and promotes the survival of breast cancer cells exposed to adverse microenvironments[J]. Oncogene, 2013, 32(19): 2412-2420. DOI:10.1038/onc.2012.264 (  0) 0) |
| [38] |
SORBARA M T, FOERSTER E G, TSALIKIS J, et al. Complement C3 drives autophagy-dependent restriction of cyto-invasive bacteria[J]. Cell Host Microbe, 2018, 23(5): 644-652. DOI:10.1016/j.chom.2018.04.008 (  0) 0) |
| [39] |
MOWERS E E, SHARIFI M N, MACLEOD K F. Functions of autophagy in the tumor microenvironment and cancer metastasis[J]. FEBS Journal, 2018, 285(10): 1751-1766. DOI:10.1111/febs.14388 (  0) 0) |
| [40] |
柯帅, 王文波, 廖红展, 等. 生物信息学方法筛选胶质母细胞瘤的核心基因[J]. 生物信息学, 2020, 18(1): 56-64. KE Shuai, WANG Wenbo, LIAO Hongzhan, et al. Identifying core genes of glioblastoma by bioinformatics analysis[J]. Chinese Journal of Bioinformatics, 2020, 18(1): 56-64. DOI:10.12113/201907004 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


