研究表明,编码蛋白质的基因只占整个基因组的一小部分,大部分都属于非编码区域,在非编码区域中有大部分会经过转录产生RNA,但是这些RNA不经过翻译的步骤,这类RNA称为非编码RNA(Non-coding RNA, ncRNA)[1]。非编码RNA不仅数量庞大,种类也有很多[2]。其中长链非编码RNA(Long non-coding RNA, lncRNA)属于分子长度大于200个碱基的调控非编码RNA[3]。最初lncRNA并不被重视,被认为是转录过程中的“噪音”,但是随着实验技术和预测算法的改进,不仅鉴定和预测到越来越多的lncRNA,而且还发现lncRNA在动植物中都具有重要的生物学功能[4][5]。目前大量的lncRNA的研究还主要集中在人和动物等物种中,相对来说对于植物lncRNA的研究还比较落后[6]。随着高通量测序技术和计算方法的发展,在过去的几十年中,大量的lncRNA在不同的植物中被发现,例如拟南芥、水稻、玉米、小麦、黄瓜、番茄等[7]。通过生物信息学分析手段可以预测和鉴定越来越多的lncRNA[8]。2017年Mohan Singh等人开发的预测植物lncRNA的工具PLncPRO[9],分别在干旱和盐胁迫条件下,在水稻和鹰嘴豆中发现了3 714和3 457个高可信度的lncRNA。2018年常征等[10]也通过机器学习方法对植物lncRNA进行了预测,从PNRD数据库中下载了2 565条lncRNA为正集数据,负集是从RefSeq数据库下载的2 500条mRNA,采用5折交叉验证的准确率为89%,用了同样的数据集在CPAT、CNCI、PLEK等软件上进行分类预测的结果分别是85.7%、82.7%、71.4%。可见计算机预测的方法对植物lncRNA的预测起到了很好的作用,但是由于lncRNA特殊的序列属性,使得lncRNA的鉴定工作仍然面临着挑战[11],到目前来说,lncRNA的精确识别仍然是植物研究领域的主要问题之一[9]。本文通过新建植物lncRNA和mRNA数据集,提取lncRNA的序列及结构特征,并将多特征融合,利用支持向量机算法对植物lncRNA进行了预测,取得了较好的预测效果。
1 材料和方法 1.1 数据集正集数据来源于数据库NONCODEv5(http://www.noncode.org/)中拟南芥的3 763条lncRNA,负集数据是从NCBI(https://www.ncbi.nlm.nih.gov/)中随机下载的3 800条多种植物的mRNA序列。考虑到序列的相似性对预测结果会造成一定的影响,通过CD-Hit软件[12]去除冗余,取相似性小于60%的序列,最后得到2 464条lncRNA,2 459条mRNA。
1.2 特征提取 1.2.1 lncRNA序列的k-mer核苷酸组分信息从lncRNA序列中提取核苷酸组分特征[13],如果将lncRNA序列表示为
| $ S = {A_1}, {A_2}, {A_3}, {A_4}, \ldots , {A_{L - 1}}, {A_L} $ | (1) |
其中Aj就是四种碱基(腺嘌呤A,胞嘧啶C,鸟嘌呤G,尿嘧啶U)中的一种。
对于一个长度为L的核苷酸序列,当k=1时就代表四种碱基出现的频数,则RNA序列就能表示成4维的向量,同样当k=4的时候,则有AAAA,AAAC,...,UUUU不同的组合,RNA序列就能表示成256维的特征向量:
| $ X=\left[x_{1}, x_{2}, \ldots, x_{256}\right] $ | (2) |
考虑到碱基的化学特性,将四种核苷酸进行了约化,约化分别是嘌呤嘧啶约化和强弱键约化,腺嘌呤和鸟嘌呤都是嘌呤,都用M来表示,胞嘧啶和尿嘧啶都是嘧啶,都用N来表示;又因为鸟嘌呤和胞嘧啶之间以3个氢键形成碱基配对,分子能量的稳定性较高,所以将鸟嘌呤和胞嘧啶都用P来表示,而腺嘌呤与尿嘧啶是以两个氢键形成碱基对,稳定性相对较弱,将腺嘌呤和尿嘧啶都用Q表示。对RNA序列进行约化后重新提取k-mer信息,此时k取4的时候RNA序列就可以表示成24=16维的特征向量。
1.2.2 开放阅读框开放阅读框(Open reading frame, ORF)的长度是常被用来区分lncRNA和mRNA的最基本的标准之一[14],然而,专门用于预测ORF的生物信息学工具很少,sORF finder[15]根据编码序列间的核苷酸组成偏见,并通过同义和非同义替换率评估的氨基酸水平上的潜在功能限制进行编码sORFs的识别,但是它是将近十年前开发的。还有一些编码潜能的评估工具,例如CPC、CPAT、CNCI、CPC2、LGC等都可以用来预测ORF[16],尤其LGC是在基于ORF长度和GC含量之间的特征关系来评估编码潜能的,它在计算分析从植物到哺乳动物等多种物种方面都具有广泛的应用潜力[17],本文就是利用LGC来识别植物lncRNA的ORF。为了找到关于开放阅读框的最佳的特征集,构建了最长开放阅读框的长度以及它的相对长度两组特征,一个开放阅读框的相对长度是由其长度除以相应的转录本长度来定义的[18]。
1.2.3 二级结构RNA序列是由四种不同的碱基组成,RNA二级结构也是由不同的结构元素组成,像是茎区和环状结构等[19]。RNA的结构在很大程度上决定了它的功能,识别RNA分子的二级结构是了解其生物学功能的有效途径[20]。结构特征的提取是利用机器学习的方法预测lncRNA的重要步骤之一,但是目前还没有合适的结构特征提取工具[21]。本文使用了RNAfold软件[22]预测了lncRNA的二级结构,得到了二级结构点括号表示形式,括号表示配对的碱基,形成茎结构,点表示没有配对的碱基,形成单链或环结构,最后的数字表示释放后的最小自由能[23]。将lncRNA的二级结构颈环个数及归一化的最小自由能等作为特征,归一化的最小自由能是由释放后的最小自由能除以对应序列的长度来定义的[24]。
1.2.4 RNA的几何柔性信息PseKNC在核苷酸序列的分类上常被作为一种特征[25],它用离散的模型或者向量表示核苷酸序列,而且通过其组成寡核苷酸的物理化学特性保留相当多的序列顺序信息,特别是全局或者局部的序列顺序信息[26]。提取了核苷酸序列的远距离和近距离的信息后,能够更加有助于对核苷酸序列进行分类预测。近几年来,PseKNC被广泛用于计算遗传学和基因组学等各个领域,像是预测DNA甲基化位点,预测启动子,预测基因组中核小体位置,鉴定microRNA前体等等[27]。考虑RNA局部结构特性,一般相邻两个碱基对的空间排列有六个参数,三个角度旋转参数(Tilt,Roll,Twist)和三个局部平移参数(Rise,Slide,Shift)[28],六个RNA结构信息参数值详细见表 1。将RNA序列表示为4k+λ维的特征向量[26]:
| $ R = {\left[ {{d_1}{d_2} \cdots {d_{{4^k}}}{d_{{4^k} + 1}} \cdots {d_{{4^k} + \lambda }}} \right]^{\rm{T}}} $ | (3) |
| $ {r_u} = \left\{ {\begin{array}{*{20}{l}} {\frac{{f_u^{k - {\rm{ }}tuple{\rm{ }}}}}{{\sum\limits_{i = 1}^{4k} {f_i^{k - {\rm{ }}tule{\rm{ }}}} + \omega \sum\limits_{j = 1}^\lambda {{\theta _j}} }}\quad \left( {1 \le u \le {4^k}} \right)}\\ {\frac{{\omega {\theta _{u - 4k}}}}{{\sum {_{i = 1}^{4k}} f_i^{k - {\rm{ }}tule{\rm{ }}} + \omega \sum\limits_{j = 1}^\lambda {{\theta _j}} }}\quad \left( {{4^k} + 1 \le u \le {4^k} + \lambda } \right)} \end{array}} \right. $ | (4) |
fik-tuple是第i个核苷酸在RNA序列中出现的频率,θj为第j级序列顺序的相关因子,ω是权重因子,用于权衡核苷酸组分和RNA局部结构性质的影响(取值为0.1~1)。
1.3 支持向量机算法(Support Vector Machine, SVM)支持向量机的基本思想是寻找两个类之间的最大边界超平面,对于非线性数据,使用核函数将它映射到线性的高维空间中,然后在高维空间中拟合一个线性函数去解决非线性分类问题[30]。文中采用的是Chan和Lin小组开发的LIBSVM软件包[31]。
1.4 预测性能评估预测算法的性能是常用的敏感性(Sensitivity, Sn)、特异性(Specificity, Sp)、总体预测成功率(Acc)以及马修相关系数(Mathew's Correlation Coefficient, MCC),定义为:
| $ {{S_{\rm{n}}} = \frac{{TP}}{{TP + FN}} \times 100\% } $ | (5) |
| $ {{S_{\rm{p}}} = \frac{{TN}}{{TN + FP}} \times 100\% } $ | (6) |
| $ {Acc = \frac{{TP + TN}}{{TP + FN + FP + FN}} \times 100\% } $ | (7) |
| $ \begin{array}{*{20}{l}} {MCC = }\\ {\frac{{TP \times TN + FP \times FN}}{{\sqrt {(TP + FP) \times (TN + FN) \times (TP + FN) \times (TN + FP)} }} \times }\\ {100\% } \end{array} $ | (8) |
其中,TP表示正集序列被预测正确的序列数,TN表示负集序列被预测正确的序列数,FN表示负集序列被预测错误的序列数,FP表示正集序列被预测错误的序列数。
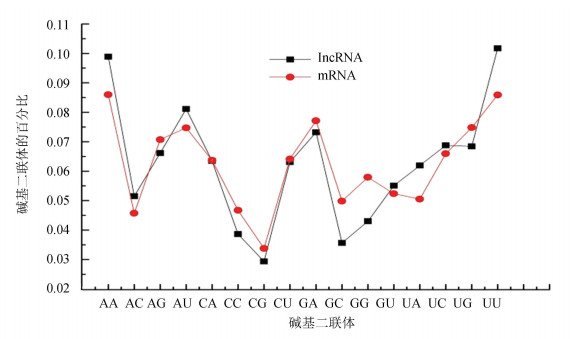
2 结果分析 2.1 植物lncRNA的序列特征分析了植物lncRNA序列特征,首先是计算2 464条植物lncRNA序列所含的单碱基的比例,与2 459条植物mRNA所含单碱基的比例相对比,见图 1。发现植物lncRNA富含碱基A和U,而mRNA富含碱基C和G。同样计算了2 464条植物lncRNA序列所含的碱基二联体的比例,与2 459条植物mRNA所含的碱基二联体的比例相对比,见图 2。发现植物lncRNA的AA/AU/ UA /UU二联体的频数也是明显比mRNA高。

|
图 1 两类序列中的四种核苷酸组分 Figure 1 Four kinds of nucleotide components in two sequences |
|
图 2 两类序列中的二联体组分 Figure 2 Percentage of diomorphic component in two sequences |
根据计算分析植物lncRNA的序列特征,发现植物lncRNA也有一定的序列偏好特征。所以,本文提取lncRNA的k-mer序列特征作为SVM的输入向量来识别植物lncRNA,基于Jackknife检验的不同特征的预测结果见图 3。k值的范围是从1取到6,从图 3中可以看出,在k取4的时候总体预测成功率是最高的。以约化后的k-mer信息作为特征向量时,取得的总体预测成功率相对来说并不是很好,但是强弱键约化后的总体预测成功率明显要比嘌呤嘧啶约化后的总体预测成功率高,可见强弱键约化在识别植物lncRNA时是比嘌呤嘧啶约化更好的一个序列特征。

|
图 3 不同k-mer组分的预测结果 Figure 3 Prediction performance of different k-mer features |
用基于ORF的两个特征识别植物lncRNA时,以最长开放阅读框的长度作为特征向量输入到SVM算法中,总体预测成功率达到87.26%,而用其相对长度作为特征向量时,总体预测成功率达到88.26%,相对长度作为特征向量比直接以其长度作为特征向量时的总体预测成功率要高,相对长度通常用作长度的补充特征,在分类算法中有更好的表现[32]。
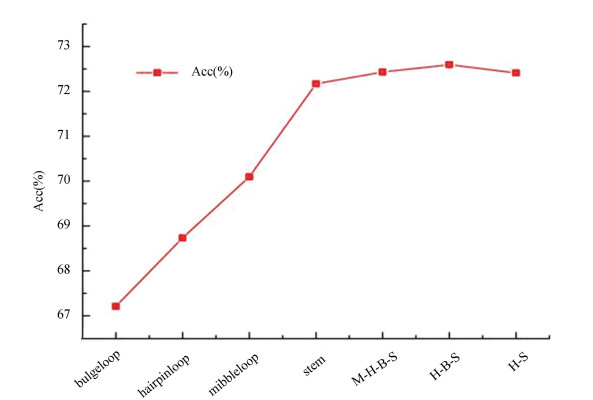
用RNAfold软件预测出的lncRNA的二级结构颈环个数及归一化的最小自由能作为特征向量进行预测时,预测结果见图 4,其中单一特征中茎的个数预测效果相对来说是比较好的,总体预测成功率为72.17%,将这些单一特征融合后进行预测,最好的预测成功率达到78.36%。
|
图 4 不同二级结构对预测结果的影响 Figure 4 Influence of different secondary structures on prediction results |
使用Pse-in-one[33]软件对序列的核苷酸之间的空间位置信息进行提取时,有两个参数ω和λ,ω是权重因子,取值范围是0到1,为了找到最佳的ω和λ的值,计算了λ的步长为5时对应的总体预测成功率,见图 5。从图 5中可以看出,λ步长为5且取值在1到30之间时,随着ω的增加,总体预测成功率逐渐降低,在ω相同时,λ值越大,预测成功率是偏小的,总体来看,当λ=5,ω=0.1时,得到的总体预测成功率最大为85.9%。

|
图 5 λ步长为5伪核苷酸特征分类准确率 Figure 5 Classification accuracy of pseudonucleotides with λ Steps 5 |
综合这些序列和结构特征信息的预测结果,结果比较好的特征有4-mer组分信息、最长开放阅读框的长度和相对长度以及PseKNC在参数λ=5,ω=0.1时的结构信息。将这些特征信息融合后进行预测,预测结果见表 2。
| 表 2 Jackknife检验下不同特征融合后的SVM预测结果 Table 2 Prediction performance of SVM model fusing different features under Jackknife test |
研究发现植物lncRNA也有一定的序列和结构偏好特征。在提取k-mer特征信息时,随着k的增加,特征向量的维数在以2k增加,由于特征向量的维数过大的时候会导致序列信息的冗余现象,因此将k的取值只取到6,结果发现在k=4的时候预测结果最好,在Jackknife检验下,总体预测成功率达到93.36%。考虑到碱基的化学特性和植物lncRNA功能相关,将碱基约化后进行预测,分析两种不同的碱基约化方式预测的结果发现,强弱键约化后预测取得的成功率比嘌呤嘧啶约化取得的成功率高,说明强/弱键约化(PQ约化)更能反应植物lncRNA的序列信息,有利于植物lncRNA的识别。在RNA序列的几何柔性信息中,用不同的参数预测结果表明,当参数ω=0.1、λ=5的时候预测结果最好,总体预测成功率达到85.9%。
虽然这些特征信息的预测结果都比较好,但是单一的特征去预测总是有一定的局限性,所以为了能够提取到更多的植物lncRNA序列中蕴藏的结构和功能的信息,将这些特征信息进行了融合,用融合后的特征再去对植物lncRNA进行预测,发现结果有了一定的提高,但是融合信息太多也会造成信息冗余,所以只融合序列和结构信息参数较优的几个特征,对植物lncRNA预测的总体成功率达到了96.14%,敏感性达到了96.51%,特异性达到了95.77%,马修相关系数的值是0.92。说明最长开放阅读框的相对长度以及4-mer组分信息的融合对植物lncRNA的预测很有效,而且发现最长开放阅读框的相对长度和其它一些序列及结构特征信息的组合对预测结果都能有一定程度的提高,由此可见,最长开放阅读框的相对长度在植物lncRNA和mRNA的分类预测中是一个重要的特征信息。
随着鉴定和预测出的lncRNA越来越多,植物lncRNA也开始越来越受到关注,虽然植物lncRNA的研究相对于动物lncRNA的研究还远远落后,但是动物lncRNA的研究策略为植物lncRNA的研究提供了很好的借鉴。下一步,为了更加准确地识别植物lncRNA,可以再深入的挖掘一些更加全面的特征信息,将有效的特征信息融合后再去预测,或者合理的去融合一些比较优越的算法提高预测结果。
| [1] |
EDDY S R. Non-coding RNA genes and the modern RNA world[J]. Nature Reviews Genetics, 2001, 2(12): 919-929. DOI:10.1038/35103511 (  0) 0) |
| [2] |
QIN Tao, LI Juan, ZHANG Keqin. Structure, regulation, and function of linear and circular long non-coding RNAs[J]. Frontiers in Genetics, 2020, 11: 150. DOI:10.3389/fgene.2020.00150 (  0) 0) |
| [3] |
KUNG J T Y, COLOGNORI D, LEE J T. Long non-coding RNAs: Past, present, and future[J]. Genetics, 2013, 193(3): 651-669. DOI:10.1534/genetics.112.146704 (  0) 0) |
| [4] |
束永俊, 张晶红, 王明波, 等. 小麦长链非编码RNA的预测及功能分析[J]. 生物信息学, 2013, 11(2): 153-157. SHU Yongjun, ZHANG Jinghong, WANG Mingbo, et al. Computational identification and functional analysis of long non-coding RNA in Triticum aestivum[J]. Chinese Journal of Bioinformatics, 2013, 11(2): 153-157. DOI:10.3969/issn.1672-5565.2013.02.15 (  0) 0) |
| [5] |
ZHANG Jian, MUJAHID H, HOU Yuxuan, et al. Plant Long ncRNAs: A new frontier for gene regulatory control[J]. American Journal of Plant Sciences, 2013, 4(5): 1038-1045. DOI:10.4236/ajps.2013.45128 (  0) 0) |
| [6] |
WU Ling, LIU Sian, QI Haoran, et al. Research progress on plant long non-coding RNA[J]. Plants, 2020, 9(4): 408. DOI:10.3390/plants9040408 (  0) 0) |
| [7] |
蔡媛, 钟灿, 刘浩, 等. 植物长链非编码RNA的生物信息学预测与分析研究进展[J]. 生物信息学, 2019, 17(3): 151-160. CAI Yuan, ZHONG Can, LIU Hao, et al. Progress of bioinformatics prediction and analysis of long non-coding RNA in plants[J]. Chinese Journal of Bioinformatics, 2019, 17(3): 151-160. DOI:10.12113/j.issn.1672-5565.201812006 (  0) 0) |
| [8] |
RAI M I, ALAM M, LIGHTFOOT D A, et al. Classification and experimental identification of plant long non-coding RNAs[J]. Genomics, 2019, 111(5): 997-1005. DOI:10.1016/j.ygeno.2018.04.014 (  0) 0) |
| [9] |
SINGH U, KHEMKA N, RAJKUMAR M S, et al. PLncPRO for prediction of long non-coding RNAs(lncRNAs) in plants and its application for discovery of abiotic stress-responsive lncRNAs in rice and chickpea[J]. Nucleic Acids Research, 2017, 45(22): e183. DOI:10.1093/nar/gkx866 (  0) 0) |
| [10] |
常征, 孟军, 施云生, 等. 多特征融合的lncRNA识别与其功能预测[J]. 智能系统学报, 2018, 13(6): 928-934. CHANG Zheng, MENG Jun, SHI Yunsheng, et al. LncRNA recognition by fusing multiple features and its function prediction[J]. CAAI Transactions on Intelligent Systems, 2018, 13(6): 928-934. DOI:10.11992/tis.201806008 (  0) 0) |
| [11] |
陈思佟, 岑益, 柳建发, 等. 鉴定和预测长非编码RNAs的生物信息学方法[J]. 生命科学, 2015, 27(7): 946-952. CHEN Sitong, CEN Yi, LIU Jianfa, et al. Bioinformatics methods of identifying and predicting long noncoding RNAs[J]. Chinese Bulletin of Life Sciences, 2015, 27(7): 946-952. DOI:10.13376/j.cbls/2015131 (  0) 0) |
| [12] |
LI Weizhong, GODZIK A. Cd-hit: A fast program for clustering and comparing large sets of protein or nucleotide sequences[J]. Bioinformatics (Oxford, England), 2006, 22(13): 1658-1659. DOI:10.1093/bioinformatics/btl158 (  0) 0) |
| [13] |
CAO Zhen, PAN Xiaoyong, YANG Yang, et al. The lncLocator: a subcellular localization predictor for long non-coding RNAs based on a stacked ensemble classifier[J]. Bioinformatics, 2018, 34(13): 2185-2194. DOI:10.1093/bioinformatics/bty085 (  0) 0) |
| [14] |
DINGER M E, PANG K C, MERCER T R, et al. Differentiating protein-coding and noncoding RNA: challenges and ambiguities[J]. PLOS Computational Biology, 2008, 4(11). DOI:10.1371/journal.Pcbi.1000176 (  0) 0) |
| [15] |
HANADA K, AKIYAMA K, SAKURAI T, et al. sORF finder: A program package to identify small open reading frames with high coding potential[J]. Bioinformatics, 2010, 26(3): 399-400. DOI:10.1093/bioinformatics/btp688 (  0) 0) |
| [16] |
SCHNEIDER H W, RAIOL T, BRIGIDO M M, et al. A support vector machine based method to distinguish long non-coding RNAs from protein coding coding transcripts[J]. BMC Genomics, 2017, 18: 804. DOI:10.1186/s12864-017-4178-4 (  0) 0) |
| [17] |
WANG Guangyu, YIN Hongyan, LI Boyang, et al. Characterization and identification of long non-coding RNAs based on feature relationship[J]. Bioinformatics, 2019, 35(17): 2949-2956. DOI:10.1093/bioinformatics/btz008 (  0) 0) |
| [18] |
DESHPANDE S, SHUTTLEWORTH J, YANG J H, et al. PLIT: An alignment-free computational tool for identification of long non-coding RNAs in plant transcriptomic datasets[J]. Computers in Biology and Medicine, 2019, 105: 169-181. DOI:10.1016/j.compbiomed.2018.12.014 (  0) 0) |
| [19] |
MCGINNIS J L, DUNKLE J A, CATE J H D, et al. The mechanisms of RNA SHAPE chemistry[J]. Journal of the American Chemical Society, 2012, 134(15): 6617-6624. DOI:10.1021/ja2104075 (  0) 0) |
| [20] |
QUAN L J, CAI L X, CHEN Y, et al. Developing parallel ant colonies filtered by deep learned constrains for predicting RNA secondary structure with pseudo-knots[J]. Neurocomputing, 2020, 384: 104-114. DOI:10.1016/j.neucom.2019.12.041 (  0) 0) |
| [21] |
TORKAMANIAN-AFSHAR M, LANJANIAN H, NEMATZADEH S, et al. RPINBASE: An online toolbox to extract features for predicting RNA-protein interactions[J]. Genomics, 2020, 112(3): 2623-2632. DOI:10.1016/j.ygeno.2020.02.013 (  0) 0) |
| [22] |
HOFACKER I L, FONTANA W, STADLER P F, et al. Fast folding and comparison of RNA secondary structures[J]. Monatshefte für Chemie, 1994, 125(2): 167-188. DOI:10.1007/BF00818163 (  0) 0) |
| [23] |
XUE Changhai, LI Fei, HE Tao, et al. Classification of real and pseudo microRNA precursors using local structure-sequence features and support vector machine[J]. BMC Bioinformatics, 2005, 6: 310. DOI:10.1186/1471-2105-6-310 (  0) 0) |
| [24] |
常征. 深度学习在植物lncRNA识别中的研究与应用[D]. 大连: 大连理工大学, 2019. DOI: 10.26991/d.cnki.gdllu.2019.001858. CHANG Zheng. Research and application of deep learning in plant lncRNA recognition[D]. Dalian: Dalian University of Technology, 2019. DOI: 10.26991/d.cnki.gdllu.2019.001858. (  0) 0) |
| [25] |
CHEN Wei, ZHANG Xitong, BROOKER J, et al. PseKNC-General: Across-platform package for generating various modes of pseudo nucleotide compositions[J]. Bioinformatics, 2015, 31(1): 119-120. DOI:10.1093/bioinformatics/btu602 (  0) 0) |
| [26] |
CHEN Wei, LEI Tianyu, JIN Dianchuan, et al. PseKNC: A flexible web server for generating pseudo K-tuple nucleotide composition[J]. Analytical Biochemistry, 2014, 456: 53-60. DOI:10.1016/j.ab.2014.04.001 (  0) 0) |
| [27] |
CHEN Wei, LIN Hao, CHOU Kuochen. Pseudo nucleotide composition or PseKNC: an effective formulation for analyzing genomic sequences[J]. Molecular BioSystems, 2015, 11(10): 2620-2634. DOI:10.1039/c5mb00155b (  0) 0) |
| [28] |
GONI J R, PEREZ A, TORRENTS D, et al. Determining promoter location based on DNA structure first-principles calculations[J]. Genome Biology, 2007, 8(12): R263. DOI:10.1186/gb-2007-8-12-r263 (  0) 0) |
| [29] |
PEREZ A, NOY A, LANKAS F, et al. The relative flexibility of B-DNA and A-RNA duplexes: database analysis[J]. Nucleic Acids Research, 2004, 32(20): 6144-6151. DOI:10.1093/nar/gkh954 (  0) 0) |
| [30] |
PAN Xiaoyong, CHEN Lei, FENG Kaiyan, et al. Analysis of expression pattern of snoRNAs in different cancer types with machine learning algorithms[J]. International Journal of Molecular Sciences, 2019, 20(9): 2185. DOI:10.3390/ijms20092185 (  0) 0) |
| [31] |
CHANG C C, LIN C J. LIBSVM: a library for support vector machines[J]. ACM Transactions on Intelligent Systems and Technology, 2011, 2(3): 27. DOI:10.1145/1961189.1961199 (  0) 0) |
| [32] |
ZHAO Jian, SONG Xiaofeng, WANG Kai. lncScore: Alignment-free identification of long noncoding RNA from assembled novel transcripts[J]. Scientific Reports, 2016, 6: 34838. DOI:10.1038/srep34838 (  0) 0) |
| [33] |
LIU Bin, LIU Fule, WANG Xiaolong, et al. Pse-in-one: a web server for generating various modes of pseudo components of DNA, RNA, and protein sequences[J]. Nucleic Acids Research, 2015, 43(W1): W65-W71. DOI:10.1093/nar/gkv458 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


