2. 河南农业大学 农学院, 郑州 450002;
3. 深圳市作物分子设计育种研究院, 广东 深圳 518107
2. College of Agronomy, Henan Agricultural University, Zhengzhou 450002, China;
3. Shenzhen Institute of Molecular Crop Design, Shenzhen 518107, Guangdong, China
MYB转录因子是植物中最大的转录因子家族之一,广泛参与植物生理生化过程,包括植物表皮组织细胞分化、外界环境因素响应、激素应答等[1-2]。MYB家族成员都具有非常保守的MYB结构域。MYB结构域在三维空间中构成3个α-螺旋,第二个和第三个螺旋以三个色氨酸残基为疏水核心构成“螺旋-转角-螺旋”结构,MYB转录因子通过该结构与DNA结合[3]。MYB结构域可分为3种,分别为R1、R2和R3,它们在碱基数目和色氨酸保守性上有所不同。根据MYB结构域数量,MYB基因可分为1R-MYB、2R-MYB、3R-MYB和4R-MYB四个基因亚家族,其中1R-MYB的MYB结构域主要为R1或R3,2R-MYB主要为R2R3,3R-MYB主要为R1R2R3,4R-MYB主要为R1和R2[4-5]。
自PAZ-ARES等[6]第一次在玉米中发现植物MYB转录因子后,研究人员已先后对拟南芥和水稻[7]、西瓜[8]、棉花[9]、大豆[10]、中国枣[11]、苜蓿[12]等植物中MYB转录因子的功能进行了相关研究。研究表明,MYB转录因子的功能多种多样,对植物的生长发育至关重要。例如,MYB转录因子可通过控制各个细胞分裂时期来实现对细胞周期的调控[13-15],也可通过调节植物次生代谢相关基因的表达来调节花青素、类黄酮等次生代谢产物的合成[16-19],在非生物胁迫和生物胁迫响应中也扮演了重要的角色[20-27]。此外,MYB转录因子还能调控植株对植物激素的响应[28-29]。
栽培花生是世界上最重要的油料和粮食作物之一[30],研究花生功能基因对提升化生产量及品质具有重要意义。目前,对栽培花生MYB转录因子家族基因的整体研究较少,仅Chen等[31]鉴定了30个MYB转录因子,鉴定数量少,鉴定方法也有改良空间,因此,有必要进行更全面的鉴定和分析工作。鉴定及分析花生MYB基因家族的功能有助于为该类基因对花生的生长发育调控功能研究提供参考。2019年,Bertioli等[32]和Zhuang等[30]分别完成了栽培花生基因组的全基因组测序及组工作,为栽培花生全基因组范围MYB转录因子基因家族的系统鉴定、功能分析提供了条件。本研究基于花生的基因组序列信息,筛选并鉴定栽培花生MYB转录因子家族成员,分析其蛋白保守结构域、系统发育和共线性等信息,同时利用花生的转录组数据,研究MYB转录因子成员在栽培花生不同组织和不同发育时期的表达模式以及部分同源基因对的表达偏向,为后续的功能研究奠定基础。
1 材料与方法 1.1 数据来源RNA-Seq数据来源于NCBI项目PRJNA419393,参考基因组来源于花生数据网站PeanutBase(https://peanutbase.org)。
1.2 AhMYB基因家族成员鉴定通过以下途径获得候选AhMYB基因:1)在PeanutBase数据库中以“MYB”为关键字搜索栽培花生的基因注释信息;2)使用blastp比对花生蛋白序列和拟南芥已知MYB蛋白(e-value<1x10-3)。获得全部候选基因后,去除重复基因,提取最长蛋白序列,采用hmmsearch鉴定候选基因MYB结构域(e-value<1x10-5),同时提交至SMART、Pfam数据库,对MYB结构域可信度进行重复确认(SMART: e-value<1x10-5或PFam: e-value<1x10-8),最终确定可靠的AhMYB基因。
1.3 AhMYB基因家族系统发育分析提取所有候选花生MYB蛋白序列,对于有多个转录本的基因选取最长蛋白序列,使用ClustalW进行序列比对;利用MEGA 7.0根据邻接法(Neighbour-joining, NJ)构建MYB基因系统发育树,依据具有的MYB保守结构域数目和特征,将栽培花生的MYB转录因子归类到1R-MYB、2R-MYB、3R-MYB、4R-MYB4个亚家族中;根据栽培花生各MYB转录因子基因在染色体上的位置顺序信息对各基因进行命名。所有AhMYB的位置分布情况使用TBtools中的Gene distribution功能进行可视化。
1.4 AhMYB基因之间的同线性与部分同源基因对的确认通过blastn比对MYB基因序列(e-value <1x10-10),初步获得MYB基因之间的相似性,利用MCScanX进一步分析MYB转录因子基因之间的同线性关系(e-value <1x10-5),使用Circos对结果进行可视化。根据Bertioli等[32]发现的部分同源基因对和AhMYB基因同线性分析结果,确认AhMYB部分同源基因对。
1.5 AhMYB基因在各组织的表达特征分析为了分析花生MYB转录因子的时空表达模式,从NCBI下载了19个花生不同组织样品的RNA-seq数据(PRJNA419393),利用Salmon v1.0.0分析MYB基因在每个样品中的表达量,计算MYB转录因子在不同时空条件中的表达水平(Transcripts Per Million mapped reads,TPM值),使用R package: pheatmap展示MYB基因在各组织中的表达情况。
1.6 AhMYB部分同源基因对表达偏向确认使用R包DESeq2对同一组织中的同源基因对表达量进行分析,鉴定同源表达偏向的MYB基因对。利用Benjamini和Hochberg方法对P值进行调整以控制错误率。部分同源偏向表达基因对筛选条件为:1)调整后P值(FDR)<0.05;2)|差异倍数log2(fold change)| ≥1。使用R包pheatmap绘制相关热图。
1.7 AhMYB基因的功能富集分析根据AhMYB是否具有同源性以及所在基因对是否具有偏向性,将AhMYB分为四类,分别为A偏向性、B偏向性、无偏向性以及不成对基因,使用AgriGOv2[33](http://systemsbiology.cau.edu.cn/agriGOv2/)逐一进行功能富集分析。
2 结果分析 2.1 栽培花生中MYB家族成员的确定根据PeanutBase参考蛋白序列,选取每个基因最长的蛋白序列作为该基因的代表序列,通过hmmsearch、blast初步获得MYB转录因子成员,随后用SMART和Pfam确认MYB结构域(SANT结构域)是否存在,最终确定443个MYB基因(见表 1)。按照每个MYB转录因子具有的MYB保守结构域数量,将鉴定到的MYB转录因子分别归类到1R-MYB、2R-MYB、3R-MYB和4R-MYB类型,最终确定1R-MYB转录因子219个,2R-MYB(R2R3-MYB)转录因子209个,3R-MYB(R1R2R3-MYB)转录因子12个,4R-MYB转录因子3个。
| 表 1 花生MYB转录因子不同亚家族在花生染色体组上的分布情况 Table 1 Different subfamilies of AhMYB transcription factors distributed on cultivated peanut chromosomes |
依据基因注释信息,获得443个MYB基因在染色体上的分布情况(见表 1),并依据分布位置信息和基因组来源命名为AhMYB1A到AhMYB443B。其中,AhMYB1A至AhMYB217A分布于A亚基因组上,AhMYB218B至AhMYB443B分布在B亚基因组上,说明B基因组有更多的MYB基因。AhMYB基因在染色体上的分布和密度并不均匀。AhMYB基因数目最多的同源染色体对为8号(A08-B08)与3号(A03-B03),均有64个AhMYB基因。成员最少的染色体对是10号染色体对(AhA10-AhB10), 仅有25个AhMYB基因。此外,大多数AhMYB基因成簇分布于染色体两端,其中有15对基因形成串联重复基因(见图 1),如A3染色体上的AhMYB51A/AhMYB52A、B5的AhMYB313B/AhMYB314B、B8染色体的AhMYB386B/AhMYB387B等,这可能与染色体两端多处于常染色质状态,在减数分裂过程中更易发生基因重组交换和基因重复有关。

|
图 1 443个AhMYB基因在栽培花生基因组上的分布情况 Figure 1 Distribution of 443 AhMYB genes on cultivated peanut chromosomes 注:红颜色字体标注的基因代表串联重复基因。 Notes: Genes highlighted in red are tandem repeat. |
植物MYB转录因子的MYB结构域共有三种类型,分别为R1,R2以及R3,其中R1主要分布于1R-MYB转录因子中,R2和R3主要分布于2R-MYB转录因子中。在MYB结构域中,色氨酸残基是DNA结合域的疏水核心残基,对结合域空间结构的维持非常重要,也是关键的MYB结构域识别碱基[34]。为分析花生MYB转录因子的MYB保守结构域,对鉴定到的209个2R-MYB转录因子及219个1R-MYB转录因子的MYB结构域进行提取和分析。结果表明(见图 2),花生MYB结构域约含51个氨基酸残基。分析结果显示,在三种MYB结构域中,R2的3个色氨酸保守度最高,R3第一个保守色氨酸残基(W)会被苯丙氨酸(F)、异亮氨酸(I)或亮氨酸(L)替代;R1结构域第三个保守色氨酸残基会被亮氨酸(L)或丙氨酸(A)替代,第二个保守色氨酸残基也存在被脯氨酸(P)替代的情况。整体而言,三种MYB结构域的色氨酸残基均存在被其它疏水氨基酸替代的情况,但相较于其它氨基酸残基,色氨酸残基的保守程度依然是极高的。除了色氨酸残基,MYB结构域中还有其它保守性较高的氨基酸残基,如首个保守疏水残基近旁的谷氨酸(E)和天冬氨酸(D)。此外,R2和R3均具有特异的保守氨基酸,如R2结构域中靠近第三个色氨酸残基的精氨酸(R)和半胱氨酸(C),R3结构域中靠近第二个色氨酸残基的甘氨酸(G)和天冬酰胺(N)等。这些保守性较高的氨基酸残基很可能与保守的色氨酸残基共同维持花生MYB转录因子的DNA结合域结构,保证转录因子的功能。总体而言,在MYB结构域识别位点的保守性上,R1结构域的保守性最低,R3结构域保守性较高,R2结构域保守性最高。

|
图 2 花生MYB转录因子DNA结构域 Figure 2 DNA binding domain of MYB transcription factors in pea nut |
根据花生MYB转录因子蛋白序列以邻接法(Neighbor Joining)构建系统发育树,同时展示花生MYB转录因子上MYB结构域的分布情况(见图 3)。发育树显示,各亚家族总体上拥有各自的进化分枝,说明花生MYB转录因子在进化上有不同程度的分化。然而部分2R-MYB转录因子虽然拥有两个MYB结构域,但结构域的分布并不典型,如AhMYB62A、AhMYB259B、AhMYB205A、AhMYB424B等,蛋白序列中两个MYB结构域之间的距离较远,在进化上也更接近1R-MYB转录因子。同样地,少数1R-MYB转录因子在序列上也与2R-MYB转录因子更为相近。3R-MYB在进化上与2R-MYB更接近。在大多数分支末端中,MYB转录因子基因分别来自A、B基因,说明花生MYB转录因子的蛋白序列在亚基因组间具有相似性。
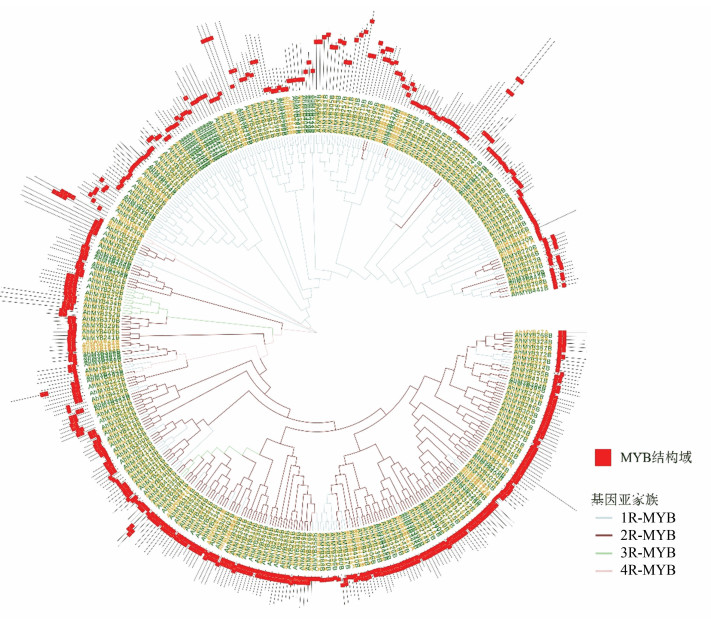
|
图 3 花生443个AhMYB基因系统发育 Figure 3 Phylogenetic analysis of 443 AhMYB genes 注:橙色基因名代表A基因组基因,绿色基因名代表B基因组基因。 Notes: Orange name represents genes from A genome, and green name represents genes from B genome. |
花生基因组内部AhMYB基因也存在大量亚基因组内的旁系同源关系,可能由染色体大片段重复而来,使AhMYB基因家族得到了进一步的扩张(见图 4(a),图 4(b))。此外,A、B基因组之间存在大量的AhMYB直系同源基因(见图 4(c))。在这些同源基因中,存在染色体定位对应关系的基因对即构成部分同源基因对(homoeologous gene pair)[35]。结合Bertioli等[32]的研究结果和同线性分析结果,我们共鉴定到170对AhMYB同源基因对(见图 4(c)),涵盖了327个AhMYB基因和13个与AhMYB基因配对但无完整MYB结构域的MYB-like基因,这说明MYB转录因子在祖先种杂交加倍进化形成的栽培花生亚基因组水平上保持了较高的相似性。
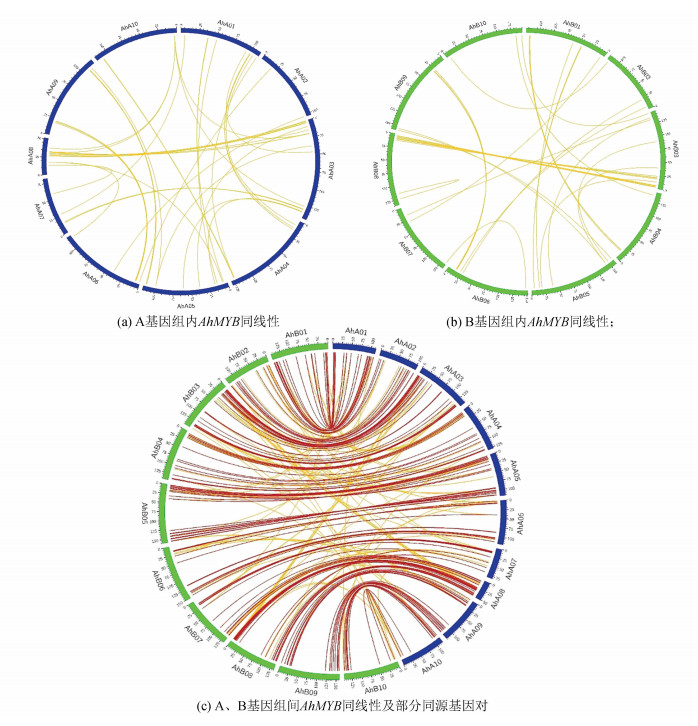
|
图 4 AhMYB基因同线性关系 Figure 4 Synteny of AhMYB genes 注:红色代表部分同源基因对,黄色代表A/B基因组不同染色体上的基因同线性。 Notes: Red lines represent homeologous gene pairs; yellow lines represent gene synteny on different chromosomes between A/B genome. |
MYB基因的表达与植物的生长发育密切相关。利用转录组数据分析MYB基因的时空表达模式能够为探究MYB转录因子的功能提供线索。转录组数据来自Clevenger等[36]的研究,其样品共有19个组织和时期,分别是属于营养生长组织的根瘤、根、茎(主茎尖、侧茎尖)、幼芽叶、叶(主茎叶、侧茎叶)和属于生殖生长组织的花、雌蕊、雄蕊、荚针(地上、地下)、荚皮(3期、5期)、荚果(3期、5期、6期、7期、8期、10期)。根据部分同源基因对配对情况,AhMYB基因被分为“配对”和“无配对”两组,共获得116个无配对的AhMYB基因和170对AhMYB部分同源基因对。结果表明,不同AhMYB基因的表达模式存在非常大的差异(见图 5)。在116个无配对的AhMYB基因中(见图 5(a)),有21个基因在19个组织中均维持低表达量(TPM <1),有20个基因在19个组织中均维持较高表达量(TPM ≥ 1),只在营养生长组织中或生殖生长组织中高或较高表达的无配对AhMYB基因分别有8个和13个。在170对AhMYB部分同源基因对中(见图 5(b)),有9对在19个组织中均维持低表达量,有29对在19个组织中均维持较高表达量,只在营养生长组织或生殖生长组织中高或较高表达的AhMYB基因对分别有21对和14对,以上结果表明,不同的AhMYB基因在不同的时空条件下发挥转录调控的功能,其功能很可能差异且互补。
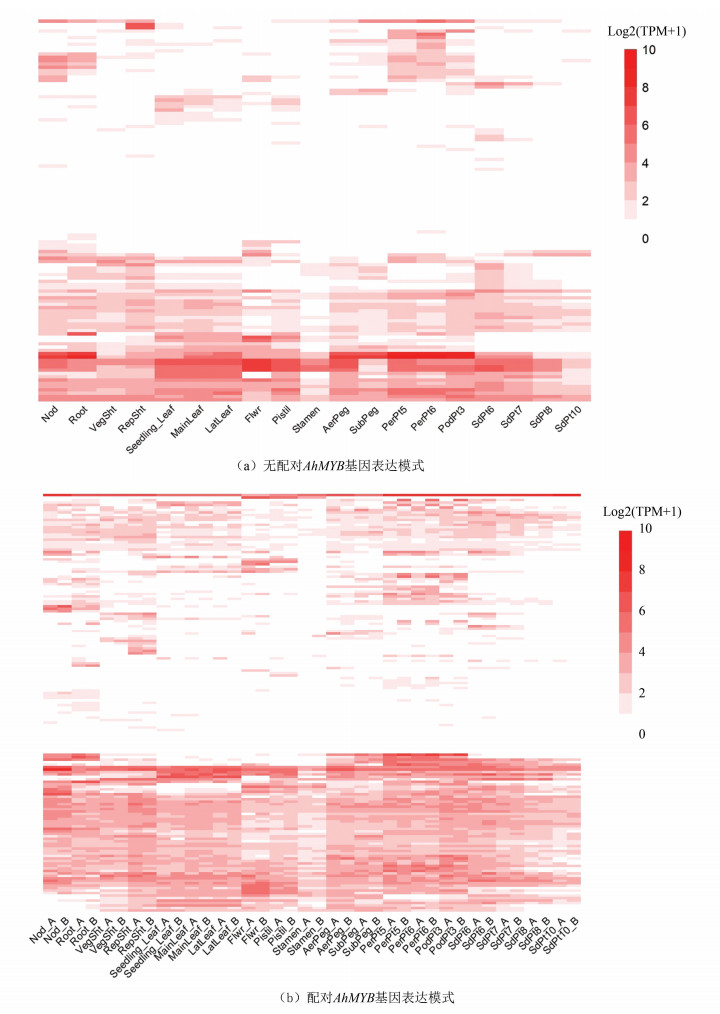
|
图 5 AhMYB基因各组织表达分析 Figure 5 Expression analysis of AhMYB genes in diverse tissues and different stages 注:TPM:每百万转录本,相对表达量计量单位。组织类型(从左至右):Nod,根瘤;Root,根;VegSht,主茎;RepSht,侧茎;Seedling_Leaf,幼芽叶;Main_Leaf,主茎叶Lat_Leaf,侧茎叶;Flwr,花;Pistil,雌蕊;Stamen,雄蕊;AerPeg,地上荚针;SubPeg,地下荚针;PerPt5,荚皮(第5时期);PerPt6,荚皮(第6时期);PodPt3,荚果(第5时期);SdPt6,荚果(第6时期);SdPt7,荚果(第7时期);SdPt8,荚果(第8时期);SdPt10,荚果(第10时期)。 Notes: TPM: transcript per million, unit of relative expression level. The tissues are (from left to right): Nod, nodule; Root, root; VegSht, vegetative shoot tip; RepSht, reproductive shoot tip; Seedling_Leaf, seedling leaf; Main_Leaf, main stem leaf; Lat_Leaf, lateral stem leaf; Flwr, flower; Pistil, pistil; Stamen, stamen; AerPeg, aerial gynophore tip; SubPeg, subterranean gynophore tip; PerPt5, pattee 5 pericarp; PerPt6, pattee 6 pericarp; PodPt3, pattee 3 pod; SdPt6, pattee 6 seed; SdPt7, pattee 7 seed; SdPt8, pattee 8 seed; SdPt10, pattee 10 seed. |
花生是一个复杂的异源四倍体,理论上,每一个花生共同祖先的基因在花生的基因组中都分别在A、B基因组上有部分同源基因,它们很可能对植株的表型有不同的贡献。在此前的研究中,Bertioli等[32]确认了花生中15 328对部分同源基因对。结合该数据和同线性分析记过,我们从中找出了由MYB组成的部分同源基因对共170对(见图 4(c))。表达偏向分析的结果显示,在170对AhMYB部分同源基因中,有72对在至少1个组织中表现出表达偏向性(见图 6)。在这72对基因中,有34对表现出完全的A基因组倾向,如AhMYB217A-AhMYB442B、AhMYB73A-AhMYB298B和AhMYB66A-AhMYB290B,它们分别在18个、7个和4个组织中表现出表达偏向性,且全部高表达A基因组基因;有33对表现出完全的B基因组倾向,如AhMYB80A-AhMYB305B、AhMYB73A-AhMYB298B和AhMYB66A-AhMYB290B,它们分别在11个、17个和9个组织中呈现表达偏向性,且全部高表达B基因组基因。还有5对基因在不同组织中呈现了不同的表达偏向性,如AhMYB94A-AhMYB318B,其在10个组织和发育时期中呈现表达偏向性,其中9个组织和发育时期内主要表达AhMYB318B,但在雌蕊中主要表达AhMYB94A,以及AhMYB56A-AhMYB282B,其在根瘤中高表达B基因组的AhMYB282B,在侧茎叶中高表达A基因组的AhMYB56A。
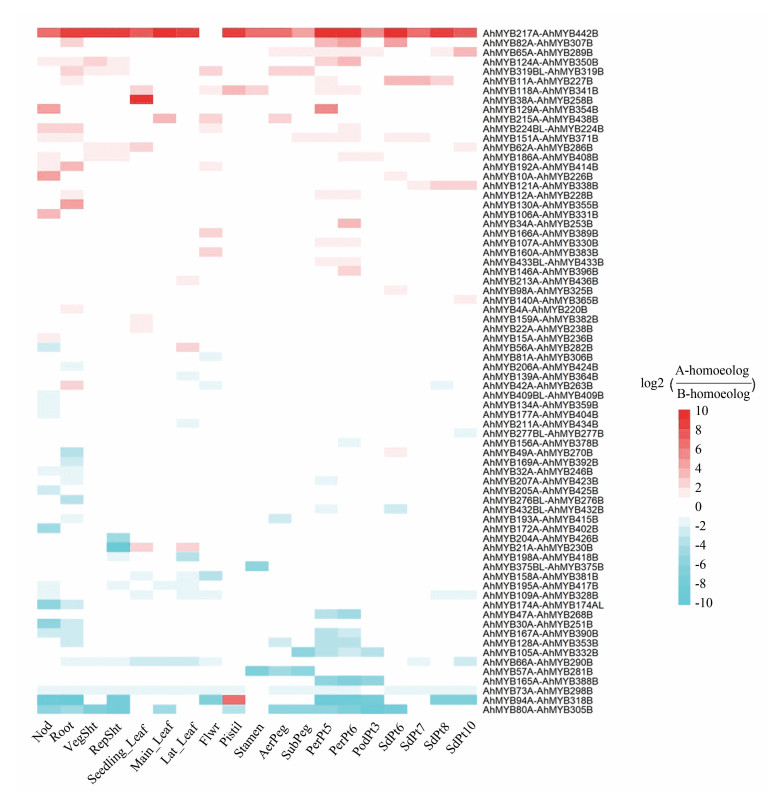
|
图 6 72对AhMYB部分同源基因对在19个组织中的部分同源表达偏向 Figure 6 Homoeologous expression bias of 72 AhMYB homoeologous gene pairs in 19 tissues |
依照是否存在部分同源基因以及所在基因对的表达偏向性,将AhMYB基因分为A基因组偏向性基因对、B基因组偏向性基因、无基因组偏向性基因、无配对基因4类。功能富集分析(见图 7)显示,四类AhMYB基因在功能上既有共同之处,也有独特之处。四类AhMYB基因在生长素响应、脱落酸响应、乙烯响应、赤霉素响应、水杨酸响应等激素响应进程中均有高富集度,意味着大多数AhMYB基因能响应植物激素的刺激,在次级细胞壁生物合成调控中也高度富集。此外,四类AhMYB基因均有特定的功能富集条目,如A偏向性AhMYB基因对在细胞分裂素依赖的信号通路中显著富集,B偏向性AhMYB基因对则在根部发育调控、分生组织调控和茎组织发育等功能中显著富集,无偏向性AhMYB基因对在聚氨类分解代谢、香豆素生物合成、花粉精细胞分化、苯丙烷代谢调控以及花药壁绒毡层发育等进程中显著富集,而无配对AhMYB基因在长日照光周期开花、节律等生物进程中显著富集。此外,无偏向性基因和无配对基因富集在赤霉素合成、细胞程序性死亡等功能,而受伤响应、缺水响应只有A偏向性AhMYB基因没有显著富集,磷酸转移信号传导功能只有B偏向性AhMYB基因没有显著富集。总体而言,每一类AhMYB都有其特有的富集功能,也存在共有的富集功能。
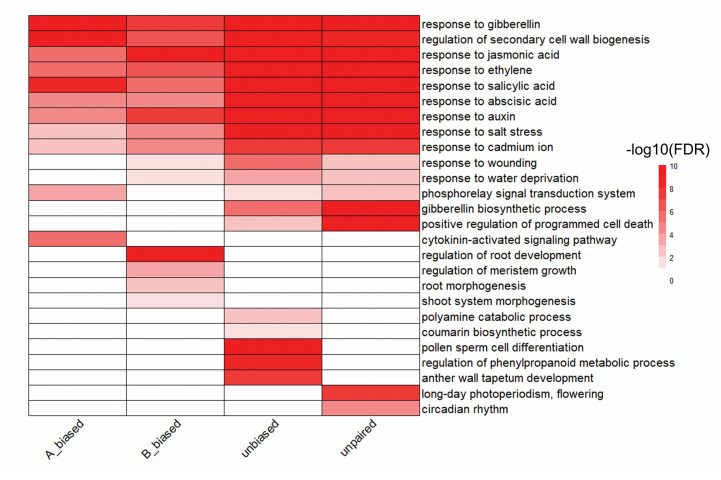
|
图 7 4类AhMYB基因的功能富集分析 Figure 7 GO enrichment analysis of four groups of AhMYB genes |
植物MYB转录因子共存在三种MYB结构域[4],其中R1在间隔R2结构域在色氨酸残基之间多为19个间隔残基,R3结构域在色氨酸残基之间多为18个间隔残基[2]。对花生MYB转录因子三种MYB结构域的序列分析结果显示,花生中R1结构域的保守程度最低,三个关键的色氨酸残基均存在被替换的现象,与大多数高等植物中的R1结构域情况相似;R2包含3个极度保守的色氨酸残基,但色氨酸残基之间的氨基酸数目为20个,而R3第一个色氨酸残基虽然存在替换情况,但第二、三色氨酸的保守程度非常高,且三个保守残基之间间隔的氨基酸残基均为18个,与多数高等植物中的R3结构域相同。经过Pfam与SMART数据库的验证,共发现443个AhMYB基因。对所有AhMYB基因的MYB结构域数量进行统计后,共发现219个1R-MYB蛋白、209个2R-MYB蛋白、12个3R-MYB蛋白以及3个4R-MYB蛋白。MYB转录因子的MYB结构域多靠近蛋白序列的N端[2, 4-5],但本次研究发现的花生MYB转录因子中,部分1R-MYB蛋白的MYB结构域位于C端附近,如AhMYB37A、AhMYB189A、AhMYB257B、AhMYB411B等。根据Du等对1R-MYB蛋白的分类[37],这些MYB转录因子很可能属于1R-MYB亚家族中的类TBP亚组(TPB-like subgroup),而MYB结构域分布于蛋白序列中部区域的1R-MYB蛋白,如AhMYB148A、AhMYB179A、AhMYB368B、AhMYB405B等,则很可能属于类CCA1/R-R亚组(CCA1-like/R-R subgroup)。这些结果说明1R-MYB转录因子无论是在MYB结构域的相对位置还是MYB结构域的序列上,其多样性均高于2R-MYB转录因子,很可能承载了更多的花生进化信息。此外,花生的MYB转录因子家族中1R-MYB成员数目高于2R-MYB成员数目,这与多数植物中MYB转录因子有所不同,表明花生很可能经历了更为特殊的进化、驯化过程。不过,少数1R-MYB转录因子在系统发育树分支上与2R-MYB转录因子相邻(见图 1),说明部分1R-MYB转录因子在序列上与2R-MYB转录因子相似。考虑到MYB结构域信息很可能由于转录本的错误拼接而缺失,导致MYB结构域数目减少,最终影响其分类并使得花生MYB转录因子家族成员中的1R-MYB多于2R-MYB,不能排除该部分1R-MYB应为2R-MYB的可能性,因此对于各类MYB转录因子数量的确认工作仍有提升空间。
3.2 AhMYB基因的表达模式基因表达模式、表达丰度与基因的功能密切相关,通过研究不同组织和发育时期中AhMYB基因的表达情况可以发现特定组织和时期中发挥作用的功能基因。已有研究均证实MYB转录因子参与多种植物的生命活动,Pan等[38]发现,只在水稻花药中相对高表达的OsMYB80有多个潜在的调控基因,敲除OsMYB80会引起绒毡层细胞凋亡,导致雄性不育;BnTT8主要在油菜种子各发育时期表达,在bntt8突变体中,油菜种子的脂肪酸生物合成受到了极大的影响[17]。基于转录组数据,本研究发现部分AhMYB基因在不同组织和种子发育时期特异表达。例如AhMYB159A和AhMYB382B在根部高表达;AhMYB114A和AhMYB347B在花中表达量极高;AhMYB25A、AhMYB143A和AhMYB388B则在荚皮中高表达。这些特定时空条件下高表达丰度的基因很可能与对应组织的发育生长或特定的次生代谢活动密切相关。此外,本研究还发现AhMYB92A和AhMYB317B在19个组织和发育时期中均呈现高或较高的表达量,同时这两个基因还组成部分同源基因对,由此推测它们很可能组成型参与多种生物学途径的调控,与花生的正常生长发育高度相关。
3.3 花生MYB转录因子的部分同源表达偏向异源多倍化能够引起编码或非编码DNA序列缺失、表观遗传修饰和基因表达模式的改变[39],且不同的基因组很可能受到不同程度的影响[40],是植物进化的重要动力[41],花生的形成就是异源多倍化成功推动物种形成的经典例子之一。异源多倍化在转录组水平上引起的变化表现为部分同源基因偏向性表达。异源多倍体中,每一对部分同源基因来源于共同祖先中的同一基因,因此理论上部分同源基因对的个体在表达水平上应该相似。然而,在多倍体物种中,普遍存在同源基因、部分同源基因的偏向性表达[41-43]。在AhMYB基因中,我们确认了170对包含MYB转录因子的部分同源基因对。在这170对花生MYB转录因子部分同源基因对中,有72对在至少一个组织中表现出部分同源表达偏向,其中A偏向性与B偏向性的基因对均为35对。功能富集分析结果显示,A基因组偏向性的AhMYB基因对特异地富集在细胞分裂素依赖的信号通路中,B基因组偏向性的AhMYB基因对则在根形态发育、茎形态发育等功能中显著富集。不同基因组偏向性的AhMYB在功能上展现了一定的富集差异,说明不同亚基因组中的AhMYB基因在部分功能上很可能受到了不同程度的选择压力,使得某种偏向性的基因对在特定功能中出现富集。此外,5对部分同源基因对在不同组织中表现不同的基因组偏向性,这说明它们的转录本序列虽然高度相似,但很可能经历了亚功能化或者产生了新功能。
4 结论本研究在栽培花生中共鉴定到443个MYB转录因子基因,包括219个1R-MYB、209个2R-MYB、12个3R-MYB以及3个4R-MYB,它们在MYB结构域具有各自独特的保守性。这些MYB转录因子基因中,共有170对部分同源基因对,其中72对在至少一个组织和发育时期中表现出偏向性表达。具有表达偏向性的基因和无偏向性的基因在功能上具有明显不同的富集。本研究为AhMYB基因功能的深入研究奠定了一定的基础。
| [1] |
MILLARD P S, KRAGELUND B B, BUROW M. R2R3 MYB transcription factors-functions outside the DNA-binding domain[J]. Trends in Plant Science, 2019, 24(10): 934-946. DOI:10.1016/j.tplants.2019.07.003 (  0) 0) |
| [2] |
DU H, ZHANG L, LIU L, et al. Biochemical and molecular characterization of plant MYB transcription factor family[J]. Biochemistry (Moscow), 2009, 74(1): 1-11. DOI:10.1134/s0006297909010015 (  0) 0) |
| [3] |
OGATA K, KANEI-ISHII C, SASAKI M, et al. The cavity in the hydrophobic core of Myb DNA-binding domain is reserved for DNA recognition and trans-activation[J]. Nature Structural Biology, 1996, 3(2): 178-187. DOI:10.1038/nsb0296-178 (  0) 0) |
| [4] |
STRACKE R, WERBER M, WEISSHAAR B. The R2R3-MYB gene family in Arabidopsis thaliana[J]. Current Opinion in Plant Biology, 2001, 4(5): 447-456. DOI:10.1016/s1369-5266(00)00199-0 (  0) 0) |
| [5] |
DUBOS C, STRACKE R, GROTEWOLD E, et al. MYB transcription factors in Arabidopsis[J]. Trends in Plant Science, 2010, 15(10): 573-581. DOI:10.1016/j.tplants.2010.06.005 (  0) 0) |
| [6] |
PAZ-ARES J, GHOSAL D, WIENAND U, et al. The regulatory c1 locus of Zea mays encodes a protein with homology to MYB proto-oncogene products and with structural similarities to transcriptional activators[J]. EMBO Journal, 1987, 6(12): 3553-3558. (  0) 0) |
| [7] |
KATIYAR A, SMITA S, LENKA S K, et al. Genome-wide classification and expression analysis of MYB transcription factor families in rice and Arabidopsis[J]. BMC Genomics, 2012, 13: 544. DOI:10.1186/1471-2164-13-544 (  0) 0) |
| [8] |
XU Q, HE J, DONG J, et al. Genomic survey and expression profiling of the MYB gene family in watermelon[J]. Horticultural Plant Journal, 2018, 4(1): 1-15. DOI:10.1016/j.hpj.2017.12.001 (  0) 0) |
| [9] |
SALIH H, GONG W, HE S, et al. Genome-wide characterization and expression analysis of MYB transcription factors in Gossypium hirsutum[J]. BMC Genetics, 2016, 17(1): 129. DOI:10.1186/s12863-016-0436-8 (  0) 0) |
| [10] |
DU H, YANG S S, LIANG Z, et al. Genome-wide analysis of the MYB transcription factor superfamily in soybean[J]. BMC Plant Biology, 2012, 12: 106. DOI:10.1186/1471-2229-12-106 (  0) 0) |
| [11] |
JI Q, WANG D W, ZHOU J, et al. Genome-wide characterization and expression analyses of the MYB superfamily genes during developmental stages in Chinese jujube[J]. PeerJ, 2019, 7: e6353. DOI:10.7717/peerj.6353 (  0) 0) |
| [12] |
ZHOU Q, JIA C, MA W, et al. MYB transcription factors in alfalfa (Medicago sativa): Genome-wide identification and expression analysis under abiotic stresses[J]. PeerJ, 2019, 7: e7714. DOI:10.7717/peerj.7714 (  0) 0) |
| [13] |
MU R L, CAO Y R, LIU Y F, et al. An R2R3-type transcription factor gene AtMYB59 regulates root growth and cell cycle progression in Arabidopsis[J]. Cell Research, 2009, 19(11): 1291-1304. DOI:10.1038/cr.2009.83 (  0) 0) |
| [14] |
XIE Z, LEE E, LUCAS J R, et al. Regulation of cell proliferation in the stomatal lineage by the Arabidopsis MYB FOUR LIPS via direct targeting of core cell cycle genes[J]. Plant Cell, 2010, 22(7): 2306-2321. DOI:10.1105/tpc.110.074609 (  0) 0) |
| [15] |
HAGA N, KOBAYASHI K, SUZUKI T, et al. Mutations in MYB3R1 and MYB3R4 cause pleiotropic developmental defects and preferential down-regulation of multiple G2/M-specific genes in Arabidopsis[J]. Plant Physiology, 2011, 157(2): 706-717. DOI:10.1104/pp.111.180836 (  0) 0) |
| [16] |
LOTKOWSKA M E, TOHGE T, FERNIE A R, et al. The Arabidopsis transcription factor MYB112 promotes anthocyanin formation during salinity and under high light stress[J]. Plant Physiology, 2015, 169(3): 1862-1880. DOI:10.1104/pp.15.00605 (  0) 0) |
| [17] |
ZHAI Y, YU K, CAI S, et al. Targeted mutagenesis of BnTT8 homologs controls yellow seed coat development for effective oil production in Brassica napus L[J]. Plant Biotechnology Journal, 2019, 18: 1153-1168. DOI:10.1111/pbi.13281 (  0) 0) |
| [18] |
ZHAO Y, MA J, LI M, et al. Whole-genome resequencing-based QTL-seq identified AhTc1 gene encoding a R2R3-MYB transcription factor controlling peanut purple testa colour[J]. Plant Biotechnology Journal, 2020, 18(1): 96-105. DOI:10.1111/pbi.13175 (  0) 0) |
| [19] |
ZONG Y, ZHU X, LIU Z, et al. Functional MYB transcription factor encoding gene AN2 is associated with anthocyanin biosynthesis in Lycium ruthenicum Murray[J]. BMC Plant Biology, 2019, 19(1): 169. DOI:10.1186/s12870-019-1752-8 (  0) 0) |
| [20] |
ZHU N, CHENG S, LIU X, et al. The R2R3-type MYB gene OsMYB91 has a function in coordinating plant growth and salt stress tolerance in rice[J]. Plant Science, 2015, 236: 146-156. DOI:10.1016/j.plantsci.2015.03.023 (  0) 0) |
| [21] |
ZHANG L, SONG Z, LI F, et al. The specific MYB binding sites bound by TaMYB in the GAPCp2/3 promoters are involved in the drought stress response in wheat[J]. BMC Plant Biology, 2019, 19(1): 366. DOI:10.1186/s12870-019-1948-y (  0) 0) |
| [22] |
ZHANG J, YIN X R, LI H, et al. Ethylene response factor 39-MYB8 complex regulates low-temperature-induced lignification of loquat fruit[J]. Journal of Experimental Botany, 2020, 71(10): 3172-3184. DOI:10.1093/jxb/eraa085 (  0) 0) |
| [23] |
SONG Z, LUO Y, WANG W, et al. NtMYB12 positively regulates flavonol biosynthesis and enhances tolerance to low Pi stress in Nicotiana tabacum[J]. Frontiers in Plant Science, 2019, 10: 1683. DOI:10.3389/fpls.2019.01683 (  0) 0) |
| [24] |
SEO P J, PARK C M. MYB96-mediated abscisic acid signals induce pathogen resistance response by promoting salicylic acid biosynthesis in Arabidopsis[J]. New Phytologist, 2010, 186(2): 471-483. DOI:10.1111/j.1469-8137.2010.03183.x (  0) 0) |
| [25] |
RAFFAELE S, RIVAS S, ROBY D. An essential role for salicylic acid in AtMYB30-mediated control of the hypersensitive cell death program in Arabidopsis[J]. FEBS Letters, 2006, 580(14): 3498-3504. DOI:10.1016/j.febslet.2006.05.027 (  0) 0) |
| [26] |
PERSAK H, PITZSCHKE A. Dominant repression by Arabidopsis transcription factor MYB44 causes oxidative damage and hypersensitivity to abiotic stress[J]. International Journal of Molecular Sciences, 2014, 15(2): 2517-2537. DOI:10.3390/ijms15022517 (  0) 0) |
| [27] |
LIU Z, LUAN Y, LI J, et al. Expression of a tomato MYB gene in transgenic tobacco increases resistance to Fusarium oxysporum and Botrytis cinerea[J]. European Journal of Plant Pathology, 2016, 144(3): 607-617. DOI:10.1007/s10658-015-0799-0 (  0) 0) |
| [28] |
WEI X, LU W, MAO L, et al. ABF2 and MYB transcription factors regulate feruloyl transferase FHT involved in ABA-mediated wound suberization of kiwifruit[J]. Journal of Experimental Botany, 2020, 71(1): 305-317. DOI:10.1093/jxb/erz430 (  0) 0) |
| [29] |
KU Y S, NI M, MUNOZ N B, et al. GmABAS1 from soybean is a 1R-subtype MYB transcriptional repressor that enhances ABA sensitivity[J]. Journal of Experimental Botany, 2020, 71(10): 2970-2981. DOI:10.1093/jxb/eraa081 (  0) 0) |
| [30] |
ZHUANG W, CHEN H, YANG M, et al. The genome of cultivated peanut provides insight into legume karyotypes, polyploid evolution and crop domestication[J]. Nature Genetics, 2019, 51(5): 865-876. DOI:10.1038/s41588-019-0402-2 (  0) 0) |
| [31] |
CHEN N, YANG Q, PAN L, et al. Identification of 30 MYB transcription factor genes and analysis of their expression during abiotic stress in peanut (Arachis hypogaea L.)[J]. Gene, 2014, 533(1): 332-345. DOI:10.1016/j.gene.2013.08.092 (  0) 0) |
| [32] |
BERTIOLI D J, JENKINS J, CLEVENGER J, et al. The genome sequence of segmental allotetraploid peanut Arachis hypogaea[J]. Nature Genetics, 2019, 51(5): 877-884. DOI:10.1038/s41588-019-0405-z (  0) 0) |
| [33] |
TIAN T', LIU Y, YAN H Y, et al. agriGO v2.0: a GO analysis toolkit for the agricultural community, 2017 update[J]. Nucleic Acids Research, 2017, 45(W1): W122-W129. DOI:10.1093/nar/gkx382 (  0) 0) |
| [34] |
ROSINSKI J A, ATCHLEY W R. Molecular evolution of the Myb family of transcription factors: evidence for polyphyletic origin[J]. Journal of Molecular Evolution, 1998, 46(1): 74-83. DOI:10.1007/pl00006285 (  0) 0) |
| [35] |
GLOVER N M, REDESTIG H, DESSIMOZ C. Homoeologs: What are they and how do we infer them?[J]. Trends in Plant Science, 2016, 21(7): 609-621. DOI:10.1016/j.tplants.2016.02.005 (  0) 0) |
| [36] |
CLEVENGER J, CHU Y, SCHEFFLER B, et al. A developmental transcriptome map for allotetraploid Arachis hypogaea[J]. Frontiers in Plant Science, 2016, 7: 1446. DOI:10.3389/fpls.2016.01446 (  0) 0) |
| [37] |
DU H, WANG Y B, XIE Y, et al. Genome-wide identification and evolutionary and expression analyses of MYB-related genes in land plants[J]. DNA Research, 2013, 20(5): 437-448. DOI:10.1093/dnares/dst021 (  0) 0) |
| [38] |
PAN X, YAN W, CHANG Z, et al. OsMYB80 regulates anther development and pollen fertility by targeting multiple biological pathways[J]. Plant & Cell Physiology, 2020, 61(5): 988-1004. DOI:10.1093/pcp/pcaa025 (  0) 0) |
| [39] |
ZHANG H, BIAN Y, GOU X, et al. Persistent whole-chromosome aneuploidy is generally associated with nascent allohexaploid wheat[J]. Proceedings of the National Academy of Sciences, USA, 2013, 110(9): 3447-3452. DOI:10.1073/pnas.1300153110 (  0) 0) |
| [40] |
LI A L, GENG S F, ZHANG L Q, et al. Making the bread: insights from newly synthesized allohexaploid wheat[J]. Molecular Plant, 2015, 8(6): 847-859. DOI:10.1016/j.molp.2015.02.016 (  0) 0) |
| [41] |
WEI J, CAO H, LIU J D, et al. Insights into transcriptional characteristics and homoeolog expression bias of embryo and de-embryonated kernels in developing grain through RNA-Seq and Iso-Seq[J]. Functional and Integrative Genomics, 2019, 19(6): 919-932. DOI:10.1007/s10142-019-00693-0 (  0) 0) |
| [42] |
YOO M J, SZADKOWSKI E, WENDEL J F. Homoeolog expression bias and expression level dominance in allopolyploid cotton[J]. Heredity, 2013, 110(2): 171-180. DOI:10.1038/hdy.2012.94 (  0) 0) |
| [43] |
WU J, LIN L, XU M, et al. Homoeolog expression bias and expression level dominance in resynthesized allopolyploid Brassica napus[J]. BMC Genomics, 2018, 19(1): 586. DOI:10.1186/s12864-018-4966-5 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


