2. 天津康复疗养中心 检验病理科,天津 300381;
3. 解放军总医院 第二医学中心血液科 国家老年疾病临床医学研究中心,北京 100853;
4. 同济大学附属东方医院 中医科,上海 200120
2. Department of Laboratory Pathology, Tianjin Rehabilitation and Sanatorium Center, Tianjin 300381, China;
3. Department of Hematology, Second Medical Center, PLA General Hospital, Beijing 100853, China;
4. Department of Traditional Chinese Medicine, Oriental Hospital of Tongji University, Shanghai 200120, China
2019年12月,武汉发生多起不明原因肺炎。目前,已确认该病的爆发是由严重急性呼吸综合征冠状病毒2(SARS-CoV-2)引起,此病毒与引起严重急性呼吸综合征(SARS)和中东呼吸综合征(MERS)的病毒高度同源。此前临床观察认为,细胞因子升高诱发的细胞因子风暴可能是导致患者病情加重的原因,但这一认识存在局限性,并不能解释全部临床症状[1-3]。目前对于危重型COVID-19患者除对症支持治疗外,缺乏有效的干预措施,尽快确定药物治疗方案对于防止轻型、普通型患者转为重型、改善危重型患者预后具有关键作用。氯雷他定作为新一代抗组胺药物,不仅能够拮抗组胺受体,而且对其他免疫细胞和免疫因子均有抑制作用[4]。既往研究表明,氯雷他定作为抗组胺药物,能够减少炎症递质如白三烯的释放,抑制细胞间粘附分子的表达以及降低IL-6,IL-8等细胞因子产生[5],并且与热毒宁联用具有降低病毒易感性和严重程度的作用[6],推测其对COVID-19可能具有潜在的治疗作用。鉴于SARS-CoV-2的组学数据分析尚不完善,为进一步探明氯雷他定的体内分子机制,我们运用临床生物信息学方法,以本课题组前期建立的EpiMed平台为基础,通过分析氯雷他定对全基因组表达谱的影响,进行临床生物信息学分析,并结合EpiMed平台的预测结果,探讨其抗SARS-CoV-2的潜在分子机制,为后续制定和优化含氯雷他定的COVID-19治疗方案提供理论依据。
1 材料和方法 1.1 材料 1.1.1 数据来源以“Loratadine”为检索词在GEO数据库中进行检索,在条目类型中选择“series”,共得到10个检索结果,为进一步提高数据的准确性和可靠性,对检索结果进行二次筛选,筛选条件为:①物种:人或与人基因同源性高的物种(小鼠、大鼠等)。②实验设计:三组或三组以上的重复实验。③数据类型:转录组数据或全基因组表达谱数据③数据质量:数据完整以及对应的平台注释文件标准、规范。在经过仔细筛选后,选出符合条件的数据:GSE59927。GSE59927数据集的作用对象均为大鼠,其中包含了超过600种不同药物或化合物样本的表达矩阵,涵盖了氯雷他定作用于大鼠心脏和原代肝细胞的全基因组表达谱数据。
1.1.2 实验设计首先,根据氯雷他定的作用部位、作用时间以及药物剂量,将筛选后的数据分为5组(见表 1)。将正常大鼠的心脏(或原代肝细胞)作为对照组,经过氯雷他定处理的大鼠心脏(或原代肝细胞)作为实验组,进行差异分析。氯雷他定作用部位、作用时间以及药物剂量皆为GSE59927中的原始信息,考虑到药物在不同细胞系、剂量以及时间的影响不同,去除5组差异基因中调控作用相反的交集基因,并使用R语言程序对5组差异基因调控作用相同的基因表达量取平均值,得到的结果用于后续分析。
| 表 1 样本分组情况表 Table 1 Sample grouping table |
在Bioconductor(http://www.bioconductor.org/)网站下载R语言程序包,用于数据处理和差异分析[7]。首先使用Impute程序包估算表达矩阵中的缺失值[8],使用biomaRt包对基因进行同源转化[9],然后使用limma程序包对数据进行标准化,并筛选差异基因[10]。差异基因筛选设定阈值为log2(Fold Change)>1,P < 0.05。
1.2.2 差异分析结果整理使用Venny 2.1(https://bioinfogp.cnb.csic.es/tools/venny/)[11]在线工具,去除5组差异基因中调控作用相反的交集基因,并使用R语言程序对5组差异基因调控作用相同的基因表达量取平均值。
1.2.3 构建差异基因的蛋白互作网络将差异基因上传至STRING v11.0(https://string-db.org/)在线分析网站进行蛋白质相互作用(protein-protein interaction,PPI)网络分析,以置信度>0.7作为阈值进行筛选[12]。关键基因是指PPI网络中具有高度相关的基因。在本研究中,关键基因为在PPI网络关系中出现频次最高的前10个基因,使用R软件,通过Degree算法筛选出关键基因,结果输出得分排名前10的基因[13]。
1.2.4 富集分析使用R语言clusterProfiler包对差异基因进行GO富集分析以及KEGG通路富集分析[14],并使用R语言程序对聚类结果进行可视化处理。
1.2.5 疾病与药物关联分析使用本研究团队基于“整合多组学分析”算法建立的临床生物信息学表观精准治疗预测平台(EpiMed),对差异基因进行多组学关联分析[15]。以|关联系数|>0.1,P < 0.05作为阈值,筛选与氯雷他定负相关的疾病以及与氯雷他定正相关的药物、化合物或中药,并预测相关作用靶点。
1.2.6 文本验证为了验证氯雷他定作用靶点的有效性,将10个关键基因作为关键靶点,以‘关键靶点’ AND ‘coronavirus’为检索词,在英文数据库中进行检索,以‘关键靶点’ AND ‘冠状病毒’为检索词,在中文数据库中进行检索,并记录相关检索结果。
2 结果 2.1 数据处理及差异表达分析通过limma程序包对5组数据进行差异分析,以|log2FC|>1,P < 0.05,为阈值,得到5组差异结果,差异基因个数依次为223个、130个、169个、207个、202个,其中上调表达的基因个数依次为79个、35个、60个、86个、69个,下调表达的基因个数依次为144个、95个、109个、121个、133个(见图 1)。

|
图 1 各组差异表达分析结果 Figure 1 Results of differential expression analysis in each group |
使用Venny 2.1工具和R语言脚本,将5组差异结果汇总,去除交集基因并求平均值后,共得到差异基因642个,其中上调230个,下调412个。
2.3 蛋白互作调控网络的构建将差异表达基因导入STRING v11.0在线分析网站进行蛋白互作网络分析,共得到642个蛋白,1 087条相互关系,平均点度为5.56。导入Cytoscape软件进行PPI网络可视化处理[16](见图 2)。
|
图 2 差异表达基因蛋白互作网络结果 Figure 2 Results of differentially expressed gene PPI networks |
利用R语言脚本,使用Degree算法对PPI网络中的基因进行节点得分计算,筛选得到排名前10位的关键基因(见图 3)。
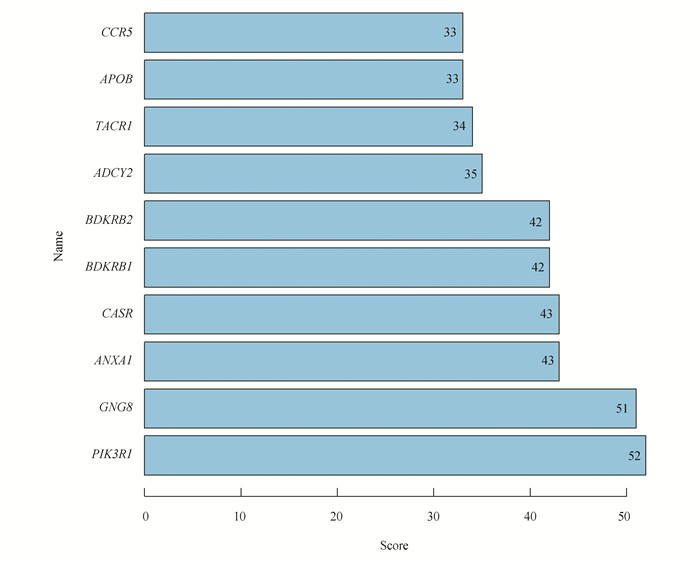
|
图 3 排名前10位的关键基因 Figure 3 Top 10 hub genes 注: 其中包括与心脏保护相关的基因,例如ADCY2、BDKRB2、BDKRB1、CASR;与免疫相关的基因,例如CCR5、TACR1、ANXA1;与激素和跨膜信号相关的基因,例如PIK3R1、GNG8;与脂质代谢相关的基因,例如APOB. |
使用R程序语言clusterProfiler包对差异基因进行GO功能富集和KEGG通路富集。GO富集分析共得到311条结果,其中包括生物学过程(Biological Process,BP)241条,细胞组分(Cellular Components,CC)29条,分子功能(Molecular Function,MF)41条(见图 4)。在生物学过程方面,富集的基因大多与免疫反应、细胞因子、趋化因子、激素作用、糖脂代谢有关,如IL18、IFNG、LRP1、TNF、TGFB1、KCNA4、GCGR、APOB、LDLR等。在细胞组分方面,富集的基因大多参与炎症调节过程,如C1S、CD3D、CD4等。在分子功能方面,富集的基因大多与免疫反应、激素调节和跨膜运输有关,如CCR5、GCGR、SLC4A1、SLC5A5、SLC5A7、SLC18A3、SLC38A2等。
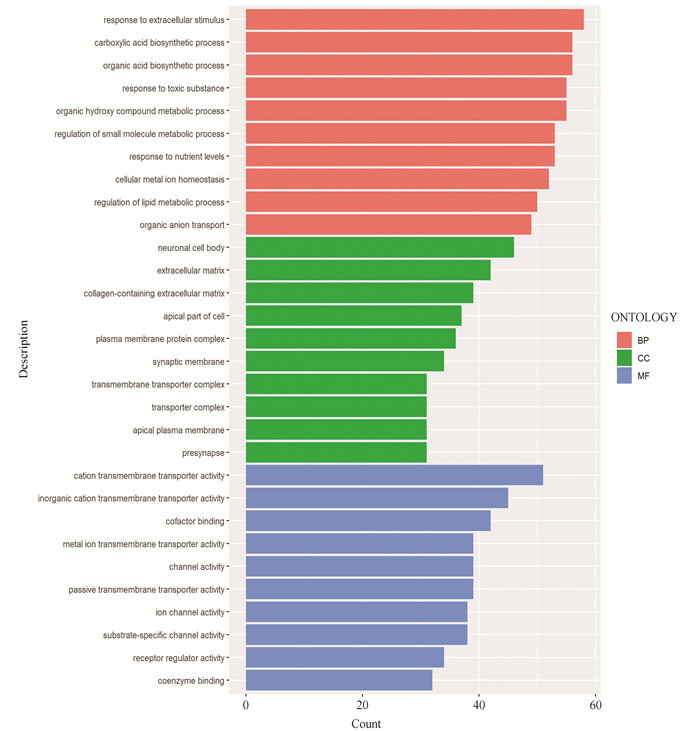
|
图 4 GO富集分析结果 Figure 4 Results of GO enrichment analysis 注: 富集基因主要参与免疫反应,趋化因子作用,激素作用,分子代谢调节和药物代谢等过程. |
KEGG通路共富集通路305条,富集的通路主要与补体和凝血系统,炎症反应,心律失常,氧化应激和病毒感染等相关功能或过程。(见图 5,图 6)。
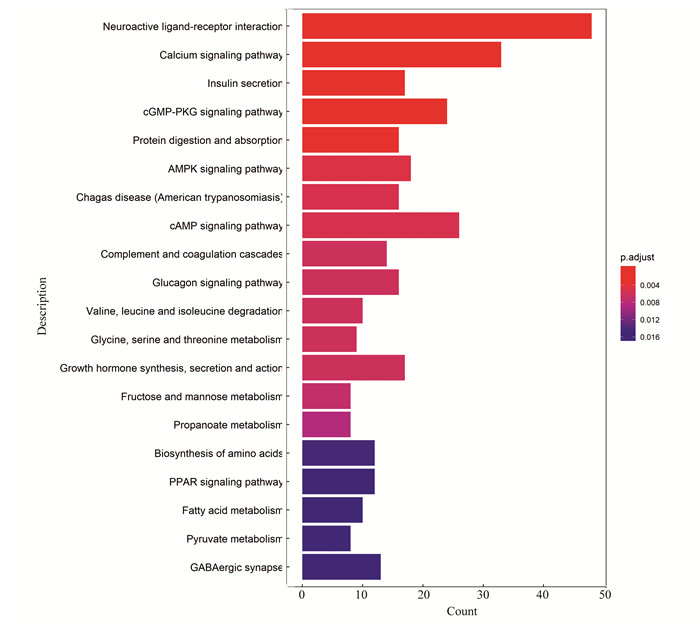
|
图 5 KEGG通路富集分析结果 Figure 5 Results of KEGG pathway enrichment analysis 注: 其富集途径主要与补体和凝血系统、免疫调节、炎症反应、心律失常、氧化应激和病毒感染等有关. |

|
图 6 部分重要信号通路 Figure 6 Some significant signal pathways |
使用EpiMed平台对疾病与药物进行关联分析,以关联系数 < -0.1,P < 0.05作为阈值,筛选出与氯雷他定负相关的疾病,预测结果多集中于炎症疾病、病毒性疾病、免疫相关疾病,包括SARS、流行性感冒、全身炎症反应综合征、呼吸道合胞体病毒感染和社区获得性肺炎,同时,以关联系数> 0.1,P < 0.05作为阈值筛选出与氯雷他定正相关的药物,预测结果包括TNF-α抑制剂(Etanercept)、抗病毒药物泛昔洛韦(Famciclovir)、奈韦拉平(Nevirapine),免疫调节药物氟伐他汀(Fluvastatin)以及抗炎药磷酸氯喹(Chloroquine)等,预测出与氯雷他定正相关的中药,包含虎杖(Polygonum cuspidatum),鱼腥草(Houttuynia cordata),连翘(Forsythia),甘草(Liquorice),知母(Common anemarrhena rhizome)(见表 2)。
| 表 2 氯雷他定的正负相关分析的部分结果 Table 2 Partial results of loratadine positive and negative correlation analysis |
为了验证EpiMed平台预测出的氯雷他定相关作用靶点的有效性,我们选取10个关键靶点,包括PIK3R1、GNG8、ANXA1、CASR、BDKRB1、BDKRB2、ADCY2、TACR1、APOB、CCR5,分别在中、英文数据库中检索冠状病毒与上述靶点作用相关的文献(见表 3)。结果表明,上述关键靶点在冠状病毒感染患者中,主要参与抗病毒反应、炎性因子的抑制、趋化因子调控、跨膜信号传导、糖脂代谢、磷酸化等过程,对冠状病毒感染及其造成的免疫损伤有一定的抑制作用。
| 表 3 文本验证结果 Table 3 Results of text mining for verification |
目前,全球为遏制SARS-CoV-2已做出了巨大努力,但病毒的传播已经达到了大流行的水平。遗憾的是,治疗新冠病毒的疗法和疫苗开发仍处于早期阶段,已有研究表明,一些药物对COVID-19患者的治疗有效,但缺乏相关临床证据,可能会导致不可预测的临床预后效果,因此,快速筛选目前已经过批准的以及效果良好的药物可能是一种更实用的方法[31]。
本研究以氯雷他定的基因表达谱数据为研究对象,经临床生物信息学分析发现,氯雷他定对COVID-19的治疗作用是个多靶点的组学网络调控,关键的基因如PIK3R1、GNG8、ANXA1、CASR、BDKRB1、BDKRB2、ADCY2、TACR1、APOB、CCR5等,与以往研究结果一致[17-30],这些基因主要参与跨膜信号传导、胰岛素的代谢、免疫监视、炎症反应、器官保护、维持钙稳态、调控细胞周期、补体和凝血等过程。Sille等[32]研究发现,GNG8参与控制白细胞跨内皮细胞迁移的趋化因子信号,并在神经系统谷氨酸能、胆碱能、GABA能和多巴胺能突触的几条通路中发挥作用。Fox等[33]研究发现,CCR5是一种表达在白细胞上的趋化因子受体,趋化因子刺激CCR5触发了趋化因子结合的CCR5分子的内在化,使CCR5下调,有助于趋化因子的抗病毒活性。Zhang等[34]研究发现,CASR下调有助于减轻缺氧/复氧(A/R)心肌细胞的钙超载,具有一定的心脏保护功能。Zhou等[35]研究发现,肝细胞AKR7A通过减少恶性肝细胞对脂质的利用来调节APOB的表达,对AFB1诱导的细胞毒性具有保护作用。综上所述,关键基因的调控在氯雷他定治疗COVID-19患者过程中可能发挥着重要作用,一方面氯雷他定上调GNG8等基因,下调CCR5等基因,可能具有控制白细胞迁移、降低中性粒细胞浸润程度、抑制细胞因子表达、防止因大量细胞因子趋化造成的细胞因子风暴,抑制炎症反应等作用。另一方面,氯雷他定通过下调CASR、APOB等基因,可能具有清除免疫复合物、减轻免疫应答、抑制钙紊乱的功能,在清除病毒、抑制免疫反应的同时,起着一定的器官保护的作用。
在GO富集分析结果中,生物学过程方面,氯雷他定主要参与细胞外刺激反应,糖脂代谢,钙离子稳态调节等过程;在细胞组分方面,主要影响跨膜转运复合物,离子通道复合体以及顶端质膜、基底外侧质膜、突触膜等多种膜的构成;在分子功能方面,主要与多种离子通道转运蛋白活性,激素调节,糖蛋白结合等功能相关。本研究认为,氯雷他定可能通过阻断跨膜运输,调控糖脂代谢,调节多种激素作用和趋化因子活性来减轻病毒引起的炎症反应与免疫失调,维持钙离子稳态并起到一定的器官保护作用。
在KEGG通路富集方面,氯雷他定主要作用于机体补体和凝血系统,氧,炎症反应,心律失常,氧化应激和病毒感染等相关功能或过程。Hunto等[36]研究发现,氯雷他定抑制诱导型一氧化氮合酶、IL-1、TNF-α和IL-6的表达,并抑制Syk/Src介导的NF-κB通路,从而抑制变态反应,并作为抑制剂,调节炎症反应中的蛋白活性。Zhou等[37]研究发现,氯雷他定能有效抑制氧化低密度脂蛋白诱导的单核细胞与内皮细胞的粘附、活性氧和血管黏附分子的产生以及VCAM-1、E-选择素、TNF-α、IL-6和IL-8等细胞因子的诱导,有效地阻断了氧化低密度脂蛋白(LDL)诱导的JNK的激活,以及AP-1和NF-κB信号通路,提示了氯雷他定在内皮保护中的作用。Tontonoz等[38]研究发现,激活PPARG可以促进单核细胞和巨噬细胞分化,减少LDL的摄取。PPARG激动剂具有抑制心肌细胞肥大和心肌炎症反应的作用,在ACE2突变小鼠中PPARG信号的激活导致心肌损伤减轻,丝裂原活化蛋白激酶(MAPK)通路在此过程中起着关键作用。总之,本研究认为,氯雷他定不仅具有传统的抗组胺作用,其作用于机体后,可能会激活PPAR信号通路、MAPK信号通路等通路,抑制NF-κB信号通路,AP-1信号通路等通路,从而抑制了IL-1、IL-6等多种细胞因子以及其他分子的表达,阻断了JNK、AP-1等通路的激活,减少对LDL的摄取,具有一定的抑制炎症,减少器官损伤的作用。
为了进一步验证氯雷他定相关作用靶点的可靠性,我们选取10个关键基因(PIK3R1、GNG8、ANXA1、CASR、BDKRB1、BDKRB2、ADCY2、TACR1、APOB、CCR5) 在中英文数据库中分别进行检索,结果显示10个关键基因均与冠状病毒密切相关,López-Cortés等[17]研究发现,在免疫系统相关蛋白和SARS-CoV-2相关的人类蛋白之间建立PPI网络,中心度最高的为UBA52,其次为GNB1、APP、FPR2、NCBP1、NCBP2、KNG1、PIK3CA、PIK3R1。Sun等[20]研究发现,在传染性支气管炎病毒感染气管中ANXA1下调,提示ANXA1的表达降低可能与呼吸道病毒感染过程中的炎症表型有关。Chen等[29]研究发现,感染SARS-CoV后,肺中的趋化因子受体CCR5上调,CCR5 mRNA转录增加9倍,这些受体转录本表达增强与其配体在SARS冠状病毒感染的肺中上调密切相关。因此,本研究认为,上述10个关键基因在氯雷他定治疗COVID-19中或许起着重要作用,其对炎症调节的相关机制以及对免疫调控网络作用有待进一步研究加以证实。
本研究基于前期建立的EpiMed表观精准治疗预测平台[39],对氯雷他定开展关联分析。在疾病关联分析方面,发现氯雷他定与SARS、流行性感冒、全身炎症反应综合征、呼吸道合胞体病毒感染和社区获得性肺炎等感染性疾病呈负相关,提示氯雷他定对COVID-19可能具有类似作用。在药物关联分析方面,发现TNF-α抑制剂依那西普、抗病毒药物泛昔洛韦、奈韦拉平,免疫调节药物氟伐他汀以及抗炎药氯喹等西药,虎杖,鱼腥草,连翘,甘草,知母等中药与氯雷他定正相关,提示其与氯雷他定可能具有潜在协同作用。综上所述,氯雷他定对COVID-19具有潜在治疗作用,在临床可考虑与上述正相关药物进行联合治疗,以上结果均有待进一步临床及基础研究验证。
总之,氯雷他定不仅是一种传统的抗组胺药物,其作用于机体能产生全基因组范围的影响,通过抑制多种细胞因子,可能会减轻COVID-19危、重患者体内细胞因子风暴造成的多器官损伤,在抑制炎症反应、器官保护等方面可能也有着重要作用。本研究使用课题组前期建立的EpiMed平台,分析预测了氯雷他定调控的分子靶点以及可能具有相似治疗作用的药物,对于制定包含氯雷他定的COVID-19危重症患者治疗方案具有一定的指导意义。
| [1] |
HUANG C L, WANG Y M, LI X W, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China[J]. The Lancet, 2020, 395(10223): 497-506. DOI:10.1016/S0140-6736(20)30183-5 (  0) 0) |
| [2] |
CHEN G, WU D, GUO W, et al. Clinical and immunologic features in severe and moderate 1 forms of Coronavirus Disease[EB/OL]. MedRxiv, (2020-02-16)[2020-03-08]. DOI: 10.1101/2020.02.16.20023903.
(  0) 0) |
| [3] |
陈蕾, 刘辉国, 刘威, 等. 2019新型冠状病毒肺炎29例临床特征分析[J/OL]. 中华结核和呼吸杂志, 2020, 43(2020-02-06)[2020-03-08]. DOI: 10.3760/cma.j.issn.1001-0939.2020.0005. CHEN Lei, LIU Huiguo, LIU Wei, et al. Analysis of the clinical characteristics of 29 cases of 2019 new coronavirus pneumonia[J/OL]. Chinese Journal of Tuberculosis and Respiratory Diseases, 2020, 43(2020-02-06)[2020-03-08]. DOI: 10.3760/cma.j.issn.1001-0939.2020.0005. (  0) 0) |
| [4] |
DAVIS B, ILLAMPERUMA C, GAUVREAU G M, et al. Single-dose desloratadine and montelukast and allergen-induced late airway responses[J]. European Respiratory Journal, 2009, 33(6): 1302-1308. DOI:10.1183/09031936.00169008 (  0) 0) |
| [5] |
杨勤萍, 陈连军, 徐金华, 等. 氯雷他定治疗慢性荨麻疹93例临床疗效观察[J]. 临床皮肤科杂志, 2004(1): 50-50. YANG Qinping, CHEN Lianjun, XU Jinhua, et al. Observation on the clinical efficacy of loratadine in the treatment of 93 patients with chronic urticaria[J]. Journal of Clinical Dermatology, 2004(1): 50-50. DOI:10.3969/j.issn.1000-4963.2004.01.034 (  0) 0) |
| [6] |
SHAHEN M, GUO Z H, SHAR A H, et al. Dengue virus causes changes of MicroRNA-genes regulatory network revealing potential targets for antiviral drugs[J]. BMC Systems Biology, 2018, 12(1): 2. DOI:10.1186/s12918-018-0536-3 (  0) 0) |
| [7] |
GENTLEMAN R C, CAREY V J, BATES D M, et al. Bioconductor: open software development for computational biology and bioinformatics[J]. Genome Biology, 2004, 5(10): R80. DOI:10.1186/gb-2004-5-10-r80 (  0) 0) |
| [8] |
HASTIE T, TIBSHIRANI R, NARASIMHAN B, et al. Impute: Imputation for microarray data[J]. Bioinformatics, 2001, 17(6): 520-525. DOI:10.18129/B9.bioc.impute (  0) 0) |
| [9] |
DURINCK S, MOREAU Y, KASPRZYK A, et al. BioMart and Bioconductor: a powerful link between biological databases and microarray data analysis[J]. Bioinformatics, 2005, 21(16): 3439-3440. DOI:10.1093/bioinformatics/bti525 (  0) 0) |
| [10] |
RITCHIE M E, PHIPSON B, WU D I, et al. limma powers differential expression analyses for RNA-sequencing and microarray studies[J]. Nucleic Acids Research, 2015, 43(7): e47-e47. DOI:10.1093/nar/gkv007 (  0) 0) |
| [11] |
OLIVEROS J C V. An interactive tool for comparing lists with Venn's diagrams[EB/OL]. https://bioinfogp.cnb.csic.es/tools/venny/index.html, 2007-2015.
(  0) 0) |
| [12] |
SZKLARCZYK D, GABLE A L, LYON D, et al. STRING v11:protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Research, 2019, 47(D1): D607-D613. DOI:10.1093/nar/gky1131 (  0) 0) |
| [13] |
张皓旻, 陈红飞, 杨波, 等. KRAS突变对人肺腺癌转录组影响的生物信息学分析[J]. 转化医学杂志, 2019, 8(2): 14-18. ZHANG Haomin, CHEN Hongfei, YANG Bo, et al. Bioinformatics analysis of the effect of KRAS mutation on the transcriptome of human lung adenocarcinoma[J]. Translational Medicine Journal, 2019, 8(2): 14-18. DOI:10.3969/j.issn.2095-3097.2019.02.003 (  0) 0) |
| [14] |
YU G, WANG L G, HAN Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. Omics: A Journal of Integrative Biology, 2012, 16(5): 284-287. DOI:10.1089/omi.2011.0118 (  0) 0) |
| [15] |
卢学春, 杨波, 张皓旻, 等. 表观功能基因组学是解开恶性肿瘤治疗的钥匙[M]. 中国肿瘤临床年鉴. 北京: 中国协和医科大学出版社, 2018. LU Xuechun, YANG Bo, ZHANG Haomin, et al. Epifunctional genomics is the key to the treatment of malignant tumors[M]. Chinese Cancer Clinical, Yearbook. Beijing: Peking Union Medical College Press, 2018. (  0) 0) |
| [16] |
SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks[J]. Genome Research, 2003, 13(11): 2498-2504. DOI:10.1101/gr.1239303 (  0) 0) |
| [17] |
LÓPEZ-CORTÉS A, GUEVARA-RAMÍREZ P, KYRIAKIDIS N C, et al. In Silico analyses of immune system protein interactome network, single-cell RNA sequencing of human tissues, and artificial neural networks reveal potential therapeutic targets for drug repurposing against COVID-19[J]. Frontiers in Pharmacology, 2020. DOI:10.26434/chemrxiv.12408074.v1 (  0) 0) |
| [18] |
詹群璋, 黄英杰, 林树红, 等. 基于网络药理学和分子对接的玉屏风散预防新型冠状病毒肺炎(COVID-19)活性化合物的研究[J]. 中草药, 2020, 51(7): 1731-1740. ZHAN Qunzhang, HUANG Yingjie, LIN Shuhong, et al. Study on the prevention of COVID-19(COVID-19) active compound of yupingfeng powder based on network pharmacology and molecular docking[J]. Chinese Traditional and Herbal Drugs, 2020, 51(7): 1731-1740. DOI:10.7501/j.issn.0253-2670.2020.07.007 (  0) 0) |
| [19] |
BRANN D, TSUKAHARA T, WEINREB C, et al. Non-neural expression of SARS-CoV-2 entry genes in the olfactory epithelium suggests mechanisms underlying anosmia in COVID-19 patients[J]. BioRxiv, 2020, 6(30): eabc5801. DOI:10.1126/sciadv.abc5801 (  0) 0) |
| [20] |
SUN J, HAN Z, SHAO Y, et al. Comparative proteome analysis of tracheal tissues in response to infectious bronchitis coronavirus, Newcastle disease virus, and avian influenza virus H9 subtype virus infection[J]. Proteomics, 2014, 14(11): 1403-1423. DOI:10.1002/pmic.201300404 (  0) 0) |
| [21] |
XIAO X, QI J, LEI X, et al. Interactions between enteroviruses and the inflammasome: New insights into viral pathogenesis[J]. Frontiers in Microbiology, 2019, 10: 1-9. DOI:10.3389/fmicb.2019.00321 (  0) 0) |
| [22] |
DU M, CAI G, CHEN F, et al. Multiomics evaluation of gastrointestinal and other clinical characteristics of COVID-19[J]. Gastroenterology, 2020, 158(8): 2298. DOI:10.1053/j.gastro.2020.03.045 (  0) 0) |
| [23] |
吴晨, 魏昀, 葛珊, 等. 夏天无对新型冠状病毒肺炎的网络药理学和分子对接研究[J]. 中医药信息, 2020, 37(3): 104-111. WU Chen, WEI Yun, GE Shan, et al. Network pharmacology and molecular docking study of corydalis amabilis against COVID-19[J]. Information on Traditional Chinese Medicine, 2020, 37(3): 104-111. DOI:10.19656/j.cnki.1002-2406.200084 (  0) 0) |
| [24] |
VERSTEEG G A, SLOBODSKAYA O, SPAAN W J M. Transcriptional profiling of acute cytopathic murine hepatitis virus infection in fibroblast-like cells[J]. Journal of General Virology, 2006, 87(7): 1961-1975. DOI:10.1099/vir.0.81756-0 (  0) 0) |
| [25] |
谢静, 田野, 刘杨, 等. 基于网络药理学探讨痰咳净方防治新型冠状病毒肺炎的可行性[J]. 药物评价研究, 2020, 43(4): 591-600. XIE Jing, TIAN Ye, LIU Yang, et al. Potential mechanism analysis of respiratory system treated by Tankejing based on network pharmacology[J]. Drug Evaluation Research, 2020, 43(4): 591-600. DOI:10.7501/j.issn.1674-6376.2020.04.002 (  0) 0) |
| [26] |
JEAN S S, HSUEH P R. Old and re-purposed drugs for the treatment of COVID-19[J/OL]. Expert Review of Anti-infective Therapy, 2020(accepted). https://doi.org/10.1080/14787210.2020.1771181.DOI: 10.1080/14787210.2020.1771181.
(  0) 0) |
| [27] |
NIE S, ZHAO X, ZHAO K, et al. Metabolic disturbances and inflammatory dysfunction predict severity of coronavirus disease 2019 (COVID-19): A retrospective study[J/OL]. MedRxiv, 2020. https://doi.org/10.1101/2020.03.24.20042283.DOI: 10.1101/2020.03.24.20042283.
(  0) 0) |
| [28] |
陈保生. 载脂蛋白的结构和功能与病毒病的预防和治疗[J]. 中国医学科学院学报, 2007, 29(3): 448-451. CHEN Baosheng. Structure and function of apolipoprotein and prevention and therapy of virus diseases[J]. Acta Academiae Medicinae Sinicae, 2007, 29(3): 448-451. (  0) 0) |
| [29] |
CHEN J, LAU Y F, LAMIRANDE E W, et al. Cellular immune responses to severe acute respiratory syndrome coronavirus (SARS-CoV) infection in senescent BALB/c mice: CD4+ T cells are important in control of SARS-CoV infection[J]. Journal of Virology, 2010, 84(3): 1289-1301. DOI:10.1128/JVI.01281-09 (  0) 0) |
| [30] |
叶子葳, 金冬雁. 2019新型冠状病毒及其诊疗与防控: 回顾与展望[J]. 生物工程学报, 2020, 36(4): 571-592. YE Ziwei, JIN Dongyan. .Diagnosis, treatment, control and prevention of SARS-CoV-2 and coronavirus disease 2019:Back to the future[J]. Chinese Journal of Biotechnology, 2020, 36(4): 571-592. DOI:10.13345/j.cjb.200115 (  0) 0) |
| [31] |
AHN D G, SHIN H J, KIM M H, et al. Current status of epidemiology, diagnosis, therapeutics, and vaccines for novel coronavirus disease 2019 (COVID-19)[J]. Journal of Microbiology and Biotechnology, 2020, 30(3): 313-324. DOI:10.4014/jmb.2003.03011 (  0) 0) |
| [32] |
SILLE F C M, THOMAS R, SMITH M T, et al. Post-GWAS functional characterization of susceptibility variants for chronic lymphocytic leukemia[J]. PLoS One, 2012, 7(1). DOI:10.1371/journal.pone.0029632 (  0) 0) |
| [33] |
FOX J M, KASPROWICZ R, HARTLEY O, et al. CCR5 susceptibility to ligand-mediated down-modulation differs between human T lymphocytes and myeloid cells[J]. Journal of Leukocyte Biology, 2015, 98(1): 59-71. DOI:10.1189/jlb.2A0414-193RR (  0) 0) |
| [34] |
ZHANG L, CAO S, DENG S, et al. Ischemic postconditioning and pinacidil suppress calcium overload in anoxia-reoxygenation cardiomyocytes via down-regulation of the calcium-sensing receptor[J]. PeerJ, 2016, 4: e2612. DOI:10.7717/peerj.2612 (  0) 0) |
| [35] |
ZHOU R, LIU M, LIANG X, et al. Clinical features of aflatoxin B1-exposed patients with liver cancer and the molecular mechanism of aflatoxin B1 on liver cancer cells[J]. Environmental Toxicology and Pharmacology, 2019, 71: 103225. DOI:10.1016/j.etap.2019.103225 (  0) 0) |
| [36] |
HUNTO S T, KIM H G, BAEK K S, et al. Loratadine, an antihistamine drug, exhibits anti-inflammatory activity through suppression of the NF-kB pathway[J]. Biochemical Pharmacology, 2020, 177: 113949. DOI:10.1016/j.bcp.2020.113949 (  0) 0) |
| [37] |
ZHOU Y, GAO C, WANG H, et al. Histamine H1 type receptor antagonist loratadine ameliorates oxidized LDL induced endothelial dysfunction[J]. Biomedicine & Pharmacotherapy, 2018, 106: 1448-1453. DOI:10.1016/j.biopha.2018.07.025 (  0) 0) |
| [38] |
PETER T, NAGY L, ALVAREZ J G A, et al. PPARγ promotes monocyte/macrophage differentiation and uptake of oxidized LDL[J]. Cell, 1998, 93(2): 241-252. DOI:10.1016/S0092-8674(00)81575-5 (  0) 0) |
| [39] |
卢学春, 迟小华, 杨波, 等. 重型再生障碍性贫血发病相关T淋巴细胞基因表达谱的生物信息学分析及作为药物筛选新方法的探索[J]. 中国实验血液学杂志, 2010, 18(2): 416-420. LU Xuechun, CHI Xiaohua, YANG Bo, et al. Gene expression profile analysis of T lymphocytes involved in pathogenesis of severe aplastic anemia by using bioinformatics method as a novel way of drug screening[J]. Journal of Experimental Hematology, 2010, 18(2): 416-420. (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


