2. 西北农林科技大学 农学院,陕西 杨凌 712100
2. College of Agronomy, Northwest A & F University, Yangling 712100, Shannxi, China
生物信息学是随着人类基因组计划(Human Genome Project, HGP)启动而兴起的一门新兴学科,它是由数学、计算机科学、信息学和生物学产生的一门交叉学科。从广义上说,生物信息学是应用信息科学的方法和技术,研究生物体系和生物过程中信息的存贮、信息的内涵和信息的传递,研究和分析生物体细胞、组织、器官的生理、病理、药理过程中的各种生物信息[1]。狭义上讲,生物信息学就是生命科学中的信息科学,就是应用信息科学的理论、方法和技术,管理、分析和利用生物分子数据,或者是一门利用计算机技术研究生物系统之规律的学科[2]。生物信息学的研究内容主要包括以发展新的数理和信息科学的技术和方法,用于管理和分析生物数据的算法开发和以收集、整理、储存、加工、发布、分析及解释生物学数据的数据挖掘与运用两个方面。伴随着人类基因组计划的完成,生命科学研究进入了后基因组时代(Post-genome area),在此过程中产生了大量的蛋白序列、结构、功能以及互作的数据,相比于基因组时代,蛋白质组更加庞大且复杂,仅仅依靠传统的生物手段无法解决问题,必须借助生物信息学技术全方位的处理所产生的生物学数据。因此,面对海量蛋白质组数据的获取、整理、注释、处理、存储以及蛋白质组数据信息的挖掘及数据的可视化,生物信息学技术成为蛋白质组学研究中不可或缺的重要工具和手段。现就简要综述一下生物信息学技术在蛋白质组学研究中的应用并初步展望了其未来的发展趋势。
1 蛋白质组学及其研究策略蛋白质组(Proteome)源于蛋白质(Protein)与基因组(Genome)两个词的组合,意指“一种基因组所表达的全套蛋白质”[1], 即包括一种细胞乃至一种生物所表达的全部蛋白质。蛋白质组学(Proteomics)从整体的角度分析细胞内动态变化的蛋白质组成成分、表达水平与修饰状态,了解蛋白质之间的相互作用与联系,从而揭示蛋白质功能与细胞生命活动规律,其逐渐成为当前生物学研究的热点和突破最快的领域。自2014年人类蛋白质组草图发表后多种蛋白质组学相关研究陆续公布,蛋白质组研究取得了大量重要成果。
蛋白质组学研究是不断发展完善的。初期,主要以2-DIGE及SDS-PAGE为主,这种方法通量较低,灵敏度不高。随着高效液相色谱技术(HPLC)和质谱技术的发展,基于液相色谱+质谱(LC-MS/MS)的3D蛋白质组学研究逐渐成为本领域的主流方法;在此基础上,伴随着各种定量技术(如SILAC、iTRAQ、SWATH和Label-Free等)的不断出现和高精密质谱仪器的成功应用,离子淌度(mobility)的引入,使得蛋白质组学研究进入了4D的高通量时代(见图 1)。
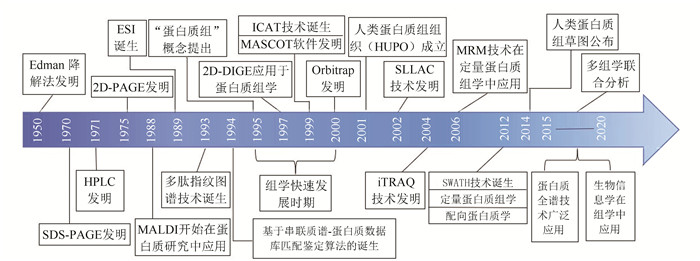
|
图 1 蛋白质组研究的简要发展历程 Figure 1 Development history of proteome research |
蛋白质组学研究的基本策略主要包括定性和定量研究,其中定性检测主要有两种策略:自底向上(Bottom-up)和自顶向下(Top-down)(见图 2)。其中Bottom-up应用的较为广泛,其中“bottom”指的是肽段,“up”是指由肽段推理为蛋白的过程,即先将蛋白酶解成肽段,然后通过色谱分离肽段混合物,再用质谱技术将肽段碎裂,根据碎裂谱图的离子峰信息进行数据库搜索来鉴定肽段,最后将鉴定的肽段进行组装、重新归并为蛋白质。Top-down的“top”指的是完整蛋白质分子的质量测定,“down”则是指对完整蛋白的碎裂。这种方法无需酶解,通过完整蛋白质的质量及其碎裂谱图信息可以实现蛋白质鉴定,序列覆盖度较高,能保留多种翻译后修饰之间的关联信息。
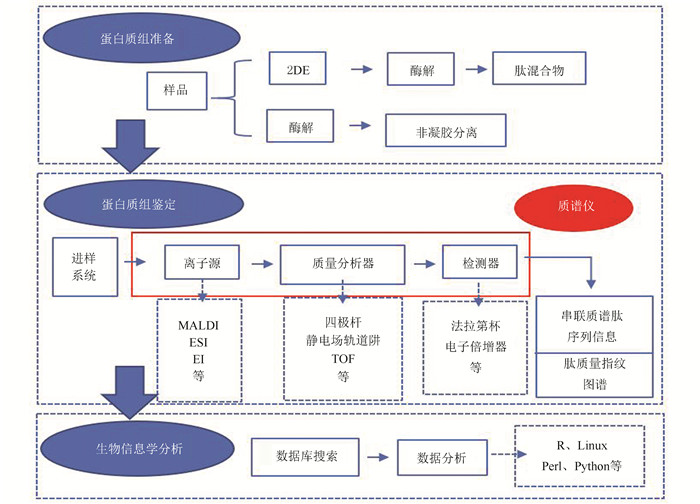
|
图 2 蛋白质组学定性分析流程 Figure 2 Qualitative analysis process of proteomics |
针对蛋白质组学研究的不同的策略、不同对象、不同方法,需要应用不同的软件和工具来进行数据的挖掘、处理和分析。现就生物信息学技术在蛋白质谱数据处理、蛋白质鉴定及蛋白质翻译后修饰以等方面的应用进展做一简要介绍。
2.1 生物信息学在质谱数据处理上的应用质谱技术已成为蛋白质组学研究的核心技术之一,也是开展蛋白鉴定与分析的主要手段。一台质谱仪可以在几天内产生数百万张的图谱。如此庞大的信息需要利用高效、易学易用的软件工具来进行质谱数据的收集、保存、搜索、鉴定与分析。主要的质谱数据分析工具包括以下几类。
2.1.1 质谱数据处理工具质谱数据搜索软件有Mascot、SEQUEST、Lutkefish、Proteome software、Profound和PepSea等(见表 1)。Mascot是质谱数据搜索的常用软件,它是英国Matrix Sciences公司开发的产品,利用分子序列数椐检索的方法,鉴定样本中蛋白质的组成以及翻译后修饰。该软件整合了先进的统计学算法,能快速,准确的得到分析结果[3]。Mascot可以进行在线检索和本地检索。在线检索免费,检索速度快,操作简单,只需将peak list文件导入即可,但文件大小受限制;而本地检索需要购买软件及安装数据库,使用方便、可以进行大规模的数据检索分析和数据库配置,功能更加强大[4]。
| 表 1 用于质谱蛋白鉴定与分析工具 Table 1 Tools for protein identification and analysis by mass spectrometry |
SEQUEST是Thermo Electron开发的基于串联质谱数据(MS/MS)的搜索软件。它将串联质谱数据与蛋白质数据库序列相联系,使研究者的质谱数据蛋白质鉴定,从费时的工作中解放出来,而且SEQUEST软件适合混合蛋白质的质谱鉴定[5-6]。
pFind(http://pfind.ict.ac.cn)是中国科学院计算技术研究所李德泉、贺思敏等开发,我国有自主知识产权的串联质谱数据搜索软件。相比Mascot、SEQUEST,它的改进是在匹配打分过程中,考虑了相关离子的匹配程度,引入了核谱向量点积(kernel spectrum dot product, KSDP) 算法,通过对普通打分算法谱向量点积(SDP)的扩展, 借助机器学习领域中的核函数技术,利用连续离子匹配信息进行匹配打分,很好地降低了质谱数据搜索的假阳性结果。
2.1.2 定量蛋白质分析工具质谱技术作为蛋白质组学研究的关键技术,在定量蛋白质组学分析中起着十分重要的作用。非标定量法(Label-free)就是通过液质联用技术对蛋白质酶解肽段进行质谱分析,然后比较质谱分析次数或质谱峰强度,分析不同来源样品蛋白的数量变化,肽段在质谱中被捕获检测的频率与其在混合物中的丰度成正相关,通过适当的数学公式可以将质谱检测技术与蛋白质的量联系起来,从而对蛋白质进行定量。目前基于生物质谱的定量蛋白质组学分析策略主要分为相对定量和绝对定量,相对定量蛋白质组是指对不同生理状态下的细胞、组织或体液蛋白质表达量的相对变化进行比较分析;绝对定量蛋白质组是测定细胞、组织或体液蛋白质组中每种蛋白质的绝对量或浓度。基于质谱数据的定量蛋白质分析软件很多,主要包括DeCyder MS、MaXIC-Q、MSQuant等(见表 1)。其中,DeCyder MSTM软件是GE公司开发的商业化软件,是运用于蛋白质非标记定量(Label-free)的主要工具;而MaXIC-Q是高通量定量蛋白质组学的通用计算平台,可用于大规模稳定同位素标记定量和液相色谱串联质谱数据的高通量、高精度定量分析;MSQuant是一款常用的定量蛋白质组学/质谱分析工具,主要用于对蛋白质和肽进行定量[7]。
2.1.3 质谱数据的de novo鉴定工具蛋白质从头测序(De novo sequencing),又叫全新蛋白测序,这项技术根据肽段与惰性气体相碰撞产生的一系列的有规律的片段离子之间的质量差来推断氨基酸序列。de novo测序方法不依赖于数据库,能明确解释串联质谱(Tandem mass spectrometry,MS/MS)图谱,对鉴定新的蛋白质和提高图谱的利用率具有重要的作用。De novo蛋白质鉴定软件有很多,包括MSNovo、Lutefisk、PEAKS、NovoHMM等(见表 1)。MSNovo是一款新的多肽de novo测序软件,不支持在线模式,但它支持多种类型仪器产出的数据,能够支持+1、+2和+3价的母离子;Lutefisk是应用于开放资源肽CID图谱从头解析的工具;PEAKS是一个综合性肽图谱分析软件包,不仅可以用于蛋白从头测序,而且可以进行蛋白质鉴定、蛋白序列同源性搜索以及标记和非标记定性、定量分析等[8];NovoHMM将隐马尔可夫模型引入蛋白序列解析中,提供了一种比其他从头测序更准确的鉴定方法[9]。
2.2 生物信息学在蛋白质翻译后修饰上的应用蛋白质的翻译后修饰(Post-translational modification, PTM)是指对翻译后的蛋白质进行共价加工的过程,通过在一个或多个氨基酸残基加上修饰基团,可以改变蛋白质的理化性质,进而影响蛋白质的空间构象和活性状态、亚细胞定位、折叠及其稳定性以及蛋白质-蛋白质相互作用,是调节蛋白质功能的重要方式。许多至关重要的生命进程不仅由蛋白质的相对丰度控制,更重要的是受到时空特异性和翻译后修饰的调控。对蛋白质翻译后修饰的研究可以帮助阐明和了解蛋白质功能及其功能变化,翻译后修饰的预测和分析也日渐成为生物信息学蛋白质序列分析中的重要的研究内容。其主要包括磷酸化、糖基化、甲基化、乙基化(如组蛋白质)、泛素化和羟基化等。
质谱是鉴定蛋白质翻译后修饰的重要方法,其原理是利用蛋白质发生修饰后的质量偏移来实现翻译后修饰位点的鉴定;同时,由于翻译后修饰的蛋白质在样本中含量低且动态范围广,检测前需要对发生修饰的蛋白质或肽段进行富集,然后再进行质谱鉴定。翻译后修饰的生物信息分析通常采用数据库检索和预测工具来进行。常见的蛋白翻译后修饰数据库主要有Swiss-Prot、Phospho.ELM、dbPTM、O-GlycBase以及RESID等数据库(见表 2)。其中,Swiss-Prot数据库世界两大蛋白序列数据库之一,收录了经实验验证的真实存在的蛋白信息资源,包括序列、功能、结构以及翻译后修饰信息;PROSITE数据库,又叫蛋白质结构分类数据库,它收录了蛋白质家族保守结构域(Domains)、包含重要生物学意义的位点(sites)、模式(Patterns)、轮廓(Profiles)和翻译后修饰位点等。Swiss-Prot和PROSITE数据库均已整合到了ExPASy数据库中。而Phospho.ELM是收录了不同生物体S/T/Y磷酸化位点的数据库,主要用于S/T/Y磷酸化位点的检索和预测;dbPTM和RESID数据库是均为综合性蛋白翻译后修饰数据库,收录了不同物种、各种不同修饰类型的修饰位点及其生物学功能,是翻译后修饰位点鉴定的重要工具;O-GlycBase是O-糖基化数据库,是糖基化预测和鉴定的重要数据库。鉴于蛋白翻译后修饰在调节蛋白质功能上的重要作用,大量的翻译后修饰工具也被开发出来,包括预测黏菌蛋白的O-糖基化位点的DictyOGlyc工具,预测哺乳动物蛋白的O-GalNAc糖基化位点的NetOGlyc工具,预测人类蛋白中的N-糖基化位点NetNGlyc工具,预测植物甲基化位点的CyMATE工具以及预测磷酸化位点的DISPHOS和Kinase Phos工具等(见表 3)。
| 表 2 翻译后修饰数据库 Table 2 Protein post-translational modification databases |
| 表 3 翻译后修饰预测工具 Table 3 Prediction tools for protein post-translational modification |
当前,生命科学已步入了后基因组时代。蛋白质组学研究是后基因组研究的重要部分,即将基因组静态的碱基序列清楚后,转而对基因组进行动态的生物学功能的研究。随着蛋白质组学研究的不断发展和深入,对生物信息学技术也提出了更高的要求,除了服务如今的蛋白质组数据的产生、处理、搜索、存储和信息挖掘,未来还需要在蛋白质从头测序(De novo sequencing)、蛋白质全谱分析、定量蛋白质组数据分析、目标蛋白质的功能预测以及蛋白修饰分析等方面逐步发展成熟。同时,还需要提高质谱数据的解析率和搜索正确率、跨平台质谱数据标准的建立(如基于XML等格式、大数据整合上有更大发展),推进蛋白质组数据分析的标准化。
同时,利用生物信息学解决蛋白质组学问题的挑战不仅仅在于数据存储分析的硬件条件,还在于运算法则(算法)和分析软件,因此开发新的更适合蛋白质组学分析的算法以及对使用者编程能力要求不高的分析软件显得十分重要。随着学科交叉的不断深入,生物信息学也在飞速发展。生物信息学技术在未来蛋白质组研究中重点包括:
(1) 物种全蛋白质组图谱绘制大规模整体性的分析蛋白质在某一个细胞、组织或个体中的含量、修饰以及动态表达。当前,在人类、小鼠以及模式植物拟南芥中取得了重要进展。2014人类蛋白质组草图由两个团队分别发表在Nature杂志上[16-17],两项研究具有互补性,可以互相印证。为了产生生物学价值,需要对各个组织的蛋白质进行精确定量,因此全世界各个国家的科研工作者启动了人体体液蛋白质组计划[18]、人类肝脏蛋白质组计划[19]、人类脑蛋白质组计划、人的糖蛋白质组计划、人类抗体组计划[20]、人类疾病小鼠模型蛋白质组计划[21-23]、人类肾脏蛋白质组计划、人类心血管蛋白质组计划、干细胞生物蛋白质组计划、疾病蛋白标志物计划、人类染色体蛋白质组计划和模式生物蛋白质组计划等,为人类蛋白质组学的研究成果在阐释生理与病理过程的分子机制的研究中发挥更加积极的作用, 并为人类的健康事业和生命科学的发展奠定基础。2020年3月基于质谱的拟南芥蛋白质组草图发表[24],这是目前最全面的拟南芥蛋白修饰与互作图谱其对拟南芥的30种组织进行蛋白质组、磷酸化修饰组以及转录组的定量分析,系统地揭示了蛋白质复合体的组织特异性和磷酸化调控的信号通路,是目前拟南芥蛋白质表达丰度与磷酸化翻译后修饰最为系统全面的研究。
(2) 大规模的蛋白质功能研究蛋白互作,包括有蛋白分子和亚基的聚合,蛋白分子杂交,蛋白分子识别,蛋白分子自组装,以及蛋白质复合体的形成等形式。通过分析一个蛋白质是否能和功能已知的蛋白质相互作用可得到揭示其功能的线索。蛋白质研究最大的挑战是鉴定每一个蛋白质以及它们的异构体的功能,如何系统整体性研究蛋白质与蛋白质之间的相互作用。2020年4月,蛋白质互作图绘制完成[25],这份蛋白互作组数据包含了约53 000种不同的蛋白互作信息,涉及超过8 000种蛋白质,虽然仅占到了所有人类蛋白质相互作用中的2%~11%,但仍然是世界上最大的互作图绘,这份图谱无论是对理解基础的生物学进程,还是对理解疾病的发生,都有着极为重要的意义。日后科研工作者将会通过更为深入的研究来扩展该蛋白质互作图谱,从而为研究人类疾病等多个领域提供重要线索和信息。
(3) 蛋白质调控网络建立蛋白质调控网,不仅可以提供蛋白质之间的相互关系的信息,而且还可以和基因组学、转录组学、代谢组学、表型组学等信息联系起来。2018年1月,发表的番茄代谢组研究论文[26]。该研究利用多重组学的大数据,揭示了在驯化和育种过程中番茄果实的营养和风味物质发生的变化,并发现了调控这些物质的重要遗传位点,为植物代谢物的分子机理研究提供了源头大数据和方法创新。近年来,随着基因组学,转录组学和蛋白质组学的发展,联合代谢物进行分析已经成为流行的趋势,不仅从现象中检测出差异的代谢物,更从基因层面解释了代谢物变化的原因,反之基因层面变化,导致的代谢物变化。
| [1] |
LESK A M. Introduction to bioinformatics[M]. Oxford: Oxford University Press, 2002. DOI:10.1007/978-1-59259-335-4
(  0) 0) |
| [2] |
陈铭. 生物信息学[M]. 北京: 科学出版社, 2012. CHEN Ming. Bioinformatics[M]. Beijing: Science Press, 2012. (  0) 0) |
| [3] |
HIROSAWA M, HOSHIDA M, ISHIKAWA M, et al. MASCOT: Multiple alignment system for protein sequences based on three-way dynamic programming[J]. Bioinformatics, 1993, 9(2): 161-167. DOI:10.1093/bioinformatics/9.2.161 (  0) 0) |
| [4] |
KENNY H, LENNART M, JOEL V, et al. MascotDatfile: An open-source library to fully parse and analyse MASCOT MS/MS search results[J]. Proteomics, 2007, 7(3): 364-366. DOI:10.1002/pmic.200600682 (  0) 0) |
| [5] |
MACCOSS M J, WU C C, YATES J R. Probability-based validation of protein identifications using a modified sequest algorithm[J]. Analytical Chemistry, 2002, 74(21): 5593-5599. DOI:10.1021/ac025826t (  0) 0) |
| [6] |
MOORE R E, YOUNG M K, LEE T D. Qscore: An algorithm for evaluating SEQUEST database search results[J]. Journal of the American Society for Mass Spectrometry, 2002, 13(4): 378-386. (  0) 0) |
| [7] |
PETER M, JOOST W, GOUU W J, et al. MSQuant: An open source platform for mass spectrometry-based quantitative proteomics[J]. Journal of Proteome Research, 2010, 9(1): 393-403. DOI:10.1021/pr900721e (  0) 0) |
| [8] |
MA B, ZHANG K, CHRISTOPHER H, et al. PEAKS: Powerful software for peptide de novo sequencing by tandem mass spectrometry[J]. Rapid Communications in Mass Spectrometry, 2003, 17(20): 2337-2342. DOI:10.1002/rcm.1196 (  0) 0) |
| [9] |
BERND F, VOLKER R, FRANZ R, et al. NovoHMM: A hidden Markov model for de novo peptide sequencing[J]. Analytical Chemistry, 2005, 77(22): 7265-7273. DOI:10.1021/ac0508853 (  0) 0) |
| [10] |
ALEXANDRE G, WILLY V B, AMOS B, et al. FindPept, a tool to identify unmatched masses in peptide mass fingerprinting protein identification[J]. Proteomics, 2002, 2(10): 1435-1444. DOI:10.1002/1615-9861(200210)2:10<1435::aid-prot1435>3.0.co;2-9 (  0) 0) |
| [11] |
CLAROS M G. MitoProt, a Macintosh application for studying mitochondrial proteins[J]. Computer Applications in the Biosciences Cabios, 1995, 11(4): 441-447. DOI:10.1093/bioinformatics/11.4.441 (  0) 0) |
| [12] |
PETERSEN T N, SOREN B, HEIJNE G V, et al. SIGNALP 4.0:discriminating signal peptides from transmembrane regions[J]. Nature Methods, 2011, 8(10): 785-786. DOI:10.1038/nmeth.1701 (  0) 0) |
| [13] |
HETZL J, FOERSTER A M, RAIDL G, et al. CyMATE: a new tool for methylation analysis of plant genomic DNA after bisulphite sequencing[J]. Plant Journal, 2007, 51(3): 526-536. DOI:10.1111/j.1365-313X.2007.03152.x (  0) 0) |
| [14] |
REN J, WEN L P, GAO X J, et al. CSS-Palm 2.0:An updated software for palmitoylation sites prediction[J]. Protein Engineering Design & Selection, 2008, 21(11): 639-644. DOI:10.1093/protein/gzn039 (  0) 0) |
| [15] |
XUE Y, ZHOU F F, FU C H, et al. SUMOsp: A web server for sumoylation site prediction[J]. Nucleic Acids Research, 2006, 34(W): 254-257. DOI:10.1093/nar/gkl207 (  0) 0) |
| [16] |
KIM M S, PINTO S M, GENTET D, et al. A draft map of the human proteome[J]. Nature, 2014, 509(7502): 575-581. DOI:10.1038/nature13302 (  0) 0) |
| [17] |
WILHELM M, SCHLEGL J, HAHNE H, et al. Mass-spectrometry-based draft of the human proteome[J]. Nature, 2014, 509(7502): 582-587. DOI:10.1038/nature13319 (  0) 0) |
| [18] |
BENJAMIN B S, JOSEPH C M, JAMES E P, et al. Genomic atlas of the human plasma proteome[J]. Nature, 2018, 558(7501): 73-79. DOI:10.1038/s41586-018-0175-2 (  0) 0) |
| [19] |
JIANG Y, SUN A, ZHAO Y, et al. Nature.Proteomics identifies new therapeutic targets of early-stage hepatocellular carcinoma[J]. Transplantation, 2019, 103(5): 855-857. DOI:10.1097/TP.0000000000002713 (  0) 0) |
| [20] |
FELIX H, CHRISTOPHER V, DEREK C, et al. Lineage tracing of human B cells reveals the in vivo landscape of human antibody class switching[J]. eLife, 2016, 5(e16578): 1-20. DOI:10.7554/eLife.16578 (  0) 0) |
| [21] |
ZHOU Q, LIU M, XIA X, et al. A mouse tissue transcription factor atlas[J]. Nature Communications, 2017, 8: 15089. DOI:10.1038/ncomms15089 (  0) 0) |
| [22] |
LI X J, ZHANG C C, GONG T Q, et al. A time-resolved multi-omic atlas of the developing mouse stomach[J]. Nature Communications, 2018, 9: 4910. DOI:10.1038/s41467-018-07463-9 (  0) 0) |
| [23] |
GONG T, ZHANG C, NI X, et al. A time-resolved multi-omic atlas of the developing mouse liver[J]. Genome Research, 2020, 30(2): 1-13. DOI:10.1101/gr.253328.119 (  0) 0) |
| [24] |
MERGNER J, FREJNO M, LIST M, et al. Mass-spectrometry-based draft of the Arabidopsis proteome[J]. Nature, 2020, 579(D1): 409-414. DOI:10.1038/s41586-020-2094-2 (  0) 0) |
| [25] |
LUCK K, KIM D K, LAMBOURNE L, et al. A reference map of the human binary protein interactome[J]. Nature, 2020, 580(D1): 1-7. DOI:10.1038/s41586-020-2188-x (  0) 0) |
| [26] |
ZHU G, WANG S, HUANG Z, et al. Rewiring of the fruit metabolome in tomato breeding[J]. Cell, 2018, 172(1/2): 249-261, e12. DOI:10.1016/j.cell.2017.12.019 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


