环状RNA是一类在前体mRNA(pre-mRNA)剪接过程中形成的新型内源RNA,与传统线性RNA不同的是其通过反向剪接使5’端和3’端共价连接,形成闭合环状结构(见图 1),因而不受核酸外切酶介导的降解的影响,比线性RNA更稳定[1-2]。多年来,环状RNA被认为是前体mRNA剪接过程中的副产品[3],但越来越多的研究表明环状RNA在生物体中通过多种途径发挥着重要的作用。环状RNA作为竞争性内源RNA,可以调控基因的转录和表达[4-5];一些环状RNA包含多个microRNA结合位点,可以充当microRNA海绵[6];环状RNA可以与蛋白相互作用,从而参与多种生物过程的调控[5, 7];另外,环状RNA还能够翻译蛋白[8]。

|
图 1 环状RNA的形成 Figure 1 Formation of circRNA |
为了更好地研究环状RNA,准确有效地鉴定环状RNA是至关重要的。多种因素可以促进环状RNA的形成,如侧翼区域的互补序列[9]、反向重复序列[10]、ALU和串联重复序列[2]以及SNP密度[11]等。这些因素与RNA分子的进化保守性和结构特征被认为是鉴定环状RNA的重要特征。目前,常见的环状RNA识别工具是通过识别高通量测序(RNA-seq)数据中的反向剪接位点来鉴定环状RNA,如find_circ[12]、CIRI[13]、circRNA_finder[14]、MapSplice[15]和CIRCexplorer[16]等。然而,已有相关研究[17-18]表明这些工具普遍存在较高的假阳性率和假阴性率,不同工具对同一个测序数据的检出重合率非常低,这是因为首先这些工具是基于高通量测序数据的,因此对表达丰度非常敏感,但大部分环状RNA通常是低表达的,在测序覆盖率较低情况下难以捕获,其次它们只利用了反向剪接位点信息来识别环状RNA,忽略了环化过程中其他因素的影响。
近年来,机器学习方法越来越多地应用于生物信息学研究。一些研究[19-20]分析环状RNA形成过程中的影响因素,通过训练传统的机器学习算法(支持向量机、随机森林和多核学习等)来鉴别环状RNA和长链非编码RNA(Long non-coding RNA,lncRNA),取得了较高的识别正确率。但是这些方法需要先进行特征分析,而且这些选取的特征不能全面充分地表征反向剪接过程。深度学习算法能够处理大规模数据并自动提取有效特征,可以弥补传统机器学习模型的不足。本文将从传统机器学习方法和深度学习方法这两个方面来介绍基于序列计算预测环状RNA的8种工具,并比较分析它们在测试数据集上的识别结果。
1 基于传统机器学习方法的工具 1.1 PredcircRNAPredcircRNA[19](https://github.com/xypan1232/predcircrna)采用了基于多种特征训练的多核学习框架模型,用来区分环状RNA和其他长链非编码RNA(其运行流程见图 2(a))。首先,从转录本中提取不同的特征—图特征、保守性分数、序列组成、ALU和串联(Tandem)重复序列、SNP密度和开放阅读框(Open Reading Frame,ORF)等。图特征用节点表示核苷酸,用边表示核苷酸之间的键关系,可以表示RNA分子的序列和结构。保守性分数是根据UCSC中下载的phyloP(Phylogenetic p-values)保守分数自定义的。序列组成特征包含三核苷酸特征、GC含量、序列长度、GT、AG、GTAG和AGGT的频率等。另外计算每个转录本的Alu重复序列数目,利用Tandem Repeats Finder检测串联重复序列并计算其频率,txCdsPredict获得每个转录本的开放阅读框并提取其长度和比例。为了融合多种特征,PredcircRNA使用多核学习方法,能有效地对环状RNA和其他lncRNA进行分类。PredcircRNA分析的这些特征(如保守性分数和GT/AG序列)对于鉴别环状RNA和lncRNA有重要作用,而且不同类型的特征相较于单一特征能相互补充从而提高模型能力。PredcircRNA正样本来自circBase数据库中的14 084条环状RNA,负样本来自GENCODE数据库中19 722条其他类型的lncRNA,随机选取10 000条环状RNA和相同数量的其他lncRNA进行模型训练,剩余的数据作为独立的测试数据集,通过5折交叉验证,准确率达到0.862。
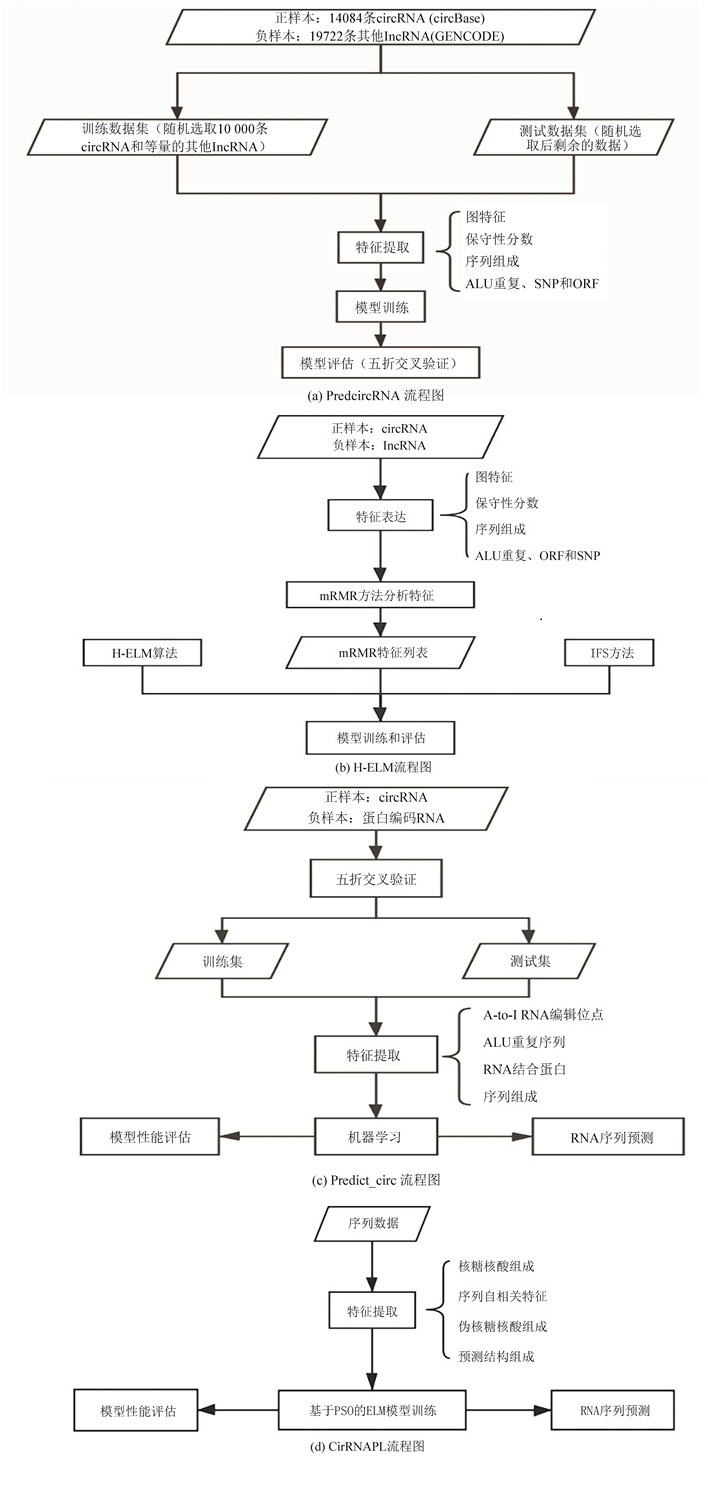
|
图 2 基于传统统计学习方法的工具流程图 Figure 2 Flowchart of tools based on statistical learning |
H-ELM[20]利用有特征选择功能的H-ELM(hierarchical extreme learning machine,层次极限学习器)算法来提取特征,进一步识别环状RNA和lncRNA(其流程原理见图 2(b))。该方法沿用了PredcircRNA中定义的特征,使用mRMR(minimum redundancy maximum relevance,最小冗余最大相关)方法对这些特征进行分析。利用获得的特征列表、IFS(incremental feature selection,增量特征选择)方法和H-ELM算法,建立最优分类模型。相较于PredcircRNA,H-ELM用mRMR方法对特征进行了分析选择,利用了IFS方法和H-ELM算法来建立模型。H-ELM使用PredcircRNA的数据,虽然在同样的数据集上通过十折交叉验证,H-ELM模型的准确率为0.789,低于PredcircRNA,但它通过特征分析,发现进化保守性、序列特征、特异性序列和结构是区分环状RNA和其他lncRNA的重要因素。
1.3 predict_circpredict_circ[21]通过选取剪接位点侧翼上下游内含子的长度、A-to-I密度、ALU重复序列和RNA结合蛋白(RBP)作用位点等100个与RNA成环相关的序列特征,建立机器学习模型来识别环状RNA(其原理见图 2(c)),并比较了随机森林和支持向量机的分类效果。结果表明,选取的序列特征能有效地鉴别RNA能否成环,同时不同序列特征对模型的分类预测能力的贡献也不同。predict_circ共选取了3组数据集和1组独立测试集,正样本来自5种环状RNA预测工具的检测结果交集、circBase中的人类环状RNA以及收集文献中的经过PCR验证的环状RNA,负样本来自UCSC人类基因数据hg19版本的编码蛋白序列(去除与环状RNA重合的转录本),随机抽取与每组正样本集数量相近的序列作为对应的负样本集。将每组数据的正负数据集中随机抽取2/3作为训练集,1/3作测试集,5折交叉验证后,每组数据的分类准确率均达到0.85以上。
1.4 CirRNAPLCirRNAPL[22](http://server.malab.cn/CirRNAPL/) 使用了基于粒子群优化(particle swarm optimization,PSO)算法的极限学习机(extreme learning machine,ELM)模型,能准确识别circRNA(其流程见图 2(d))。首先,提取序列数据的四个特征:核糖核酸组成、序列自相关特征、伪核糖核酸组成和预测结构组成。核糖核酸组成包括k-mer(k参数为2和3)、错配和子序列;序列自相关特征主要表征序列中核苷酸间的相关性;预测结构组成主要表示序列的结构特征。然后使用ELM识别环状RNA,并且通过利用PSO算法优化ELM的参数,提高其泛化能力,达到更高的识别精度。CirRNAPL选取了三组数据对模型进行了训练。第一组和第二组的正样本均使用PredCircRNA方法中使用的14 084条环状RNA数据,第一组和第二组的负样本分别为GENCODE v19版本的9 533条编码蛋白基因(protein-coding genes,PCGs)和1 973条lncRNAs;第三组数据的正样本是H1hsec干细胞中表达的2 082条circRNA,反之,负样本为H1hsec干细胞中未表达的相同数量的circRNA。将三组数据分为了训练集和测试集,并进行了十折交叉验证,最终在三组数据上的准确率分别为0.815,0.802和0.782。
2 基于深度学习方法的工具 2.1 DeepCirCodeDeepCirCode[23](https://github.com/BioDataLearning/DeepCirCode)是第一个采用深度学习模型来预测mRNA是否能反向剪接形成环状RNA的分类工具(其流程见图 3(a))。该方法采用卷积神经网络自动地从序列中学习相关特征——序列基序(Sequence motif)。因为有研究表明某些RBP等作用因子能通过特定的结合位点(序列基序)来促进RNA环化。DeepCirCode将候选反向剪接位点侧翼的内含子和外显子序列转换成二进制向量作为网络输入,通过识别侧翼序列中是否存在能促进环化的序列基序来预测环状RNA。通过分析DeepCirCode检测出的序列基序,发现其中一些确实与已知的RNA剪接、转录或翻译的基序相匹配。此外,通过对小鼠和果蝇数据进行测试,发现一些人类序列基序在小鼠和果蝇的序列中也存在,这说明这些基序在进化过程中存在保守性,很可能在环状RNA的生物发生过程中起着重要作用。DeepCirCode将circBase和circRNADb[24]两个公共数据库的环状RNA作为正样本数据集,按条件筛选出共7 964条人类外显子环状RNA,负样本是从GENCODE的人类参考基因组注释信息中随机选取相应的剪接位点,10折交叉验证的准确率为0.852 4,AUC为0.905 8。
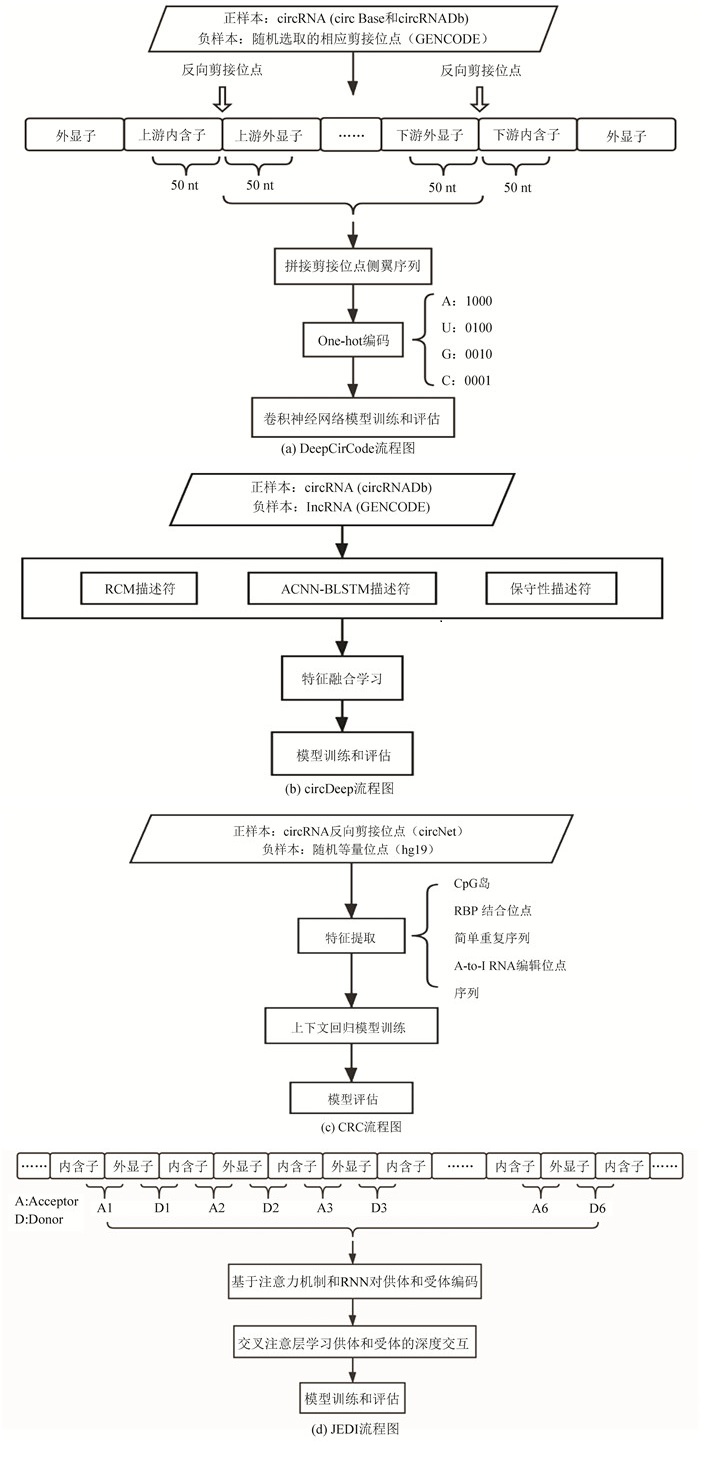
|
图 3 基于深度学习方法的工具流程图 Figure 3 Flowchart of tools based on deep learning |
circDeep[25](https://github.com/UofLBioinformatics/circDeep)采用端到端(End-to-End)的深度学习框架来区别环状RNA和lncRNA(流程原理见图 3(b))。circDeep引入了三种描述符(descriptor):RCM(Reverse Complement Matching,反向互补匹配)描述符、ACNN-BLSTM序列描述符和保守性描述符。RCM描述符目的是选取促进环化过程的潜在侧翼序列。ACNN-BLSTM序列描述符结合了ACNN(Asymmetric Convolution Neural Network,非对称卷积神经网络)和BLSTM(Bidirectional Long Short-Term Memory Network,双向长短期记忆网络),能够从每个序列中提取局部模式和远程作用(Long-range dependencies)。保守性描述符包含物种间特殊序列的保守信息和保守基序特征。为了融合三种不同的描述符,它使用了一种从不同方面的信息构建非线性表示的深度学习架构。其正样本来自circRNADb数据库的31 939条人类环状RNA,负样本来自GENCODE的19 683条其他类型lncRNA。将每个数据集划分为训练集(75%)、验证集(10%)和测试集(15%),模型训练后,测试集上的结果准确率达到0.941 7。
2.3 CRC(Contextual Regression for CircRNA)CRC[26](https://github.com/chl556/Contextual_Regression_for_CircRNA)基于环状RNA反向剪接位点的侧翼区域特征—CpG岛(Where long noncoding RNAs meet DNA methylation.)、RBP结合位点、简单重复序列、A-to-I RNA编辑位点和序列,通过上下文回归(contextual regression)模型来预测环状RNA的形成(流程原理见图 3(c)),接着还通过特征提取(feature extraction technique)和PCA获得10个特征主成分,在此基础上运用K均值聚类,将环状RNA分成7种亚型,这些亚型分别对应于已有的环状RNA生物发生机制。因此作者认为人类环状RNA具有多种不同的生物发生机制,可以分成多个不同的亚型。此外,CRC还发现环状RNA生物发生与侧翼区域CpG岛之间新的关联以及鉴定了相关的RNA结合蛋白。CRC从circNet数据库中收集55 689个人类环状RNA反向剪接位点作为正样本,在hg19人类基因组上随机选择等量的位点作为负样本,然后将数据随机划分为训练集和测试集(比例为7∶3),通过十次训练,达到平均准确率为0.726和AUC值为0.801。
2.4 JEDIJEDI(Junction Encoder with Deep Interaction)[27](https://github.com/hallogameboy/JEDI) 用深度学习方法对剪接位点及其深层相互作用建立模型,直接从基因或转录本序列中预测环状RNA。JEDI对序列里每个外显子和内含子连接位点进行基于深度双向循环神经网络的编码,然后用交叉注意层(Cross-attention layer)对反向剪接位点的深层相互作用建模(见图 3(d))。JEDI不仅能够预测环状RNA,而且能够解释剪接位点间的关系,从而发现基因内的反向剪接。另外对小鼠环状RNA的研究结果表明,JEDI预测人类环状RNA的模型也适用于小鼠环状RNA数据。JEDI选取了三组数据。第一组:正样本来自circRNADb的31 939条人类环状RNA,负样本来自GENCODE参考注释的19 683条其他lncRNA,进行5次交叉验证后所得准确率达到0.989 9;第二组:正样本来自circRNADb的每条环状RNA对应的7 777条基因,对于负样本,去除了正样本中选择的基因,得到7 000条基因,经过5折交叉验证,模型达到准确率0.964 6;第三组:正样本来自circBase的1 522条小鼠环状RNA,负样本来自GENCODE参考注释的1 522条其他lncRNA,模型训练后,准确率为0.886 8。
3 环状RNA预测工具的性能比较为了在同一标准下比较分析以上算法的功能,我们利用公共数据库circRNADb中人类环状RNA数据对以上工具进行测试。其中,H-ELM未提供代码下载链接,PredcircRNA工具所需比对数据库目前不提供下载,CRC需要对数据计算一组特征值,但相应的计算代码并未提供,CirRNAPL虽然提供了网站服务,但是网站上不方便处理大量的数据,也没有提供工具的下载,所以本文最后只进行了基于深度学习算法的三种工具(DeepCirCode,circDeep和JEDI)的比较。circRNADb公共数据库中共有32 194条人类环状RNA,去除长度短于200 nt的环状RNA,并过滤掉剪接位点侧翼内含子序列和两端外显子序列短于50 nt的环状RNA,最终获得13 264条序列作为正样本数据集。提取GENCODE[28]v19版本人类参考基因组注释的其他类型的lncRNA,剔除与circBase[29]和circRNADb中环状RNA序列相重叠的序列,得到8 125条lncRNA作为负样本数据集。分别将正负样本集中75%的数据作为训练集,25%的数据作为测试数据,对以上三种模型进行训练和测试,并采用以下指标来评估模型在测试集上的性能:准确度(Acc)、灵敏度(Sn)、特异性(Sp)和马修斯相关系数(MCC),分别定义如下:
| $ Acc = \frac{{TP + TN}}{{TP + FP + TN + FN}} $ | (1) |
| $ Sn = \frac{{TP}}{{TP + FN}} $ | (2) |
| $ Sp = \frac{{TN}}{{TN + FP}} $ | (3) |
| $ Mcc = \frac{{TP*TN - FP*FN}}{{\sqrt {\left( {TP + FP} \right)\left( {TP + FN} \right)\left( {TN + FP} \right)\left( {TN + FN} \right)} }} $ | (4) |
结果见表 1和图 4,这表明这三种基于深度学习算法的工具对于识别环状RNA都有较好的效果,尤其是JEDI,在测试集上的识别正确率达到了97.89%。这三个工具中,circDeep运行时间最长,因为该算法需要耗费大量时间提取特征。
| 表 1 模型分类性能打分 Table 1 Performance in model classification |

|
图 4 测试分类结果ROC曲线 Figure 4 ROC curves of prediction results of testing dataset |
环状RNA通过非经典方式进行反向剪接而成,通常认为剪接位点侧翼区域的反向互补序列和RBP结合位点等是促进内含子区域配对从而介导反向剪接形成环状RNA分子。本文主要介绍了8种基于序列预测环状RNA的工具。这8种工具均基于RNA序列来挖掘其内在特征,利用不同的机器学习算法来识别环状RNA,其优缺点见表 2所示。PredcircRNA、H-ELM以及predict_circ发展了不同的策略来提取特征,并使用了传统的统计学习算法(PredcircRNA的多核学习,H-ELM的层次极端学习机,predict_circ的支持向量机和随机森林及CirRNAPL的基于粒子群优化算法的极限学习机)来构建分类器。这一类方法需要预先进行选择和计算特征,而且提取的特征是专门用于描述序列某方面的性质,因此需要一定的先验知识为基础。DeepCirCode、circDeep、CRC和JEDI使用深度学习算法可以自动地从原始序列中学习复杂模式。DeepCirCode使用卷积神经网络对反向剪接位点的侧翼序列进行学习,circDeep使用卷积神经网络和双向长短时记忆网络对序列进行编码,CRC对反向剪接位点的侧翼区域特征建立基于卷积神经网络的上下文回归模型,JEDI使用深度双向循环神经网络编码序列并通过交叉注意层构建反向剪接的深层相互作用模型。卷积神经网络能够获得重要的序列局部模式来进行预测,但是无法识别每个剪接位点的位置信息。circDeep通过应用循环神经网络学习序列信息,弥补了卷积神经网络的不足,但是忽略了一些基本的信息(如剪接位点)。CRC虽然能通过深度学习方式识别环状发生过程,但输入特征中包含了基于统计的信息。JEDI相较于前几种深度学习预测工具,只对序列剪接位点周围的侧翼区域进行建模,不需要其他特征信息,充分挖掘了序列的剪接位点信息及其深度相互作用信息,可以自动发现反向剪接的位点,而无需任何注释,还能够很好地保留形成环状RNA的剪接位点信息和其他重要信息,因此在模型评估的各衡量指标中都取得了最好的表现。
| 表 2 预测环状RNA工具优缺点 Table 2 Advantages and disadvantages of circRNA prediction tools |
无论是基于传统机器学习方法还是深度学习方法,以上模型都是从序列中挖掘局部信息,但受限于知识和方法的不足,已经利用的序列信息(序列基序,ALU序列和剪接位点等)还是不足以完全地解释RNA成环机制。序列的上下游调控信息、远程调控信息、RNA与蛋白质互作信息和RNA结构等是现有工具未挖掘到的一些信息。如何更全面地挖掘信息并有效地表征,是环状RNA识别工具开发的一个可能方向。
本文通过比较分析现有工具各自特征提取的侧重点和方法的优劣,目的在于帮助大家在研究过程中选择合适的工具,也希望能对开发出更好的预测环状RNA的算法和工具有所启发,从而推进对环状RNA形成机制的研究和功能的探索。
| [1] |
SALZMAN J, GAWAD C, WANG P L, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J]. PLoS One, 2012, 7(2): e30733. DOI:10.1371/journal.pone.0030733 (  0) 0) |
| [2] |
JECK W R, SORRENTINO J A, WANG K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats[J]. RNA, 2013, 19(2): 141-157. DOI:10.1261/rna.035667.112 (  0) 0) |
| [3] |
DANAN M, SCHWARTZ S, EDELHEIT S, et al. Transcriptome-wide discovery of circular RNAs in Archaea[J]. Nucleic Acids Research, 2012, 40(7): 3131-3142. DOI:10.1093/nar/gkr1009 (  0) 0) |
| [4] |
QU S, YANG X, LI X, et al. Circular RNA: A new star of noncoding RNAs[J]. Cancer Letters, 2015, 365(2): 141-148. DOI:10.1016/j.canlet.2015.06.003 (  0) 0) |
| [5] |
ASHWAL-FLUSS R, MEYER M, PAMUDURTI N R, et al. circRNA biogenesis competes with pre-mRNA splicing[J]. Molecular Cell, 2014, 56(1): 55-66. DOI:10.1016/j.molcel.2014.08.019 (  0) 0) |
| [6] |
HANSEN T B, JENSEN T I, CLAUSEN B H, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384-388. DOI:10.1038/nature11993 (  0) 0) |
| [7] |
CONN S J, PILLMAN K A, TOUBIA J, et al. The RNA binding protein quaking regulates formation of circRNAs[J]. Cell, 2015, 160(6): 1125-1134. DOI:10.1016/j.cell.2015.02.014 (  0) 0) |
| [8] |
WANG Y, WANG Z. Efficient backsplicing produces translatable circular mRNAs[J]. RNA, 2015, 21(2): 172-179. DOI:10.1261/rna.048272.114 (  0) 0) |
| [9] |
IVANOV A, MEMCZAK S, WYLER E, et al. Analysis of intron sequences reveals hallmarks of circular RNA biogenesis in animals[J]. Cell Reports, 2015, 10(2): 170-177. DOI:10.1016/j.celrep.2014.12.019 (  0) 0) |
| [10] |
DUBIN R A, KAZMI M A, OSTRER H. Inverted repeats are necessary for circularization of the mouse testis Sry transcript[J]. Gene, 1995, 167(1/2): 245-248. DOI:10.1016/0378-1119(95)00639-7 (  0) 0) |
| [11] |
THOMAS L F, SAETROM P. Circular RNAs are depleted of polymorphisms at microRNA binding sites[J]. Bioinformatics, 2014, 30(16): 2243-2246. DOI:10.1093/bioinformatics/btu257 (  0) 0) |
| [12] |
MEMCZAK S, JENS M, ELEFSINIOTI A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333-338. DOI:10.1038/nature11928 (  0) 0) |
| [13] |
GAO Y, WANG J, ZHAO F. CIRI: An efficient and unbiased algorithm for de novo circular RNA identification[J]. Genome Biology, 2015, 16(1): 4. DOI:10.1186/s13059-014-0571-3 (  0) 0) |
| [14] |
WESTHOLM J O, MIURA P, OLSON S, et al. Genome-wide analysis of drosophila circular RNAs reveals their structural and sequence properties and age-dependent neural accumulation[J]. Cell Reports, 2014, 9(5): 1966-1980. DOI:10.1016/j.celrep.2014.10.062 (  0) 0) |
| [15] |
WANG K, SINGH D, ZENG Z, et al. MapSplice: accurate mapping of RNA-seq reads for splice junction discovery[J]. Nucleic Acids Research, 2010, 38(18): e178. DOI:10.1093/nar/gkq622 (  0) 0) |
| [16] |
ZHANG X O, WANG H B, ZHANG Y, et al. Complementary sequence-mediated exon circularization[J]. Cell, 2014, 159(1): 134-147. DOI:10.1016/j.cell.2014.09.001 (  0) 0) |
| [17] |
HANSEN T B, VENO M T, DAMGAARD C K, et al. Comparison of circular RNA prediction tools[J]. Nucleic Acids Research, 2016, 44(6): e58. DOI:10.1093/nar/gkv1458 (  0) 0) |
| [18] |
ZENG X, LIN W, GUO M, et al. A comprehensive overview and evaluation of circular RNA detection tools[J]. PLoS Computational Biology, 2017, 13(6): e1005420. DOI:10.1371/journal.pcbi.1005420 (  0) 0) |
| [19] |
PAN X, XIONG K. PredcircRNA: Computational classification of circular RNA from other long non-coding RNA using hybrid features[J]. Molecular Biosystems, 2015, 11(8): 2219-2226. DOI:10.1039/c5mb00214a (  0) 0) |
| [20] |
CHEN L, ZHANG Y H, HUANG G, et al. Discriminating cirRNAs from other lncRNAs using a hierarchical extreme learning machine (H-ELM) algorithm with feature selection[J]. Molecular Genetics & Genomics, 2018, 293(1): 137-149. DOI:10.1007/s00438-017-1372-7 (  0) 0) |
| [21] |
周晶, 谢雪英, 顾万君. 基于序列特征的环状RNA识别[J]. 生物信息学, 2018, 16(2): 113-118. ZHOU Jing, XIE Xueying, GU Wanjun. Identification of circular RNAs using genomic sequence features[J]. Chinese Journal of Bioinformatics, 2018, 16(2): 113-118. DOI:10.3969/j.issn.1672-5565.201709002 (  0) 0) |
| [22] |
NIU M, ZHANG J, LI Y, et al. CirRNAPL: A web server for the identification of circRNA based on extreme learning machine[J]. Computational and Structural Biotechnology Journal, 2020, 18: 834-842. DOI:10.1016/j.csbj.2020.03.028 (  0) 0) |
| [23] |
WANG J, WANG L. Deep learning of the back-splicing code for circular RNA formation[J]. Bioinformatics, 2019, 35(24): 5235-5242. DOI:10.1093/bioinformatics/btz382 (  0) 0) |
| [24] |
CHEN X, HAN P, ZHOU T, et al. circRNADb: A comprehensive database for human circular RNAs with protein-coding annotations[J]. Scientific Reports, 2016, 6(1): 34985. DOI:10.1038/srep34985 (  0) 0) |
| [25] |
CHAABANE M, WILLIAMS R M, STEPHENS A T, et al. circDeep: deep learning approach for circular RNA classification from other long non-coding RNA[J]. Bioinformatics, 2020, 36(1): 73-80. DOI:10.1093/bioinformatics/btz537 (  0) 0) |
| [26] |
LIU C, LIU Y C, HUANG H D, et al. Biogenesis mechanisms of circular RNA can be categorized through feature extraction of a machine learning model[J]. Bioinformatics, 2019, 35(23): 4867-4870. DOI:10.1093/bioinformatics/btz705 (  0) 0) |
| [27] |
JIANG J, JU C J T, HAO J, et al. JEDI: Circular RNA prediction based on junction encoders and deep interaction among splice sites[J]. BioRxiv, 2020, 2020-2022. DOI:10.1101/2020.02.03.932038 (  0) 0) |
| [28] |
FRANKISH A, DIEKHANS M, FERREIRA A M, et al. GENCODE reference annotation for the human and mouse genomes[J]. Nucleic Acids Research, 2019, 47(D1): D766-D773. DOI:10.1093/nar/gky955 (  0) 0) |
| [29] |
GLAZAR P, PAPAVASILEIOU P, RAJEWSKY N. circBase: A database for circular RNAs[J]. RNA, 2014, 20(11): 1666-1670. DOI:10.1261/rna.043687.113 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


