2. 清华大学 人工智能研究院,北京 100084
2. Institute for Artificial Intelligence, Tsinghua University, Beijing 100084, China
DNA作为生物重要的遗传物质,一直被生物学家在不同层面进行深入研究。从发现DNA的结构开始,人类通过研究复杂多样的基因组,在健康和疾病的研究中已取得了长足的进步。为了研究DNA序列,DNA测序是必不可少的环节。由于现有技术仅能测得较短的DNA序列,因此将DNA测序获得的读段(Read)进行从头拼接(De novo assembly)成为较长的重叠群(Contig)、进而拼装成更长的骨架(Scaffold)、最终获得完整的基因组(Genome)一直是生物信息学的重要课题。而随着测序技术的发展,测序试剂和仪器不断更新换代,测序的速度、价格,以及测序获得的读段在质量、长度、碱基偏好等方面特性也有变化,针对新的测序数据设计更适应数据的拼装算法也是不断推陈出新。
1977年,由Sanger等人提出的链终止法测序方法开创了测序技术的先河[1],这种测序方法后来通常被称为Sanger测序法,即第一代测序技术。第一代测序技术的主要特点是获得的读段序列长度较长,通常可达1 000 bp,准确性高,可达99.999%,但是测序的成本比较高,通量也比较低。这些特点决定了第一代测序技术通常被应用于长度较短,或者十分重要的基因组测序任务中。
为了解决第一代测序技术具有通量低、成本高的局限性,454焦磷酸测序[2],Illumina[3]等测序仪器相继诞生。这些新一代测序仪可以获得每次运行上百万上亿次读数的更高输出,但是读取长度更短,最新一代的Illumina机器现在输出大约250-300 bp的高质量读数。这些测序技术现在被称为第二代测序技术。第二代测序技术还包括Ion Torrents的Ion Proton测序仪。与Sanger,454和Illumina相比,这种技术不依赖于光学方法,可以使用购买成本较低的机器进行快速而便宜的测序。尽管第二代测序仪具有高通量、相对便宜的价格,同时具有高质量的碱基和低错误率,但它们测得的读段较短是主要问题。现在,第二代测序技术可以测得包括复杂的哺乳动物在内的绝大多数生物的基因组,并保证成本控制在可接受的范围内。这意味着几乎所有生物的基因组都向人类打开了解码的道路,并允许人们对这些基因组做进一步的分析。
随着技术的发展,第三代测序技术应运而生。由Pacific Biosciences(PacBio)公司开发的测序仪(如RS Ⅰ,RS Ⅱ和Sequel机器)使用单分子实时(SMRT)测序技术[2],而牛津纳米孔技术(ONT)开发了用于纳米孔测序的装置[4]。与第二代技术(如454,Illumina,Ion Torrent)相比,这些第三代测序方法在测序文库制备过程中不包括扩增步骤,因此能够进行单分子测序,去除了扩增过程带来的偏倚(Bias)。此外,第三代测序技术预期的读段长度远远高于第二代技术,平均读段长度可以超过6-8 kbp,最大读段长度超过30-150 kbp[5]。使用第三代测序长读段文库可以定向、排序、间隔或连接基因组拼接中的重叠群,以提升这些基因组草图的质量。PacBio的SMRT长读段已经证明在解析长重复区域方面是有效的[6],并且可能成为原核生物基因组测序的金标准。此外,SMRT长读段已被用于解决黑猩猩[7]和人类[8]的复杂基因组区域。但是第三代测序仪的比较高的错误率决定了它们还不能完全取代第二代测序测序平台。对于许多组学问题研究,比如宏基因组学,第二代技术至少在未来几年仍将是最先进的技术[9]。
尽管从头拼接算法取得了长足的进展,但短读段或长读段技术的固有问题仍然阻碍了完整的基因组的构建。一方面,由于第二代测序数据误差小,组装第二代测序数据集可以产生准确的重叠群,但由于它们读段尺寸太小而无法识别更大的基因组重复序列。另一方面,由于第三代测序数据的长度优势,纯第三代测序数据装配算法(例如HGAP[7])可以轻松地解决较大的重复区域,但是为了最小化错误率的影响以得到较高的准确率,需要巨大的测序深度。自第一个SMRT测序平台发布,就出现了利用这两种测序方法的互补性产生各种组装混合数据集的想法以利用两者的混合拼接算法。
1 现有拼接算法尽管测序手段不断升级,但无论是二代测序数据拼接还是二三代混合拼接,对测序数据进行拼接的算法思路是整体一致的。由于基因组中绝大部分的区域是没有互相重复的,所以通过比对读段上的碱基序列,找到他们的重叠区域(Overlap),然后通过将这些读段通过重叠区域连接起来以得到更长的序列,即重叠群(Contig)。由于拼接得到的重叠群数量通常远多于实际的染色体数量,因此需要第二阶段的拼接骨架以将部分碎片拼接为更长的序列。拼接骨架(Scaffolding)是将重叠群按照正确的顺序和方向进行集合并进行连接的过程,其中间可能存在一些没有具体测序数据间隙(Gap)以N(即未知碱基)进行填充,最终得到的序列被称为骨架(Scaffold)。通过包括对特定的一些间隙进行填充(Gap fill)等方式,对拼接得到的进一步进行基因组后处理(Genome finishing),即可以得到相应物种的基因组草图。
无论是将读段连接为重叠群,还是进一步的骨架拼接,都可以归纳为如下数学模型:在读段/重叠群间重叠信息的约束条件下,确认所有读段/重叠群的顺序和方向。可以证明,确定所有读段/重叠群的顺序等价于最优线性排列问题,这一问题是NP难的(NP-hard)[10]。因此,寻求最优解在计算复杂度上目前而言是不可行的,拼接算法需要寻求某种近似解,以在合理的时间和空间复杂度下获得准确可以率接受的解。现有的拼接算法大体可以按以下分类:
1.1 贪心法贪心法[11]是最早采用于基因拼接算法的方法(见图 1)。贪心法的策略是选取某一问题的初始解,然后选择最小代价的步骤,转移到下一个逼近最终目标的解,如此反复以达到最终目标或者其他终止条件。在基因组拼接问题中,贪心法的具体步骤如下:首先选取一个读段作为初始重叠群,然后寻找与当前重叠群重叠区域相似度得分最高的读段,与当前重叠群进行拼接,得到新的重叠群,直到没有读段与当前重叠群足够相似。思路简单、复杂度低是贪心法的主要优势,但该方法只能找到局部最优解。如何从局部最优逼近全局最优是该方法面临的主要挑战。使用这一方法的软件有SSPACE等。

|
图 1 贪心法图解 Figure 1 An example of the greedy method 注:由于测序误差、不同区域间的重复序列等原因,贪心法可能选择错误读段扩展. |
OLC算法(Overlap-Layout-Consensus)[12]如其名字所示,主要可分为以下三个步骤:(1)Overlap:对所有读段进行两两比对,找到读段间的重叠信息。但由于两两比对的复杂度太高,实际算法通常先采取某种方式筛选可能比较相似的读段对,仅比较这些可能相似的读段,其余读段对相似度视为0。(2)Layout:根据得到的重叠信息将测序读段视为图上的点,读段间的相似关系视为连线,构建OLC图(见图 2)。(3)Consensus:利用OLC图上的连接信息,找到遍历图上所有节点的最佳路径,即寻找图上的哈密顿道路,这也导致该算法复杂度通常较高,通常用于读段较长、总读段数较少的拼接。使用这一方法的软件有Celera Assembler等。
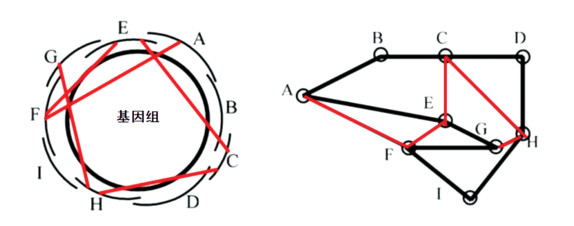
|
图 2 OLC法图解 Figure 2 An example of the OLC method 注:图中左侧为基因组上序列实际对应的位置,红色连接是因重复序列等原因产生的的重叠信息。右图为实际得到的OLC图,OLC算法需要排除错误的红色边,找到真实的读段间顺序,即图中黑色边构成的路径。 |
De Bruijn图[13]是另外一种被许多流行的拼接软件使用的图模型算法,该算法首先被拼接软件EULER提出。该算法首先将每个读段分解为一个k-mer(此处使用的长度k远远小于OLC法中使用的长度),并以这些k-mer为边,(k-1)-mer为节点构建一张图。如果两个(k-1)-mer在k-mer上连续出现,则他们在图上会连接一条边,每条读段均会形成一条链,将这些链上的点和边的信息整合到一张图上,就得到了De Bruijn图(见图 3)。这种方法没有显式地对读段间的重叠信息进行计算,而是通过k-mer建图的方式进行有效的捕获,从而节省了大量的计算时间。接下来,算法通过遍历DBG中的无分支路径的方式来构造重叠群。然而,由于测序错误,或者存在重复序列,或者基因组的多样性如多条染色体,都会导致高度相似的序列中存在局部差异,这些差异在图中就表现为一些“分叉”和“气泡”,导致得到的高质量的重叠群非常短。为了解决这一问题,程序会预先在图中搜索分叉和气泡并检测对应读段是否可能是错误序列,尽可能地剪除分叉和气泡。如果路径对应的读段非常少,则说明该路径很可能是测序错误导致的,因而应该删去。实践证明,如果在实际装配过程前加入错误校正步骤,可以得到更好的拼接结果。该方法的主要缺点是在将读段转化为k-mer路径的过程中,大量k-mer间的信息丢失,导致图上路径的正确性很容易受到其他k-mer连入边的干扰。同时,由于该方法使用了大量基于图的计算,导致难以进行并行化处理,因而在处理哺乳动物等复杂生物的基因组测序数据时不能有效拓展。除此之外,图的存储需要极其大量的内存,因而限制了该方法的应用范围。使用这一方法的软件有SPAdes等。
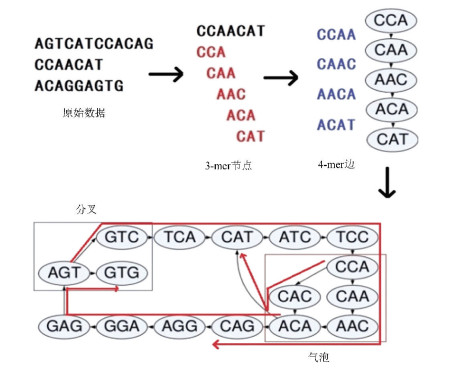
|
图 3 De Bruijn图法图解 Figure 3 An example of the De Bruijn graph method 注:图中三条读段作为原始数据,分别先以长度为3的k-mer为节点进行分割、再用长度为4的k-mer为边连接节点,形成一条读段链;然后整合三条读段链上k-mer的连接信息生成De Bruijn图的示例。三条读段链以折线标注在图中。图中右下的方框内为一个“气泡”,左上的方框内为一个“分叉”。 |
除了以上基本方法之外,还有一些工具通过结合以上方法的优势,提出组合式的新算法。例如Pacific Biosciences公司开发的软件FALCON提出的序列图(String graph)算法[14]。该方法首先使用OLC中的方法计算读段间的重叠信息,然后根据重叠区域的边缘将序列切割成长度不等子序列,并以这些子序列为节点,在同一读段中的连接顺序为边建立类似DBG的图结构。然后在该图上遍历无分支路径获得重叠群。
2 混合拼接软件介绍自第三代测序问世以来,已经有许多混合拼接工具问世。这些工具除了在拼接算法主要思路的选取上有所不同,如何结合使用二、三代测序数据的方式也各有异同。大体而言,可以主要分为以下几种:
2.1 三代骨架拼接用二代数据拼接为重叠群,然后结合三代数据进行骨架拼接(见图 4)。这一方式的主要创新点在骨架拼接阶段。该方式的优点在于可以利用之前二代测序单独拼接、骨架拼接的相关成果。但是如果由于重复区域等原因,二代拼接结果有较多错误,难以利用三代测序数据对二代拼接结果进行直接校正,这是该方法的不足。采用这种方式的软件有:AHA[15],Cerulean[16]和SSPACE-LongRead[17]等。
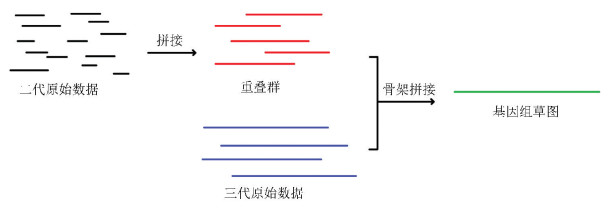
|
图 4 三代骨架拼接 Figure 4 Third-generation sequencing scaffolding 注:先以二代数据拼接为重叠群,然后结合三代数据进行骨架拼接成基因组草图。 |
将二代测序数据比对到三代测序数据上,对三代测序数据进行校正,再使用校正后的三代数据进行拼接(见图 5)。这种方法的创新点主要在比对和校正三代测序数据上。校正后的流程与前代拼接流程需求基本相同,但由于读段长度变化极大,拼接方法的选择相对比较受限,Celera Assembler (runCA)[18]是通常的选择。采用这种方式的软件有:PBcR pipeline[19],此外还有专门的纠错软件Proovread[20]。

|
图 5 校正-拼接 Figure 5 Correction-assembly 注:先将二代测序数据比对到三代测序数据上,对三代测序数据进行校正,再使用校正后的三代数据进行拼接。 |
将二代测序数据进行预处理,再将重叠群比对到三代测序数据上,对三代测序数据进行校正,再使用校正后的三代数据进行拼接。由于第二代测序的读段较短,而第三代读段错误率较高,直接将第二代测序数据比对到第三代测序数据通常会得到较多可能的正确位置,直接选出正确位置是一个相当具有挑战性的问题。为了优化这一问题,先对第二代测序数据进行预处理,如拼接成重叠群、构建de-Bruijn图、将相邻的相同碱基压缩等方式。以拼接成重叠群为例,这相当于增加了读段的长度,可以减少可能比对到的位置,提高了比对和校正的准确性。但是如何矫正可能的二代数据错误拼接是这一方法新引入的问题。ECTools[21]采取的方式是将二代数据拼接为重叠群,LoRDEC[22]采取的方式是先用二代测序数据构建de-Bruijn图,LSC[23]采取的方式是将相邻的相同碱基压缩为一个以应对三代数据插入、删除错误较多的问题。拼接流程图见图 6。
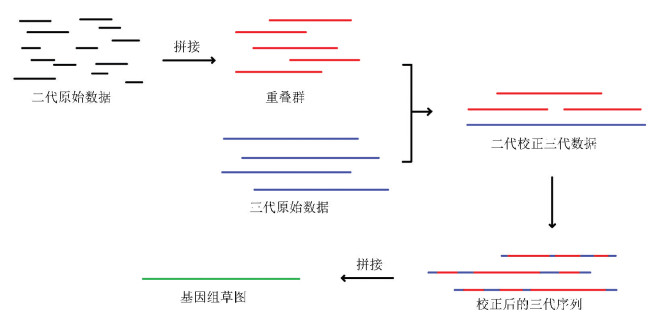
|
图 6 拼接-校正-拼接 Figure 6 Assembly-correction-assembly 注:先将二代测序数据进行拼接,再将二代重叠群比对到三代测序数据上,对三代测序数据进行校正,最后使用校正后的三代数据进行拼接。 |
即直接将第二代、第三代测序数据混合输入,设计综合使用两种数据的拼接算法。这种方案理论上可以更好地应用两种数据的优势,但算法通常复杂。采取这种方式的有SPAdes-Hybrid[24]的混合拼接,其主要思路为先用二代测序数据构建de-Bruijn图,然后利用三代测序数据在图上的比对结果计算得到重叠群。
3 性能比较为了比较不同混合拼接软件的拼接效果、拼接性能等指标,Hsin-Hung Lin等人[25]使用Escherichia coli K12 MG1655 (E. coli), 151-bp paired end二代真实测序数据,S. Koren等人研究中发布的第三代SMRT测序数据数据(SMRT1)[19],Pacific Biosciences’ DevNet网站(http://pacificbiosciences.github.io/DevNet/)发布的第三代SMRT E. coli测序数据(SMRT2) 进行测试。其中,SMRT1读段平均长度为2.0 Kbp,测序深度为16X;SMRT2读段的平均长度为2.6 Kbp,测序深度23X。测试中,二代测序数据拼接使用软件Abyss[26]先进行二代数据单独从头拼接,为需要二代测序数据拼接的软件提供支持。拼接结果使用QUAST[27]软件与参考基因组(NCBI reference sequence NC000913.2)进行评估,该软件是被广泛使用对组装结果的质量进行评估的软件。评估的主要指标为NGA50。NGA50是将所有拼接得到的重叠群在其错误拼接处打断,然后统计这些打断后的重叠群按长度从大到小排列,然后依次相加,当相加结果恰好超过参考基因组总长度的50%时,取该重叠群的长度作为NGA50的值。使用该指标是评估拼接结果准确性和连续性的指标。对该文章中的测试结果进行整合后,绘制出图 7。其中部分软件运行结果给出了更详尽的数据,整理后列在表 1中。

|
图 7 以NGA50度量的不同拼接软件结果对比 Figure 7 Comparison of NGA50 results of different assembly software tools 注:Abyss为二代数据单独拼接的结果,AHA、Cerulean和SSPACE-LongReads使用Abyss拼接得到的重叠群和三代SMRT数据(SMRT1和SMRT2)进行骨架拼接。PBcR pipeline直接以二代和三代数据作为输入,并输出拼接结果。ECTools + runCA、LoRDEC + runCA、LSC + runCA、Proovread + runCA:分别为使用ECTools、LoRDEC、LSC、Proovread对三代测序数据进行校正,然后用runCA进行从头拼接。 |
| 表 1 不同长读段校正工具参与的混合拼接结果比较 Table 1 Comparison of hybrid assembly results with different assembly strategy softwares |
从图 7中,可以看出,在本次实验中,AHA,Cerulean和SSPACE-LongRead这三款三代骨架拼接的软件中,AHA和SSPACE-LongRead的效果稳定,受二代拼接结果和三代测序读段情况影响较小,但是结果最差;而Cerulean拼接结果受二代拼接结果和三代测序读段情况的影响较大,拼接性能较为不稳定,在测序深度和平均长度较小的SMRT1的数据中表现较好,这可能是由于在骨架拼接中,长读段的测序深度不需要太高,过高的测序深度反而引入更多的噪音影响软件性能。校正-拼接软件PBcR pipeline受三代测序读段的情况影响非常大,结果很不稳定,好的结果超过前三者,但是差的结果甚至不如二代数据单独拼接。PBcR在三代测序深度更高的SMRT2上效果更好,这可能是由于其需要使用矫正后的三代序列进行单独拼接,因此更高的三代测序深度可以得到更好的结果。运行时间相对较短是该方法的优点。ECTools + runCA等拼接-校正-拼接的软件组合,都可以得到较好的结果,相较之下ECTools + runCA和Proovread + runCA的结果好于其他两个组合。此外,这ECTools + runCA也在覆盖深度较高的SMRT2数据上结果较好,可以说明长读段测序深度的增加这类软件组合最终拼接质量。相比而言,这类算法运行时间整体偏长。
为了对完全混合拼接方法的软件Hybrid-SPAdes进行性能测试,本文在大肠杆菌数据集(E.colistr.K12)上对其进行了测试。使用的大肠杆菌数据集包括:2代WGS配对数据(测序仪器Illumina HiSeq 2000),读段长度150 bp,数据总量37.5 Gbp,下载自NCBI SRA数据库,编号SRR8365224;3代SMRT WGS数据(测序仪器PacBio RS Ⅱ),读段平均长度1 027 bp,测序深度22X,数据总量1.2 Gbp,下载自NCBI SRA数据库,编号SRR2063112。此外本文还使用了PBSIM []模拟器生成了不同深度的3代SMRT测序数据以测试。拼接参数选择默认参数(16线程并行),最后拼接得到结果以QUAST软件评估,具体结果如表二所示。从模拟数据的结果上,可以看到NGA50指标变化较小,说明Hybrid-SPAdes对三代测序深度的鲁棒性较好,但综合其他指标,还是可以看到拼接结果随着测序深度增加而变好。另外真实数据上的结果远远好于同等测序深度下的拼接结果,关键指标NGA50比深度更深(40X)的模拟数据结果,说明现有模拟器生成的模拟数据与实际测序数据仍然有较大偏差,比较应以真实数据为主。真实数据上,Hybrid-SPAdes的NGA50指标为492.2 kbp,拼接结果质量较为一般,与PBcR pipeline的NGA50较为接近。内存消耗方面,Hybrid-SPAdes内存消耗相对较小且非常稳定,但在运算时间方面有一定波动,但整体用时大约在3 h左右,相对较少。结果见表 2。
| 表 2 Hybrid-SPAdes拼接结果 Table 2 Hybrid-SPAdes assembly results |
本文分析了在二代、三代测序数据混合拼接领域的现有软件,并比较了他们测试性能,希望为需要使用二代、三代测序数据进行相关生物学研究的团队提供参考,方便选出更合适的拼接软件。通过文中的比较,拼接-校正-拼接的软件组合性能较好,可以优先考虑使用,其中以ECTools + runCA和Proovread + runCA的组合最为推荐。此外,如果三代测序数据深度较低,三代骨架拼接的Abyss+Cerulean的软件组合也是可以考虑尝试的。如果三代测序深度较高质量较好,则可以考虑尝试校正-拼接的软件PBcR pipeline。而无论三代数据测序深度如何,完全混合拼接软件Hybrid-SPAdes都是值得尝试的。不同混合拼接策略的比较整理在表 3中。
| 表 3 不同拼接策略混合拼接软件比较 Table 3 Comparison of hybrid assembly softwares with different assembly strategies |
另外,对于致力于通过优化拼接算法,得到更好的拼接结果的研究者,希望通过本文的分析,能帮助有志于优化混合拼接算法的团队提供参考和比较的方向,以便更好地设计出更加高效、高质量的混合拼接软件。同时,也为需要使用二代、三代测序数据进行相关生物学研究的团队提供参考,方便选出更合适的拼接软件。随着测序技术的发展,测序数据通量更高、价格更便宜、测序深度更长,这些对基因组拼接软件即是数据优势,也是计算挑战。通过优化算法、提高并行度等方式,提高计算效率、降低计算复杂度,应该是未来一段时间二三代数据混合拼接算法的发展方向。
| [1] |
SANGER F, NICKLEN S, COULSON A R. DNA sequencing with chain-terminating inhibitors[J]. Proceedings of the National Academy of Sciences, 1977, 74(12): 5463-5467. DOI:10.1073/pnas.74.12.5463 (  0) 0) |
| [2] |
EID J, FEHR A, GRAY J, et al. Real-time DNA sequencing from single polymerase molecules[J]. Science, 2009, 323(5910): 133-138. DOI:10.1126/science.1162986 (  0) 0) |
| [3] |
ROTHBERG J M, HINZ W, REARICK T, et al. An integrated semiconductor device enabling non-optical genome sequencing[J]. Nature, 2011, 475(7356): 348-352. DOI:10.1038/nature10242 (  0) 0) |
| [4] |
BRANTON D, DEAMER D W, MARZIALI A, et al. The potential and challenges of nanopore sequencing[J]. Nature Biotechnology, 2008, 26(10): 1146. DOI:10.1038/nbt.1495 (  0) 0) |
| [5] |
BLEIDORN C. Third generation sequencing: Technology and its potential impact on evolutionary biodiversity research[J]. Systematics and Biodiversity, 2016, 14(1): 1-8. DOI:10.1080/14772000.2015.1099575 (  0) 0) |
| [6] |
CHIN C S, ALEXANDER D H, MARKS P, et al. Nonhybrid, finished microbial genome assemblies from long-read SMRT sequencing data[J]. Nature Methods, 2013, 10(6): 563. DOI:10.1038/nmeth.2474 (  0) 0) |
| [7] |
HUDDLESTON J, RANADE S, MALIG M, et al. Reconstructing complex regions of genomes using long-read sequencing technology[J]. Genome research, 2014, 24(4): 688-696. DOI:10.1101/gr.168450.113 (  0) 0) |
| [8] |
CHAISSON M J P, HUDDLESTON J, DENNIS M Y, et al. Resolving the complexity of the human genome using single-molecule sequencing[J]. Nature, 2015, 517(7536): 608-611. DOI:10.1038/nature13907 (  0) 0) |
| [9] |
WEI N A, BEMMELS J B, DICK C W. The effects of read length, quality and quantity on microsatellite discovery and primer development: From I llumina to PacBio[J]. Molecular Ecology Resources, 2014, 14(5): 953-965. DOI:10.1111/1755-0998.12245 (  0) 0) |
| [10] |
MEDVEDEV P, GEORGIOU K, MYERS G, et al. Computability of models for sequence assembly[C]//International Workshop on Algorithms in Bioinformatics. Heidelberg: Springer, 2007: 289-301. DOI: 10.1007/978-3-540-74126-8_27.
(  0) 0) |
| [11] |
POP M. Genome assembly reborn: Recent computational challenges[J]. Briefings in Bioinformatics, 2009, 10(4): 354-366. DOI:10.1093/bib/bbp026 (  0) 0) |
| [12] |
PEVZNER P A, TANG H, WATERMAN M S. An Eulerian path approach to DNA fragment assembly[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(17): 9748-9753. DOI:10.1073/pnas.171285098 (  0) 0) |
| [13] |
IDURY R M, WATERMAN M S. A new algorithm for DNA sequence assembly[J]. Journal of Computational Biology, 1995, 2(2): 291-306. DOI:10.1089/cmb.1995.2.291 (  0) 0) |
| [14] |
CHAISSON M J P, WILSON R K, EICHLER E E. Genetic variation and the de novo assembly of human genomes[J]. Nature Reviews Genetics, 2015, 16(11): 627-640. DOI:10.1038/nrg3933 (  0) 0) |
| [15] |
BASHIR A, KLAMMER A, ROBINS W P, et al. A hybrid approach for the automated finishing of bacterial genomes[J]. Nature Biotechnology, 2012, 30(7): 701-707. DOI:10.1038/nbt.2288 (  0) 0) |
| [16] |
DESHPANDE V, FUNG E D K, PHAM S, et al. Cerulean: A hybrid assembly using high throughput short and long reads[C]//International Workshop on Algorithms in Bioinformatics. Heidelberg: Springer, 2013: 349-363. DOI: 10.1007/978-3-642-40453-5_27.
(  0) 0) |
| [17] |
BOETZER M, PIROVANO W. SSPACE-LongRead: Scaffolding bacterial draft genomes using long read sequence information[J]. BMC Bioinformatics, 2014, 15(1): 211. DOI:10.1186/1471-2105-15-211 (  0) 0) |
| [18] |
KOREN S, HARHAY G P, SMITH T P L, et al. Reducing assembly complexity of microbial genomes with single-molecule sequencing[J]. Genome Biology, 2013, 14(9): R101. DOI:10.1186/gb-2013-14-9-r101 (  0) 0) |
| [19] |
JVNEMANN S, SEDLAZECK F J, PRIOR K, et al. Updating benchtop sequencing performance comparison[J]. Nature Biotechnology, 2013, 31(4): 294. DOI:10.1038/nbt.2522 (  0) 0) |
| [20] |
HACKL T, HEDRICH R, SCHULTZ J, et al. Proovread: Large-scale high-accuracy PacBio correction through iterative short read consensus[J]. Bioinformatics, 2014, 30(21): 3004-3011. DOI:10.1093/bioinformatics/btu392 (  0) 0) |
| [21] |
LEE H, GURTOWSKI J, YOO S, et al. Error correction and assembly complexity of single molecule sequencing reads[J]. BioRxiv, 2014, 006395. DOI:10.1101/006395 (  0) 0) |
| [22] |
SALMELA L, RIVALS E. LoRDEC: Accurate and efficient long read error correction[J]. Bioinformatics, 2014, 30(24): 3506-3514. DOI:10.1093/bioinformatics/btu538 (  0) 0) |
| [23] |
AU K F, UNDERWOOD J G, LEE L, et al. Improving PacBio long read accuracy by short read alignment[J]. PloS One, 2012, 7(10): e46679. DOI:10.1371/journal.pone.0046679 (  0) 0) |
| [24] |
ANTIPOV D, KOROBEYNIKOV A, MCLEAN J S, et al. HybridSPAdes: An algorithm for hybrid assembly of short and long reads[J]. Bioinformatics, 2016, 32(7): 1009-1015. DOI:10.1093/bioinformatics/btv688 (  0) 0) |
| [25] |
LIN H H, LIAO Y C. Evaluation and validation of assembling corrected PacBio long reads for microbial genome completion via hybrid approaches[J]. PLoS One, 2015, 10(12): e0144305. DOI:10.1371/journal.pone.0144305 (  0) 0) |
| [26] |
SIMPSON J T, WONG K, JACKMAN S D, et al. ABySS: A parallel assembler for short read sequence data[J]. Genome Research, 2009, 19(6): 1117-1123. DOI:10.1101/gr.089532.108 (  0) 0) |
| [27] |
GUREVICH A, SAVELIEV V, VYAHHI N, et al. QUAST: Quality assessment tool for genome assemblies[J]. Bioinformatics, 2013, 29(8): 1072-1075. DOI:10.1093/bioinformatics/btt086 (  0) 0) |
| [28] |
ONO Y, ASAI K, HAMADA M. PBSIM: PacBio reads simulator-toward accurate genome assembly[J]. Bioinformatics, 2013, 29(1): 119-121. DOI:10.1093/bioinformatics/bts649 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


