全球数据信息总量将由2018年的30 ZB增长至2025年的163 ZB, 该趋势将很快超过现有硬盘等存储介质的承受能力。脱氧核糖核酸(Deoxyribonucleic Acid, DNA)数据存储技术开辟了一种新的存储模式, 其发展对于节省存储能源及推进大数据存储发展有着重要作用。利用DNA分子进行信息存取的想法早在60年代就已出现,由于DNA信息的读写较为困难,直到1988年才开始出现利用DNA保存少量信息的实验性工作,信息量极小,缺乏实际应用。随着二代测序技术的发展,出现了真正具有突破性进展的DNA存储工作。2012年,哈佛医学院的Church团队通过在DNA中存储650 KB的数据,第一次以体外存储方式实现了较大数据的DNA存储,实现了DNA存储的实际应用[1]。之后DNA数据存储逐渐成为全球研究的热点,包括哈佛大学、哥伦比亚大学、微软研究院、华盛顿大学和剑桥大学等国内外多家研究机构均展开对DNA存储的研究,并取得一定的进展,但仍有许多难题需要攻克。
DNA是一种天然的信息存储介质,DNA具有存储密度高、存储时间长、损耗率低等特点,在传统存储方式不能满足信息增长的需求时,DNA数据存储技术逐渐成为生物信息领域的研究热点。DNA存储是将数据通过DNA编码算法转换为DNA分子链中不同碱基的序列信息并存储于相应的存储载体,需要时通过特定的DNA解码算法进行读取操作,重新生成原始数据。DNA存储最明显的优势是存储量巨大,1 kg的DNA可以存储全世界所有的信息,同时具有安全性高、存储时间长、保存稳定等优点。
DNA编码是DNA存储中的关键技术, 它的目的是用尽可能少的碱基序列无错的存储数据信息。DNA编码的结果直接影响存储性能的优劣和数据读写的完整。整个DNA存储编码过程包括压缩(尽可能少的占用空间)、纠错(无错存储)和转换(数字信息转为碱基序列)3部分组成。其中转换为DNA存储编码的核心,压缩、纠错早期研究中涉及较少[2],但在近年的研究中已成为必须步骤[3-5],有效的提高了存储密度和准确性。本文主要对DNA存储中的编码技术进行综述。
1 DNA存储的发展随着信息技术的飞速发展,数据信息的含量呈指数级增长,预计到2025年,全球数据信息总量将达到163 ZB, 约相当于87.5亿张2 TB常用硬盘,这一数据增长趋势将很快超过现有硬盘等存储介质的承受能力[2]。而且现阶段使用的主要存储方式包括磁带、硬盘驱动器、蓝光存储器和闪存等,都存在有效存储时间短、数据易丢失缺损、能源消耗大、维护成本高以及污染环境等缺陷弊端[5-8]。因此寻求一种新的数据存储介质势在必行,而DNA数据存储技术开辟了一种新的存储模式, 其发展对于节省存储能源及推进大数据存储发展有着重要作用。
DNA是一种天然的信息存储介质,保证生物体内海量遗传信息安全的存储和一代代稳定的复制遗传,作为已知最密集、稳定的数据存储介质之一,DNA具有存储密度高、存储时间长、能量消耗低、并行存取性好,损耗率低和兼容性强等特点[9]。1 g的DNA可存储455 EB信息, 4 g DNA即可存储全球一年产生的信息量,而1 kg的DNA可以存储人类所有的信息[10]。DNA单位体积的存储密度是硬盘和存储器的106倍, 是闪存的103倍,DNA存储时长至少为硬盘、闪存的10倍。同时, 它还可以通过聚合酶链反应较容易地实现扩增以获取所需数量的拷贝副本。DNA作为最稳定的储存设备之一, 对于外部环境, 如高温、震荡等具有极强的抗干扰能力。即使经历数千年自然环境的考验,DNA信息依旧能够被有效地读取[8-9]。研究表明在-5 ℃的条件下,DNA每6.8×106年只降解1 bp[11]。由于DNA可隐匿在任何生物体当中,肉眼难以察觉,其又具有超高的安全性能。表 1列举了DNA和传统数据存储介质各种性能的比较。所有传统的数据存储媒体(DVD、软盘、CD、磁带等)在几年内就会开始失去完整性。相比之下,DNA作为数据存储介质的寿命要长得多,而且很容易通过聚合酶链反应技术(PCR,一种可对特定DNA片段进行放大扩增的生物技术)放大,从而获得所需的拷贝数。因此,有研究者认为一旦未来发生全球灾难,DNA将能够作为一本“启示录”记载所有人类的文明[12]。此外,DNA存储与现有的计算机存储有共同之处:①类似的编解码方式对存储信息进行写入和读取;②存储的信息是可定位、识别和还原的;③为了确保信息的正确性和存储效率,均可引入压缩码、纠错码等不同的数学算法。基于以上特点,DNA数据存储技术应运而生。
| 表 1 传统存储设备与DNA存储的性能参数 Table 1 Performance parameters of traditional storage device and DNA storage |
DNA数据存储技术作为下一代存储技术的热门,尤其是作为生物和信息等学科深度交叉发展的新技术,仍然有许多难题需要攻克。当前阶段首先要解决的就是高存储成本,存储1 MB的数据大约需要2 000-3 000美元,远远高于目前的硬盘存储成本,很难进入实际应用阶段;其次是DNA存储中的DNA序列合成和测序耗时太长,由此导致数据读取和写入需要至少以小时为单位,读写效率远低于现有硅基存储设备。除了上述两个核心难题外,仍有其他多个难题需要解决,DNA存储的错误率较高、冗余较大,主要是DNA合成、存储、测序技术的限制;现有DNA存储编解码算法来自计算机领域的简单改变和应用,与DNA存储所需的生化技术不完全适应,存在不稳定性和高错误率;DNA数据实现随机读取较为困难,为了读取某一部分数据,需要将整个DNA库中的序列测序解吗;将大数据转换为DNA碱基序列需要消耗大量计算资源等。
DNA存储技术优势明显,需要解决的难题也较多,但DNA数据存储技术是生物、信息等多学科交叉发展的成果,各个研究团队乃至科技巨头纷纷进入这一领域[13],最近5年DNA存储发展逐渐被众多的研究者和企业关注。2012年,哈佛医学院的Church团队通过在DNA中存储650 KB的数据,第一次以体外存储方式实现了较大数据的DNA存储,成为DNA信息存储领域的一个里程碑[2],2017年该团队又将更大的视频文件存入大肠杆菌的DNA中,完成了体内存储的DNA信息存储[14];2013年欧洲生物信息研究所的Nick Goldman及其团队在DNA中采用三进制编码方式实现了20 MB数据可行、高容量的存储,并申请了相关专利,这使DNA数据存储又迈出了一大步,逐步开始向应用阶段迈进[15-16];2016年,微软研究院和华盛顿大学联合将200 MB数据存入DNA[17],同时微软计划于2020年在数据中心建立基于DNA的数据存储系统;2017年纽约哥伦比亚大学将喷泉码引入DNA存储,这种方法可将2.15亿千兆的数据存入到仅1 g的DNA中[3],此后多项DNA存储研究均在此基础上展开[18-19];同年Shipman等人成功利用CRISPR Cas系统在DNA中存储信息,并将像素值编码到一个活细菌种群的基因组中[14],2019年,以色列理工学院的研究团队在喷泉码的基础上利用复合的DNA碱基“字母”进行编码,从而减少合成循环数,降低合成成本,使得DNA存储技术的发展有了新突破[18]。同年,Erlich等通过喷泉码编码后,3D打印出一只存有遗传信息的斯坦福兔子,并实现了DNA蓝图的稳定复制和遗传[19]。DNA信息存储领域目前已得到了各行各业的广泛关注。
2 DNA存储框架DNA是通过A(腺嘌呤)、T(胸腺嘧啶)、C(胞嘧啶)、G(鸟嘌呤)4种脱氧核糖核苷酸连接形成的长链分子。A与T,C与G之间两两配对能够形成稳定的双链结构,无论是单链DNA还是双链DNA均可用于以二进制代码的形式存储信息。图 1为DNA单链和双链的结构模型,其中单链一般用于体外合成,而双链用于体内合成。
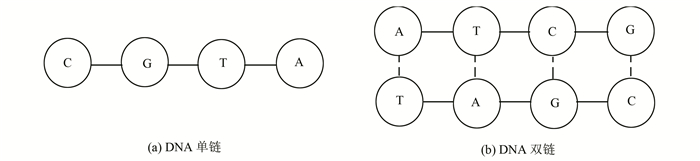
|
图 1 DNA模型 Figure 1 DNA model |
DNA作为存储设备对信息进行保存及读取的整体流程如图 2所示,主要框架包括3部分:编码写入, 数据存放及解码读取部分。首先通过计算机算法将二进制数据映射成碱基序列,然后合成特定序列的DNA完成编码的写入;随后以溶液或干粉的形式对DNA进行保存,外部封装的形式多种多样,常见的为瓶装,也可以像斯坦福兔子[19],3D打印为任意形状;最后利用PCR扩增来实现数据拷贝,并通过测序仪器测得目标DNA的所有碱基序列,进而再通过解码转换成二进制数据完成数据的读取。

|
图 2 DNA存储流程图 Figure 2 flow chart of DNA storage |
受限于现有的DNA合成技术,编码写入的碱基序列会分割为长度相同的短序列,一般单条序列长度不超过200 bp。每一条需要合成的序列里包括引物、数据、地址位、纠错码等,其中地址位用于各条序列的快速定位、拼接和查找。引物是专门设计,合成前添加到序列两端,用于提取所需的DNA序列。纠错码包括序列内纠错码和序列间纠错码,如图 3所示,序列内纠错码用于纠正单条序列内的错误,序列间纠错码用于纠正整条序列缺失等错误。

|
图 3 DNA存储序列示意图 Figure 3 Schematic diagram of DNA sequences |
DNA编码是通过一定的算法和映射关系, 将需要存储的信息以码流的形式转变成DNA碱基序列的排列组合, 从而实现文件信息与DNA之间的关系转换。不同的DNA模型适用于不同类型信息的存储, 虽然模型之间存在差别, 但DNA信息编码写入的流程大致都是一致的, 主要包括数据压缩-引入纠错-转换为碱基序列的过程, 整体的流程如图 4所示。

|
图 4 DNA编码流程 Figure 4 Encoding process in DNA storage |
DNA解码是由存储的DNA序列中获取数字信息的过程,是编码的逆过程。整个DNA解码的读取过程如图 5所示。解码前通过PCR扩增得到多个DNA拷贝,从而不会对原始存储造成影响。再对拷贝进行DNA测序,获取DNA序列的碱基排列方式。获取碱基序列后对序列纠错、去冗余、解码,读取原始数据。
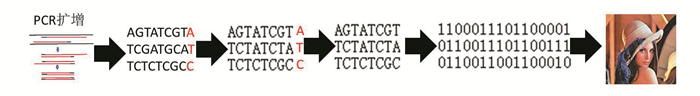
|
图 5 DNA解码流程 Figure 5 Decoding process in DNA storage |
现有的数据均可以二进制形式存储在计算机硬盘等硅存储介质内,因此将信息存储至DNA中实质上就是将二进制数据编码为碱基序列存入DNA。碱基序列是由A, T, C和G 4种碱基组成, 根据DNA的组成及结构, 基本的DNA存储编码模型有3种:二进制模型[2]、三进制模型[16]和四进制模型[3]。在此基础上,还有混合模型(如二、四进制组合在一起等)[20]、含简并碱基的模型[18]等。
3.1.1 二进制模型二进制模型是DNA存储中最简单的模型,根据二进制0、1和四种碱基A、T、C、G之间的可能映射关系,将任意两种碱基定义为0,另两个则为1,共有6种可能的组合形式。早期的DNA存储研究采用这种转换模型进行数据编码。Church[2]在2012年按A或G等于0, C或T等于1,使用二进制模型将0.65 MB的数据编码成长度为单条长度159 nt的8.8 MB的DNA序列。鉴于大量的数字数据成功地存储在DNA中,这被认为是一项里程碑式的研究,同时也证明了基于DNA的数据存储在应对信息爆炸挑战方面的潜力。
这种二进制模型相对简单, 具有较高的碱基变换灵活性,能够较好地控制GC含量、均聚物数量等条件, 降低DNA合成难度,减少合成和测序错误。但就编码效率而言, 该编码方案通过将每个二进制码转换成1碱基,牺牲了信息密度,相同长度的碱基序列二进制模型能存储的信息量较少, 编码效率不高。在后续的研究中,已经很少采用二进制模型,研究者通过开发和引入新的编码方式,在保证可靠性的前提下,进一步提升存储密度。
3.1.2 三进制模型三进制编码模型是将数据转换为三进制,以0、1、2的形式表示,接着按对应关系转换为相应碱基,如图 6所示。这种对应关系下,下一位碱基的确定依赖于前一位碱基,而碱基与数据之间没有明确的对应映射关系。三进制模型由Goldman[16]团队在2013年提出,并在国内外均申请了相关专利[15, 21]。

|
图 6 三进制转换模型 Figure 6 Ternary transformation model |
三进制模型相较于二进制模型,提高了存储密度,也能够控制GC含量、均聚物数量等条件,降低后期合成难度。但三进制模型也没有充分利用DNA的存储能力,除了Goldman团队外,其他研究较少采用三进制模型。
3.1.3 四进制模型将碱基A, T, C, G看作0, 1, 2, 3,则DNA序列可视为天然的四进制编码模型。对于任意二进制数据, 将按两位二进制数一组就可以编码为碱基序列。例如将二进制数据00, 01, 10, 11编码为A, T, C, G,即可一一对应进行数据编码。这种映射关系并不唯一, 共有24种组合方案, 理论上这24种方案彼此是等价的,但考虑到实际编码时GC含量等条件,存在部分更优化组合方案。
四进制模型相对于其他两种模型存储能力最强,理论存储极限为2 bit/nt,达到了碱基序列存储效率的极限。在目前以提高存储密度为导向的研究中,四进制模型是应用最广泛的DNA存储转换模型[3, 9, 18, 22]。但需要指出的是,这种模型易出现GC含量过高、均聚物较多等影响后续的DNA合成和测序的情况。为了克服这些情况,研究者引入纠错码等冗余数据进行数据质量控制,实际存储效率都低于理论值。
3.1.4 混合模型二进制模型能够较好地控制GC含量、均聚物数量等条件, 降低DNA合成难度,减少合成和测序错误;而四进制模型理论存储极限为2 bit/nt,达到了碱基序列存储效率的极限。综合两者的优点,有研究者[20]提出了混合模型,在四进制模型的基础上加入二进制模型,在保证存储效率的同时,控制合成条件,降低合成难度。如图 7所示,前三组二进制数采用四进制模型,最后一组二进制数采用二进制模型,8 bit的数据存入5个碱基之中。类似的研究还有将5 bit的数据存入3个碱基之中[23]。
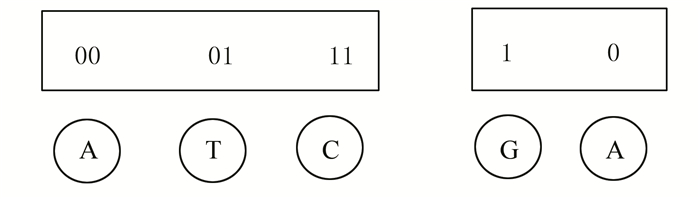
|
图 7 混合模型示意图 Figure 7 Schematic diagram of mixture model |
混合模型基本都是以四进制模型为主,在保证存储效率的前提下,引入二进制模型降低合成难度,也可视为四进制模型的一个变种。在单一的四进制模型合成困难的缺点没有完全克服前,混合模型可以降低对纠错码等冗余数据的需求量,从降低冗余的角度提高存储密度。这种模型可以根据实际数据的情况灵活组合,在存储密度和合成难度之间取得较好的平衡,是一种较为常用的模型。
3.1.5 含简并碱基的模型最新研究[18]首次在编码阶段引入简并碱基,这种复合DNA字母表是由四种DNA核苷酸按预定比例混合而成的序列中位置的表示(见图 8)。利用现有的DNA存储技术中涉及多个相同分子的并行合成和排序所导致的信息冗余,用更少的合成周期来编码数据,每单位数据使用的合成周期减少了20%。模拟编码表明,合成周期可能减少达75%。
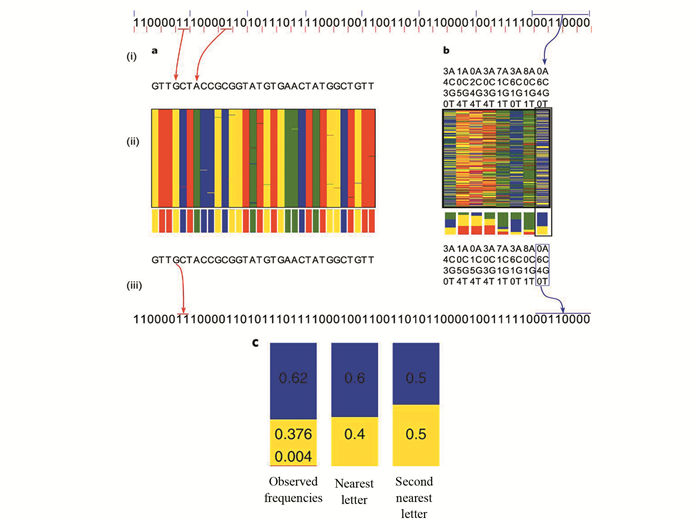
|
图 8 含简并碱基模型示意图(来自Anavy等的工作[18]) Figure 8 Schematic diagram of model with degenerate bases (from work of aNavy et al[18]) |
压缩的目的在于尽可能地减少数据冗余,用尽可能少的空间存储尽可能多的数据,最大化的利用DNA存储序列。DNA存储利用目前已有的信息领域的编码方法进行数据压缩,主要有霍夫曼编码[15]和喷泉吗[3],此外还有LZMA[24]等编码方法。其中,霍夫曼编码是DNA存储领域最常见的编码方法,而喷泉码则是可能的未来主流编码方法。
3.2.1 霍夫曼编码霍夫曼编码是一种由David Huffman在20世纪50年代开发的,基于最小冗余编码的无损数据压缩算法,广泛应用于数据文件压缩。2013年,Goldman[16]首次在DNA存储中采用了霍夫曼编码,有效地将编码潜力提高到1.58 bit/nt。二进制数据首先由值霍夫曼编码压缩,然后通过三进制模型转换为DNA序列,将每8 bit的数据存储进5到6个碱基之中。通过霍夫曼编码和三进制模型,可以压缩原始数据25%~37.5%,并避免了均聚物的产生。霍夫曼编码适用于多类数据,并能取得较好的压缩效果。
然而,在处理某些二进制数据时,霍夫曼编码可以控制,但不能完全避免均聚物的产生,也不能防止异常的GC分布。此外,霍夫曼编码对部分数据的压缩效果不佳。
3.2.2 喷泉码喷泉码是通信系统中广泛使用的一种信息编码方法,以其鲁棒性和高效率而著称。喷泉码又称无速率擦除码,其存储的数据分为k个段,即资源包,可以从这些资源包派生出无限数量的编码包。当它返回n (n > k)个编码包时,原始资源数据将完全恢复。在实际应用中,只要n比k稍大一点,就可以获得更好的编码效率和信息通信的鲁棒性。
在2017年,Erilich和Zielinski[3]在首次在DNA存储中使用了喷泉码,采用四进制转换模型,00, 01, 10, 11分别映射到A, C, G, T。将原始的二进制信息分割成若干小块,这些块是根据预先设计的伪随机序列选择的。然后,通过按位添加所选的带有随机种子的块,并根据四进制模型映射关系创建新的数据块(见图 9)。最后进行筛选防止单核苷酸重复和GC含量异常。该编码方案中的引物是相关的,具有网格状的拓扑结构,实现极低但必要的冗余。该研究将编码潜力的理论极限提高到前所未有的高值1.98 bit/nt,并显著降低了源文件无错误恢复所需的冗余。此外,随机选择和有效性验证机制确保了长单核苷酸均聚物不会出现在编码序列中。
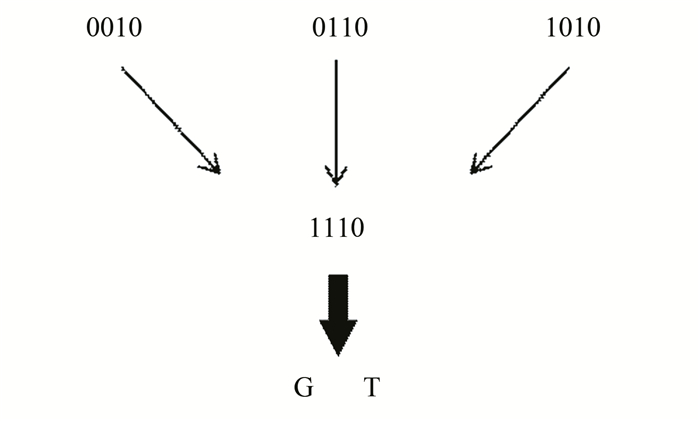
|
图 9 喷泉码编码示意图 Figure 9 Schematic diagram of fountain code |
然而,在这种编码方案中,编码和解码的复杂度与数据大小并不是线性相关的。因此,解码可能很复杂,并且可能需要更多的资源和更长的计算时间。有研究[25]认为,尽管Erilich的文章表示丢失总包数4%不会影响原始文件的恢复,但就DNA喷泉码的特征而言,丢失更多的包数可能会导致恢复完全失败。如果最终目标是永久存储数据,则必须增加冗余的数量以确保信息的完整性。
在基于DNA的数据存储和检索中,最常见的错误是由碱基突变引起的。为了解决这个问题,大多数编码方案都创建了高冗余度来进行错误纠正。然而,这些纠错算法需要复杂的译码过程和大量的计算资源。在这里,喷泉编码方案的使用表明,它不必要使用错误检测/纠正算法,可以有效提高DNA编码的性能。
3.2.3 其他算法DNA存储编码中,也有研究采用其他压缩编码,但是相对较少,效果也一般。例如,Yim研究团队[24]于2012年对一张BMP图片的二进制码流利用LZMA算法压缩后存储于DNA中,但该方法并不适用于高通量数据, 且压缩过程的耗时较长。也有研究采用TAR和LZMA算法联合进行数据压缩和DNA存储[23]。
采用其他压缩算法的DNA存储研究较少,彼此之间相对孤立,也缺乏连贯性和验证。这些方法可能存在进一步研究的空间,但目前尚无报道。
3.3 纠错在DNA存储信息的过程中, 无论是DNA编码、合成、存储,还是DNA测序、解码, 均有可能出现错误, 导致最终出现信息的损失。为了尽量保证信息的无错读取, 在DNA存储过程中引入相应的纠错机制来提高存储的准确性。
纠错机制多种多样,在合成、存储和测序阶段都有相应的措施,例如可以通过提高测序深度来减少错误率。但上述措施都意味着DNA存储成本的上升,而在编码阶段引入纠错码则是在控制成本的前提下保证准确率的最有效方式。值得注意的是,纠错码本身属于冗余,纠错是通过引入冗余的方式提高准确率,在冗余和准确率之间取得平衡,是非常关键的一点。目前的DNA存储中使用的纠错方式以RS码为主[3, 18, 26],少量文献采用LDPC码[24]、汉明码[27]、前向纠错[22]、多倍冗余[16]、XOR计算[9]等纠错方式。
3.3.1 RS码RS码是一种典型的线性循环码, 即源文件编码后向左或向右移动后仍为有限组码组中的一组, 它可对随机错误、突发错误及二者的组合进行纠错。Grass[26]在2015年年将基于有限域的RS代码引入DNA存储领域,特别强调错误检测和校正,将潜在的数据密度提高到1.78 bits/nt。该编码方案以2字节(8×2位)的基本信息块为基础,引入一个有限域作为其元素。为了防止编码过程中产生长度大于3 nt的均聚物,三联体的最后2个核苷酸是不同的,可以产生48个不同的三联体。因为47是比48小的最大质数,所以用了GF(47)。然后将信息块映射到GF(47)中的3部分元素,即,2562至473。该方案采用RS码来检测和纠正错误。对GF转码生成的矩阵分别进行水平方向和垂直方向的2轮RS编码。在这项初步研究中,83 KB的文本数据被编码。虽然数据量不是很大,但是引入了一种有效的纠错机制,大大提高了编码和合成的效率。
RS码能用较小的冗余恢复更多的数据信息, 此后的研究中大部分均采用RS码作为纠错机制。但由于涉及有限域,其计算量较大,对大数据编码的计算机硬件要求较高。
3.3.2 其他纠错机制Goldman[16]在2013年的研究中采用四倍重叠冗余进行纠错,保证存储的准确性,在DNA存储领域引入纠错的概念。但这种纠错机制带来了巨大的冗余,存储密度只有0.33 bit/nt。2016年,Bornholt等人[9]利用异或(XOR)编码原理改进了Goldman的编码方案,每2个原始序列,A和B,将由A⊕B产生一个冗余序列C。因此,任意2个序列(AB、AC或BC),都可以很容易地恢复到第三个序列。这种编码方案还根据特定数据链的重要程度提供了冗余的灵活性,即“可调冗余”。它将原始数据的冗余度从三倍降低到一半,存储效率上升到0.88 bit/nt。
此外,Blawat[22]采用“前向纠错”机制,预先指定两个参考编码表,将一个1字节(8位)的基本信息块分配给一个5碱基DNA序列,并交换第三个和第四个碱基,并满足条件:前3个碱基不相同,且最后两个碱基不相同。22 Mb的数据被成功地编码并存储,且这些数据被无错误地检索,存储密度达到0.92 bit/nt。然而,这种方法不能检测和纠正单个突变的情况。
在DNA信息存储中也有研究团队将LDPC码[24]、汉明码[27]用于纠错环节, 以防止在DNA合成及测序中出现随机错误, 提高文件读取的准确性。然而,虽然简单的LDPC码可以检测错误,但它不能纠正错误。此外,冗余度的增加不可避免地降低了编码效率。
4 DNA存储编码技术的发展方向目前为止,DNA存储编码已经形成压缩+纠错+转换的较为稳定的模式,其中四进制转换模型成为主流,在此基础上,改进的混合模型[20]、含简并碱基模型[18]等进一步得到发展。而喷泉码有取代霍夫曼编码成为DNA存储领域最常见编码方法的趋势。DNA存储中使用的纠错方式仍然以RS码为主[3, 18, 26],没有新的突破,其他纠错方式研究者较少,如表 2所示。
| 表 2 DNA存储主要方案的参数 Table 2 Parameters of main DNA storage schemes |
DNA存储编码技术未来发展方向是比较明确的,首先是编码算法的进一步深化,其次是将编码技术扩展到DNA存储的合成、测序环节,在实现DNA存储编码的基础上,进一步对整个存储流程进行编码算法优化,提高存储效率和准确率,降低成本和合成周期。
4.1 编码算法 4.1.1 压缩编码目前的主要研究方向仍然是将现有信息领域的算法与DNA存储相结合,寻找更适合DNA的编码方法, 尽可能充分地利用DNA存储空间, 引入较少的冗余。现有的压缩方法不对数据类型进行区分,压缩质量参差不齐,DNA存储的成本仍居高不下,尤其是存储数据规模逐渐扩大,需要针对不同类型的数据选择不同的压缩方法以尽可能的提高存储效率,降低成本。
4.1.2 纠错编码RS码是现阶段效果最好的纠错编码,被大部分研究所采用,但计算量较大。RS码的高效性在小规模数据存储中被证明,未来大数据存储需要有效降低RS码的计算量,同时寻找更合适、冗余更小的纠错编码。
4.1.3 转换模型现有理论存储效率最高的四进制模型为2 bit/nt,但因为地址码、纠错码等冗余的引入,存储效率无法达到理论值。未来需要考虑在四进制模型的框架内合理设置冗余,做到冗余与纠错之间的平衡。此外,简并碱基在编码中的引入,虽然不是真正意义上的突破了2 bit/nt的限制,但在目前合成阶段存在大量相同序列的前提下,引入的简并碱基越多,存储效率越高,现阶段达到了2.5 bit/nt,理论可达到10 bit/nt[18]。但这也对编码和合成技术提出了较高的要求。
4.2 合成与测序编码优化算法既往算法研究主要在满足低均聚物和适当的GC含量的基础上展开,没有更进一步考虑更多的DNA特性和生化技术特点,缺乏对DNA存储中合成与测序错误的优化算法,仅靠编码阶段的纠错机制被动减少错误率。究其原因,DNA存储尚在初级研究阶段,现阶段研究者主要关注高密度数据存储的实现,对合成与测序中的错误通过增加冗余的简单处理方式进行被动控制和纠正,效果不稳定,成本和周期也大幅上升。
DNA存储载体一般为溶液或干粉,进行信息提取时则必须为溶液形式,受合成技术与成本的影响,不同DNA存储样品的单位密度存在差异。密度高,包含的DNA序列多,信息存储完整,但重复冗余较大,合成周期长,成本高昂;密度低,DNA序列少,冗余小,成本和合成周期低,但信息可能丢失。既往研究发现,完全相同的数据和算法的重复性实验中,由于合成技术的可能差异,会导致密度发生变化,进而影响编解码参数设置,并导致信息冗余或者丢失。
目前的DNA存储算法无法对这些基于DNA特性的问题进行调控优化,而只能通过合成前添加冗余纠错的方式进行被动校正,纠错码属于冗余,纠错是通过引入冗余的方式提高准确率,在冗余和准确率之间取得平衡,是非常关键的一点,但现有的纠错机制无法做到这一点,再加上各种合成与测序技术的不同,相关研究重复性较差,使得错误率的控制机制完全属于经验判断,不稳定性很高。
综上所述,现有DNA存储算法研究主要集中在输入文件转换为DNA序列的编码部分,对于合成与测序阶段的错误缺乏客观的识别、控制、优化和评价的模型算法,单靠前期的纠错码进行错误校正,只能被动的等待结果输出,对合成与测序过程无法进行基于生物学特性的优化评价,且目前的DNA合成和测序技术主要为生物学服务,对于数字信息编码而成的DNA序列的效果并不稳定,仍有待探索。通过算法优化模型,提高合成和测序的成功率是必须解决的问题。
4.3 DNA存储的计算机适配系统现阶段DNA存储算法主要来自计算机等领域的简单改编,研究重点均集中在编码技术上,较少涉及之后的合成、存储、测序和解码等步骤,且相关研究是零散的,不成体系的,基本只包括了将输入文件转换为DNA合成序列这部分,甚至只关注其中的压缩、转换或纠错等某一步骤。究其原因,DNA存储尚在初级研究阶段,现阶段研究者主要关注高密度数据存储,而对解码技术的要求较低,只需保证数据可以完整读取即可。
由于缺乏完整的DNA存储计算机适配系统,不同的研究采用的编解码算法、合成、测序技术和存储条件各不相同,相应的软件适配系统差异很大。DNA存储的计算机适配系统应考虑存储效率、鲁棒性、准确率和成本等多方面因素,目前算法较多的只考虑存储效率和准确率,且不同的合成与测序技术对DNA存储效果影响很大,缺乏一个全面的、具有一致性的软件适配系统,导致研究的可重复性不高,难以重复实现。
针对上述问题,在目前编码研究基础上,需要首先确定DNA存储的完整流程,并实现模块化,并延伸至DNA存储流程的每一步,针对DNA存储编码、合成、测序、解码等主要阶段分别进行算法设计和优化,建立完整的DNA存储算法适配系统,有效提高存储效率和准确率,降低合成周期和成本,增强研究的可重复性和实际应用性。
5 总结DNA存储编码算法经过近十年的发展,压缩以霍夫曼编码和喷泉码为主,纠错主要为RS码,转换大多使用四进制模型,整体编码模式已经形成,未来将逐步向大规模编码存储和商业化应用发展。但现有的DNA存储的编解码算法主要来自计算机等领域的简单改编,缺乏对DNA分子特性的研究和匹配,适应性和可靠性不高,基于现有算法编码得到的DNA合成序列,直接用于DNA存储合成较不稳定,错误率较高。未来需要考虑更多的DNA特性和生化技术特点,通过建立参数优化模型对DNA合成序列实现优化,在目前编码研究基础上,针对DNA存储编码、合成、测序、解码、评价等主要阶段分别进行算法设计和优化,建立完整的DNA存储算法适配系统,为大规模的DNA存储研究奠定基础。
| [1] |
CHURCH G M, GAO Yuan, KOSURI S. Next-Generation Digital Information Storage in DNA[J]. Science, 337(6102): 1628-1628. DOI:10.1126/science.1226355 (  0) 0) |
| [2] |
ZHIRNOV V, ZADEGAN R M, SANDHU G S, et al. Nucleic acid memory[J]. Nature Materials, 2016, 15(4): 366-370. DOI:10.1038/nmat4594 (  0) 0) |
| [3] |
ERLICH Y, ZIELINSKI D. DNA fountain enables a robust and efficient storage architecture[J]. Science, 3355(6328): 950-954. DOI:10.1126/science.aaj2038 (  0) 0) |
| [4] |
FATIMA A, IKRAM U H, AIMAN T L, et al. Trends to store digital data in DNA:An overview[J]. Molecular Biology Reports, 2018, 45(10): 1479-1490. DOI:10.1007/s11033-018-4280-y (  0) 0) |
| [5] |
PANDA D, MDLLA K A, BAIG M J, et al. DNA as a digital information storage device:hope or hype?[J]. Biotech, 2018, 8(5): 239. DOI:10.1007/s13205-018-1246-7 (  0) 0) |
| [6] |
GODA K, KITSUREGAWA M. The history of storage systems[J]. Proceedings of the IEEE, 2012, 100(Special Centennial Issue): 1433-1440. DOI:10.1109/JPROC.2012.2189787 (  0) 0) |
| [7] |
WILLIAMS E D, AYRES R U, HELLER M. The 1.7 kilogram microchip:Energy and material use in the production of semiconductor devices[J]. Environmental Science & Technology, 2002, 36(24): 5504-5510. DOI:10.1021/es025643o (  0) 0) |
| [8] |
EXTANCE A. How DNA could store all the world's data[J]. Nature, 2016, 537(7618): 22-24. DOI:10.1038/537022a (  0) 0) |
| [9] |
BORNHOLT J, LOPEZ R, CARMENN D M, et al. A DNA-based archival storage system[J]. IEEE Micro, 2016, 51(4): 637-649. DOI:10.1145/2954679.2872397 (  0) 0) |
| [10] |
JACQVES B, COLOTTE M, COUDY D, et al. Chain and conformation stability of solid-state DNA:implications for room temperature storage[J]. Nucleic Acids Research, 2009, 38(5): 1531-1546. DOI:10.1093/nar/gkp1060 (  0) 0) |
| [11] |
ALLENTOFT M E, COLLINS M J, HARKER D, et al. The half-life of DNA in bone:Measuring decay kinetics in 158 dated fossils[J]. Proceedings of the Royal Society B:Biological Sciences, 2012, 279(1748): 4724-4733. DOI:10.1098/rspb.2012.1745 (  0) 0) |
| [12] |
YONG E D. Synthetic double-helix faithfully stores Shakespeare's sonnets[J]. Nature News, 2013. DOI:10.1038/nature.2013.12279 (  0) 0) |
| [13] |
HAKAMI H A, CHACZKO Z, KALE A.Review of big data storage based on DNA computing[C]//Proceedings of 2015 Asia-Pacific Conference on Computer Aided System Engineering, Piscataway, IEEE, 2015.DOI: 10.1109/APCASE.2015.27.
(  0) 0) |
| [14] |
SHIPMAN S L, NIVALA J, MACKLIS J D, et al. CRISPR-Cas encoding of a digital movie into the genomes of a population of living bacteria[J]. Nature, 2017, 547(7663): 345. DOI:10.3389/fbioe.2017.00057 (  0) 0) |
| [15] |
GOLDMAN N, BIRNEY J.High-capacity storage of digital information in DNA: JP2019023890[P].2019-02-14.
(  0) 0) |
| [16] |
GOLDMAN N, BERTONE P, CHEN Siyuan, et al. Towards practical, high-capacity, low-maintenance information storage in synthesized DNA[J]. Nature, 2013, 494(7435): 77-80. DOI:10.1038/nature11875 (  0) 0) |
| [17] |
BORNHOLT J, LOPEZ R, CARMEAN D M, et al. Toward a DNA-based archival storage system[J]. IEEE Micro, 2017, 37(3): 98-104. DOI:10.1145/2954679.2872397 (  0) 0) |
| [18] |
ANAVY L A, INBAL A, ORNA A, et al. Data storage in DNA with fewer synthesis cycles using composite DNA letters[J]. Nature biotechnology, 2019, 37(10): 1229-1236. DOI:10.1038/s41587-019-0240-x (  0) 0) |
| [19] |
KOCH J, GANTENBEIN S, MASANIA K, et al. A DNA-of-things storage architecture to create materials with embedded memory[J]. Nature Biotechnology, 2019, 12(9): 1-5. DOI:10.1038/s41587-019-0356-z (  0) 0) |
| [20] |
ORGANICK L, ANG S D, CHEN Y J, et al. Random access in large-scale DNA data storage[J]. Nature Biotechnology, 2018, 36(3): 242-248. DOI:10.1038/nbt.4079 (  0) 0) |
| [21] |
尼克·高曼, 约翰·伯尼.DNA中数字信息的高容量存储: CN201611110091.X[P].2017-08-18. GOLDMAN N, Birney J.High-capacity storage of digital information in DNA: CN201611110091.X[p].2017-08-18. (  0) 0) |
| [22] |
BLAWAT M, CHVRCH G M. Forward error correction for DNA data storage[J]. Procedia Computer Science, 2016, 80: 1011-1022. DOI:10.1016/j.procs.2016.05.398 (  0) 0) |
| [23] |
樊隆, 蒋浩君, 刘家栋, 等.一种DNA数据存储编码解码方法: CN201710611123.2[P].2019-02-01. FAN Long, JIANG Haojun, LIU Jiadong, et al.A method of encoding and decoding in DNA data storage: CN201710611123.2[P].2019-02-01. (  0) 0) |
| [24] |
YIM A K, YU A C, LI J W, et al. The essential component in DNA-based information storage system:Robust error-tolerating module[J]. Frontiers in Bioengineering & Biotechnology, 2014, 2: 49. DOI:10.3389/fbioe.2014.00049 (  0) 0) |
| [25] |
PING Zhi, MA Dongzhao, HUANG Xiaoluo, et al. Carbon-based archiving:current progress and future prospects of DNA-based data storage[J]. GigaScience, 2019, 8(6). DOI:10.1093/gigascience/giz075 (  0) 0) |
| [26] |
GRASS R N, HECKEL R, PUDDU M, et al. Robust chemical preservation of digital information on DNA in silica with error-correcting codes[J]. Angewandte Chemie International Edition, 2015, 54(8): 2552-2555. DOI:10.1002/anie.201411378 (  0) 0) |
| [27] |
宋香明.基于Huffman编码的DNA信息存储方法研究[D].天津: 天津大学, 2018. SONG Xiangming.Research on DNA Information Storage Method Based on huffman Coding[D].Tianjin: Tianjin University, 2018. (  0) 0) |
 2020, Vol. 18
2020, Vol. 18


