多型性胶质母细胞瘤(GBM)又称为胶质母细胞瘤,是最常见也最具侵袭性的原发恶性脑肿瘤。经过标准化治疗后的胶质母细胞瘤患者,中位总生存期为14.6个月,诊断为胶质母细胞瘤就预示了患者的不良预后[1]。由于其低存活率、短生存期、高复发的特点,让胶质母细胞瘤已经成为令人闻风丧胆的可怕疾病[2]。传统的手术联合术后大剂量放化疗的治疗方案无法有效提高胶质母细胞瘤患者的生存时间,保障患者预后[3]。因此,针对胶质母细胞瘤药物治疗靶点的研究成为全世界研究者们臻待解决的问题。
1986年,Renato Dulbecco首次提出全基因组测序的概念,为全世界癌症研究点亮了一束光[4]。通过生物信息手段,分析不同个体基因组间的结构差异,同时完成SNP及基因组结构注释,可以由宏观到微观来认识恶性肿瘤的发生机制[5]。Cameron W. Brennan等提出在一个统计支持的队列中,系统的基因组分析可以定义核心生物学通路的概念,以此来促进在分子层面上对胶质母细胞瘤的了解[6]。全面阐明胶质母细胞瘤中的基因变化可以提供新的靶点,达到诊断、预后预测和治疗的目的,基因组学亚型的确定,更可以将胶质母细胞瘤类型细化,进行分类诊疗。从最开始的基因组学亚分型研究,到药物靶点的确定,再到目前药物通过血脑屏障、药物载体的研发,针对胶质母细胞瘤的研究一直没有间断过[7]。但是,即便如此,影响胶质母细胞瘤疾病进展的因素和作用机制,仍然是困扰研究者们的难题。
在转录水平分析胶质母细胞瘤的基因表达水平,试图寻找一个新的标记物,有利于从分子层面了解胶质母细胞瘤。数据均来自TCGA(the Cancer Genome Atlas,https://cancergenome.nih.gov/)数据库。在TCGA数据库中搜索胶质母细胞瘤mRNA样本,经过仔细筛查,从中选取符合研究标准的样本集,其中共有胶质母细胞瘤169例和正常脑组织5例,随之进行差异基因的筛选。将筛选出来的差异基因利用GO(Gene Ontology)和KEGG(Kyoto Encyclopedia of Gene and Genomes pathway enrichment analysis)富集分析进行功能注解。然后,通过构建差异基因的蛋白互作网络筛选出连通性最高的核心基因。最后,利用Kaplan-Meier法对各个核心基因进行生存分析,从而获得与胶质母细胞瘤生存时间相关的核心基因KCNAB2。
1 材料与方法 1.1 筛选差异基因胶质母细胞瘤样本均来自TCGA,下载肿瘤样本和正常对照样本的mRNA表达数据。将基因的表达量转换为数值,对重复的基因取平均值并过滤表达量低的基因,然后构建设计矩阵和对比矩阵,计算标准化因子,估计散度,随后对两组数据间进行双马尾检验,矫正p值,导出基因。根据所获得的各个基因的p值及logFC值筛选差异基因。以上计算均依靠R软件(R 3.5.2)进行。
1.2 GO和KEGG富集分析GO富集分析是一种常用的基因功能分析方法,通过将数量庞大的基因按照生物学进程、分子功能、细胞组分进行聚类富集调查的方法,对基因功能进行注解。KEGG富集分析是一种现在广泛使用的系统分析基因产物在细胞中代谢途径及基因产物功能的分析方法。KEGG可以将基因按照所在的生物学通路进行富集调查,从而对目标基因做进一步注解[8]。利用DAVID (https://david.ncifcrf.gov/tools.jsp)工具[9]对差异基因进行注解和探索。
1.3 构建蛋白互作网络和筛选核心基因蛋白互作网络(Protein-protein Interaction,PPI)是由单个蛋白间的相互作用关系连接而成,蛋白互作网络的构建可以帮助我们了解疾病状态下生物信号、能量代谢以及蛋白之间的功能联系[13]。利用STRING(The search tool for the retrieval of interacting genes,https://string-db.org/)平台分析差异基因的蛋白互作信息。将蛋白互作网络导入cytoscape软件,制作蛋白互作网络鸟瞰图。利用cytohubba计算各差异基因之间连通性得分,得分高的选定为核心基因。鉴于BottleNeck在大数量基因数据计算时具有更好的表现, 并且,BottleNeck算法得到的核心基因(Hub-bottlenecks)更倾向于神经信号转导通路,BottleNeck得分被选用来选取核心基因[10]。
1.4 ONCOMINEONCOMINE(https://www.oncomine.org/)被用来对所获得的核心基因进行进一步差异分析。ONCOMINE癌基因芯片数据挖掘平台是目前世界上最大的癌基因芯片数据库和整合数据挖掘平台,可用于比较主要癌症类型和各自正常组织的差异表达分析,进行一个或多个基因的查询[11]。借助已有的研究,综合回顾、分析核心基因在各个探针中的表达情况。
1.5 生存分析为探索核心基因与胶质母细胞瘤患者生存时间的联系,评估核心基因对胶热质母细胞瘤患者生存时间的影响,利用患者的生存数据进行生存分析。将核心基因表达量的改变与胶质母细胞瘤患者的生存时间和生存几率的关系绘制为生存曲线,计算p值,p值<0.05为具有统计学的生存曲线。
2 结果分析 2.1 差异基因从TCGA中下载获取胶质母细胞瘤转录组数据169例,正常对照脑组织标本5例,基因17 848个。共获得差异基因3 183个,上调基因1 582个,下调基因1 601个,所获结果可视化为热图(见图 1)。

|
图 1 差异基因分布热图 Figure 1 DEGs distribution heatmap 注:X轴代表TCGA样本号,Y轴代表基因名。黑色代表非差异基因,红色代表上调差异基因,黄色代表下调差异基因。由于基因数量巨大,只在Y轴显示部分不同表达量的基因。 |
为适应DAVID对基因数量的限制,将差异基因的截取指标进一步调整,将p < 0.05并|logFC|≥2.0并FDR < 0.01的基因截取为差异基因,共获得2 932个基因,并将其导入DAVID。GO富集分析的结果表明,这些基因主要富集于细胞组分上,包括膜的组成部分、质膜、质膜组分等;分子功能主要富集于钙离子结合;生物学过程主要富集于化学性突触传递和细胞黏附,主要结果列为表 1。KEGG富集分析结果表明,差异基因主要富集于hsa04080信号通路,hsa04080通路的功能为神经活性配体-受体相互作用,主要结果列为表 2。
| 表 1 GO富集分析结果 Table 1 Results of GO enrichment analysis |
| 表 2 KEGG富集分析结果 Table 2 Results of KEGG enrichment analysis |
利用STRING得到的蛋白互作网络,共有1 951个节点,6 967条边(见图 2)。将结果导入cytoscape。利用cytohubba对差异基因连通性进行计算,将BottleNeck得分前十的差异基因选定为核心基因,得分第一为HIST1H2BH(Histone Cluster 1 H2B Family Member H),BottleNeck得分64分,得分第二为AR(Androgen Receptor),BottleNeck得分54分。KCNAB2(Potassium Voltage-Gated Channel Subfamily A Regulatory Beta Subunit 2)排在第九位,BottleNeck得分26分。在后续的生存分析中,发现只有AR与KCNAB2与胶质母细胞瘤患者生存时间存在统计学意义的相关性,因此将研究重点放在尚未被验证过的KCNAB2基因上。
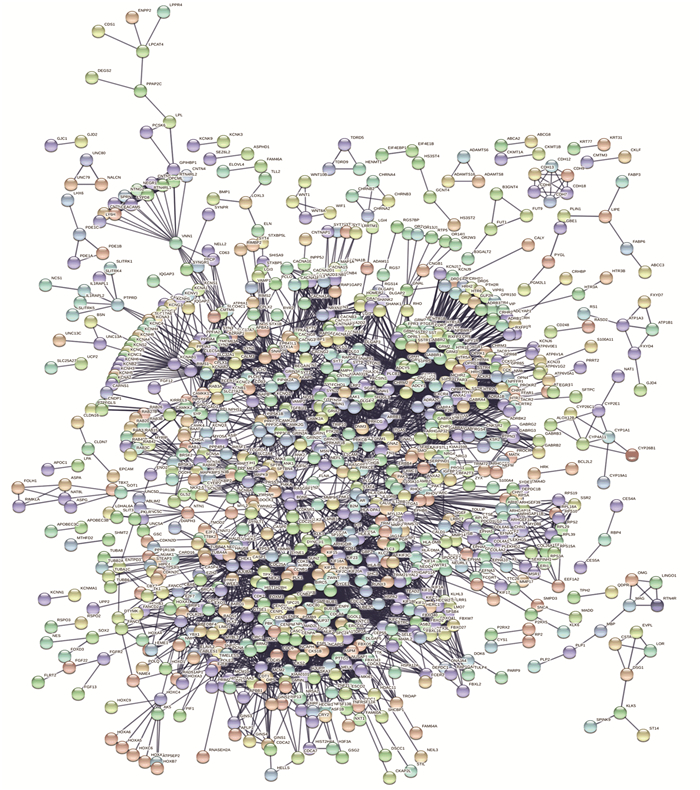
|
图 2 差异基因蛋白互作网络鸟瞰图 Figure 2 Bird's eye view of protein-protein interaction network 注:图中共有1 951个节点,6 967条边。 |
将KCNAB2基因导入ONCOMINE,各研究中KCNAB2表达量箱线图输出结果见图 3,结果提示KCNAB2在各个研究的不同探针结果里均存在不同程度的低表达。综合三个已有的研究进行meta分析。meta分析的结果提示,在Bredel Brain2的研究中KCNAB2的低表达程度最显著,综合分析的p值为2.67X10-10。
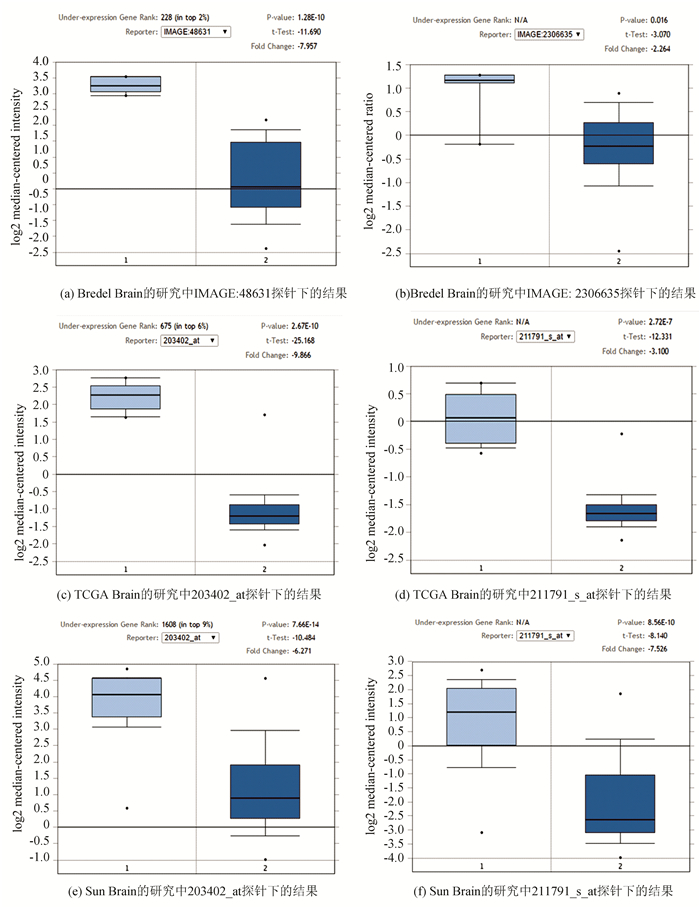
|
图 3 ONCOMINE联合分析结果 Figure 3 Result of ONCOMINE analysis 注:KCNAB2在不同探针下的表达量,x轴的1代表KCNAB2在正常脑组织中的表达量,2代表KCNAB2在GBM中的表达量。(a)中p值1.28X10-10,FC值-7.957;(b)中p值0.016,FC值-2.264;(c)中p值2.67X10-10,FC值-9.866;(d)中p值2.72X10-7,FC值-3.100;(e)中p值7.6 X10-14,FC值-6.271;(f)中p值8.56X10-10,FC值-7.526。 |
利用Rstudio将168例胶质母细胞瘤样本的基因表达信息与生存信息进行匹配,绘制核心基因生存曲线,其中只有KCNAB2和AR具有统计学意义,结果可视化输出见图 4。

|
图 4 AR和KCNAB2的存活曲线 Figure 4 Survival curves of AR and KCNAB2 注:x轴为存活时间(d),y轴为存活率,红色曲线代表基因的高表达,绿色曲线代表基因的低表达。 |
与胶质母细胞瘤生存相关的核心基因主要为AR和KCNAB2。AR基因最早在前列腺癌的研究中被发现,在Zalcman, N等的文献里已经证实其可作为胶质母细胞瘤的一个重要化学药物治疗靶点,AR的表达沉默可以导致胶质母细胞瘤的细胞活力降低;在细胞系中,AR的沉默会导致胶质母细胞瘤细胞在体内和体外的凋亡[12-13]。KCNAB2胶质母细胞瘤的表达差异关系及对患者生存时间的影响均为首次提出,通过分析KCNAB2的生存曲线可以发现,KCNAB2过表达时胶质母细胞瘤患者表现出更低的生存率。这提示我们,在胶质母细胞瘤中存在着一条KCNAB2相关的作用机制,可以影响胶质母细胞瘤患者的生存率。
3 讨论脑胶质瘤来源于神经上皮细胞,占颅脑肿瘤的40%~50%,是最常见的颅内原发肿瘤。多型性胶质母细胞瘤又称为胶质母细胞瘤(GBM),由星形胶质瘤恶变而来, 占全部颅脑肿瘤的10.2%[14]。是成人最常见也最具侵袭性的恶性颅脑肿瘤,grade Ⅳ级[15]。胶质母细胞瘤患者中位生存期仅为12~16个月,5年生存率不足5%[16],诊断为胶质母细胞瘤就预示了患者的不良预后[17]。目前,针对胶质母细胞瘤的方案,主要是手术联合术后大剂量放疗和化疗,尽管近年来,手术、放疗和化疗的综合治疗手段得到了长足的发展,但是胶质母细胞瘤的预后仍取决于首次手术切除程度。胶质母细胞瘤具有浸润性生长的特性,这使得手术完全切除肿瘤组织成为几乎不可能的事情[18]。随着分子生物学的飞速发展,针对胶质母细胞瘤的基因治疗被全世界的研究者寄予厚望,基因治疗有望在胶质母细胞瘤患者中取得良好的治疗效果,甚至最终治愈这一可怕疾病。
筛选得到的十个核心基因分别是:HIST1H2BH、AR、SNAP25、VAMP2、GNAI1、TP53、CDK1、DYNC1I1, KCNAB2 and SLC04C1。相较于正常脑组织,SNAP25、VAMP2、GNAI1、DYNC1I1、KCNAB2,SLC04C1在胶质母细胞瘤中低表达,而HIST1H2BH、AR、TP53、CDK1在胶质母细胞瘤中高表达。
离子通道基因KCNAB2,别名AKR6A5、HKvbeta2、HKvbeta2.1、HKvbeta2.2、KCNA2B,KV-BETA-2。编码电压门控K+通道β亚单位蛋白Kvb2,位于1号染色体1p36区域。1p36缺失综合征的患者,在受精卵形成时,由于该染色体片段的缺失,导致部分患者KCNAB2基因的缺失,这一部分缺失KCNAB2基因的患者出现了联想学习和联想记忆功能为主要表现的认知功能障碍,通过研究发现,KCNAB2基因的缺失可以导致杏仁核外侧核投射神经元的神经生理学改变,包括动作电位的慢后超极化(SAHP)的降低和神经元兴奋性的增加,从而导致患者联想记忆和联想学习能力受[19-20]。另外,KCNAB2基因缺失的患者中绝大多数出现了严重癫痫表型,由于离子通道的异常可以增加癫痫的易感性,可以认为KCNAB2基因作为癫痫表型基因家族的一个候选基因。但是仍有很大一部分1p36缺失综合征的患者出现了癫痫表型,却未发现存在KCNAB2基因的缺失,提示:KCNAB2基因的缺失是患者发生严重癫痫的一个重要危险因素[21]。除了KCNAB2基因,在胶质母细胞瘤患者中一定还存在一个潜在的机制,或其他责任基因的共同作用,从而导致患者严重癫痫表型的出现。在胶质母细胞瘤的患者中,KCNAB2基因的低表达是否也会导致患者出现严重癫痫和认知功能障碍有待我们进一步的实验去寻求实践支持。
4 结论1) KCNAB2基因在胶质母细胞瘤患者中存在显著的低表达;
2) KCNAB2基因的高表达与胶质母细胞瘤患者生存时间的缩短存在相关性,可能作为胶质母细胞瘤药物治疗的有效靶标;
3) Kvb2为钾离子通道蛋白,KCNAB2基因的低表达可能是导致胶质母细胞瘤细胞膜电位改变的因素,甚至是导致胶质母细胞瘤癫痫表型的因素之一。
| [1] |
INDER Z A, THORNE A H, BAKAS S, et al. Epidermal growth factor receptor extracellular domain mutations in glioblastoma present opportunities for clinical imaging and therapeutic development[J]. Cancer Cell, 2018, 34(1): 163-177, e7. DOI:10.1016/j.ccell.2018.06.006 (  0) 0) |
| [2] |
BREHMER S, GRIMM M A, FORSTER A, et al. Study protocol: Early stereotactic gamma knife radiosurgery to residual tumor after surgery of newly diagnosed glioblastoma (gamma-GBM)[J]. Neurosurgery, 2019, 84(5): 1133-1137. DOI:10.1093/neuros/nyy156 (  0) 0) |
| [3] |
BRADLEY C A. Glioblastoma: Stem cells-masters of their fates[J]. Nature Reviews Cancer, 2017, 17(10): 574-575. DOI:10.1038/nrc.2017.88 (  0) 0) |
| [4] |
DULBECCO R. A turning point in cancer research:Sequencing the human genome[J]. Science, 1986, 231(4742): 1055-1056. DOI:10.1126/science.3945817 (  0) 0) |
| [5] |
EYRE S, BOWES J, DIOGO D, et al. High-density genetic mapping identifies new susceptibility loci for rheumatoid arthritis[J]. Nature Genetics, 2012, 44(12): 1336-1340. DOI:10.1038/ng.2462 (  0) 0) |
| [6] |
BRENNAN C W, VERHAAK R G W, MCKENNA A, et al. The somatic genomic landscape of glioblastoma[J]. Cell, 2013, 155(2): 462-477. DOI:10.1016/j.cell.2013.09.034 (  0) 0) |
| [7] |
TANG W, FAN W, LAU J, et al. Emerging blood-brain-barrier-crossing nanotechnology for brain cancer theranostics[J]. Chemical Society Reviews, 2019, 48(11): 2967-3014. DOI:10.1039/c8cs00805a (  0) 0) |
| [8] |
ASHBURNER M, BALL C A, BLAKE J A, et al. Gene ontology: Tool for the unification of biology[J]. The Gene Ontology Consortium. Nature Genetics, 2000, 25(1): 25-29. DOI:10.1038/75556 (  0) 0) |
| [9] |
GAO X, CHEN Y, CHEN M, et al. Identification of key candidate genes and biological pathways in bladder cancer[J]. PeerJ, 2018, 6: e6036. DOI:10.7717/peerj.6036 (  0) 0) |
| [10] |
HAN J D, BERTIN N, HAO T, et al. Evidence for dynamically organized modularity in the yeast protein-protein interaction network[J]. Nature, 2004, 430(6995): 88-93. DOI:10.1038/nature02555 (  0) 0) |
| [11] |
YANG X M, CAO X Y, HE P, et al. Over expression of Rac GTPase activating protein 1 contributes to proliferation of cancer cells by reducing hippo signaling to promote cytokinesis[J]. Gastroenterology, 2018, 155(4): 1233-1249. DOI:10.1053/j.gastro.2018.07.010 (  0) 0) |
| [12] |
ZALCMAN N, CANELLO T, OVADIA H, et al. Androgen receptor: A potential therapeutic target for glioblastoma[J]. Oncotarget, 2018, 9(28): 19980-19993. DOI:10.18632/oncotarget.25007 (  0) 0) |
| [13] |
CRAWFORD E D, SCHELLHAMMER P F, MCLEOD D G, et al. Androgen receptor targeted treatments of prostate cancer: 35 years of progress with antiandrogens[J]. The Journal of Urology, 2018, 200(5): 956-966. DOI:10.1016/j.juro.2018.04.083 (  0) 0) |
| [14] |
SIDDIQUI J, KRISHNAN A S. Butterfly glioma[J]. The New England Journal of Medicine, 2018, 378(3): 281. DOI:10.1056/NEJMicm1704713 (  0) 0) |
| [15] |
FURNARI F B, FENTON T, BACHOO R M, et al. Malignant astrocytic glioma: Genetics, biology, and paths to treatment[J]. Genes Development, 2007, 21(21): 2683-710. DOI:10.1101/gad.1596707 (  0) 0) |
| [16] |
SMOLL N R, SCHALLER K, GAUTSCHI O P. Long-term survival of patients with glioblastoma multiforme (GBM)[J]. Journal of Clinical Neuroscience, 2013, 20(5): 670-675. DOI:10.1016/j.jocn.2012.05.040 (  0) 0) |
| [17] |
WICK W, GORLIA T, BENDSZUS M, et al. Lomustine and bevacizumab in progressive glioblastoma[J]. The New England Journal of Medicine, 2017, 377(20): 1954-1963. DOI:10.1056/NEJMoa1707358 (  0) 0) |
| [18] |
DING Z, ROOS A, KLOSS J, et al. A novel signaling complex between TROY and EGFR mediates glioblastoma cell invasion[J]. Molecular Cancer Research, 2018, 16(2): 322-332. DOI:10.1158/1541-7786.MCR-17-0454 (  0) 0) |
| [19] |
GAJECKA M, MACKAY K L, SHAFFER L G. Monosomy 1p36 deletion syndrome[J]. American Journal of Medical Genetics Part C, Seminars Medical Genetics, 2007, 145C(4): 346-56. DOI:10.1002/ajmg.c.30154 (  0) 0) |
| [20] |
PERKOWSKI J J, MURPHY G G. Deletion of the mouse homolog of KCNAB2, a gene linked to monosomy 1p36, results in associative memory impairments and amygdala hyperexcitability[J]. Journal of Neuroscience, 2011, 31(1): 46-54. DOI:10.1523/JNEUROSCI.2634-10.2011 (  0) 0) |
| [21] |
LI M, HE Q, MA J, et al. PPICurator: A tool for extracting comprehensive protein-protein interaction information[J]. Proteomics, 2019, 19(4): e1800291. DOI:10.1002/pmic.201800291 (  0) 0) |
 2020, Vol. 18
2020, Vol. 18


