2. 河南大学 基础医学院病理学教研室,河南 开封 475004
2. Department of Pathology, Basic Medical College, Henan University, Kaifeng 475004, Henan, China
乳腺癌是全球范围内最常见的肿瘤之一[1],在女性肿瘤患者中,乳腺癌致死率排名第二[2]。如今,乳腺癌的治疗包括局部手术、全身化疗、精确放疗、内分泌治疗、生物靶向制剂治疗等手段,而生物标志物可用于建立病人预后和预测的结果。因此,寻找更有效、灵敏的乳腺癌预后生物标志物是乳腺癌研究的迫切需要[3]。过去的几十年,新型治疗靶点的发现和新型靶向药物的开发已大大改变了抗乳腺癌战场的局面[4-5]。
Src homology 2 domain containing(Shc)家族包含ShcA、ShcB,ShcC和ShcD 4个亚基因家族[6],SHC SH2-domain binding protein 1(SHCBP1)在酵母双杂交实验中首次被鉴定出来,目的是寻找适配蛋白ShcA新的结合伴侣[7]。因为SHCBP1在T细胞活化时表达上调,所以它最初被命名为鼠活化淋巴细胞蛋白(mPAL)[7]。
SHCBP1可以通过其他途径来影响多种恶性肿瘤如肝癌、神经胶质瘤、滑膜肉瘤等的生长[8-9]。有研究表明,SHCBP1在乳腺癌细胞中过度表达[10]。但目前对于SHCBP1的表达与预后作用方面的关系研究匮乏。利用多种常用公共数据库中乳腺癌临床数据,评估分析了SHCBP1在不同亚型乳腺癌中的表达水平和潜在的临床价值,以期为SHCBP1靶向治疗的未来发展和乳腺癌的预后和预测提供理论指导。
1 材料和方法 1.1 Oncomine分析Oncomine数据库是一个可公开访问的在线癌症表达谱数据库(www.oncomine.org)。在数据库中,通过查询SHCBP1,通过选择乳腺癌与正常组织进行比较。在本次研究中,P值由studentt检验产生,用于乳腺癌和正常组织中SHCBP1 mRNA水平的比较。有效倍数变化阈值定义为2,P值设置为0.01[11]。
1.2 bc-GenExMiner v4.2分析bc-GenExMinerv4.2(bcgenex.centregauducheau.fr/BC-GEM/)在2019年1月进行数据更新,是一种数据挖掘工具,包含36个已发表的注释基因组数据[12-13]。根据临床标准(如激素受体、淋巴结状态等),用三个模块来呈现统计的数据,使用表达模块分析SHCBP1基因的临床参数,如Scarff Bloom & Richardson分级(SBR)、诺丁汉预后指数(Nottingham Prognositc Index, NPI)、雌激素受体(ER)、孕酮受体(PR)、表皮生长因子受体-2 (HER-2)、基底细胞样(Basal-like);使用预后模块评估SHCBP1表达模块与转移复发事件的相关性;使用相关模块评估SHCBP1与CDC45的相关性。
1.3 Kaplan-Meier plotter分析Kaplan-Meier plotter(www.kmplot.com)是一个微阵列数据集的在线数据库,数据来源于GEO, EGA和TCGA三个数据库。使用Kaplan-Meier plotter评价无转移生存期(R)和无复发生存率(RFS),显著性利用log-rank P值表示[14]。
1.4 STRING分析STRING(http://string-db.org/)是一个可用于确定蛋白质与蛋白质的相互作用的在线数据库[15]。本文通过该数据库对SHCBP1进行蛋白质网络分析,以发现可能与SHCBP1存在相互作用的蛋白质。
2 结果分析 2.1 与正常乳腺组织相比,SHCBP1在乳腺癌中显著高表达大量研究表明,SHCBP1在多种癌症中高表达,如白血病、肝细胞癌等[8]。而Oncomine分析结果也证实了该表型(见图 1a)。与正常组织相比,SHCBP1在乳腺癌组织中高表达。在乳腺癌中,有6组数据显示乳腺癌组织高表达SHCBP1,而仅有1组数据显示正常组织内SHCBP1高表达。癌症基因组图谱(The Cancer Genome Atlas, TCGA)数据和Richardson研究表明,几种类型的乳腺癌中SHCBP1转录水平均高于正常组织(见图 1b~1g)。
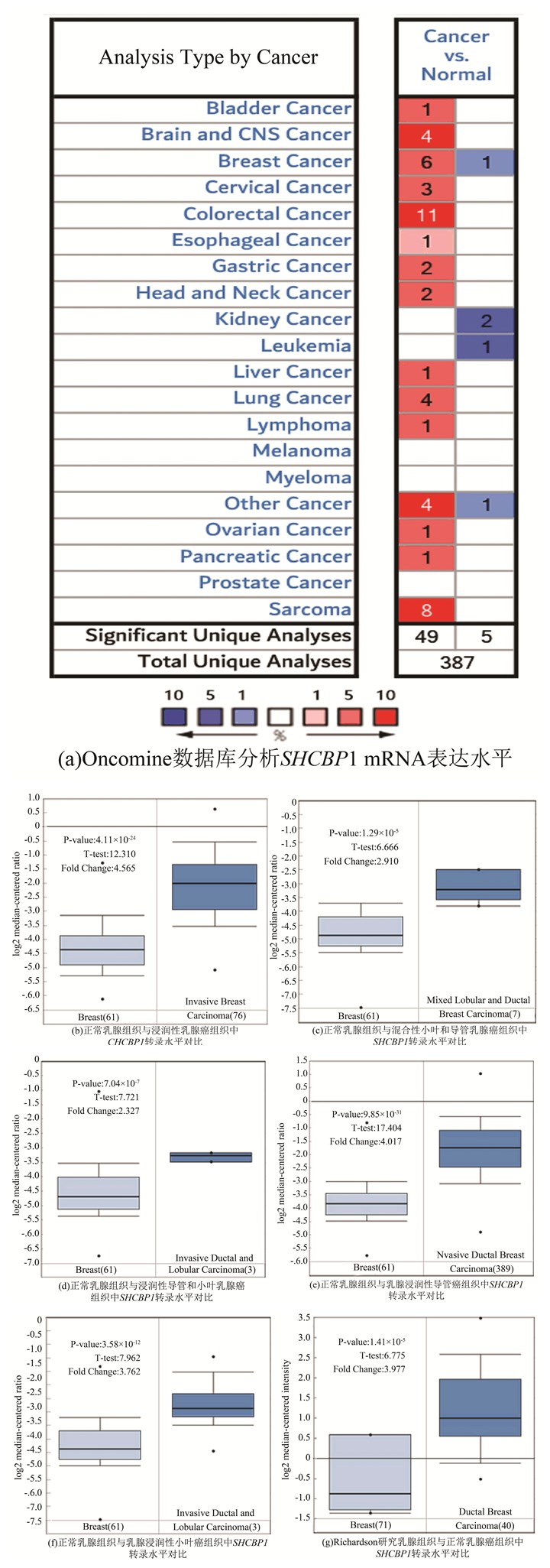
|
图 1 SHCBP1在乳腺癌组织中的表达明显高于正常组织 Figure 1 The expression of SHCBP1 in breast cancer tissue is significantly higher than that in normal tissue 注:(a)图显示了具有统计学意义的mRNA过度表达(红色)或靶基因的下调表达(蓝色)的数据集的数目。P值阈值为0.01。每个方框中的数字代表在这些分析和癌症类型中达到阈值的分析数量。在每一项研究中,以所有基因中所测得的目标基因的百分比来分析基因等级。分析结果颜色由方框内分析的最佳基因等级百分位数决定。(b-g)TCGA数据库(b-f),(g)中SHCBP1 mRNA表达的比较。利用Oncomine数据库比较SHCBP1在正常组织和乳腺癌组织中的表达得出箱式图。P值设为0.01,有效倍数变化阈值定义为2。 |
如表 1所示,使用bc-GenExminer v4.2进行Welch试验,根据不同的临床病理特征,比较各组患者SHCBP1的转录水平。结果表明,≤51岁组与>51岁组间无显著性差异(P=0.975)。淋巴结阳性(Nodal status+)的乳腺癌患者中SHCBP1 mRNA水平与淋巴结阴性者(Nodal status-)相比无显著差异(P=0.053)。ER(+)(P=0.000)、PR(+)(P=0.000)与SHCBP1 mRNA表达呈负相关(见表 1)。相反,与HER2(-)患者相比,SHCBP1 mRNA水平在HER2(+)患者中表达较高(P=0.000)(见表 1)。组间比较还表明,在三阴性乳腺癌(TNBC)中,SHCBP1 mRNA表达显著高于非TNBC(P=0.000),而在Basal-like亚型中SHCBP1 mRNA表达显著高于其他亚型(P=0.000)(见表 1)。在分子亚型分析中,SHCBP1在HER-2亚型和Basal-like亚型中的表达明显高于乳腺癌的Luminal A亚型和luminal B亚型(Dunnett-Tukey-Kramer’s test,P=0.000)(见图 2a)。而在诺丁汉预后指数(NPI)分析中,NPI水平越高,SHCBP1的mRNA表达越高(P=0.000)(见图 2b)。在Scarff Bloom & Richardson分级标准(SBR)中,SBR分级越高,SHCBP1 mRNA水平越高(P=0.000)(见图 2c)。
| 表 1 SHCBP1 mRNA表达与乳腺癌临床特征参数的关系 Table 1 Relationship between SHCBP1 mRNA expression and clinical characteristic parameters of breast cancer |

|
图 2 SHCBP1在不同分子亚型乳腺癌患者中的表达 Figure 2 SHCBP1 expression in breastcancer patients with different molecular subtypes |
SHCBP1的表达增高可以预测乳腺癌患者的无转移生存期(DMFS)较短,尤其是Luminal A亚型的乳腺癌患者中评估了SHCBP1对乳腺癌远处转移的预后价值。Kaplan-Meier plotter分析表明,在乳腺癌患者中,SHCBP1 mRNA的高表达与DMFS的缩短有关(HR=1.85, P=0.000)(见图 3a),尤其是在Luminal A亚型乳腺癌患者中(HR=1.96, P=0.000)(见图 3b)时。然而,在Luminal B亚型乳腺癌患者(HR=1.06, P=0.75) (见图 3c),HER-2(+)亚型乳腺癌患者(HR=1.28, P=0.44) (见图 3d)或Basal-like亚型乳腺癌患者(HR=0.9, P=0.69) (见图 3e)中,SHCBP1 mRNA的高表达或低表达与DMFS的缩短无显著差异。
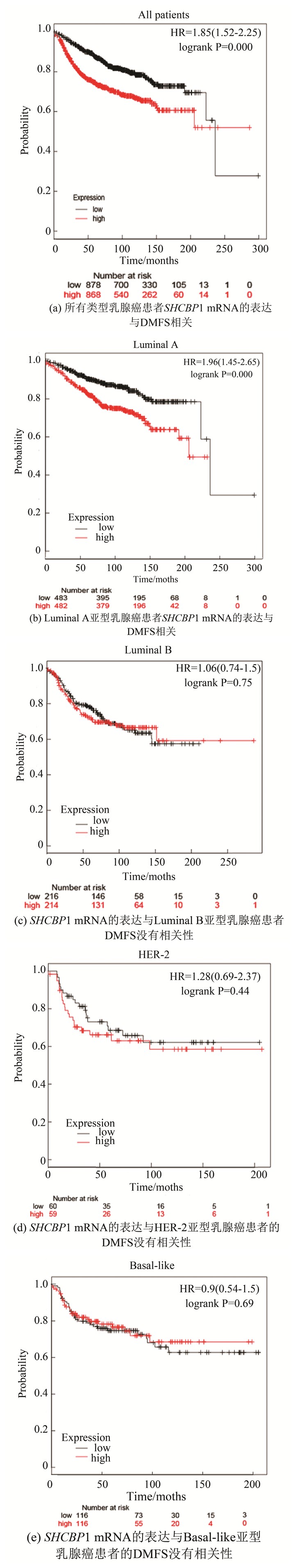
|
图 3 高表达SHCBP1的乳腺癌患者具有较短的DMFS,尤其是Luminal A亚型乳腺癌患者 Figure 3 Breast cancer patients with high SHCBP1 expression have shorter DMFS, especially those with Luminal A subtype breast cancer |
Kaplan-Meier plotter分析表明:所有类型乳腺癌患者SHCBP1 mRNA的表达与DMFS相关; Luminal A亚型乳腺癌患者SHCBP1 mRNA表达与DMFS相关; SHCBP1 mRNA表达与Luminal B亚型乳腺癌患者的DMFS没有相关性; SHCBP1 mRNA表达与HER-2亚型乳腺癌患者的DMFS没有相关性; SHCBP1 mRNA表达与Basal-like亚型乳腺癌患者的DMFS没有相关性。
2.4 SHCBP1表达高的Luminal A亚型乳腺癌患者具有较短的无病生存期(DFS)为进一步探究SHCBP1在不同亚型乳腺癌中的预后情况,对GSE41994数据集[16]中的Luminal A亚型病人临床数据展开分析,发现SHCBP1高表达的乳腺癌患者具有较差的DFS(HR=2.93, P=0.002)(见图 4a),而在Luminal B亚型中,SHCBP1的表达与DFS的长短无关(HR=1.32, P=0.786)(见图 4b)。由于HER-2(+)亚型,Basal-like亚型乳腺癌数据库样本数量不足,因此无法分析SHCBP1的表达对这两类亚型的乳腺癌DFS的影响。

|
图 4 高表达SHCBP1的Luminal A亚型乳腺癌患者具有较短的无病生存期(DFS) Figure 4 Patients with Luminal A subtype of breast cancer with high expression of SHCBP1 have a short DFS |
SHCBP1的表达升高可表现出更短的无复发生存率(RFS), 尤其是在ER(+)、PR(+)、仅接受辅助化疗, 和仅接受辅助化疗治疗的ER(+)与接受包括或排除内分泌治疗乳腺癌患者中,且SHCBP1的高表达与较高的转移复发风险相关。所有的乳腺癌患者的SHCBP1 mRNA高表达与RFS缩短显著相关(HR=1.76, P=0.000)(见图 5a),特别是在ER(+)亚型(HR=1.52, P=0.000)(见图 5b)和PR(+)亚型(HR=1.74, P=0.002)(见图 5c)的乳腺癌患者中。值得注意的是,对接受不同治疗的乳腺癌患者,SHCBP1 mRNA的高表达与仅接受辅助化疗的患者的RFS缩短显著相关(HR=2.15, P=0.000)(见图 5d)。在ER(+)亚型中具有显著相关性(HR=2.13, P=0.003)(见图 5e),而在ER(-)亚型中无显著相关性(HR=1.3, P=0.23)(数据未展示)。这些结果表明SHCBP1对乳腺癌耐药性起关键的作用,可能与ER的表达有关。在接受内分泌治疗(HR=1.4, P=0.007)(见图 5f)或无内分泌治疗(HR=1.74, P=0.000)(见图 5g)的乳腺癌患者中,SHCBP1 mRNA的高表达与RFS的缩短显著相关。在仅接受他莫昔芬治疗的乳腺癌患者中,这种相关性并不显著(HR=1.23, P=0.16)(数据未显示)。
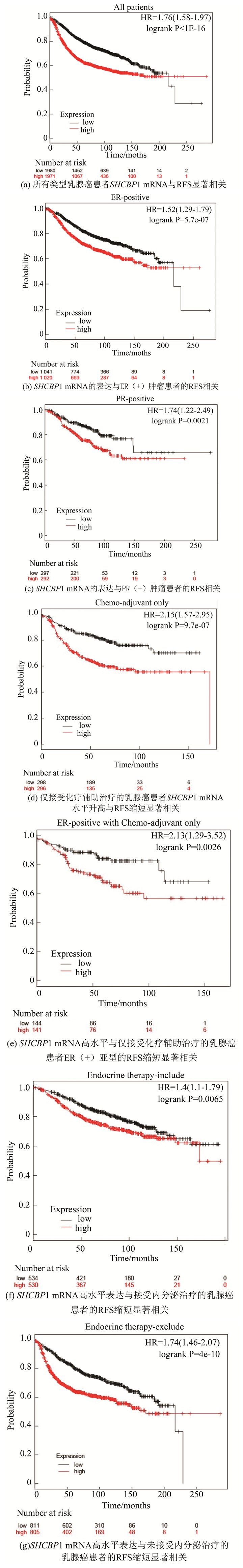
|
图 5 Kaplan-Meier plotter分析表明SHCBP1高表达的乳腺癌患者生存率低 Figure 5 Low survival rate of breast cancer patients with high SHCBP1 expression indicated by Kaplan-Meier plotter |
Oncomine分析SHCBP1的共表达基因,发现CDC45与SHCBP1显著正相关(见图 6a)。研究发现,人类肿瘤来源细胞的CDC45蛋白水平始终较高,CDC45的表达与增殖细胞群密切相关[17]。利用bc-GenExMiner v4.2数据挖掘,证实了SHCBP1和CDC45 mRNA之间的确实存在正相关性(见图 6b)。
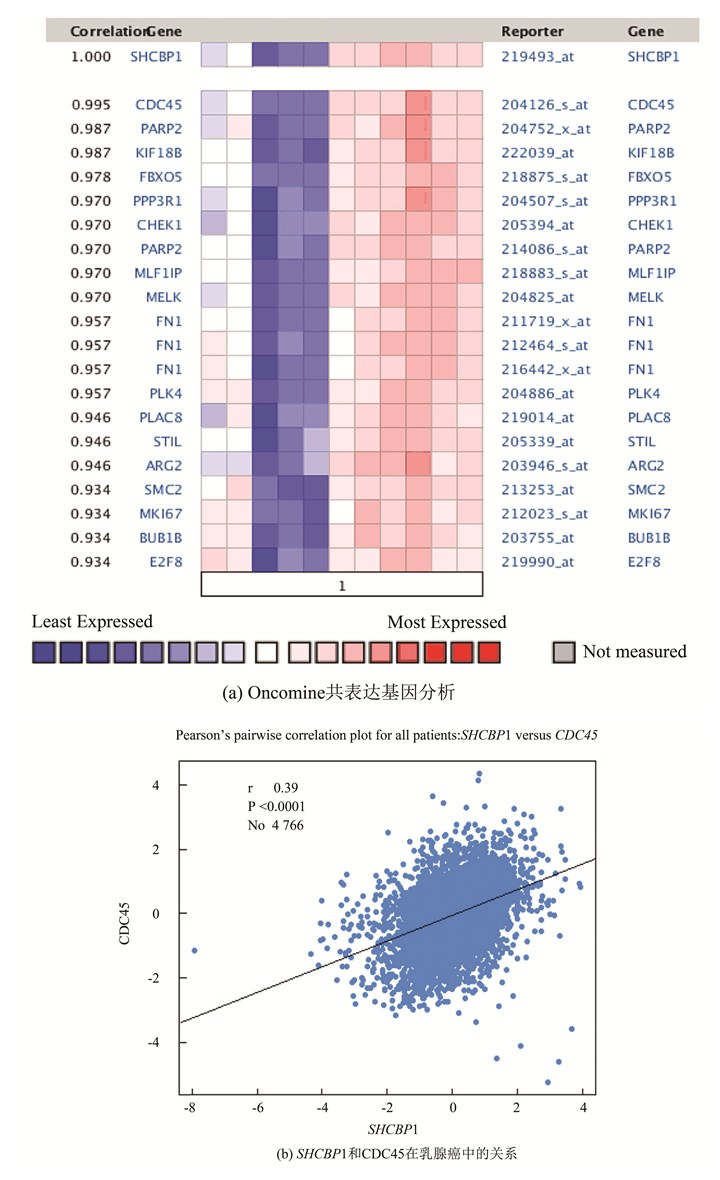
|
图 6 SHCBP1和CDC45的表达呈正相关 Figure 6 Positive correlated of the expression of SHCBP1 and CDC45 |
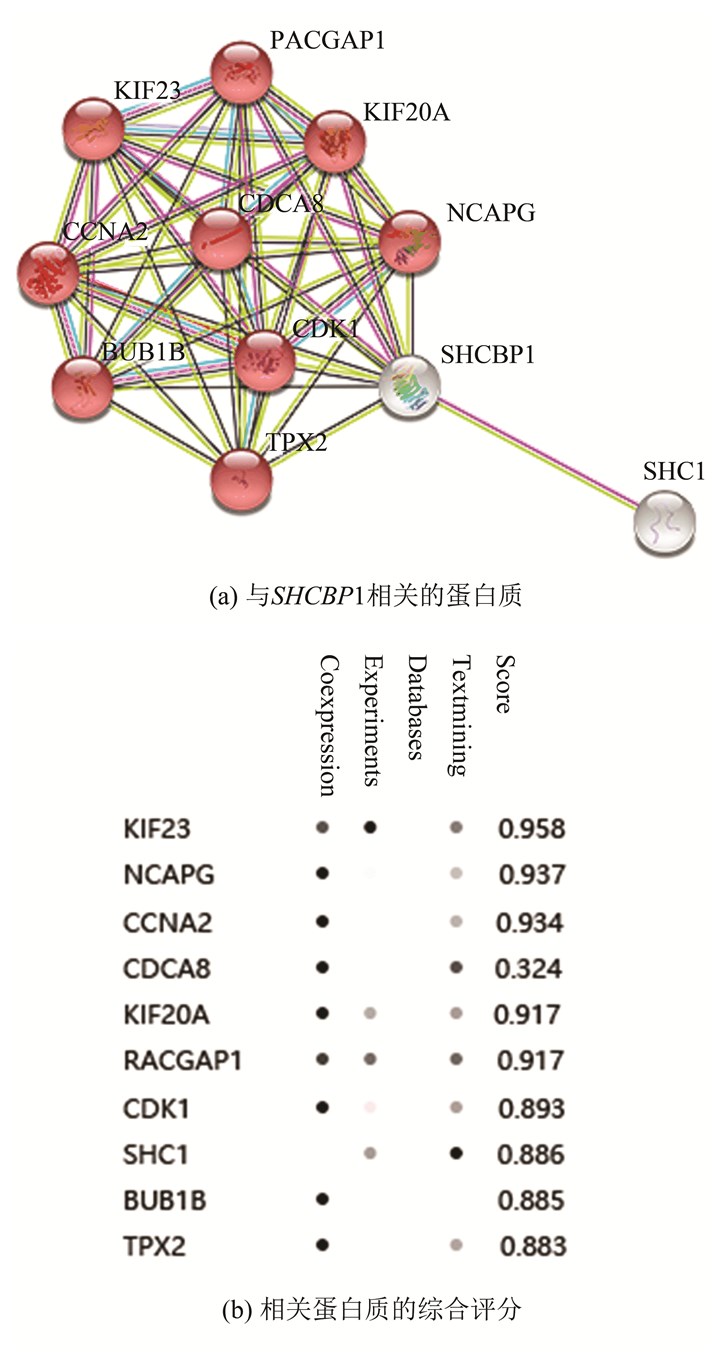
仅仅通过共表达分析并不能完全揭示出SHCBP1作用于乳腺癌的机制。因此,通过STRING对SHCBP1进行蛋白质相互作用网络分析,获得了潜在的与SHCBP1作用的10个蛋白质:KIF23、NCAPG、CCNA2、CDCA8、KIF20A、RACGAP1、CDK1、SHC1、BUB1B、TPX2。除SHC1外,其余蛋白质均与细胞分裂密切相关。根据STRING分析,KIF23与SHCBP1具有最强的相互作用关系(Score=0.958)(见图 7)。
|
图 7 SHCBP1的蛋白质相互作用网络 Figure 7 Protein-protein interaction netaork of SHCBP1 注:(a)以上10个蛋白质与SHCBP1相关,红色标志的9个蛋白质与细胞分裂相关。(b)通过评价这10个蛋白质在共表达、实验、文字挖掘方面和SHCBP1的相关性给出综合的相关性评分。表中圆点颜色越深,说明相关性越强。 |
Shc基因家族在哺乳动物细胞凋亡和耐药性调控中具有重要作用[18],SHCBP1是ShcA的结合伴侣基因。一个好的药物靶点应该具有高度特异性和对正常细胞毒性小的特点。通过慢病毒介导SHCBP1基因的敲除实验,Western blot检测显示,sgRNA感染乳腺癌细胞中SHCBP1蛋白表达明显降低,sgRNA慢病毒敲除SHCBP1显著抑制乳腺癌细胞MDA-MB-231和MCF-7的增殖,表明SHCBP1可能成为乳腺癌治疗的有效靶点[10]。
根据图 2a所示,Luminal B亚型乳腺癌患者中SHCBP1 mRNA高表达,但Kaplan-Meier plotter及GSE41994数据集分析表明SHCBP1 mRNA表达与Luminal B亚型乳腺癌患者的DMFS, DFS没有相关性,这里存在两种可能:(1) SHCBP1在Luminal B亚型患者总体表达水平较高所以不能作为Luminal B亚型的预后标志物;(2) Luminal B亚型本身预后较差,因其低预后使得DMFS按SHCBP1区分不明显。根据临床研究,Luminal B亚型较Luminal A亚型具有更差的预后,但这是由于Luminal B型乳腺癌具有更多的不良预后因素,在肿瘤分期、组织分级及肿瘤大小等基线特征基本一致的情况下,Luminal A亚型与Luminal B亚型乳腺癌对预后的影响没有差异[19],Luminal B亚型并不能作为独立的预后因素。因此,SHCBP1是由于在Luminal B亚型患者总体表达水平较高所以不能作为Luminal B亚型的预后标志物。
通过分析多种公共数据库中的数据集,发现与其他辅助因素协同作用导致肿瘤发生的基础。这样的规律在临床对乳腺癌的诊治中具有极大的价值。在确定患者的肿瘤亚型后,可以通过此规律来大致推测患者的预后。如患者的肿瘤是ER(-)或PR(-)亚型,就需要医护人员更频繁的平时监护以及术后监护,更密切地观察患者情况,一旦出现预后不良的征兆,及时发现并处理,以提升患者的存活率,延长存活时间。而在对各种亚型的乳腺癌患者的预后分析中,如果为Luminal A亚型,SHCBP1 mRNA高表达与DMFS(HR=1.96, P=0.000), DFS(HR=2.93, P=0.002)的缩短有关。所以对于结束住院治疗的乳腺癌患者,应定期复查,检验医师可通过SHCBP1的表达量来推断Luminal A亚型乳腺癌患者的预后情况,这对于改善乳腺癌患者的预后有很大的价值。
通过共表达相关分析发现,在乳腺癌中,SHCBP1可能与CDC45信号通路密切相关。有研究发现CDC45在DNA复制的起始和延长过程中起关键作用[20-21],而CDC45在长期静止或衰老的细胞中沉默,但在不同组织来源的人癌细胞株中过度表达[17]。这提示SHCBP1可能通过调控CDC45表达来参与肿瘤的迁移和侵袭,但此前从未有过文献表明CDC45与乳腺癌之间有关联。因此,这非常有必要通过生物化学和分子生物学手段探究SHCBP1是否与CDC45作用共同参与乳腺癌发生发展过程。
STRING相互作用网络分析进一步揭示了SHCBP1参与乳腺癌发生发展的机制:通过调节一些与细胞分裂关系密切的蛋白质,来调控乳腺癌细胞的分裂。分析表明,蛋白质KIF23与SHCBP1相互作用关系最强,而已知研究证明KIF23 mRNA高表达与ER(-)乳腺癌患者的不良预后具有极强的相关性[22];NCAPG可作为ER(+)乳腺癌的预后标志物[23];CCNA2可作为ER(+)乳腺癌的预后标志物,其高表达与ER(+)乳腺癌的较差预后以及他莫昔芬抗性相关[24]。据图 7所示,以上三个基因都与SHCBP1的表达具有极高的相互作用关系。
利用多种数据库进行数据挖掘,发现与正常组织相比,SHCBP1在乳腺癌组织中表达较高,且该基因高表达的Luminal A亚型乳腺癌患者预后较差。此外,通过共表达分析及蛋白质分析,初步探究SHCBP1作用于乳腺癌的潜在机制。这些证据均表明SHCBP1是一种很有前景的乳腺癌预后指标及潜在治疗靶点。
4 结论1) SHCBP1的表达量可用于预测Luminal A亚型乳腺癌患者的预后情况。
2) SHCBP1可作为乳腺癌潜在的治疗靶点。
3) SHCBP1和CDC45 mRNA表达水平之间呈正相关,并可能通过与KIF23等10种蛋白相互作用来参与乳腺癌发生发展。
| [1] |
TORRE L A, BRAY F, SIEGEL R L, et al. Global cancer statistics, 2012[J]. CA: A Cancer Journal for Clinicians, 2015, 65(2): 87-108. DOI:10.3322/caac.21262 (  0) 0) |
| [2] |
ZUO Y, LI Y, ZHOU Z, et al. Long non-coding RNA MALAT1 promotes proliferation and invasion via targeting miR-129-5p in triple-negative breast cancer[J]. Biomedicine & Pharmacotherapy, 2017, 95: 922-928. DOI:10.1016/j.biopha.2017.09.005 (  0) 0) |
| [3] |
DUFFY M J, WALSH S, MCDERMOTT E W, et al. Biomarkers in breast cancer: Where are we and where are we going?[J]. Advances in Clinical Chemistry, 2015, 71: 1-23. DOI:10.1016/bs.acc.2015.05.001 (  0) 0) |
| [4] |
MARUTHANILA V L, ELANCHERAN R, KUNNUMAKKARA A B, et al. Recent development of targeted approaches for the treatment of breast cancer[J]. Breast Cancer, 2017, 24(2): 191-219. DOI:10.1007/s12282-016-0732-1 (  0) 0) |
| [5] |
JAITAK V. Drug target strategies in breast cancer treatment: Recent developments[J]. Anti-Cancer Agents in Medicinal Chemistry (Formerly Current Medicinal Chemistry-Anti-Cancer Agents), 2014, 14(10): 1414-1427. DOI:10.2174/1871520614666140407114853 (  0) 0) |
| [6] |
JONES N, HARDY W R, FRIESE M B, et al. Analysis of a Shc family adaptor protein, ShcD/Shc4, that associates with muscle-specific kinase[J]. Molecular and Cellular Biology, 2007, 27(13): 4759-4773. DOI:10.1128/MCB.00184-07 (  0) 0) |
| [7] |
SCHMANDT R, LIU S K, MCGLADE C J. Cloning and characterization of mPAL, a novel Shc SH2 domain-binding protein expressed in proliferating cells[J]. Oncogene, 1999, 18(10): 1867-1879. DOI:10.1038/sj.onc.1202507 (  0) 0) |
| [8] |
TAO H C, WANG H X, DAI M, et al. Targeting SHCBP1 inhibits cell proliferation in human hepatocellular carcinoma cells[J]. Asian Pacific Journal of Cancer Prevention APJCP, 2013, 14(10): 5645-5650. DOI:10.18632/oncotarget.11651 (  0) 0) |
| [9] |
PENG C, ZHAO H, CHEN W, et al. Identification of SHCBP1 as a novel downstream target gene of SS18-SSX1 and its functional analysis in progression of synovial sarcoma[J]. Oncotarget, 2016, 7(41): 66822-66834. DOI:10.7314/APJCP.2013.14.10.5645 (  0) 0) |
| [10] |
FENG W, LI H C, XU K, et al. SHCBP1 is over-expressed in breast cancer and is important in the proliferation and apoptosis of the human malignant breast cancer cell line[J]. Gene, 2016, 587(1): 91-97. DOI:10.1016/j.gene.2016.04.046 (  0) 0) |
| [11] |
LIN H Y, ZENG D, LIANG Y K, et al. GATA3 and TRPS1 are distinct biomarkers and prognostic factors in breast cancer: Database mining for GATA family members in malignancies[J]. Oncotarget, 2017, 8(21): 34750-34761. DOI:10.18632/oncotarget.16160 (  0) 0) |
| [12] |
JÉZÉQUEL P, FRÉNEL J S, CAMPION L, et al. bc-GenExMiner 3.0: New mining module computes breast cancer gene expression correlation analyses[J]. Database, 2013, 2013(1): bas060. DOI:10.1093/database/bas060 (  0) 0) |
| [13] |
JÉZÉQUEL P, CAMPONE M, GOURAUD W, et al. bc-GenExMiner: An easy-to-use online platform for gene prognostic analyses in breast cancer[J]. Breast Cancer Research & Treatment, 2012, 131(3): 765-775. DOI:10.1007/s10549-011-1457-7 (  0) 0) |
| [14] |
GYÖRFFY B, LANCZKY A, EKLUND A C, et al. An online survival analysis tool to rapidly assess the effect of 22, 277 genes on breast cancer prognosis using microarray data of 1, 809 patients[J]. Breast Cancer Research Treatment, 2010, 123(3): 725-731. DOI:10.1007/s10549-009-0674-9 (  0) 0) |
| [15] |
SZKLARCZYK D, MORRIS J H, COOK H, et al. The string database in 2017: Quality-controlled protein-protein association networks, made broadly accessible[J]. Nucleic Acids Research, 2017, 45(Database issue): D362-D368. DOI:10.1093/nar/gkw937 (  0) 0) |
| [16] |
JANSEN M P H M, THEO K, REIJM E A, et al. Hallmarks of aromatase inhibitor drug resistance revealed by epigenetic profiling in breast cancer[J]. Cancer Research, 2013, 73(22): 6632-6641. DOI:10.1158/0008-5472.can-13-0704 (  0) 0) |
| [17] |
POLLOK S, BAUERSCHMIDT C, SÄNGER J, et al. Human Cdc45 is a proliferation-associated antigen[J]. Febs Journal, 2010, 274(14): 3669-3684. DOI:10.1111/j.1742-4658.2007.05900.x (  0) 0) |
| [18] |
张培, 池岛乔, 森望. Shc相关磷酸化酪氨酸适配蛋白在衰老过程中的调控作用[J]. 药学学报, 2008, 43(8): 793-800. ZHANG P, IKEJIMA T, MORI N. Regulatory effects of Shc-related phosphotyrosine adaptor proteins on aging[J]. Acta Pharmaceutica Sinica, 2008, 43(43): 793-800. DOI:10.16438/j.0513-4870.2008.08.005 (  0) 0) |
| [19] |
SU Yinghao, ZHENG Ying, ZHENG Wei, et al. Distinct distribution and prognostic significance of molecular subtypes of breast cancer in Chinese women: A population-based cohort study[J]. BMC Cancer, 2011, 11(1): 292. DOI:10.1186/1471-2407-11-292 (  0) 0) |
| [20] |
MIMURA S, MASUDA T, MATSUI T, et al. Central role for cdc45 in establishing an initiation complex of DNA replication in Xenopus egg extracts[J]. Genes to Cells, 2000, 5(6): 439-452. DOI:10.1093/emboj/19.9.2082 (  0) 0) |
| [21] |
TERCERO J A, LABIB K, DIFFLEY J F. DNA synthesis at individual replication forks requires the essential initiation factor Cdc45p[J]. Embo Journal, 2014, 19(9): 2082-2093. DOI:10.1046/j.1365-2443.2000.00340.x (  0) 0) |
| [22] |
KATO T, LEE D, WU L, et al. Kinesin family members KIF11 and KIF23 as potential therapeutic targets in malignant pleural mesothelioma[J]. International Journal of Oncology, 2016, 49(2): 448-456. DOI:10.3892/ijo.2016.3566 (  0) 0) |
| [23] |
MARTIN K J, PATRICK D R, BISSELL M J, et al. Prognostic breast cancer signature identified from 3d culture model accurately predicts clinical outcome across independent datasets[J]. PLOS One, 2008, 3(8): e2994. DOI:10.1371/journal.pone.0002994 (  0) 0) |
| [24] |
GAO T, HAN Y, YU L, et al. CCNA2 is a prognostic biomarker for er+ breast cancer and tamoxifen resistance[J]. PLOS One, 2014, 9(3): e91771. DOI:10.1371/journal.pone.0091771 (  0) 0) |
 2020, Vol. 18
2020, Vol. 18


