多聚半乳糖醛酸酶(polygalacturonases,PGs)属于一个大的水解酶家族,它通过降解果胶在植株生长发育过程中的细胞分离事件中发挥重要作用[1]。果胶是一种杂多糖,它是双子叶植物体细胞初生细胞壁的主要成分,同时也是花粉内壁和花粉管壁的主要成分,因此,参与果胶代谢相关酶和蛋白对植物的生长和生殖发育至关重要[2]。PG已被鉴定出具有四个保守的功能结构域(Ⅰ-Ⅳ),依据PG降解途径的不同,可以把PG分为内切PG (endo-PG)、外切PG (exo-PG)、鼠李糖PG (rhamno-PG),它们分别能够在不同部位催化水解α-(1-4)聚半乳糖苷键的断裂[3]。目前,已经在拟南芥[4]、黄瓜[5]、桃[1]、苹果[6]等众多植物中对PG基因家族进行了鉴定,对其进行功能分析发现,PGs调控着果实成熟软化、器官脱落、宿主相互作用、荚果和花药开裂、花粉管生长、种子萌发和幼苗下胚轴伸长等多个生物学过程[7]。
紫花苜蓿是当今世界上栽培面积最大的牧草作物,具有适应性强、产草量高、品质优等特点,素有“牧草之王”之称。但是,紫花苜蓿为自交不亲和的同源四倍体植物,基因组极其复杂,严重限制了其遗传育种研究。蒺藜苜蓿(Medicago truncatula)是一种自花授粉的苜蓿,其基因组较小(约470 Mb),且为二倍体,已经完成基因组测序,使其成为研究豆科,特别是苜蓿属(如紫花苜蓿)基因功能的模式植物[8]。对蒺藜苜蓿中PG基因家族进行全基因鉴定,通过序列比对和系统进化分析,完成PG基因家族成员的分类,同时,根据PG基因家族的染色体定位信息,明确其在基因组的分布特征。最后,利用蒺藜苜蓿的转录组测序数据,分析PG基因家族在蒺藜苜蓿生长发育过程的表达谱,为解析蒺藜苜蓿中PG基因家族的重要作用提供参考。
1 材料和方法 1.1 蒺藜苜蓿PG基因家族的鉴定和分类从JCVI数据库(http://www.jcvi.org/medicago/)下载蒺藜苜蓿基因组测序数据,构建本地BLAST数据库。从TAIR数据库中收集拟南芥PG家族蛋白序列,利用这些蛋白序列作为BLAST查询序列,对蒺藜苜蓿基因组进行搜索[9],evalue设置为:1×10-5,覆盖率大于80%。在Pfam数据库中下载PG结构域的HMM文件(Glyco_hydro_28,PF00295),利用HMMER (evalue:1×10-2)[10]搜索候选PG蛋白的结构域信息;同时,采用软件SMART检测,确认候选基因含有PG的结构域。最后,根据拟南芥PG蛋白基因的分类信息对蒺藜苜蓿PG基因家族进行分类。
1.2 蒺藜苜蓿PG基因家族的系统进化分析利用ClustalW2[11]对拟南芥和蒺藜苜蓿PG蛋白进行多序列联配比对分析,采用MEGA4[12]构建PG基因家族系统发育树,系统进化分析参数如下:(1)建树方法为邻近法(neighbor-joining,NJ);(2)遗传距离为泊松距离(Poisson correction);(3)抽样次数为1 000次(bootstrap: 1 000 replications)。
1.3 蒺藜苜蓿PG基因家族的染色体定位分析从蒺藜苜蓿基因组中检索到所有PG基因的位置信息和CDS序列,利用BLAST进行两两对比,当两个PG基因的一致性超过80%时,则这两个基因之间存在基因复制。根据PG基因在蒺藜苜蓿基因组中的位置信息及基因间的复制情况,利用软件CIRCOS[13]绘制PG基因家族在蒺藜苜蓿基因组中的分布情况。
1.4 蒺藜苜蓿PG基因家族的表达分析蒺藜苜蓿转录组测序(RNA-seq)数据(SRR350517-SRR350521,SRR350538和SRR349692)从NCBI的SRA数据库下载,转录组数据包含蒺藜苜蓿的根部(Root)、根部结瘤(Nodule)、叶片(Blade)、芽(Bud),心皮(Seedpod)和花(Flower)六个组织或部位。利用TopHat将所有高通量序列映射到蒺藜苜蓿基因组,并通过Cufflinks进行基因表达[14]。提取PG基因的表达数据,利用R软件对其进行分析、聚类和可视化。
2 结果与分析 2.1 蒺藜苜蓿PG基因家族的鉴定和分类通过相似性比对和结构域搜索,从蒺藜苜蓿基因组中共鉴定出74个MtPG基因家族成员,根据它们所属的亚家族和染色体位置,依次命名为MtPG01-MtPG74,如表 1所示。这些MtPG基因编码的推测蛋白长度为236-775 aa,内含子2-9个,其中,2个MtPG基因(MtPG39和MtPG47)只含有两个内含子,60个MtPG含有4个以上内含子。在已报道的PG基因家族的研究中,蒺藜苜蓿PG的数量是高于其他植物,如拟南芥(68)、番茄(54)、黄瓜(53)、桃子(45)。根据PGs的分子系统进化关系,将这些PG基因分为六个亚家族(A-F),分别含有9、7、4、11,18和25个PG基因家族成员。
| 表 1 蒺藜苜蓿基因组中PG基因 Table 1 PG genes identified in Medicago truncatula |
为了详细地研究蒺藜苜蓿PG基因家族的分子系统进化关系,利用74个MtPG基因的蛋白质序列进行了多序列比对和系统进化树构建,结果显示,它们可以分为六个亚家族,如图 1所示。六个亚家族(A-F)分别含有9、7、4、11, 18和25个PG基因家族成员,这与水稻、杨树[15]、桃、苹果等植物中PG基因家族成员分布类似,都为6亚家族。但是,在拟南芥中,PG基因家族总共有68个成员,可以分为7个亚家族,即A-G,其中亚家族G含有7个成员,研究表明它们在花粉发育过程中执行降解花粉母细胞细胞壁的生物学功能。同时,对比几种植物的PG基因家族成员分布发现,蒺藜苜蓿的亚家族E和F明显拥有更多的成员,这可能是蒺藜苜蓿PG基因家族成员明显增多的主要原因。
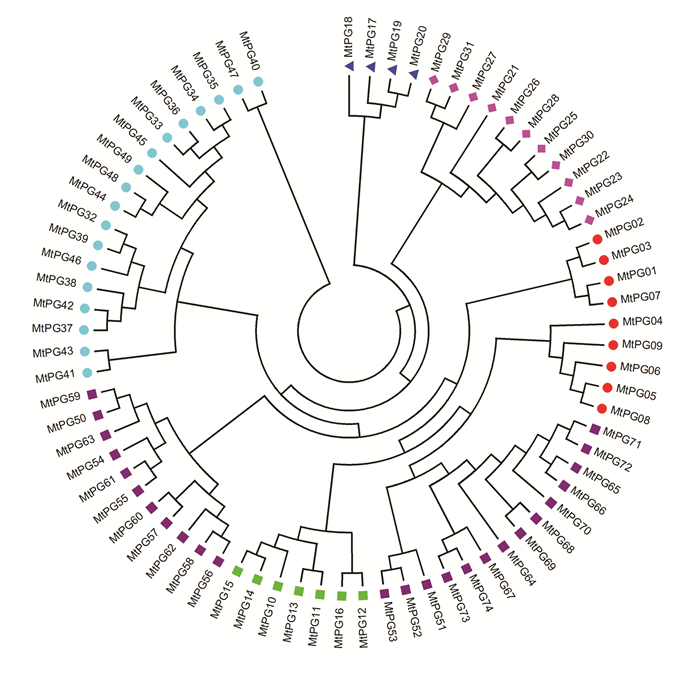
|
图 1 蒺藜苜蓿PG基因家族的系统进化分析 Figure 1 Phylogenetic analysis of PG gene family in Medicago truncatula 注:红色圆形代表亚家族A;绿色方形代表亚家族B;蓝色三角形代表亚家族C;粉红色菱形代表亚家族D;青色圆形代表亚家族E;紫色方形代表亚家族F。 |
为了探究蒺藜苜蓿PG基因家族扩增和进化的机制,本研究对蒺藜苜蓿PG基因进行染色体定位分析。通过提取蒺藜苜蓿PG基因的染色体定位信息发现,73个MtPG基因(MtPG50除外)定位在8条染色体上,如图 2所示。图中结果显示,这些MtPG基因在染色体上不是均匀分布,其中,8号和2号染色体上分布成员最多,分别为16个和15个,6号染色体最少,只有2个MtPG基因。此外,这些MtPG基因在单条染色体上也不是均匀分布,呈现明显的聚集分布,如1号和8号染色体上都形成了MtPG基因簇。通过比对发现,这些MtPG基因簇都是基因复制形成的;同时,染色体之间也存在MtPG基因间的复制事件,这些复制事件可能是MtPG基因家族的主要扩增动力。

|
图 2 蒺藜苜蓿PG基因在染色体定位 Figure 2 Chromosomal locations of PG genes in Medicago truncatula 注:红色连线代表亚家族A成员间的复制事件;蓝色连线代表亚家族B成员间的复制事件;紫色连线代表亚家族C成员间的复制事件;绿色连线代表亚家族D成员间的复制事件;黄色连线代表亚家族E成员间的复制事件;黑色连线代表亚家族F成员间的复制事件。 |
为了研究MtPG基因家族成员在蒺藜苜蓿生长发育过程中生物学功能,本研究从NCBI数据库中下载了蒺藜苜蓿转录组测序数据,获得了31个MtPG基因在六种组织(或器官)中的表达模式,对它们的表达模式进行了聚类分析,结果如图 3所示。结果显示,31个MtPG基因成员可以分成四组(Cluster Group Ⅰ-Ⅳ)。其中,Ⅰ组含有7个MtPG基因(MtPG21、22、23、26、28,35和52),它们主要在蒺藜苜蓿的心皮和花组织中表达,推测这些MtPG基因主要与蒺藜苜蓿的生殖发育过程相关;Ⅱ组含有11个MtPG基因(MtPG08、09、12、37、41、42、43、46、55,61和63),它们主要在根或心皮组织中表达,可能参与了这两种组织(或器官)的发育和形成过程;Ⅲ组含有10个MtPG基因家族成员(MtPG02、03、05、16、32、40、44、47,48和49),它们主要集中在根和根部的结瘤中表达,表明它们可能与蒺藜苜蓿的根组织形态建成相关;最后,Ⅳ组包含3个MtPG基因(MtPG01,06和19),它们主要在根、结瘤和花组织中表达,表明它们主要参与这两个组织的发育调控过程。

|
图 3 蒺藜苜蓿PG基因表达的聚类分析 Figure 3 Cluster analysis of PG gene expression in Medicago truncatula 注:粉色为聚类Ⅰ;黄色为聚类Ⅱ;蓝色为聚类Ⅲ;绿色为聚类Ⅳ。 |
多聚半乳糖醛酸酶属于植物最大的水解酶家族之一,它参与到了果实成熟、器官脱落、花粉成熟等过程,对植物的生长发育具有重要意义。在不同植物中,PG基因的进化和分类已经被大量的研究。通过生物信息学方法分析,在蒺藜苜蓿基因组中总共发现了74个MtPG基因,这些基因被聚类到六个亚家族(A-F),且这几个亚家族之间基因成员数目差异较大,其中亚家族F成员最多,高达25个;亚家族C成员最少,只有4个。通过序列比对发现,MtPG基因家族各成员之间存在大量的基因复制事件,按照产生原因,它们可以分为串联复制和片段复制两类。在蒺藜苜蓿基因组范围内,总共发生了25次MtPG基因复制事件,其中串联复制事件11次,片段复制事件14次,这些复制事件促使了MtPG基因家族的扩张,超过了其他植物基因组中的数量。此外,串联复制事件促使MtPG基因在染色体上分布发生富集,形成MtPG基因簇,如1号染色体上的MtPG32-36基因簇。同时,片段复制事件促进了MtPG基因家族成员在不同染色体上的扩张,如5号染色体和8号染色体间发生了MtPG04-05与MtPG08-09的片段复制,引起亚家族A的基因成员增多。
通过蒺藜苜蓿的基因表达谱分析发现,MtPG基因在蒺藜苜蓿的根器官形成和生殖生长过程中承担重要的调控作用。研究结果显示,部分MtPG基因成员(MtPG21、22、23、26、28,35和52)主要在心皮和花组织中表达,可能与蒺藜苜蓿的雌雄蕊发育相关,这与梁颖[2]的报导是一致,明确了它们在花蕊发育过程中调控作用。同时,这些MtPG基因分布较为集中在1号和2号染色体,表明它们可能通过同一遗传机制参与花蕊的生长发育调控过程。类似的还有MtPG48和MtPG49,它们都在根器官和根部的结瘤组织中表达,说明它们可能参与蒺藜苜蓿根部形态的建成过程。同时,这两个MtPG基因是通过片段复制事件形成的,而且两者的表达模式类似,略有差别,说明MtPG基因复制扩展基因家族的规模,为MtPG基因功能的分化创造了条件。
4 结论采用相似性比对和结构域搜索相结合的方法,从蒺藜苜蓿基因组中鉴定出了74个MtPG基因家族成员,并通过系统进化分析,明确了蒺藜苜蓿MtPG基因家族的分类和进化关系。同时,对蒺藜苜蓿PG基因家族进行染色体定位分析,解析了基因复制在MtPG基因进化过程中的作用。最后,通过蒺藜苜蓿基因表达谱分析,初步阐述了MtPG基因在蒺藜苜蓿根器官生长和生殖发育过程中的重要作用,这将为进一步解析MtPG基因的分子调控作用机制提供基础。
| [1] |
霍如雪, 刘振宁, 杨青, 等. 桃PG基因家族的鉴定与分析[J]. 江苏农业科学, 2016, 44(6): 33-40. HUO Ruxue, LIU Zhenning, YANG Qing, et al. Identification and analysis of the PG gene gamily in peach[J]. Jiangsu Agricultural Sciences, 2016, 44(6): 33-40. DOI:10.15889/j.issn.1002-1302.2016.06.008 (  2) 2) |
| [2] |
梁颖.植物PG基因进化的研究与白菜雄蕊发育相关的两个PG基因的表达分析和功能鉴定[D].杭州: 浙江大学, 2016. LIANG Ying. Molecular evolution of polygalacturonase genes in plants and the expression analysis and functional characterization of two stamen PG genes in brassica campestris[D]. Hangzhou: Zhejiang University, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10335-1016124801.htm (  2) 2) |
| [3] |
钱铭.桃PG (Polygalacturonase)家族成员在果实成熟软化过程中的表达特性及质地形成中的功能研究[D].杨凌: 西北农林科技大学, 2017. QIAN Ming. Expression characteristics and function of peach PG(polygalacturonase) family members during fruit ripening and softening[D]. Yangling: Northwest Agricultural & Forestry University, 2017. http://cdmd.cnki.com.cn/Article/CDMD-10712-1017101202.htm (  1) 1) |
| [4] |
GONZALEZ-CARRANZA Z H, ELLIOTT K A, ROBERTS J A. Expression of polygalacturonases and evidence to support their role during cell separation processes in Arabidopsis thaliana[J]. Journal of Experimental Botany, 2007, 58(13): 3719-3730. DOI:10.1093/jxb/erm222 (  1) 1) |
| [5] |
YU Y, LIANG Y, LV M, et al. Genome-wide identification and characterization of polygalacturonase genes in Cucumis sativus and Citrullus lanatus[J]. Plant Physiology and Biochemistry, 2014, 74: 263-275. DOI:10.1016/j.plaphy.2013.11.022 (  1) 1) |
| [6] |
陈鸿飞, 邵红霞, 樊胜, 等. 苹果全基因组多聚半乳糖醛酸酶基因家族的鉴定及进化分析[J]. 园艺学报, 2016, 43(10): 1863-1877. CHEN Hongfei, SHAO Hongxia, FAN Sheng, et al. Identification and phylogenetic analysis of the polygalacturonase gene family in apple[J]. Horticultural Plant Journal, 2016, 43(10): 1863-1877. DOI:10.16420/j.issn.0513-353x.2016-0224 (  1) 1) |
| [7] |
HADFIELD K A, BENNETT A B. Polygalacturonases: Many genes in search of a function[J]. Plant Physiology, 1998, 117: 337-343. DOI:10.1104/pp.117.2.337 (  1) 1) |
| [8] |
YOUNG N D, DEBELLE F, OLDROYD G E, et al. The medicago genome provides insight into the evolution of rhizobial symbioses[J]. Nature, 2011, 480(7378): 520-524. DOI:10.1038/nature10625 (  1) 1) |
| [9] |
ALTSCHUL S F, MADDEN T L, SCHAFFER A A, et al. Gapped BLAST and PSI-BLAST: A new generation of protein database search programs[J]. Nucleic Acids Research, 1997, 25(17): 3389-3402. DOI:10.1093/nar/25.17.3389 (  1) 1) |
| [10] |
FINN R D, CLEMENTS J, EDDY S R. HMMER web server: Interactive sequence similarity searching[J]. Nucleic Acids Research, 2011, 39(suppl 2): W29-W37. DOI:10.1093/nar/qkr367 (  1) 1) |
| [11] |
THOMPSON J D, HIGGINS D G, GIBSON T J. CLUSTAL W: Improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucleic Acids Research, 1994, 22(22): 4673-4680. DOI:10.1093/nar/22.22.4673 (  1) 1) |
| [12] |
TAMURA K, DUDLEY J, NEI M, et al. MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4: Molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24(8): 1596-1599. DOI:10.1093/molbev/msm092 (  1) 1) |
| [13] |
KRZYWINSKI M I, SCHEIN J E, BIROL I, et al. Circos: An information aesthetic for comparative genomics[J]. Genome Research, 2009, 19(9): 1639-1645. DOI:10.1101/qr.092759.109 (  1) 1) |
| [14] |
TRAPNELL C, WILLIAMS B A, PERTEA G, et al. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology, 2010, 28(5): 511-515. DOI:10.1038/nbt.1621 (  1) 1) |
| [15] |
YANG Z L, LIU H J, WANG X R, et al. Molecular evolution and expression divergence of the Populus polygalacturonase supergene family shed light on the evolution of increasingly complex organs in plants[J]. New Phytologist, 2013, 197(4): 1353-1365. DOI:10.1111/nph.12107 (  1) 1) |
| [16] |
RHEE S Y, OSBORNE E, POINDEXTER P D, et al. Microspore separation in the quartet 3 mutants of Arabidopsis is impaired by a defect in a developmentally regulated polygalacturonase required for pollen mother cell wall degradation[J]. Plant Physiology, 2003, 133(3): 1170-1180. DOI:10.1104/pp.103.028266 |
 2020, Vol. 18
2020, Vol. 18



