不孕不育日渐成为现代社会生殖健康面临的严重问题,其中男性不育约占50%。从生物学的角度来看,男性生殖问题的关键是如何产生正常的生殖细胞,而精子发生又是生殖细胞产生的核心环节。深入研究并认知精子发生的分子机制,对人类的生殖健康、男性的生育调节、男性不育的诊治均有十分重要的理论和实践意义。临床样品上发现的与精子发生障碍相关的遗传突变通常需要在小鼠等重要模式动物中进行功能验证和机制探索,因此对于小鼠精子发生不同时期细胞的基因表达调控研究可为人类精子发生调节机制的阐明提供重要线索。除了信使RNA,新近研究还发现了不少lncRNA(long non-coding RNA,长非编码RNA)在生殖细胞增殖、分化过程中起到重要作用[1]。环状RNA(circular RNA, circRNA)作为lncRNA家族的一个新成员,其是否在精子发生过程中发挥特定的生物学功能也引起了广泛的关注。
circRNA和过去研究的许多种类RNA不同,其序列并不以基因组外显子正常的顺序排列组成,而是RNA的5'末端和3'末端头尾相连,形成一个像质粒一样共价闭合的单链环状分子[2]。按形成circRNA的序列在原始母基因中的类别,circRNA可分为外显子、内含子和基因间区域来源,其中,目前发现的circRNA绝大多数来源于外显子[2]。研究表明外显子侧翼的内含子中的反向重复序列(比如人中的Alu序列)可促进两个线性外显子的反向成环而形成circRNA[3]。
近年来的研究也不断发现:circRNA具有多种基因调控功能,如高丰度的circRNA可以竞争所在母基因正常的剪接或可变剪接,从而使得正常剪接的RNA水平下降,引起下游的一系列反应[4];circRNA可作为microRNA(miRNA)海绵吸附特定miRNA,继而影响该miRNA对其下游靶基因的调控[5]。此外,circRNA上的m6A(腺嘌呤6位甲基化)修饰可以促进翻译[6]。circRNA还可以通过结合RNA结合蛋白(RBP)[7]后与Pol Ⅱ互作调控宿主基因的表达[8]等方式体现其功能。但是在精子发生过程中,circRNA的生成机制和下游功能尚不清楚。
精子发生是一个高度有序的过程,它的每一个阶段都被精细调控。目前已知有一些RNA结合蛋白如ELAVL1/HuR、hnRNP G-T、NANOS2等在精子发生过程中起着重要调控作用[7, 9-10]。同时,多种miRNA如miR-21[11]、miR-17-92簇[12]、mir-184[13]等也被证实在精子发生过程中起重要调节作用。有研究发现circRNA在睾丸中高度富集[14],与合作者的研究也发现小鼠精子发生过程中lncRNA和circRNA均表现出了显著的表达变化[15],提示circRNA在精子发生过程中也可能起着重要作用,其上游的形成机制值得深入研究。利用之前发表的数据,系统分析了小鼠精子发生五个时期(小鼠精原干细胞、原始精原细胞、前细线期精母细胞,粗线期精母细胞及圆形精子细胞)中的circRNA及其可能成环机制,并分析探讨了随精子发生不同时期表达显著变化的circRNA行使功能的可能机制。
1 材料和方法 1.1 数据获取所用数据来自于本实验室之前发表的数据[15],其生物学样本均来自于DBA/2和C57BL6两种品系的老鼠产生的杂交后代,样本获取、RNA提取及RNA文库构建流程参见文献[15]。原始数据也可从NCBI SRA数据库下载(SRP067167)。每个时期样品(处理)均有两份生物学重复,各样本及它们对应的原始reads数据见表 1。为了让后续分析更加精准,利用NGSQCToolkit[16]去除了原始测序数据中的低质量reads,并用fastuniq对质量过滤后的reads进行去冗余。
| 表 1 参试样本信息及测序reads数 Table 1 Reference sample information and sequencing reads |
CIRI[17]是一款常用的从RNA-seq测序数据中预测鉴定circRNA的软件,使用CIRI的默认参数,预测各参试样本中的circRNA,并将得到的circRNA和CIRCpedia数据库[18]中的circRNA进行比对分析。采用sailfish-cir软件[19](默认参数)对circRNA在各时期的表达进行定量分析。同时,借助sailfish_cir计算出的circRNA read数目,分析获得到各个时期的环形-线性比。最后,为了确保circRNA差异表达分析的可靠性,仅对Ribozero文库来源的circRNA使用Next maSigPro[20]分析五个时期中差异表达的circRNA,并采用R中的clusterProfiler包[21]对差异表达circRNA的来源基因进行了Gene Ontoloty(GO)功能类分析。
1.3 小鼠精子发生过程中circRNA上游生成机制分析为了分析在小鼠精子发生过程中哪些因素可能影响了外显子型circRNA的生成,首先分析了circRNA侧翼内含子的长度,并将它和随机抽取的内含子的长度进行比较。借助RepeatMasker[22]分析获得基因组上重复序列,并归类到几种常见的反向重复序列类型;从它们在基因组中的位置信息判定哪些circRNA的侧翼内含子含有反向重复序列,并计算每种重复序列在circRNA侧翼内含子中的反向互补配对情况。
1.4 小鼠精子发生过程中circRNA功能机制预测使用CIRI-FULL[23]和CIRI-AS[24]从基因组信息中获取circRNA的序列信息。随后用miRanda-3.3a预测circRNA中miRNA的结合位点(设置miRanda的参数为-sc 170 -en 25,相比较miRanda的默认参数-sc 140和-en 1,提升了score和energy阈值,减少了预测结果的假阳性)。结合circRNA序列信息和m6A基序(RRm5ACH),利用IRESfinder[25]和自己编写的程序搜索circRNA上的m6A潜在位点预测circRNA作为翻译模板的可能性。结合POSTAR2数据库[26]收集的RBP结合基序,搜索circRNA中潜在的RBP结合位点。
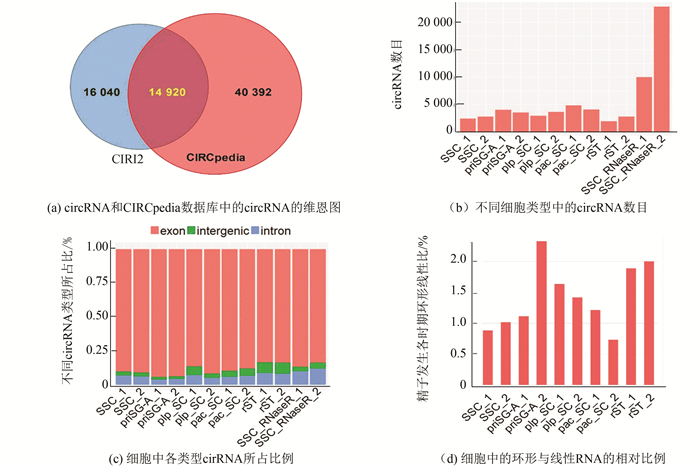
2 结果分析 2.1 小鼠精子发生5个时期中的环形RNA利用CIRI2系统分析了小鼠精子发生各参试样本的RNA-seq数据,共发现30 960个circRNA,其中有14 920个(48.2%)circRNA可被CIRCpedia数据库中的circRNA交互验证(见图 1a)。由于CIRCpedia数据库中只收集了小鼠睾丸的circRNA,并未收集完整小鼠精子发生各个时期的所有数据[18],因此推测余下的circRNA(约50%)可能是小鼠精子发生特有的circRNA。RNaseR处理可以大量去除线性RNA从而富集circRNA,分析结果显示,ribozero文库来源的RNA-seq数据鉴定出的circRNA的数量在5个不同细胞类型中变化幅度不大;而在精原干细胞中,RNaseR处理文库中发现的circRNA数要大大多于从ribozero文库中发现的circRNA数目(见图 1b),这也提示了CIRI发现circRNA的可靠性。
|
图 1 小鼠精子发生各时期细胞中circRNA的数目、类型及环形线性比 Figure 1 Number, type, and circular-linear ratio of circRNA in mouse spermatogenesis 注:(b)中1和2指同一细胞类型的两个生物学重复。RNaseR指对SSC细胞进行RNaseR酶处理去除线性RNA的样品。(c)中外显子来源即exon、内含子来源即intron、基因间区域即intergenic。 |
对精子发生各个时期的circRNA进行序列来源分析发现,它们绝大多数主要来源于外显子区域,即外显型circRNA(见图 1c)。由于circRNA可能通过竞争性剪接影响所在母基因的正常线性mRNA的生成,计算了各时期环形-线性比,发现十个样本的环形线性比均小于3%(见图 1d)。但有趣的是,圆形精子细胞中环形线性比要明显高于其他精子发生时期(见图 1d)。因为circRNA不易降解,所以推测圆形精子细胞中的circRNA增多更可能是circRNA上游生成机制的改变引起的。
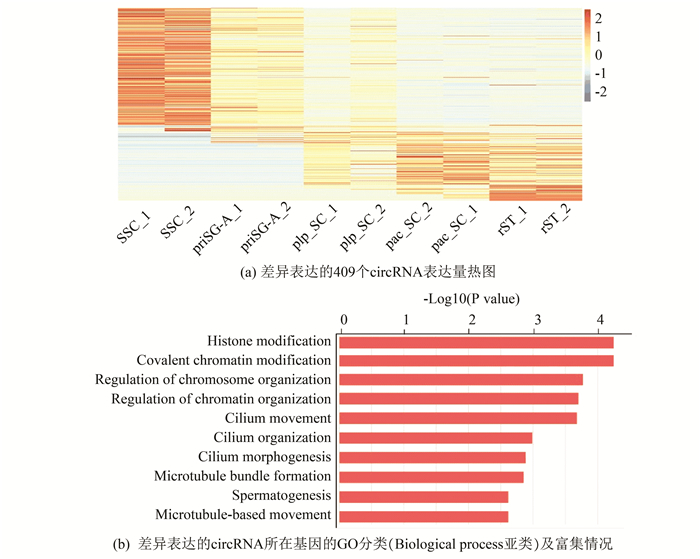
2.2 精子发生过程中差异表达的circRNA及其所在基因的功能富集分析分析小鼠精子发生不同时期circRNA本身的丰度差异和所属母基因的功能情况可为circRNA在精子发生中的功能推测提供重要线索。判别差异表达的基础是对circRNA丰度的定量,之前circRNA定量都是直接计算circRNA反向剪切连接处的读长(Junction reads)数目,Li等开发了sailfish-cir软件[19],该软件应用改进过的EM(Expectation-Maximization)算法对circRNA进行定量,能校正多种已知的系统性偏差,也克服了之前定量方法存在依赖测序深度、数据离散的缺陷,提高了circRNA定量的准确度。采用sailfish-cir算法对小鼠精子发生不同时期circRNA的稳态表达量进行了定量,随后我们使用maSigPro基于线性回归的方法分析了在不同时期样本中表达量显著不同(P≤0.001,软件默认差异判断标准)的circRNA,共得到409个差异表达circRNA。从这409个差异circRNA的热图中可以看出,circRNA表达量上升和下降的趋势表现出了极高的一致性,并且组间差异较小(见图 2a)。为了探索这些差异表达的circRNA与小鼠精子发生过程的联系,对这些circRNA来源的母基因做了基因本体(GO)富集分析,结果发现它们富集的生物学过程包括精子发生、纤毛运动和形态、微管束的形成和运动以及染色质修饰和组蛋白修饰等(见图 2b),暗示相应circRNA可能在精子发生中起作用。上述结果暗示circRNA在精子发生过程中呈动态变化且所在基因与精子发生功能具有相关性。
|
图 2 小鼠精子发生不同时期circRNA的差异表达及所在基因功能富集分析 Figure 2 Differential expression of circRNA and functional enrichment analysis of the gene in mouse spermatogenesis at different stages 注:(a)中颜色表示circRNA的相对表达量,颜色从蓝到红表示相对表达量越来越高。每一行代表一个circRNA,每一列代表一个样本。(b)中横坐标为-log10(P value),值越大表示越显著。 |
已有研究表明,在人细胞中侧翼内含子中反向重复序列的配对可促进外显子来源的circRNA的生成,外显子型circRNA的两个侧翼内含子中Alu重复序列的反向配对(Inverted repeated across,即IRacross)数目与同一内含子内部的Alu序列的配对(Inverted repeated within,即IRwithin)数目之间的竞争是circRNA形成的重要因素(见图 3),IRacross配对数目越大,越能促进circRNA的生成[3]。

|
图 3 circRNA形成及侧翼内含子中重复序列反向互补配对示意图 Figure 3 CircRNA formation and reverse complementary repeated sequences in flanking introns 注:一个circRNA由绿色和蓝色两个exon组成,位于两个侧翼内含子中的重复序列反向互补配对(IRacross)可以促进circRNA的生成,而位于同一内含子中的重复序列反向互补配对(IRwithin)则会抑制circRNA的生成。 |
小鼠中除了SINE/Alu序列,还有SINE序列(SINE/B2和SINE/B4)、LINE/L1、ERVL-MaLR和ERVK等内源逆转录病毒来源的重复序列。那么小鼠精子发生过程中这些不同类型的重复序列间的互补配对是否也对circRNA的形成有贡献呢?为了回答这个问题,对小鼠精子发生中外显子来源的circRNA的侧翼内含子进行了系统分析。首先将circRNA侧翼内含子的长度和随机抽取的内含子进行了比较,结果发现,小鼠精子发生相关细胞中所存在的circRNA的侧翼内含子的长度明显比随机抽取的内含子更长(见图 4a)(Wilcoxon Signed Rank Test,P < 0.001)。接着评估外显子来源的circRNA两个侧翼的内含子之间所形成的反向互补配对的数目,结果显示,小鼠精子发生过程中circRNA侧翼内含子中各种类型的IRacross数目均显著多于随机获取的内含子中的IRacross数目(见图 4b),提示小鼠精子细胞中多种重复序列对circRNA的形成均有潜在贡献。比较了circRNA的侧翼内含子和随机抽取的内含子中IRacross-IRwithin差值,发现成环的外显子中该值也显著大于对照(见图 4c),进一步提示侧翼内含子中反向重复序列的配对可能是小鼠精子发生过程中细胞内circRNA生成的重要促进因素。

|
图 4 circRNA侧翼内含子长度及其中的反向互补配对的重复序列分析 Figure 4 Length of circRNA flanking introns and repeated sequence analysis of their reverse complementary pairs 注:(a)中random组表示经随机选取多次的与circRNA侧翼内含子数目相当的内含子(作为对照);circRNA组表示小鼠精子发生中外显子型circRNA所对应的侧翼内含子。(b)中横坐标为不同的反向重复序列类型。对每种重复序列类型,灰色为对照组,表示随机选取的侧翼内含子(random);黄色代表circRNA侧翼内含子(total_circ);橙色为差异表达的circRNA的侧翼内含子(diff_circ);蓝色为来源于精子发生相关基因且差异表达的circRNA的侧翼内含子(diff_sperm)。(c)差异检验是基于和随机对照组的比较。#:P value < 0.001,Wilcoxon Signed Rank Test。 |
在小鼠精子发生过程中发现了许多circRNA,这些circRNA在精子发生进程中的潜在生物学功能是值得探讨的问题。circRNA可以通过多种方式来发挥其生物学功能,如促进所在基因的转录、竞争所在基因成熟mRNA的产生、作为microRNA(miRNA)的分子海绵、作为多个蛋白质结合的分子海绵、作为翻译模板等[5-6]。主要从两个层面来分析这些circRNA潜在的功能,即作为miRNA分子海绵的circRNA和具有翻译潜能的circRNA,同时对circRNA和RBP的互作进行了初步分析。
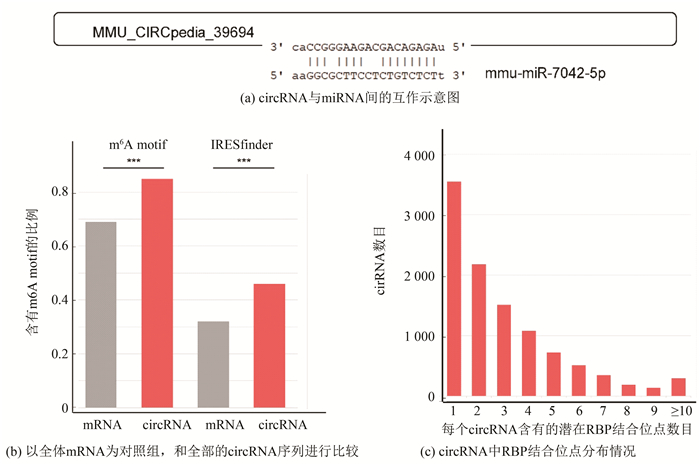
2.4.1 circRNA与miRNA结合作为miRNA“海绵”的功能预测精子发生中miRNA可通过转录后调控调节其靶基因的表达进而影响精子发生进程,如miR-19a、miR-19b通过调节PTEN的表达来影响原始生殖细胞的增殖[27-28],miR-122a通过结合TNP2对精子生成的后期阶段发挥调控作用[29]等。那么研究发现的409个精子发生中差异表达的circRNA中有多少可能通过miRNA分子海绵的方式起作用呢?利用miRanda-3.3a[30]对这409个差异表达的circRNA进行了miRNA结合位点预测,结果发现137个circRNA具有miRNA结合位点,涉及124个miRNA。值得提及的是,研究发现具有miRNA结合位点的circRNA中都只有一个miRNA结合位点(见图 5a)。虽然早期的研究曾报导circRNA可结合很多的miRNA,但研究表明,只有一个miRNA结合位点的circRNA也可调控相应miRNA的效应浓度并对表型产生影响[31]。发现MMU_CIRCpedia_39694(chr4:45987462|45990230)这个circRNA来源于精子发生相关基因TDRD7,预测分析发现它可以结合mmu-miR-7042-5p,从而有可能减少该miRNA对有功能的TDRD7线性mRNA的影响。精子发生过程中这些潜在的circRNA-miRNA互作在小鼠精子发生中的调控作用有待后续实验进一步验证。
|
图 5 小鼠精子发生相关细胞中circRNA的m6A基序及miRNA、RBP结合位点分析 Figure 5 Analysis of m6A motif and miRNA/RBP binding sites of circRNA in mouse spermatogenesis-related cells 注:图(b)中左边两组为自写脚本计算的mRNA和circRNA中含有m6A基序(RRm5ACH motif)的比例;右边两组为IRESfinder的结果。灰色为对照组,来自总mRNA的碱基序列。***:P value < 0.001,Wilcoxon Signed Rank Test。 |
有研究表明,一些circRNA可作为翻译模板产生蛋白质,这些可被翻译的circRNA具有m6A修饰和特定的基序(RRm5ACH),这一特征可招募翻译起始复合物并促进核糖体组装,使circRNA最终可以翻译出蛋白质[6]。为了考察精子发生过程中是否存在具有潜在翻译功能的circRNA,以普通的mRNA序列作为对照组,对全体circRNA、差异表达的circRNA和来自精子发生相关基因的差异表达circRNA分别进行m6A基序预测分析,发现全体circRNA中有85%的序列含有RRm5ACH基序,而作为对照的全部mRNA的序列中该基序的比例仅为69%,两者间存在显著差异(见图 5b左,p值< 0.001,Wilcoxon Signed Rank Test)。而使用IRESfinder[25]发现所有circRNA中有46%的序列含有IRES(Internal ribosome entry site,内部核糖体进入位点)位点,而作为对照的全部mRNA的序列中仅有32%,两者间亦存在显著差异(见图 5b右,p值< 0.001,Wilcoxon Signed Rank Test)。这一结果暗示精子发生过程中的部分circRNA有潜在的蛋白编码功能。对相应circRNA进行更为深入的实验和功能验证或许有可能为深入理解精子发生的分子调控机制开辟新的视角。
2.4.3 circRNA与RNA结合蛋白(RBP)的互作分析circRNA可与RBP结合从而影响特定的生物过程。Du等发现circ-FOXO3与CDK2蛋白结合可抑制细胞周期进程[32],Abdelmohsen等发现circ-PABPN1可竞争性结合HuR并进而抑制HuR与PABPN1的mRNA结合,从而降低PABPN1翻译效率[33]。NF90/NF110能和成熟的circRNA直接结合形成circRNA-蛋白复合体(circRNP),并在抗病毒过程中发挥重要的免疫功能[34]。小鼠精子发生过程中的circRNA是否也有结合RBP的潜力?为了回答这个问题,利用公共数据库中的RBP数据库POSTAR2对精子发生过程中的circRNA进行了系统分析。结果发现,共有10 517个circRNA具有RBP结合位点,其中除了约3 547个circRNA只有1个RBP结合位点外,其余circRNA都有2个以上的潜在RBP结合位点(见图 5c),甚至部分circRNA有10个以上的潜在RBP结合位点(见图 5c),如chr19:5800494|5800738含有31个,chr17:39845133|39845215含有30个。另外,除了一个circRNA可以结合多个RBP,一个RBP也可能被多个circRNA所吸附。比如我们发现总共有558个circRNA含有MSI2蛋白结合位点,而MSI2蛋白被报导在精子发生过程中起重要作用[35]。上述结果强烈暗示,circRNA可能通过“RBP海绵”的作用来调节细胞中相应RBP的效应浓度。circRNA-RBP互作在精子发生中的作用很值得后续深入研究。
3 讨论circRNA是一种特殊形式的内源性RNA,其闭环结构使之可能逃脱细胞内核酸外切酶的作用,并可能通过多种方式发挥其生物学功能。circRNA的上游生成机制虽已在人类细胞中进行了分析和部分功能证明,但小鼠精子发生过程中大量产生的circRNA是否通过类似的机制生成仍不清楚。通过系统的生物信息学分析发现各种类型的反向重复序列(SINE/L1、SINE/B2、SINE/B4、ERVL-MaLR和ERVK)在小鼠精子发生过程中产生的circRNA侧翼内含子中均有富集(见图 4b、4c),提示它们也可能具有类似人circRNA侧翼内含子中Alu序列促进circRNA生成的功能。本研究结果拓展了外显子型circRNA生成机制的顺式作用元件类别,为理解circRNA生成的分子调控机制提供了新的线索,但具体的作用机制还需进一步的实验验证(如在circRNA表达载体中引入这些不同类别的重复序列)。
目前尚未有circRNA在小鼠精子发生中起作用的报道。虽然我们发现了精子发生过程中有大量的circRNA产生,并初步推测了其形成机制,但它们中哪些circRNA对精子发生有调控作用,以何种方式起作用仍不清楚。功能验证需要寻找有潜能的候选circRNA,而本研究预测分析发现的部分circRNA的miRNA海绵功能、翻译模板潜能预测和RBP分子海绵等可为筛选功能性circRNA提供有价值的参考线索。虽然生信分析不能直接证明这些circRNA在精子发生中的作用,但其分析结果暗示部分circRNA有可能通过多种方式来参与精子发生的调节,后续的研究可在此基础上筛选候选circRNA,通过设计反向引物并结合定量PCR(qRT-PCR)验证相应circRNA的存在,继而构建circRNA过表达载体并注射小鼠睾丸,使得相应细胞中过表达该circRNA;或者在小鼠中通过基因编辑删除配对的反向重复序列来下调circRNA的产生,从而验证其是否具有调控精子发生表型的功能。
| [1] |
NOLASCO S, BELLIDO J, GONCALVES J, et al. The expression of tubulin cofactor A (TBCA) is regulated by a noncoding antisense TBCA RNA during testis maturation[J]. PLoS One, 2012, 7(8): e42536. DOI:10.1371/journal.pone.0042536 (  0) 0) |
| [2] |
VICENS Q, WESTHOF E. Biogenesis of circular RNAs[J]. Cell, 2014, 159(1): 13-14. DOI:10.1016/j.cell.2014.09.005 (  0) 0) |
| [3] |
ZHANG X O, WANG H B, ZHANG Y, et al. Complementary sequence-mediated exon circularization[J]. Cell, 2014, 159(1): 134-147. DOI:10.1016/j.cell.2014.09.001 (  0) 0) |
| [4] |
ASHWAL-FLUSS R, MEYER M, PAMUDURTI N R, et al. circRNA biogenesis competes with pre-mRNA splicing[J]. Molecular Cell, 2014, 56(1): 55-66. DOI:10.1016/j.molcel.2014.08.019 (  0) 0) |
| [5] |
MEMCZAK S, JENS M, ELEFSINIOTI A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333-338. DOI:10.1038/nature11928 (  0) 0) |
| [6] |
YANG Y, FAN X, MAO M, et al. Extensive translation of circular RNAs driven by N(6)-methyladenosine[J]. Cell Research, 2017, 27(5): 626-641. DOI:10.1038/cr.2017.31 (  0) 0) |
| [7] |
CHI M N, AURIOL J, JEGOU B, et al. The RNA-binding protein ELAVL1/HuR is essential for mouse spermatogenesis, acting both at meiotic and postmeiotic stages[J]. Molecular Biology Cell, 2011, 22(16): 2875-2885. DOI:10.1091/mbc.E11-03-0212 (  0) 0) |
| [8] |
ZHANG Y, ZHANG X O, CHEN T, et al. Circular intronic long noncoding RNAs[J]. Molecular Cell, 2013, 51(6): 792-806. DOI:10.1016/j.molcel.2013.08.017 (  0) 0) |
| [9] |
LIU Y, BOURGEOIS C F, PANG S, et al. The germ cell nuclear proteins hnRNP G-T and RBMY activate a testis-specific exon[J]. PLoS Genetics, 2009, 5(11): e1000707. DOI:10.1371/journal.pgen.1000707 (  0) 0) |
| [10] |
SADA A, SUZUKI A, SUZUKI H, et al. The RNA-binding protein NANOS2 is required to maintain murine spermatogonial stem cells[J]. Science, 2009, 325(5946): 1394-1398. DOI:10.1126/science.1172645 (  0) 0) |
| [11] |
NIU Z, GOODYEAR S M, RAO S, et al. MicroRNA-21 regulates the self-renewal of mouse spermatogonial stem cells[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(31): 12740-12745. DOI:10.1073/pnas.1109987108 (  0) 0) |
| [12] |
XIE R, LIN X, DU T, et al. Targeted disruption of mir-17-92 impairs mouse spermatogenesis by activating mTOR signaling pathway[J]. Medicine (Baltimore), 2016, 95(7): e2713. DOI:10.1097/MD.0000000000002713 (  0) 0) |
| [13] |
WU J, BAO J, WANG L, et al. MicroRNA-184 downregulates nuclear receptor corepressor 2 in mouse spermatogenesis[J]. BMC Developmental Biology, 2011, 11(1): 64. DOI:10.1186/1471-213X-11-64 (  0) 0) |
| [14] |
LIANG G, YANG Y, NIU G, et al. Genome-wide profiling of Sus scrofa circular RNAs across nine organs and three developmental stages[J]. DNA Research, 2017, 24(5): 523-535. DOI:10.1093/dnares/dsx022 (  0) 0) |
| [15] |
LIN X, HAN M, CHENG L, et al. Expression dynamics, relationships, and transcriptional regulations of diverse transcripts in mouse spermatogenic cells[J]. RNA Biology, 2016, 13(10): 1011-1024. DOI:10.1080/15476286.2016.1218588 (  0) 0) |
| [16] |
PATEL R K, JAIN M. NGS QC Toolkit: A toolkit for quality control of next generation sequencing data[J]. PLoS One, 2012, 7(2): e30619. DOI:10.1371/journal.pone.0030619 (  0) 0) |
| [17] |
GAO Y, WANG J, ZHAO F. CIRI: An efficient and unbiased algorithm for de novo circular RNA identification[J]. Genome Biology, 2015, 16(1): 4. DOI:10.1186/s13059-014-0571-3 (  0) 0) |
| [18] |
DONG R, MA X K, LI G W, et al. CIRCpedia v2: An updated database for comprehensive circular RNA annotation and expression comparison[J]. Genomics Proteomics Bioinformatics, 2018, 16(4): 226-233. DOI:10.1016/j.gpb.2018.08.001 (  0) 0) |
| [19] |
LI M, XIE X, ZHOU J, et al. Quantifying circular RNA expression from RNA-seq data using model-based framework[J]. Bioinformatics, 2017, 33(14): 2131-2139. DOI:10.1093/bioinformatics/btx129 (  0) 0) |
| [20] |
NUEDA M J, TARAZONA S, CONESA A. Next maSigPro: Updating maSigPro bioconductor package for RNA-seq time series[J]. Bioinformatics, 2014, 30(18): 2598-2602. DOI:10.1093/bioinformatics/btu333 (  0) 0) |
| [21] |
YU G, WANG L G, HAN Y, et al. ClusterProfiler: An R package for comparing biological themes among gene clusters[J]. OMICS, 2012, 16(5): 284-287. DOI:10.1089/omi.2011.0118 (  0) 0) |
| [22] |
TEMPEL S. Using and understanding RepeatMasker[J]. Methods Molecular Biology, 2012, 859: 29-51. DOI:10.1007/978-1-61779-603-6_2 (  0) 0) |
| [23] |
ZHENG Y, JI P, CHEN S, et al. Reconstruction of full-length circular RNAs enables isoform-level quantification[J]. Genome Medicine, 2019, 11(1): 2. DOI:10.1186/s13073-019-0614-1 (  0) 0) |
| [24] |
GAO Y, WANG J, ZHENG Y, et al. Comprehensive identification of internal structure and alternative splicing events in circular RNAs[J]. Nature Communications, 2016, 7: 12060. DOI:10.1038/ncomms12060 (  0) 0) |
| [25] |
ZHAO J, WU J, XU T, et al. IRESfinder: Identifying RNA internal ribosome entry site in eukaryotic cell using framed k-mer features[J]. Journal of Genetics and Genomics, 2018, 45(7): 403-406. DOI:10.1016/j.jgg.2018.07.006 (  0) 0) |
| [26] |
ZHU Y, XU G, YANG Y T, et al. POSTAR2: Deciphering the post-transcriptional regulatory logics[J]. Nucleic Acids Research, 2019, 47(D1): D203-D211. DOI:10.1093/nar/gky830 (  0) 0) |
| [27] |
KIMURA T, SUZUKI A, FUJITA Y, et al. Conditional loss of PTEN leads to testicular teratoma and enhances embryonic germ cell production[J]. Development, 2003, 130(8): 1691-1700. DOI:10.1242/dev.00392 (  0) 0) |
| [28] |
MOE-BEHRENS G H, KLINGER F G, ESKILD W, et al. Akt/PTEN signaling mediates estrogen-dependent proliferation of primordial germ cells in vitro[J]. Molecular Endocrinology, 2003, 17(12): 2630-2638. DOI:10.1210/me.2003-0006 (  0) 0) |
| [29] |
YU Z, RAABE T, HECHT N B. MicroRNA Mirn122a reduces expression of the posttranscriptionally regulated germ cell transition protein 2 (Tnp2) messenger RNA (mRNA) by mRNA cleavage[J]. Biology of Reproduction, 2005, 73(3): 427-433. DOI:10.1095/biolreprod.105.040998 (  0) 0) |
| [30] |
ENRIGHT A J, JOHN B, GAUL U, et al. MicroRNA targets in Drosophila[J]. Genome Biology, 2003, 5(1): R1. DOI:10.1186/gb-2003-5-1-r1 (  0) 0) |
| [31] |
BAI N, PENG E, QIU X, et al. circFBLIM1 act as a ceRNA to promote hepatocellular cancer progression by sponging miR-346[J]. Journal of Expermental & Clinical Cancer Research, 2018, 37(1): 172. DOI:10.1186/s13046-018-0838-8 (  0) 0) |
| [32] |
DU W W, YANG W, LIU E, et al. Foxo3 circular RNA retards cell cycle progression via forming ternary complexes with p21 and CDK2[J]. Nucleic Acids Research, 2016, 44(6): 2846-2858. DOI:10.1093/nar/gkw027 (  0) 0) |
| [33] |
ABDELMOHSEN K, PANDA A C, MUNK R, et al. Identification of HuR target circular RNAs uncovers suppression of PABPN1 translation by CircPABPN1[J]. RNA Biology, 2017, 14(3): 361-369. DOI:10.1080/15476286.2017.1279788 (  0) 0) |
| [34] |
LI X, LIU C X, XUE W, et al. Coordinated circRNA biogenesis and function with NF90/NF110 in viral infection[J]. Molecular Cell, 2017, 67(2): 214. DOI:10.1016/j.molcel.2017.05.023 (  0) 0) |
| [35] |
SUTHERLAND J M, FRASER B A, SOBINOFF A P, et al. Developmental expression of Musashi-1 and Musashi-2 RNA-binding proteins during spermatogenesis: Analysis of the deleterious effects of dysregulated expression[J]. Biology Reproduction, 2014, 90(5): 92. DOI:10.1095/biolreprod.113.115261 (  0) 0) |
 2020, Vol. 18
2020, Vol. 18


