2. 长春市朝阳区清和社区卫生服务中心,长春 130000;
3. 空军杭州特勤疗养中心 康复理疗科,杭州 310000;
4. 东北师范大学 信息科学与技术学院,长春 130000
2. Qinghe Community Health Service Center, Chaoyang District, Changchun 130000, China;
3. Department of Rehabilitation Physiotherapy, Hangzhou Special Service Recuperation Center of the Air Force, Hangzhou 310000, China;
4. Information and Technology School, Northeast Normal University, Changchun 130000, China
在高通量测序等生物技术的发展下,精准医疗模式已开始为临床诊疗提供新的方法与技术。精准医疗是指与患者分子生物病理学特征相匹配的个体化诊断和治疗策略[1]。在对癌症患者的个体化诊断和治疗过程中,精准医疗模式希望通过患者的基因型与基因表达等个体化数据为治疗方案提供指导。碍于有限的数据及治疗方法,这项工作仍充满争议与挑战[2-3]。
癌症药物治疗是目前治疗癌症的主要手段之一。通过将患者的个体数据结合系统生物学的研究成果,实现对不同抗癌药物治疗效果的精准预测是癌症精准医疗的发展趋势。基于功能性实验的一些实验方法存在耗时长(如类器官培养法、循环肿瘤细胞增殖法),成本昂贵和对实验设备要求高等局限性(如四甲基偶氮唑盐比色法)[4],而使用生物信息学中基于计算机算法的计算方法可以在仅获取部分患者基因组信息的前提下,为问题提供新的实验方法和研究思路。目前已有大量的研究成果证实了使用计算方法预测抗癌药物作用效果的可行性。
1 数据源目前研究的数据源多数是使用克隆培养的癌症细胞系(Cancer cell line)数据。癌症细胞系有着无限繁殖、易于测序的特点。除了在医学研究试验中有着广泛应用,其多组学概况和细胞增殖实验数据也被计算实验所青睐。在上世纪90年代,由美国国家癌症研究所(National Cancer Institute)发表的NCI-60汇总了多种抗癌药物对60种癌症细胞系的作用效果,并在多个计算方法实验中验证了使用癌症细胞系概况数据预测抗癌药物作用的可行性[5-7]。而近些年发表并不断更新的CCLE(Cancer Cell Line Encyclopedia,癌细胞系百科全书)[8]、GDSC(Genomics of Drug Sensitivity in Cancer,癌症药物敏感性基因组学)[9]和CTRP(Cancer Therapeutics Response Portal,癌症治疗反应门户网)[10]等为计算方法实验提供了更为丰富有效的素材。CCLE汇集了多个细胞系对24种抗癌药物的作用,而GDSC汇集了200余种药物对1 001个细胞系的作用效果,并且在两个数据源中也包含了癌症细胞系的基因表达、编码基因突变、甲基化和拷贝数(变异)等数据。CTRP也是以小分子药物和探针对细胞系影响为研究对象的数据库,相较于前两者,CTRP更加重视药物和靶标的关系而非细胞系本身的概况。
除细胞系数据以外,根据研究者研究问题的不同角度,有一些成熟的数据库可以应用在研究之中。在研究癌症问题常用的基因表达关系数据中,由美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)发布的GEO(Gene Expression Omnibus)数据库[11]常被使用。而在分析单点突变时,单核苷酸多态数据库dbSNP[12]也常用于作为背景数据库之一。研究之中常见的数据源还有生物通路如KEGG数据库[13]和蛋白质组学相关的,如包含蛋白质互作(Protein-protein interaction)关系的数据库STRING[14]、BioGRID[15]等。在研究非细胞系问题上,以临床病人样本为核心的TCGA(The Cancer Genome Atlas)数据库包含了多样本、多分型和多角度的相关数据,在本问题中也常被使用。
2 计算方法区别于临床研究中常用的数学统计方法(如表达相关性研究[16]),计算方法(Computational method)更加强调整体性, 有着明确的步骤与输入输出(见图 1)。计算方法对抗癌药物作用效果的预测目的是提供一个临床前研究模型或系统,一般通过使用患者(或细胞系)的基因型数据作为模型的输入,以该模型或系统根据患者(或细胞系)基因型所预测出来的单种或多种药物敏感性作为输出。根据模型所基于的不同算法类别,可分为三类。
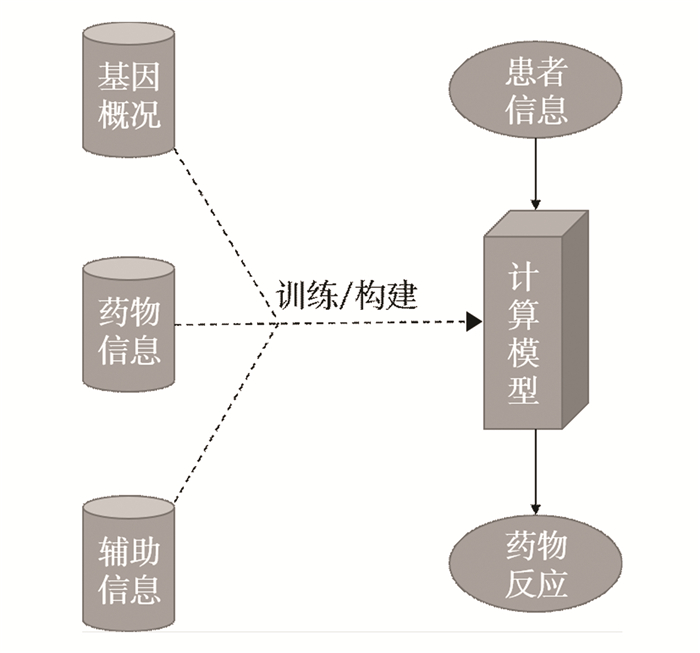
|
图 1 计算方法的研究流程 Figure 1 Research flow of calculation method |
在生物的生命活动机制中,网络这一概念广泛存在。如蛋白质互作网络、基因调控网络(Gene regulatory network),生物通路和共表达网络(Co-expression network)等。在研究癌症问题时,将生物数据以网络的方式建模可以更好地描述患者基因、蛋白间的关系,并应用适用于网络结构的算法进行分析从而实现预测对抗癌药物预测的功能。
在目前已有的研究成果中,多是将癌症细胞系与药物构建成异质性网络再结合多组学数据进行分析。Wei[17]等观察到具有相似基因型的细胞系和结构相似的药物间的作用关系存在高度关联,以此为依据构建了一个通过皮尔逊相关系数(Pearson correlation coefficient)来表示细胞系间关系的混合网络模型。Zhang[18]等根据药物和细胞系的敏感与抵抗关系构建异质性网络,再将网络关联上基因表达、药物化学结构相似度等信息,然后在网络上执行信息流算法(Information flow-based algorithm),求出原始数据中没有关联的药物与细胞系间的打分。Yang[19]等通过将基因组数据整合蛋白质互作网络和细胞系的功能注释,开发了一个反应网络模型来预测药物反应的工具。Stanfield[20]等提出了一个将基因根据蛋白质互作网络相连,再将癌症细胞系数据和基因相连建成异质性网络的方法。以上均是构建异质性网络直接预测的研究,也有通过网络分析细胞系数据得出辅助结论的研究成果。Choi[21]等提出了将癌症基因组学结合网络动力学的方法进行分析,通过使用吸引子景观分析(Attractor landscape analysis)方法,以p53的基因调控网络为例揭示了其中可能作为药物靶点的重要基因。Speyer[22]等分析了CTRP上368个小分子药物对810个细胞系上的影响,通过通路数据和与通路数据对应的差异依赖网络来发现细胞系对药物作用的特异性基因。
相较于其他算法,网络分析方法解释性相对更强,更符合目前对生物数据的认知。但是碍于构建网络的数据源并不完备、部分关系未得到检测且已知的偏向于已被大量研究的基因和疾病[23],由此会使某些潜在驱动基因得不到算法的重视从而影响对药物反应的预测结果。
2.2 机器学习方法机器学习是人工智能领域的重要方法,旨在让模型从已知的数据特征出发,通过学习得到规律以预测未知数据。在本问题的研究中,应用机器学习方法是主要的研究手段,创新点主要集中于提出新的算法、新的特征或降低预测所需的数据维度。
Riddick[6]等对NCI-60数据使用了随机森林(Random forest)算法,为机器学习方法在本问题上的应用做出了铺垫。Menden[24]等使用了基因组数据和药物的化学性质共同预测药物敏感性,使用的是人工神经网络(Artificial neural network,ANN)模型。Supahvilai[25]等提出一种基于推荐系统(Recommender system)思想的方法,根据药物和细胞系对潜在“药物基因组”空间的学习,预测未知细胞系的抗癌药物作用。Zhang[26]等在推荐系统的基础之上,采用混合内插加权协同过滤(Hybrid interpolation weighted collaborative filtering)的方法。Ruffalo[27]等使用了多任务学习的方法重构了药物作用网络,证实了多任务学习在对药物敏感性预测上的可行性。Wang[28]等使用了DiffRank、GSVA、PLAGE和Z-score四种方法基于通路的活性进行了无监督的学习,该工作减少了预测所需的特征。HUSSAIN[29]等尝试使用了Apache Spark算法并取得了良好的表现。Knowles[30]等基于贝叶斯非参数方法提出了一个多任务回归模型。Dhruba[31]等使用了迁移学习解决了CCLE和GDSC在数据源上的差异问题。CHEN[32]等为了融合问题中涉及到的多组学特征,提出了一种双层选择的方法。Kim[33]等在朴素贝叶斯(Naive Bayes)模型中使用了蛋白质组学的反相蛋白质矩阵作为特征,为多组学研究提供了参照。以上研究表明机器学习算法在本研究中已取得了一定的成果,但在对问题的定义、特征的选择等方面仍有一定的研究空间。
2.3 深度学习方法深度学习技术是通过增加人工神经网络算法中的网络层数,从而加强模型的学习能力。使用深度学习方法,可以让网络以原始数据作为训练样本自提特征以跳过传统的特征选择步骤。近些年深度学习技术在图像识别、自然语言处理等领域均取得了较大的突破。而在对抗癌药物作用的预测中,深度学习的各种网络结构在被研究者尝试使用并有了一部分的研究成果。Chiu[34]等使用了体细胞突变数据和基因表达数据,通过使用自编码器(Autoencoder)进行了预训练,再将预训练网络的中间层结果作为特征构建了预测网络模型。Chang[35]等将基因突变数据结合药物分子指纹数据使用了卷积神经网络(Convolutional neural network,CNN)进行预测。Su[36]等使用了深度非网络模型——深度森林(Deep forest)对CCLE和GDSC上的数据进行了预测。Xia[37]等为了缓解训练中的梯度消失尝试了深度残差网络(Deep residual network)。
因为不同工作在数据选择和测试算法上有所差异,所以无法直接评价算法间的优劣程度,但是从算法指标上看深度学习确实在本问题上有所突破,如Xia[37]等的工作在NCI-60数据集上将预测的均方误差降到了0.015 8;Chang[35]等在工作的比照实验中使用了SVM算法和随机森林算法但效果均不如深度学习。而对深度学习算法的研究是热点与前沿,近年常有大量新改进的和新提出的算法,其网络设计的基本原理也在被不断发现。所以虽然目前并没有太多研究成果,但未来本问题的研究在深度学习方法上将有很大的提升空间。
3 总结与展望在研究抗癌药物作用效果的问题上,相较于传统的基因检测和功能性实验的方法[38-40],使用计算方法有着时间短、成本低的优点。然而使用计算方法仍存在瓶颈。首先,细胞系不同于癌症患者身上的肿瘤细胞,缺少空间结构、微环境等重要影响因素。其次,癌症具有异质性,预测的药物作用结果只能通过患者细胞样本与细胞系的相似程度,通过计算模型预测患者对某种药物的敏感或抵抗,这显然有因误差而出现错误判断的可能。最后,一些计算模型虽然表现良好,但是其缺乏可解释性,研究者无法通过其算法的计算机理为解释癌症进行指导。
而在精准医疗与癌症诊疗结合的路上也并不平坦,因为缺乏产出和通过一些随机性试验结果[41]的验证,癌症精准医疗的意义曾受到质疑[2-3]。但随着越来越多有积极意义研究成果的发表,癌症精准医疗的理念得到了更多的侧面验证,虽然离它的彻底实现研究者们仍任重道远。
对基于计算方法视角的抗癌药物作用预测以及整个癌症精准医疗的发展有如下展望:
1) 尝试前沿计算机算法,与算法发展相辅相成。计算方法的核心是计算机算法,研究者通过尝试前沿的算法有提高模型表现的可能性。如深度学习是目前生物学信息学方向的应用热点之一,如上文所述,已经有研究成果证实深度学习可以在对抗癌药物作用效果预测上取得良好的效果。而随着研究人员对深度学习算法的不断创造与改进,在本问题上可以预见会有较大的提升空间。
2) 标准化实验数据,普及精准医疗概念。所参考的文献证实了可以直接使用计算方法分析标准化后的癌症临床数据。而能否获取更多可以用于计算分析的数据,取决于医疗从业人员是否有意识地采集有一定规范性、可以用于计算实验的数据。提高医疗从业人员对精准医疗概念的认识可以加快其发展进程。同样由数据科学家更新发布的数据库数据也可为计算方法提供更多的实验数据来训练更符合实际情况的计算模型。对本问题的研究从早期的NCI-60数据源发展到GDSC和CCLE等大型的数据库阶段,其模型的泛化能力已有了显著的提高。
3) 多角度分析癌症并设计实验。对抗癌药物作用的影响因素有很多,如点突变、染色体变异、基因表达量变化等。一般计算方法只选取其中一种或几种作为模型的输入特征,很少有实验能够全面综合多组学和多因素来训练模型。所以如何融合多角度数据训练出能多方面观测患者(或细胞系)基因型的模型是未来可以突破的关键点之一。但与此而来的数据量少、不全,特征权重平衡等问题也需要在实验设计阶段有足够的理论支持。
4) 消除临床实验与计算实验的隔阂。一直以来,临床实验和计算实验都不能很好地融合。临床实验一般倾向于数据来源和采样方法的独特性,而在分析过程中多使用数学统计方法,分析结果仅能揭示现象缺乏直接应用价值。计算实验强调模型的表现和特征选择的合理性,最终的实验结果有一定应用价值但因为缺乏解释性容易不被临床所接受。笔者认为,如何能在设计实验时将临床与计算两者结合是本问题乃至整个癌症精准医疗取得突破进展的关键点。如用计算方法将临床研究结果转化为临床前模型和用临床方法检验计算实验发现的生物标志物等,都值得研究人员尝试。
综上所述,计算方法有着一定的局限性,但在抗癌药物作用的预测问题上有着良好的表现和可以预期的提升空间。如何更好地使用计算方法是癌症精准医疗未来发展的重点之一。
| [1] |
杭渤, 束永前, 刘平, 等. 肿瘤的精准医疗:概念、技术和展望[J]. 科技导报, 2015, 33(15): 14-21. HANG Bo, SHU Yongqian, LIU Ping, et al. Precision cancer medicine:Concept, technology and perspectives[J]. Science & Technology Review, 2015, 33(15): 14-21. DOI:10.3981/j.issn.1000-7857.2015.15.001 (  0) 0) |
| [2] |
PRASAD V. Perspective:The precision-oncology illusion[J]. Nature, 2016, 537(7619): S63. DOI:10.1038/537S63a (  0) 0) |
| [3] |
TANNOCK I F, HICKMAN J A. Limits to personalized cancer medicine[J]. The New England Journal of Medicine, 2016, 375(13): 1289-1294. DOI:10.1056/NEJMsb1607705 (  0) 0) |
| [4] |
刘东岩, 叶开琴, 王宏志, 等. 基于功能性试验预测抗肿瘤药物敏感性研究进展[J]. 转化医学电子杂, 2017(01): 1-6. LIU Dongyan, YE Kaiqin, WANG Hongzhi, et al. Research progress on the prediction of sensitivity of anti-cancer agents based on functional assays[J]. E-Journal of Translational Medicine, 2017(01): 1-6. DOI:10.3969/j.issn.2095-6894.2017.01.001 (  0) 0) |
| [5] |
STAUNTON J E, SLONIM D K, COLLER H A, et al. Chemosensitivity prediction by transcriptional profiling[J]. Proceedings of National Academy of Sciences of the Unite States of America, 2001, 98(19): 10787-10792. DOI:10.1073/pnas.191368598 (  0) 0) |
| [6] |
RIDDICK G, SONG H, AHN S, et al. Predicting in vitro drug sensitivity using random forests[J]. Bioinformatics, 2011, 27(2): 220-224. DOI:10.1093/bioinformatics/btq628 (  0) 0) |
| [7] |
COHEN A L, SOLDI R, ZHANG H, et al. A pharmacogenomic method for individualized prediction of drug sensitivity[J]. Molecular Systems Biology, 2011, 7: 513. DOI:10.1038/msb.2011.47 (  0) 0) |
| [8] |
BARRETINA J, CAPONIGRO G, STRANSKY N, et al. The cancer cell line encyclopedia enables predictive modelling of anticancer drug sensitivity[J]. Nature, 2012, 483(7391): 603-607. DOI:10.1038/nature11003 (  0) 0) |
| [9] |
YANG W, SOARES J, GRENINGER P, et al. Genomics of drug sensitivity in cancer (GDSC): A resource for therapeutic biomarker discovery in cancer cells[J]. Nucleic Acids Research, 2013, 41(Database issue): D955-961. DOI:10.1016/s0959-8049(16)32839-8 (  0) 0) |
| [10] |
SEASHORELUDLOW B, REES M G, CHEAH J H, et al. Harnessing connectivity in a large-scale small-molecule sensitivity dataset[J]. Cancer Discovery, 2015, 5(11): 1210-1223. DOI:10.1158/2159-8290 (  0) 0) |
| [11] |
BARRETT T, WILHITE S E, LEDOUX P, et al. NCBI GEO: Archive for functional genomics data sets-update[J]. Nucleic Acids Research, 2013, 39(Database issue): 1005-1010. DOI:10.1093/nar/gks1193 (  0) 0) |
| [12] |
SHERRY S T, WARD M H, KHOLODOV M, et al. dbSNP: the NCBI database of genetic variation[J]. Nucleic Acids Research, 2001, 29(1): 308. DOI:10.1093/nar/29.1.308 (  0) 0) |
| [13] |
KANEHISA M, FURUMICHI M, TANABE M, et al. KEGG: New perspectives on genomes, pathways, diseases and drugs[J]. Nucleic Acids Research, 2017, 45(D1): D353-D361. DOI:10.1093/nar/gkw1092 (  0) 0) |
| [14] |
SZKLARCZYK D, GABLE A L, LYON D, et al. STRING v11: Protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Research, 2019, 47(D1): D607-D613. DOI:10.1093/nar/gky1131 (  0) 0) |
| [15] |
OUGHTRED R, CHATR-ARYAMONTRI A, BREITKREUTZ B J, et al. BioGRID: A resource for studying biological interactions in yeast[J]. Cold Spring Harb Protocols, 2016, 2016(1): pdb.top080754. DOI:10.1101/pdb.top080754 (  0) 0) |
| [16] |
WACHTERS F M, WONG L S, TIMENS W, et al. ERCC1, hRad51, and BRCA1 protein expression in relation to tumour response and survival of stage Ⅲ/Ⅳ NSCLC patients treated with chemotherapy[J]. Lung Cancer, 2005, 50(2): 211-219. DOI:10.1016/j.lungcan.2005.06.013 (  0) 0) |
| [17] |
WEI D, LIU C, ZHENG X, et al. Comprehensive anticancer drug response prediction based on a simple cell line-drug complex network model[J]. BMC Bioinformatics, 2019, 20(1): 44. DOI:10.1186/s12859-019-2608-9 (  0) 0) |
| [18] |
ZHANG F, WANG M, XI J, et al. A novel heterogeneous network-based method for drug response prediction in cancer cell lines[J]. Scientific Reports, 2018, 8(1): 3355. DOI:10.1038/s41598-018-21622-4 (  0) 0) |
| [19] |
YANG J, LI A, LI Y, et al. A novel approach for drug response prediction in cancer cell lines via network representation learning[J]. Bioinformatics, 2019, 35(9): 1527-1535. DOI:10.1093/bioinformatics/bty848 (  0) 0) |
| [20] |
STANFIELD Z, COŞKUN M, KOYUTVRK M. Drug response prediction as a link prediction problem[J]. Scientific Reports, 2017, 7: 40321. DOI:10.1038/srep40321 (  0) 0) |
| [21] |
CHOI M, SHI J, ZHU Y, et al. Network dynamics-based cancer panel stratification for systemic prediction of anticancer drug response[J]. Nature Communications, 2017, 8(1): 1940. DOI:10.1038/s41467-017-02160-5 (  0) 0) |
| [22] |
SPEYER G, MAHENDRA D, TRAN H J, et al. Differential pathway dependency discovery associated with drug response across cancer cell lines[J]. Pacific Symposium Biocomputing, 2017, 22: 497-508. DOI:10.1142/9789813207813_0046 (  0) 0) |
| [23] |
MENCHE J, SHARMA A, KITSAK M, et al. Disease networks. Uncovering disease-disease relationships through the incomplete interactome[J]. Science, 2015, 347(6224): 1257601. DOI:10.1126/science.1257601 (  0) 0) |
| [24] |
MENDEN M P, IORIO F, GARNETT M, et al. Machine learning prediction of cancer cell sensitivity to drugs based on genomic and chemical properties[J]. PLoS One, 2013, 8(4): e61318. DOI:10.1371/journal.pone.0061318 (  0) 0) |
| [25] |
SUPHAVILAI C, BERTRAND D, NAGARAJAN N. Predicting cancer drug response using a recommender system[J]. Bioinformatics, 2018, 34(22): 3907-3914. DOI:10.1093/bioinformatics/bty452 (  0) 0) |
| [26] |
ZHANG L, CHEN X, GUAN N N, et al. A hybrid interpolation weighted collaborative filtering method for anti-cancer drug response prediction[J]. Frontiers Pharmacology, 2018, 9(9): 1-11. DOI:10.3389/fphar.2018.01017 (  0) 0) |
| [27] |
RUFFALO M, STOJANOV P, PILLUTLA V K, et al. Reconstructing cancer drug response networks using multitask learning[J]. BMC Systems Biology, 2017, 11(1): 96. DOI:10.1186/s12918-017-0471-8 (  0) 0) |
| [28] |
WANG X, SUN Z, ZIMMERMANN M T, et al. Predict drug sensitivity of cancer cells with pathway activity inference[J]. BMC Medical Genomics, 2019, 12(Suppl 1): 15. DOI:10.1186/s12920-018-0449-4 (  0) 0) |
| [29] |
HUSSAIN S, FERZUND J, UL-HAQ R. Prediction of drug target sensitivity in cancer cell lines using apache spark[J]. Journal of Computational Biology, 2019, 26(8): 882-889. DOI:10.1089/cmb.2018.0102 (  0) 0) |
| [30] |
KNOWLES D A, BOUCHARD G, PLEVRITIS S. Sparse discriminative latent characteristics for predicting cancer drug sensitivity from genomic features[J]. PLoS Computional Biology, 2019, 15(5): e1006743. DOI:10.1371/journal.pcbi.1006743 (  0) 0) |
| [31] |
DHRUBA S R, RAHMAN R, MATLOCK K, et al. Application of transfer learning for cancer drug sensitivity prediction[J]. BMC Bioinformatics, 2018, 19(Suppl 17): 497. DOI:10.1186/s12859-018-2465-y (  0) 0) |
| [32] |
CHEN T H, SUN W. Prediction of cancer drug sensitivity using high-dimensional omic features[J]. Biostatistics, 2017, 18(1): 1-14. DOI:10.1093/biostatistics/kxw022 (  0) 0) |
| [33] |
KIM D C, WANG X, YANG C R, et al. A framework for personalized medicine: Prediction of drug sensitivity in cancer by proteomic profiling[J]. Proteome Science, 2012, 10(Suppl 1): S13. DOI:10.1186/1477-5956-10-s1-s13 (  0) 0) |
| [34] |
CHIU Y C, CHEN H H, ZHANG T, et al. Predicting drug response of tumors from integrated genomic profiles by deep neural networks[J]. BMC Medical Genomics, 2019, 12(Suppl 1): 18. DOI:10.1186/s12920-018-0460-9 (  0) 0) |
| [35] |
CHANG Y, PARK H, YANG H J, et al. Cancer drug response profile scan (CDRscan): A deep learning model that predicts drug effectiveness from cancer genomic signature[J]. Scientific Reports, 2018, 8(1): 8857. DOI:10.1038/s41598-018-27214-6 (  0) 0) |
| [36] |
XIA F, SHUKLA M, BRETTIN T, et al. Predicting tumor cell line response to drug pairs with deep learning[J]. BMC Bioinformatics, 2018, 19(Suppl 18): 486. DOI:10.1186/s12859-018-2509-3 (  0) 0) |
| [37] |
SU R, LIU X, WEI L, et al. Deep-Resp-Forest: A deep forest model to predict anti-cancer drug response[J]. Methods, 2019, 166: 91-102. DOI:10.1016/j.ymeth.2019.02.009 (  0) 0) |
| [38] |
IRISH J M, HOVLAND R, KRUTZIK P O, et al. Single cell profiling of potentiated phospho-protein networks in cancer cells[J]. Cell, 2005, 7(S2): 217-228. DOI:10.1016/j.cell.2004.06.028 (  0) 0) |
| [39] |
UGUREL S, SCHADENDORF D, PFÖHLER C, et al. In vitro drug sensitivity predicts response and survival after individualized sensitivity-directed chemotherapy in metastatic melanoma: A multicenter phase Ⅱ trial of the dermatologic cooperative oncology group[J]. Clinical Cancer Research An Official Journal of the American Association for Cancer Research, 2006, 12(18): 5454-63. DOI:10.1158/1078-0432.CCR-05-2763 (  0) 0) |
| [40] |
DAI H, DING H, MENG X W, et al. Constitutive BAK activation as a determinant of drug sensitivity in malignant lymphohematopoietic cells[J]. Genes & Development, 2015, 29(20): 2140-2152. DOI:10.1101/gad.267997.115 (  0) 0) |
| [41] |
LE T C, DELORD J P, GONÇALVES A, et al. Molecularly targeted therapy based on tumour molecular profiling versus conventional therapy for advanced cancer(SHIVA): A multicentre, open-label, proof-of-concept, randomised, controlled phase 2 trial[J]. Lancet Oncology, 2015, 16(13): 1324-1334. DOI:10.1016/S1470-2045(15)00188-6 (  0) 0) |
 2020, Vol. 18
2020, Vol. 18


