2. 电子科技大学 信息生物学研究中心,成都 611731
2. Center for Informational Biology, University of Electronic Science and Technology of China, Chengdu 611731, China
合成生物学最初在1980年提出来的,用来表述基因重组技术,之后2000年在美国化学年会上重新提出来,涉及生物、化学、物理、工程,计算机和信息技术多个领域。随着计算机、生物信息, 基因合成与基因测序等技术的快速发展,合成生物学结合各领域成果得到了突飞猛进地发展[1-3, 6-7]。全基因乃至染色体人工合成等原来只能停留人脑中的想法一个一个被实现[4-5, 8-10]。目前合成生物学已成为科学界大力研究发展的学科,被广泛应用于各个领域,比如癌症治疗、环境治理等,其思路也被广泛借鉴与应用在各种完全不同的领域中(例如系统科学与自动通讯技术),有望成为21世纪引领生命科学领域乃至整个科学领域的重要学科[11-12]。
合成生物学的实验流程包含设计、构建和测试三个步骤。目前,基因回路的设计还是主要以手动设计为主,但随着合成生物学的发展,其应用范围也飞速的拓展,对基因回路的需求规模不断地扩大,这种“订制”式的设计方式不再能满足合成生物学研究人员的需求,亟需一款类似于机械工程师或电工程师使用的自动计算机辅助设计系统(CAD或者EAD)的基因线路设计软件工具。但是机械或者电子的研究对象为非生命体,严格按照力学和电学原理,而生命体的复杂性远远高于非生命体,目前对于生命体的认识还远远不如非生命体那么清晰。比如基因回路的设计到底应该遵循什么原理?可以用那些合适的数学形式描述生物呢?这些问题使得自动计算机辅助基因设计系统发展缓慢[13]。
基因回路的设计目前常用的线路设计工具有:Cello[14]、j5[15],GenoCAD[16]和iBioSim[17]等。Cello是一个基于Verilog语言的基因线路设计环境,根据用户指定的功能提供所需的核酸序列,并预测性能的好坏,目前主要用于大肠杆菌。其他几种工具:j5、GenoCAD和iBioSim,都需要用户提供回路相关信息,也不能预测其性能。可见目前的基因回路设计工具的功能还非常有限,远远不及CAD或者EAD。其主要原因之一就是目前的标准生物部件(Biobricks)还非常有限。在这种背景下,iGEM诞生了。
国际遗传工程机器竞赛(International Genetically Engineered Machine Competition, iGEM)由麻省理工学院于2003年创办,2005年发展成为国际性学术竞赛,是合成生物学领域的最高国际性学术竞赛。iGEM的目的,就是希望通过学术竞赛的模式,实现生物学的系统化、工程化,促进生物工具的开源化、透明化发展,帮助构建一个可以安全、有效地应用生物技术的工程体系,推动合成生物学及相关领域的科学发展。设计、建模和模拟是合成生物学必不可少的环节,iGEM针对此问题开设软件设计比赛项目,希望结合计算机技术,简化或优化合成生物学方面的活动或实验,使得合成生物学研究者达到更高的效率。
iGEM举办至今,每年会诞生不少优秀的合成生物学基因回路设计软件,虽然由于种种原因,比如iGEM的参赛队伍大部分是本科生,没有很好的延续性,很多项目并没有被很好的维护,这些软件实际应用到科学研究中的案例并不多,但是这些项目设计中好的思路和想法可以被运用到更系统的基因设计软件中。所以在这里,详细总结了往届优秀的软件设计参赛项目,试图从中学习他人的独特思路,总结现今科研过程中亟待解决的问题,找出其发展趋势。发现近几年的iGEM软件设计参赛项目主要有以下四个设计思路。
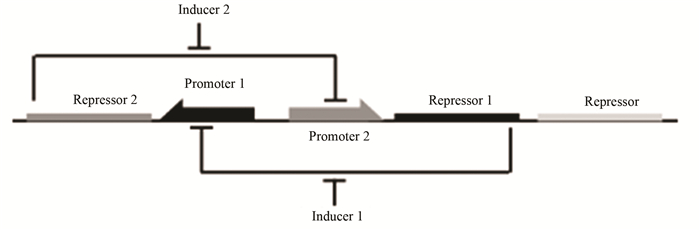
1 辅助设计辅助设计类型的项目着眼于改进基因回路的设计与测试,使生物工程更具预测性(见表 1)。合成生物学家常采用重建的方法,即通过构建具有相似功能的遗传回路,来深入了解自然回路的潜在机制。例如Gardner等[18]在2000年报道了一种拨动开关的设计。这种设计可以用作“框架”,或用作用户使用不同生物部件设计拨动开关的指南(见图 1)。2014年SYSU-Software(http://2014.igem.org/Team:SYSU-Software)开发了一款基于框架的遗传回路设计软件FLAME。当选择理想的框架时,FLAME会提供几种解决方案,每种解决方案的机制和效率都不相同。根据每个解决方案的性能,用户可以选择其中一个微调电路的细节,并通过软件仿真功能模拟其性能。
| 表 1 辅助设计类软件对比 Table 1 Comparison of softwares for auxiliary design |
SYSU-Software通过软件构建了一个IPTG控制GFP的表达的基因电路,从而控制荧光强度的简单电路,并且通过湿实验验证了其模拟的结果。这一设计是令人兴奋的,尤其是对于合成生物学家来说,软件可以在短时间内设计出想要的遗传回路。对于初学者来说他们不需要自己构建框架只需向框架中添加生物砖即可。这极大提高了合成生物学家的工作效率。但对于有较深造诣的合成生物学家来说,他们想要的可能更多是新的未发掘的框架,不同的遗传回路组成方式,这一点是这个软件可以加以改进的地方。
到目前为止,相当数量的合成生物砖(Biobricks)和装置已被表征和标准化。于是,一种基于标准化生物砖数据库的辅助设计方法出现了。2014-SJTU-Software(http://2014.igem.org/Team:SJTU-Software)创造了以生物砖为核心的软件Easy BBK,他们在iGEM官方提供的数据源的基础上搭建了新的生物砖数据库,还加入了生物砖更多信息以衡量每一个生物砖在实验中的可靠程度,并通过打分评估生物砖的优良程度。用户可通过搜索引擎获得所需并且已按优良排序的生物砖,并组成所需遗传回路。通过软件让用户了解每一块生物砖的性能,快速选择遗传回路所需的每一部分,不必通过查阅大量文献或是实验了解每一个生物砖,极大的提高了用户设计的效率。但是该软件缺乏模拟仿真功能,对于设计出来的遗传回路,用户不知道其性能究竟如何,不得不采用实验验证。而且由该软件推荐的若干优质的生物砖组成的遗传回路也未必能在实验中有好的表现,低分的生物砖在特定的遗传回路中可能发挥着重要的作用。
SYSU-Software同样注意到了遗传回路中生物砖的重要性。他们在2017年开发了一款名为S-Din的软件(http://2017.igem.org/Team:SYSU-Software,见图 2),在S-Din的数据库中建立并存储关键字,项目和零件数据之间的网络分析关系。用户搜索关键字时S-Din将提供有关生物砖、零件数据及项目。当用户在搜索产生灵感时可立即在平台中设计,通过自由组合不同的生物砖来形成全新的遗传回路,并在数学上模拟遗传回路的动态表现。设计完成后可将其转化为质粒以便合成。

|
图 2 S-Din搜索结果页面 Figure 2 S-Din webpage of search engine results (http://2017.igem.org/wiki/images/3/33/T--SYSU-Software--project-wetlab.png) |
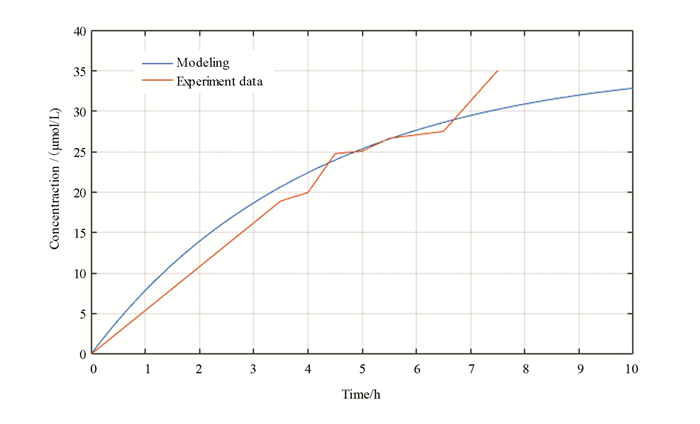
为了验证该软件,他们在S-Din中搜索“UV detection”,找到了之前相关项目——2012年ETH_Zurich的参赛项目(http://2012.igem.org/Team:ETH_Zurich),并提供了项目相关信息。根据S-Din的结果,删除了ETH_Zurich设备的一些额外部件后在软件中运行数学模型并通过湿实验验证了其设计结果(见图 3)。这表示软件的仿真性能很好。相较于SJTU-Software来说SYSU-Software给出了更多生物砖的信息并且能为设计模拟仿真,用户能很快了解所设计的遗传回路的大致性能。对于合成生物学家来说这是一个不错的喜讯。
|
图 3 模型验证结果 Figure 3 Results of model verification (http://2017.igem.org/wiki/images/e/ed/T--SYSU-Software--project_simulation-result.png) |
受到自然进化规律的启发,2016-SYSU-Software开发了一款名为CRAFT的软件(http://2016.igem.org/Team:SYSU-Software)。用户可以输入多个目标并设计限制条件来模拟自然选择条件。CRAFT可以枚举所有可能的解决方案,模拟生物变化,制造某种在自然环境下最适合保存的合成生物学系统。CRAFT将在后端自动生成调节元件。基于这些限制,每一个解决方案系统都包含一系列由CRAFT推荐的表达元件,用户可以通过操作拖动条来改变元件的强度,并且软件将估算用适当的元件替换以前的元件的效果。
这一设计为其他iGEM软件设计者提供了一个全新的想法,模拟自然选择,生成的解决方案更可靠,更容易让人接受。这种方法不仅新颖而且成为软件辅助设计的另一突破口,也能给其他iGEMers不少启发。但通过人为模拟自然环境得到的遗传设计是否能适应善变的环境发生有效的变异仍是未知的,值得探索。
辅助设计类软件因其对实验效率及方向指导的高贡献性受到科学家的青睐,在科研中也常被参考使用,各种软件侧重方向的不同也提高了对不同研究的问题的适应性。但由于目前数据仍不够丰富,且软件仿真并没有足够的可靠性和适应各类问题的能力,这类软件的表现与预期效果相去甚远,在科研中无法满足科研人员的要求,因此使用率及关注度提高困难。
目前辅助合成生物学家设计遗传回路的软件越来越多,方式各不相同,但在复杂、易变的生物环境中仍显得不够成熟。创造一款更加智能的软件,可以在用户搜索相关设计的同时不断自主学习,智能的产生一种新的人们未发掘的遗传回路也许是下一阶段要努力去实现的。
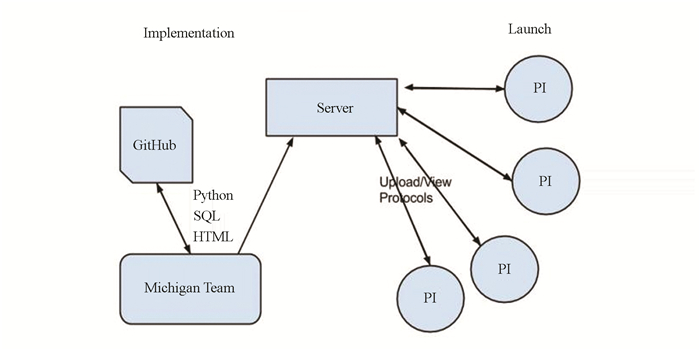
2 资料整合共享整合共享类型项目旨在通过构建或整合相关数据库和搜索引擎,以期提高iGEM和合成生物学现有成果的透明度和可重复性(见表 2)。有相当一部分iGEM软件队在他们设计的软件功能中包含了对已有成果的整合以及共享功能。通过构建相关数据库和搜索引擎,以提高iGEM和合成生物学现有成果的透明度和可重复性。合成生物学实验可重复性低是一个当下合成生物学领域遇到的令人头痛的问题。为了解决因实验方案表述不清,步骤缺失等因素而造成的实验不可重复等问题,2014年密歇根大学iGEM软件队Michigan_Software构建了一个用于清晰地记录并储存实验方案的数据库(http://2014.igem.org/Team:Michigan_Software,见图 4),供科研人员下载并查看,使用此数据库的科研人员都可以上传数据并维护自己的实验方案,其他人可以复制并生成自己的实验方案版本。2017年,Michigan_Software进一步开发了一个工具,可以调用现有的保存实验方案的数据库protocal.io的API,来导入protocal.io的实验方案,进一步加强了在这方面的资源整合程度。这一项工作也使得他们在2017年iGEM比赛中拿到了金牌。
| 表 2 资料整合共享类软件对比 Table 2 Comparison of softwares for data sharing |
|
图 4 软件设计架构 Figure 4 Software design architecture (http://2014.igem.org/wiki/images/2/2d/Concept_map.png) |
与之类似的还有很多项目,如USTC-Software 2014年的项目(http://2014.igem.org/Team:USTC-Software)。在他们的项目的数据共享部分,允许使用者把自己设计的生物部件上传到主数据库,供他人使用。这一类的设计大同小异,主要功能是允许用户上传自己的数据到数据库中,经由数据库整合后供所有人使用。但是这一类设计最大的问题在于初期数据量小的时候,并不能很好地发挥它的作用。
除此以外,还有另一类的设计不需要用户上传数据,而是整合网络上已有的数据,来提高现有项目的透明度和利用率。如SJTU-Software 2014年的项目(http://2014.igem.org/Team:SJTU-Software)就建立了一个整合现有生物砖的数据库,其数据主要来源于iGEM官网上保存的每年注册的部件的Registry。基于这个数据库,用户可以搜索高质量的生物砖,或比较生物砖的质量。同时,用户也可根据一定的标准去上传自己的生物砖。类似的,还有SJTU-Software 2016年的项目IMAP,整合并优化了iGEM官网上的信息,并放在他们的软件中,供iGEMer浏览。通过整合这些资料,使得iGEMer可以更好地了解iGEM和使用iGEM网站上的信息。
3 合作交流合作交流类项目则旨在加强iGEM团队和合成生物学家间的交流合作,以提高工作效率和项目效果(见表 3)。有一点不能忽略的是,越来越多的队伍注意到,用户之间的交流与合作可以显著的提高工作效率和产品效果,所以很多队伍在他们设计的软件中添加了社区功能,用户可以在社区中发布自己的设计,以寻求他人的改进意见;或者是在社区中寻求帮助或合作,以求解决当下研究中遇到的障碍。
| 表 3 合作交流类软件对比 Table 3 Comparison of softwares for cooperation and communication |
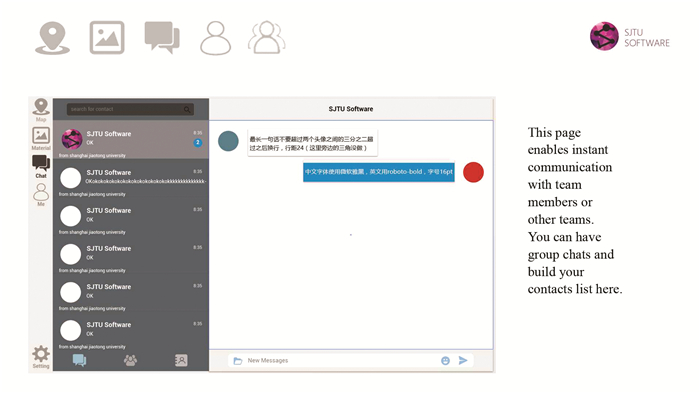
SJTU-Software 2016年的项目就实现了交流的功能。每个团队都有自己的小组,每个成员都有自己的主页。在团队页面中,每个团队成员的联系信息将被显示以方便沟通,而且可以在软件中直接发送消息(见图 5)。Michigan软件队在2017年(http://2017.igem.org/Team:Michigan_Software)推出的protocat4.0中提出的群组账号、收藏夹以及聊天功能,这些提供了一个可供合作团队进行交流分享的平台。但其功能还是局限于普通聊天软件所能提供的功能,没能在辅助iGEM设计上更进一步。这一点上,其他队伍提供了更好的思路和设计。USTC在2017年推出的Biohub2.0中(http://2017.igem.org/Team:USTC-Software)给出的功能插件系统,除了平台自身提供的一些功能插件以外,用户可以上传自己设计的功能插件在社区中共享。这样,用户不仅可以享受到更多的功能,还可以在共享中发现问题和改善插件。还有SYSU在2015推出的CORE(http://2015.igem.org/Team:SYSU-Software)中提出的CORE bank和CORE design功能。用户可以将自己的设计上传到CORE bank当中,还可以通过CORE design功能对CORE bank中已有的设计提出意见,或者进行改进升级,然后再次上传到CORE bank中覆盖原有的旧版本。与此同时,CORE还提供了用户评分系统(见图 6),来甄别相同功能的不同设计的优劣。但用户评分系统如今还并不完善,存在诸多问题。如果用户数目不够多,用户评分的可信度就会大大降低;而且没有一个统一的评价标准,仅仅凭借用户的经验进行评分也会对评价产生偏差。这点上,USTC在2015的BioBLESS(http://2015.igem.org/Team:USTC-Software)中提出了另一种思路。就是在用户评分的同时,系统辅助评分。在评价基因电路的功能时,系统会根据多个标准对电路进行综合评价,最后根据基因电路在各个标准上的表现情况进行最后评分。虽然这个评分系统目前只能针对软件自身生成的基因电路,但它提供了一种系统提供统一标准进行评分的思路。或许未来可以将这种思路和用户评分系统相结合,在注重客户的个人体验的同时,也增加统一的标准,这样的评分就变得更加科学也不失人性化。
|
图 5 社区交流页面 Figure 5 Webpage for communication (http://2016.igem.org/wiki/images/6/62/SJTUsoftware-tutorial12.jpg) |
|
图 6 Biohub的用户评价系统 Figure 6 User rating system of Biohub (http://2017.igem.org/wiki/images/a/a6/Forum-pop.png) |
数据分析类的项目主要是通过设计算法来解析和格式化生物数据,以便更好地使用。从历年的项目中我们看到,基因序列分析、挖掘,也是合成生物学中相当重要的且极具分析价值的研究方向。对基因序列这类生物数据的巧妙处理,可以让它服务于记录、追踪整个合成生物学领域的数据变化;同时,这样的工作也有可能对其他领域的发展提供新的思考方向。2014年Vanderbilt_Software制作了软件Darwin(http://2014.igem.org/Team:Vanderbilt_Sof-ware),可以系统的追踪基因序列细微变化,并记录到数据库。每次追踪到的新改变又通过算法更新数据库,从而生成更高效和安全的跟踪系统。这种设计将在未来避免合成生物学家陷入令人困扰的逻辑复杂性中。2016年,UESTC-Software(http://2016.igem.org/Team:UESTC-software)提出基于DNA的文件编辑技术,首次将生物信息转化成计算机信息。他们以0、1两个数字的不同组合定义DNA的四种碱基,使一段DNA序列成为一长串计算机二进制代码来储存信息。DNA作为信息存储介质,具有高容量(每克DNA相当于重量超过151公斤的14千张50 GB蓝光光盘或233 x 3 TB硬盘),低维护,自我复制,在数千年后仍然可读等优点。这个项目的提出是合成生物学在其他领域的开创性尝试,也是其他领域发展的新方向。
5 总结分析了近几年的iGEM软件队项目,总结出iGEM的软件设计项目主要以辅助设计、资料整合、合作交流、数据分析为设计方向,帮助合成生物学家更合理的设计回路、更高效的寻找资源、更便捷的交流探讨。也有一些队伍着眼于合成生物学某一方面的问题,提出优化的方案并设计出工具。无论角度如何,他们的工作都践行着iGEM的目标,促进了生物工具的开源化、透明化发展,推动着合成生物学及相关领域的发展和普及。我们希望本篇综述通过总结以往项目的设计思路和发展趋势,能为今后参加软件队的iGEMer提供参考的构思想法及立题方向;或者在此基础上延伸思考,激发出更好的创新与灵感,为合成生物学的发展做出贡献。
| [1] |
BENNER S A, SISMOUR A M. Synthetic biology[J]. Nature Reviews Genetics, 2005, 6(7): 533-543. DOI:10.1038/nrg1637 (  0) 0) |
| [2] |
CAMERON D E, BASHOR C J, COLLINS J J. A brief history of synthetic biology[J]. Nature Reviews Microbiology, 2014, 12(5): 381-390. DOI:10.1038/nrmicro3239 (  0) 0) |
| [3] |
KHALIL A S, COLLINS J J. Synthetic biology: Applications come of age[J]. Nature Reviews Genetics, 2010, 11(5): 367-379. DOI:10.1038/nrg2775 (  0) 0) |
| [4] |
DUAN Z, ANDRONESCU M, SCHUTZ K, et al. A three-dimensional model of the yeast genome[J]. Nature, 2010, 465(7296): 363-367. DOI:10.1038/nature08973 (  0) 0) |
| [5] |
MERCY G, MOZZICONACCI J, SCOLARI V F, et al. 3D organization of synthetic and scrambled chromosomes[J]. Science, 2017, 355(6329): eaaf4597. DOI:10.1126/science.aaf4597 (  0) 0) |
| [6] |
NANDAGOPAL N, ELOWITZ M B. Synthetic biology: Integrated gene circuits[J]. Science, 2011, 333(6047): 1244-1248. DOI:10.1126/science.1207084 (  0) 0) |
| [7] |
PURNICK P E M, WEISS R. The second wave of synthetic biology: from modules to systems[J]. Nature Reviews Molecular Cell Biology, 2009, 10(6): 410-422. DOI:10.1038/nrm2698 (  0) 0) |
| [8] |
RICHARDSON S M, MITCHELL L A, STRACQUADANIO G, et al. Design of a synthetic yeast genome[J]. Science, 2017, 355(6329): 1040-1044. DOI:10.1126/science.aaf4557 (  0) 0) |
| [9] |
SHAO Y, LU N, WU Z, et al. Creating a functional single-chromosome yeast[J]. Nature, 2018, 560(7718): 331-335. DOI:10.1038/s41586-018-0382-x (  0) 0) |
| [10] |
ZHANG W, ZHAO G, LUO Z, et al. Engineering the ribosomal DNA in a megabase synthetic chromosome[J]. Science, 2017, 355(6329): eaaf3981. DOI:10.1126/science.aaf3981 (  0) 0) |
| [11] |
FRENCH K E. Harnessing synthetic biology for sustainable development[J]. Nature Sustainability, 2019, 2(4): 250-252. DOI:10.1038/s41893-019-0270-x (  0) 0) |
| [12] |
WU M R, JUSIAK B, LU T K. Engineering advanced cancer therapies with synthetic biology[J]. Nature Reviews Cancer, 2019, 19(4): 187-195. DOI:10.1038/s41568-019-0121-0 (  0) 0) |
| [13] |
NOWOGRODZKI A. Gene design goes automatic[J]. Nature, 2018, 564(7735): 291-292. DOI:10.1038/d41586-018-07662-w (  0) 0) |
| [14] |
NIELSEN A A K, DER B S, SHIN J, et al. Genetic circuit design automation[J]. Science, 2016, 352(6281): aac7341. DOI:10.1126/science.aac7341 (  0) 0) |
| [15] |
HILLSON N J, ROSENGARTEN R D, KEASLING J D. j5 DNA assembly design automation software[J]. ACS Synthetic Biology, 2012, 1(1): 14-21. DOI:10.1021/sb2000116 (  0) 0) |
| [16] |
CAI Y, WILSON M L, PECCOUD J. GenoCAD for iGEM: A grammatical approach to the design of standard-compliant constructs[J]. Nucleic Acids Research, 2010, 38(8): 2637-2644. DOI:10.1093/nar/gkq086 (  0) 0) |
| [17] |
MYERS C J, BARKER N, JONES K, et al. IBioSim: A tool for the analysis and design of genetic circuits[J]. Bioinformatics, 2009, 25(21): 2848-2849. DOI:10.1093/bioinformatics/btp457 (  0) 0) |
| [18] |
GARDNER T S, CANTOR C R, COLLINS J J. Construction of a genetic toggle switch in Escherichia coli[J]. Nature, 2000, 403(6767): 339-342. DOI:10.1038/35002131 (  0) 0) |
 2020, Vol. 18
2020, Vol. 18