异柠檬酸脱氢酶(Isocitrate dehydrogenases, IDH)在细胞内主要催化D-异柠檬酸(D-isocitrate)氧化脱羧成α-酮戊二酸(α-oxoglutarate)和CO2的反应。该酶是三羧酸循环中的关键酶,对其活性的调节将直接影响该酶生物学功能的发挥[1]。该酶具有NAD(+)与NADP(+)两种辅助因子,根据辅助因子的不同,IDH可分为NAD(+)依赖型的IDH(NAD-IDH, EC:1.1.1.41)和NADP(+)依赖型的IDH(NADP-IDH, EC:1.1.1.42) [1]。IDHs广泛分布于原核生物以及真核生物中[2-6]。在哺乳动物组织中存在3种IDHs同工酶,分别为胞质NADP(+)依赖型IDH(IDH1)、线粒体NADP(+)依赖型IDH(IDH2)以及线粒体NAD(+)依赖型IDH(IDH3),其中IDH3被认为在三羧酸循环异柠檬酸脱羧反应中起主要作用,IDH3活性受许多变构调节剂的调节[3]。
目前的研究表明,在各类肿瘤患者中IDH的突变率较高。IDH1与IDH2的突变会抑制宿主免疫应答并且诱导α-酮戊二酸(α-KG)转化为D型2-羟基戊二酸(D2HG),造成D2HG大量积累进而诱发胶质瘤的发生[4]。IDH3β作为E3泛素连接酶后期促进复合体(APC/C)的新型底物对癌细胞的大量增值具有较为重要的作用[7]。IDH3α在缺氧诱导因子1(HIF-1)介导的癌细胞代谢以及血管生成中发挥作用[24]。该研究还表明,IDH3α的异常表达会促进血管瘤的生长[24]。因此对IDH家族进行深入研究将为癌症等疾病的治疗、分子病理分析和预后提供良好的视角[8, 30]。IDH是目前国际上公认的研究蛋白质结构与功能关系、酶的催化与调节机制以及蛋白质分子进化机制的最好模型之一[9]。目前有关牛IDH的研究报道较少。文献报道表明,牛心的烟酰胺腺嘌呤二核苷酸依赖性异柠檬酸脱氢酶(NAD-IDH)是由subunit-3/4(α)、subunit1(β)和subunit2(γ)三种亚基构成的异四聚体, 其中各亚基占比分别为2:1:1[10]。后来的研究进一步证实哺乳动物中1/β和2/γ亚基是调节性的,而包含一半四聚体的3, 4 /α亚基是催化性的[11-12]。在前人研究的基础上,本研究采用生物信息学方法分析了牛线粒体NAD-依赖型IDH的理化性质、亲疏水性、跨膜区、信号肽、亚细胞定位、二级结构预测、三级结构预测、表面电荷分布以及辅助因子结合位点,从而为进一步研究IDH家族尤其是哺乳动物NAD(+)IDH提供了重要理论依据。
1 材料与方法 1.1 材料从Uniprot数据库中查询并获得已注册的牛NAD-IDH的三种亚基IDH3A_BOVIN(P41563)、IDH3B_BOVIN(O77784)以及IDH3G_BOVIN(Q58CP0)的蛋白序列。
1.2 方法利用生物信息学分析工具(见表 1)分析了牛NAD-IDH的理化性质、亲疏水性、跨膜区、信号肽、亚细胞定位、二级结构预测、三级结构预测,表面电荷分布以及辅助因子结合位点。
| 表 1 生物信息学分析工具 Table 1 Bioinformatic analysis tools |
牛NAD(+)IDH三种亚基分析结果表明,该蛋白残基总数为1 509个,原子总数为23 207个,分子总质量为165 kDa,总分子式为C7259H11670N2048O2144S86。其中占比最高的残基为Ala(9.7%),占比最低的残基为Trp(0.6%)。三种亚基中IDH3G_BOVIN和IDH3B_BOVIN两个亚基呈碱性,IDH3A_BOVIN一个亚基呈酸性且正负电荷残基仅相差一个,据此可以推断牛NAD(+)IDH为碱性蛋白质。不稳定系数分析结果表明,三种亚基均不稳定,据此推断牛NAD(+)IDH蛋白为不稳定蛋白质。亲水性分析结果表明,三种亚基均呈亲水性,据此推断牛NAD(+)IDH为亲水性蛋白质(见表 2)。
| 表 2 牛NAD(+)IDH理化性质分析 Table 2 Analysis of physical and chemical properties of NAD(+)IDH in bovin |
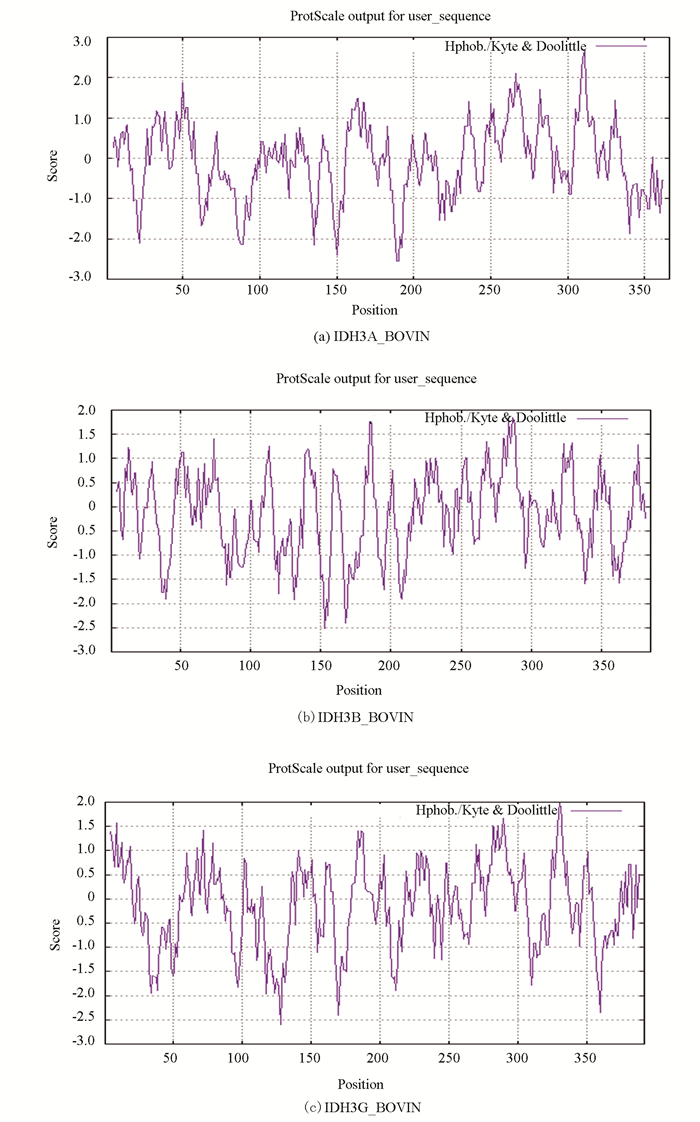
K-D预测算法分析的牛NAD(+)IDH亲水性/疏水性结果表明,IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三个亚基均为亲水性,据此推断牛NAD(+)IDH为亲水性蛋白质,这与理化性质分析结果一致。同时分析结果表明,牛NAD(+)IDH各亚基均无明显疏水区段,表明该蛋白很可能没有跨膜区,为细胞游离蛋白(见图 1,表 3)。
|
图 1 牛NAD(+)IDH的亲水性/疏水性分析(Kety and Doolittle算法) Figure 1 Hydrophlicity/hydrophobicity analysis of NAD(+)IDH in bovin (Kety and Doolittle algorithm) |
| 表 3 牛NAD(+)IDH的亲水性/疏水性分析 Table 3 Hydrophlicity/hydrophobicity analysis of NAD(+)IDH in bovin |
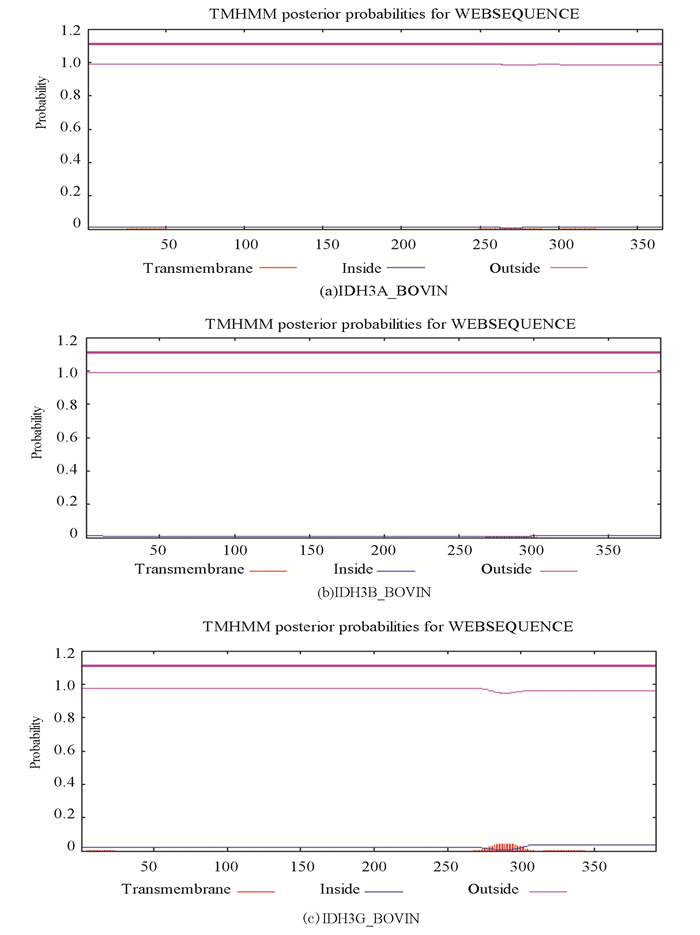
运用隐马尔可夫模型分别对三个亚基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN进行跨膜区预测分析。结果表明,该蛋白的三个亚基均无跨膜区,据此推断牛NAD(+)IDH不是跨膜蛋白,在三羧酸循环中以游离状态发挥作用,这与该蛋白亲水性/疏水性分析结果一致(见图 2)。
|
图 2 牛NAD(+)IDH跨膜区预测(隐马尔可夫模型) Figure 2 Transmembrane structure prediction of NAD(+)IDH in bovin (Hidden Markov Models) |
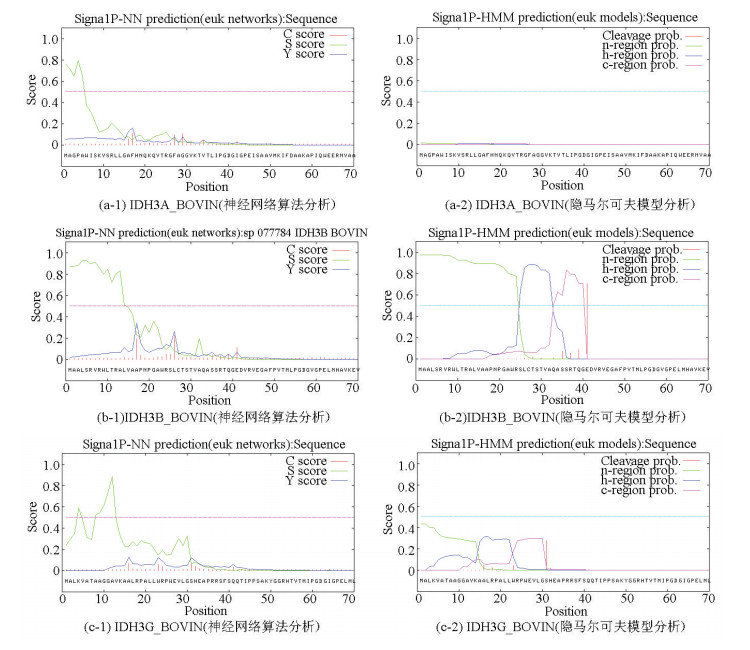
基于两种算法(神经网络算法和隐马尔可夫模型)分别对牛NAD(+)IDH三个亚基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN进行信号肽预测分析。神经网络算法分析结果中C-score表示信号肽酶切位点值;S-score表示信号肽值;Y-score表示综合得出的剪切位点分值。mean S-score作为判断蛋白质是否具有信号肽的标准,当mean S-score>0时可以认为是分泌蛋白,具有信号肽,mean S-score < 0时则认为是非分泌蛋白,没有信号肽。结合隐马尔可夫模型的预测结果,可以推断出IDH3A_BOVIN和IDH3G_BOVIN两个亚基没有信号肽,IDH3B_BOVIN亚基具有信号肽。根据文献报道,信号肽一般长度为5-30个氨基酸[15],因此排除本研究中隐马尔可夫模型对IDH3B_BOVIN亚基的预测结果(见图 3,表 4)。综上所述,可以推断牛NAD(+)IDH存在信号肽,并且该信号肽位于β亚基N端第1至第16位氨基酸,具体序列为MAALSRVRWLTRALVA。
|
图 3 牛NAD(+)IDH信号肽预测 Figure 3 Signal peptide prediction of NAD(+)IDH in bovin |
| 表 4 牛NAD(+)IDH信号肽预测 Table 4 Signal peptide prediction of NAD(+)IDH in bovin |
基于两种工具的亚细胞定位预测结果表明,IDH3A_BOVIN、IDH3B_BOVIN与IDH3G_BOVIN三个亚基均定位在线粒体中。与上述信号肽预测结果相吻合,据此推断牛DNA(+)IDH定位在线粒体中(见表 5)。
| 表 5 牛NAD(+)IDH亚细胞定位预测 Table 5 Prediction of subcellular localization of NAD(+)IDH in bovin |
蛋白质二级结构预测是联系一级结构与三级空间结构的桥梁和纽带[16-17]。本研究分别运用目前广泛使用的Jpred和SOPMA两种二级结构在线预测工具对牛NAD(+)IDH的三个亚基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN进行二级结构预测。
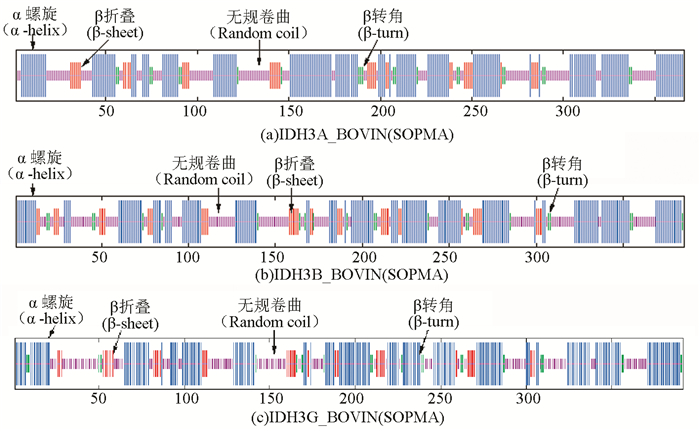
SOPMA预测结果表明,IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三个亚基的α螺旋占比分别为49.18%、47.01%和49.23%,β折叠占比分别为10.16%、11.43%和10.71%。Jpred和SOPMA预测结果比对表明,两种预测工具所预测的结果基本吻合,其中可靠性最强的α螺旋区在表 6中以深色底纹显示,可靠性最强的β折叠区以浅色底纹显示(见表 6,图 4)。
| 表 6 牛NAD(+)IDH二级结构预测(Jpred/SOPMA) Table 6 Secondary structure prediction of NAD(+)IDH in bovin (Jpred/SOPMA) |
|
图 4 牛NAD(+)IDH二级结构预测(SOPMA) Figure 4 Secondary structure prediction of NAD(+)IDH in bovin (SOPMA) |
空间结构预测对蛋白质结构与功能研究具有较为重要的作用[20]。
2.7.1 同源建模法预测三级结构采用SWISS-MODEL在线预测工具同源建模法分别预测了牛NAD(+)IDH三个亚基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN的三级结构。其中IDH3B_BOVIN亚基预测结果得到了以PDB ID 2d1c为模板的预测模型,相似度为34.03%,但该预测模型的预测结果表明IDH3B_BOVIN亚基为双肽链蛋白质(见图 5(b)),这一预测结果与文献报道的经实验验证的亚基大小不符合[10],由此推断同源建模法无法预测IDH3B_BOVIN亚基的三级结构。相比较而言,亚基IDH3A_BOVIN与IDH3G_BOVIN分别得到了以PDB ID 5grh和PDB ID 5grf为模板的高质量预测模型,相似度分别为97.34%和98.31%,全局模型质量评估得分分别为0.90和0.86,均明显高于其他预测模型。另外,QMEAN得分分别为-0.24和-0.55,比其他预测模型更接近0。进一步分析了预测模型与同源蛋白质三级结构的相似性散点分布图。结果表明,IDH3A_BOVIN与IDH3G_BOVIN模型(见图 6(a)和(b)中以红色五角星标记)Z得分均 < 0,显示该预测模型结构稳定,是最佳的理想结构(见图 6)。据此可以推断以PDB ID 5grh为模板的预测结果可以作为IDH3A_BOVIIN亚基的最可能的三级结构(见图 5(a)),以PDB ID 5grf为模板的预测结果可以作为IDH3G_BOVIIN亚基的最可能的三级结构(见图 5(d))。
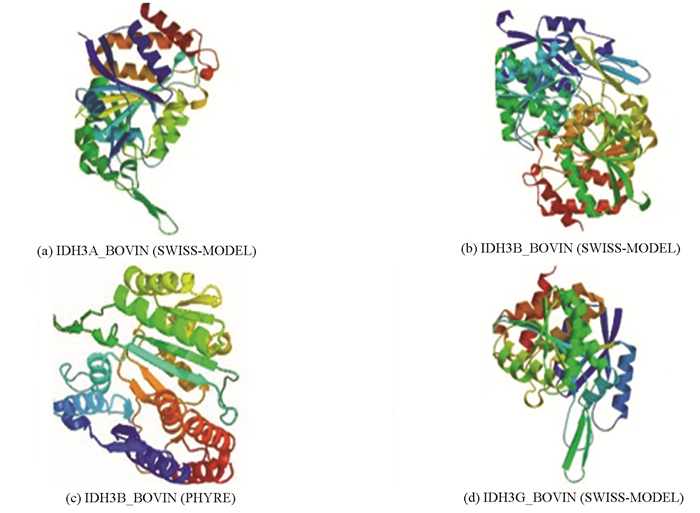
|
图 5 牛NAD(+)IDH的三级结构 Figure 5 Tertiairy structure of NAD(+)IDH predicted model |
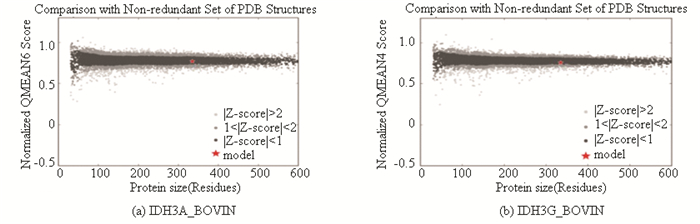
|
图 6 三级结构相似性散点分布图 Figure 6 Similarity scattergram of the predicted model and the homologous protein |
同源建模法预测结果表明,采用同源建模法无法精确预测IDH3B_BOVIN亚基的三级结构,因此,本研究采用折叠识别法进一步对IDH3B_BOVIN亚基的三级结构进行预测。预测结果显示有46个模型的准确度达到了90%以上,其中一个模板的折叠子c5grhB_是人异柠檬酸脱氢酶α与γ异二聚体中的γ亚基,该折叠子与IDH3B_BOVIN亚基序列匹配性最好,准确度达到了100%,相似性达到了54%,据此推断以该折叠子X射线晶体衍射结构折叠识别出来的三级结构可以作为IDH3B_BOVIIN亚基最可能的三级结构(见图 5(c))。
2.7.3 模型质量评估为了进一步验证预测模型的可靠性,将预测得到的模型PDB文件分别上传至SAVES v5.0平台上的Verify3D和PROCHECK进行在线评估。拉曼图中从白色到红色,颜色越深的区域表明Cα的二面角越合理。拉曼图分析结果表明IDH3A_BOVIN和IDH3G_BOVIN两个亚基的预测模型中90%以上的残基都位于红色最佳允许区域,IDH3B_BOVIN亚基中87.9%的氨基酸残基(269个)都位于红色最佳允许区域,10.1%的氨基酸残基(31个)位于正黄色额外允许区域(见图 7)。表明模型中所有的氨基酸残基均形成了一种合理的二面角,构成的蛋白质结构稳定。3D-1D得分结果表明,IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三个亚基中3D-1D值大于0.2的残基均占80%以上,且低质量部分不明显(见图 8),这表明模型质量合格。综上所述,可以推断出上述IDH3A_BOVIN、IDH3B_BOVIN与IDH3G_BOVIN三个亚基的三级结构预测结果稳定可靠。
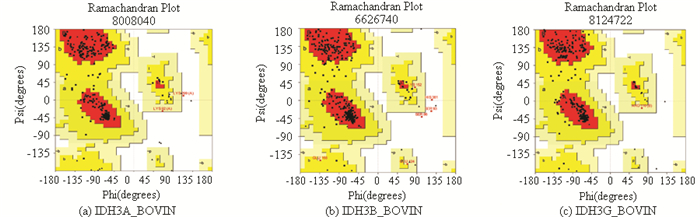
|
图 7 拉曼图 Figure 7 Ramachandran plot of the predicted model |

|
图 8 3D-1D得分散点图 Figure 8 3D-1D score scattergram of the model |
将牛NAD(+)IDH三个亚基IDH3A_BOVIN、IDH3B_BOVIN与IDH3G_BOVIN三级结构预测模型进行DSSP二级结构指认,并将指认结果与Jpred和SOPMA两种二级结构在线预测工具所预测的二级结构信息进行吻合度分析。结果表明,亚基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三级结构模型中指认的二级结构与Jpred和SOPMA所预测二级结构吻合度分别为92%、88%与95%,这表明亚基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三级结构模型中指认的二级结构分布与Jpred和SOPMA所预测的三个亚基二级结构结果是一致的,同时也进一步表明Jpred和SOPMA所预测的二级结构信息是可信的。
2.9 牛NAD(+)IDH蛋白质结构表面电荷分布及辅酶结合位点分析研究表明,高等动物中NAD(+)IDH不能单独发挥作用,需要各类辅助因子协同作用促进酶与异柠檬酸的结合并形成α-酮戊二酸,亚基IDH3B_BOVIN和IDH3G_BOVIN具有ADP结合位点,两者很可能与NAD(+)IDH蛋白活性调节有关。此外,IDH3A_BOVIN亚基为催化亚基,拥有一部分异柠檬酸结合位点[9, 12, 23]。将获得的IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三个亚基三级结构数据利用VMD软件进行分析,通过该软件中的APBS插件分别计算三个亚基的表面电荷分布。在分析的过程中,用蓝色表示所分析蛋白质带正电荷的区域,红色表示带负电荷的区域,灰色表示不带电荷的区域(见图 9)。分析结果表明,三个亚基三级结构正面均带有大量正电荷(见图 9(a),(c)和(e)),在β折叠和α螺旋形成的凹槽处尤为明显,图中以箭头所示,推断该处结构最有可能与异柠檬酸(带负电)或ADP(带负电)结合。与正面相比,三个亚基三级结构背面电荷分布均不明显,且正负电荷分布散乱,推断这些区域不太可能与异柠檬酸或ADP结合(见图 9(b),(d)和(f))。

|
图 9 牛NAD(+)IDH表面电荷分布图 Figure 9 Surface charge distribution of NAD(+)IDH in bovin |
异柠檬酸脱氢酶(IDH)催化生物体内的氧化脱羧反应,具有重要的生理作用。该酶受到多种辅助因子的调节,在三羧酸循环反应中发挥着重要作用。自2009年首次在弥漫性胶质瘤中发现IDH突变以来,IDH突变在诊断、预后和胶质瘤预测方面发挥越来越重要的作用[25]。2016年,世界卫生组织(WHO)首次将分子参数用于脑肿瘤诊断,将IDHl与IDH2突变分子病理检测作为胶质瘤新的分型诊断标准[27]。本研究利用多个生物信息学工具从多个方面分析了牛NAD(+)IDH的理化性质及空间结构。研究发现牛NAD(+)IDH无跨膜结构,具有信号肽且位于β亚基N端第1-16位的氨基酸上,属于亲水性蛋白质,整体呈碱性,亚细胞定位在线粒体中。异柠檬酸脱氢酶是三羧酸循环反应中的关键酶之一,在三羧酸循环中发挥着重要作用。三羧酸循环在线粒体中进行[28],据此推断牛NAD(+)IDH的肽链在核糖体上合成之后经进一步折叠组装,α、β和γ亚基以2:1:1的比例相结合,并在β亚基N端1-16位氨基酸残基信号肽的介导下进入线粒体参与三羧酸循环反应。这一推断与Chen等(1990)对哺乳动物NAD(+)IDH的研究结果相吻合[13]。
二级结构预测显示IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三个亚基的二级结构主要为α螺旋,并且α螺旋与β折叠排列紧凑有序,可以形成一定的结构域。此外还发现β亚基与γ亚基的二级结构中α螺旋和β折叠的位置相似,这种相似性与功能的关系有待进一步研究。三级结构预测与二级结构预测吻合度分析结果表明亚基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三级结构模型中指认的二级结构分布与Jpred和SOPMA所预测的三个亚基二级结构结果相一致,同时也表明Jpred和SOPMA所预测的二级结构信息是可信的。文献报道表明,Rushbrook等人在牛心线粒体中分离出NAD(+)IDH[10],在本研究中牛NAD(+)IDH亚细胞定位在线粒体中,本研究有关牛NAD(+)IDH亚细胞定位的结果与Rushbrook等人的实验研究结果相一致。除此之外,本研究中有关牛NAD(+)IDH的二级结构预测、三级结构预测、亲/疏水性、信号肽预测以及跨膜区预测等目前尚未发现实验研究报道,对于本研究中有关牛NAD(+)IDH蛋白的预测结果有待进一步的实验研究。
哺乳动物中存在的NAD(+)IDH蛋白的各个亚基在进化过程中表现出较高的同源性,然而有关NAD(+)IDH蛋白各个亚基残基的改变对亚基构象有怎样的影响以及这种影响的生物学意义目前尚无文献报道。本研究有关牛NAD(+)IDH蛋白IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三个亚基的生物信息学分析将有助于从生化和分子角度阐明哺乳动物尤其是人的IDH如何影响肿瘤的形成。
此外,有助于阐明DH3β与APC/C的分子对接机制、IDH3α的活性结构域的靶向调节以及肿瘤生长机制等[7, 8, 24, 30]。
| [1] |
STEEN I H, LIEN T, BIRKELAND N K. Biochemical and phylogenetic characterization of isocitrate dehydrogenase from a hyperthermophilic archaeon, Archaeoglobus fulgidus[J]. Archives of Microbiology, 1997, 168(5): 412-420. DOI:10.1007/s002030050516 (  0) 0) |
| [2] |
XU X, ZHAO J, XU Z, et al. Structures of human cytosolic NADP-dependent isocitrate dehydrogenase reveal a novel self-regulatory mechanism of activity[J]. Journal of Biological Chemistry, 2004, 279(32): 33946-33957. DOI:10.1074/jbc.M404298200 (  0) 0) |
| [3] |
KIM Y O, OH I U, PARK H S, et al. Characterization of a cDNA clone for human NAD-specific isocitrate dehydrogenase, α-subunit and structural comparison with its isoenzymes from different species[J]. Biochemical Journal, 1995, 308(1): 63-68. DOI:10.1042/bj3080063 (  0) 0) |
| [4] |
ALBERTO P, GIULIA B, ANNA L D S, et al. The clinical use of IDH1 and IDH2 mutations in gliomas[J]. Expert Review of Molecular Diagnostics, 2018, 18(12): 1041-1051. DOI:10.1080/14737159.2018.1548935 (  0) 0) |
| [5] |
EIKMANNS B J, EL-MANSI M, SHILOACH J, et al. Control of carbon flux through enzymes of central and intermediary metabolism during growth of Escherichia coli on acetate[J]. Current Opinion in Microbiology, 2006, 9(2): 173-179. DOI:10.1016/j.mib.2006.02.002 (  0) 0) |
| [6] |
LIU W, CAPUCO A V, ROMAGNOLO D F. Expression of cytosolic NADP+-dependent isocitrate dehydrogenase in bovine mammary epithelium: Modulation by regulators of differentiation and metabolic effectors[J]. Experimental Biology and Medicine, 2006, 231(5): 599-610. DOI:10.1177/153537020623100515 (  0) 0) |
| [7] |
WU Q N, ZHANG W M, XUE L Y, et al. APC/C-CDH1-regulated idh3β coordinates with the cell cycle to promote cell proliferation[J/OL]. Cancer Research, 2019. http://cancerres.aacrjournals.org/content/79/13/3281. DOI: 10.1158/0008-5472.can-18-2341.
(  0) 0) |
| [8] |
LIU L P, LI X J. IDH gene mutation in glioma[J]. Cancer Translational Medicine, 2018, 4(5): 129-133. DOI:10.4103/ctm.ctm_27_18 (  0) 0) |
| [9] |
王鹏, 赵旵军, 翟羽佳, 等. 植物NADP+依赖型异柠檬酸脱氢酶的分子进化及功能研究进展[J]. 生物学杂志, 2009, 26(3): 45-48. WANG Peng, ZHAO Chanjun, ZHAI Yujia, et al. Advances in molecular evolution and functions of NADP+-dependent isocitrate dehydrogenase in higher plants[J]. Journal of Biology, 2009, 26(3): 45-48. DOI:10.3969/j.issn.1008-9632.2009.03.045 (  0) 0) |
| [10] |
RUSHBROOK J I, HARVEY R A. Nicotinamide adenine dinucleotide dependent isocitrate dehydrogenase from beef heart: Subunit heterogeneity and enzyme dissociation[J]. Biochemistry, 1978, 17(25): 5339-5346. DOI:10.1021/bi00618a003 (  0) 0) |
| [11] |
ZENG Y, WEISS C, YAO T T, et al. Isocitrate dehydrogenase from bovine heart: Primary structure of subunit 3/4[J]. Biochemical Journal, 1995, 310(2): 507-516. DOI:10.1042/bj3100507 (  0) 0) |
| [12] |
WEISS C, ZENG Y, HUANG J, et al. Bovine NAD+-dependent isocitrate dehydrogenase: Alternative splicing and tissue-dependent expression of subunit 1[J]. Biochemistry, 2000, 39(7): 1807-16. DOI:10.1021/bi991691i (  0) 0) |
| [13] |
CHEN R D. Structure, functions and regulation of NAD and NADP dependent isocitrate dehydrogenases in higher plants and other organisms[J]. Plant Physiology and Biochemistry, 1990, 28: 411-427. DOI:10.1017/S002221510003680X (  0) 0) |
| [14] |
GURUPRASAD K, REDDY B V B, PANDIT M W. Correlation between stability of a protein and its dipeptide composition: A novel approach for predicting in vivo stability of a protein from its primary sequence[J]. Protein Engineering, 1990, 4(2): 155-161. DOI:10.1093/protein/4.2.155 (  0) 0) |
| [15] |
谢勇, 洪晓昆, 鄢仁祥, 等. 重组琼胶酶rAgaN3基因的生物信息学分析[J]. 生物信息学, 2017, 15(1): 16-26. XEI Yong, HONG Xiaokun, YAN Renxiang, et al. Bioinformatics analysis of the recombinant rAgaN3 gene of agarase[J]. Chinese Journal of Bioinformatics, 2017, 15(1): 16-26. DOI:10.3969/j.issn.1672-5565.2017.01.201608003 (  0) 0) |
| [16] |
王志新. 蛋白质结构预测的现状与展望[J]. 生命的化学, 1998, 18(6): 21-24. WANG Zhixin. The present situation and prospect of protein structure prediction[J]. Chemistry of Life, 1998, 18(6): 21-24. (  0) 0) |
| [17] |
赵国屏. 生物信息学[M]. 北京: 科学出版社, 2002. ZHAO Guoping. Bioinformatics[M]. Beijing: Science Press, 2002. (  0) 0) |
| [18] |
BARTON G J, CUFF J A. Application of multiple sequence alignment profiles to improve protein secondary structure prediction[J]. Proteins, 2000, 40(3): 502-511. DOI:10.1051/epn:2006401 (  0) 0) |
| [19] |
KANDAVELMAN A, SHANMUGHAVEL P. Evaluation of in silico protein secondary structure prediction methods by employing statistical techniques[J]. Biomedical and Biotechnology Research Journal (BBRJ), 2017, 1(1): 29-36. DOI:10.4103/bbrj.bbrj_28_17 (  0) 0) |
| [20] |
SCHWEDE T, BENKERT P, BIASINI M. Toward the estimation of the absolute quality of individual protein structure models[J]. Bioinformatics, 2011, 27(3): 343-350. DOI:10.1093/bioinformatics/btq662 (  0) 0) |
| [21] |
MARYAM D, SAMIN S. Structural, functional, and phylogenetic characterization of phosphoenolpyruvate carboxylase (PEPC) in C4 and CAM plants[J]. Caryologia, 2018, 71(3): 272-288. DOI:10.1080/00087114.2018.1465762 (  0) 0) |
| [22] |
LAWRENCE A K, STEFANS M, CHRISTOPHER M Y, et al. The Phyre2 web portal for protein modeling, prediction and analysis[J]. Nature Protocols, 2015, 10(6): 845-858. DOI:10.1038/nprot.2015.053 (  0) 0) |
| [23] |
HUANG Y C, SOUNDAR S, COLMAN R F. Affinity cleavage at the divalent metal site of porcine NAD-specific isocitrate dehydrogenase[J]. Protein Science, 2000, 9(1): 104-111. DOI:10.1110/ps.9.1.104 (  0) 0) |
| [24] |
ZENG L, MORINIBU A, KOBAYASHI M, et al. Aberrant IDH3α expression promotes malignant tumor growth by inducing HIF-1-mediated metabolic reprogramming and angiogenesis[J]. Oncogene, 2015, 34(36): 4758-4766. DOI:10.1038/onc.2014.411 (  0) 0) |
| [25] |
PARSONS D W, JONES S, ZHANG X, et al. An integrated genomic analysis of human glioblastoma multiforme[J]. Science, 2008, 321(5897): 1807-1812. DOI:10.1126/science.1164382 (  0) 0) |
| [26] |
陈剑峰, 王君, 张琛琳, 等. 异柠檬酸脱氢酶在胶质瘤临床研究中的进展[J]. 中华实验外科杂志, 2018, 35(5): 974-977. CHENG Jianfeng, WANG Jun, ZHANG Chenling, et al. Progress in clinical research of citrate dehydrogenase in glioma[J]. Chinese Journal of Experimental Surgery, 2018, 35(5): 974-977. DOI:10.3760/cma.j.issn.1001-9030.2018.05.057 (  0) 0) |
| [27] |
PIERRACHE L H M, KIMCHI A, RATNAPRIYA R, et al. Whole-exome sequencing identifies biallelic IDH3A variants as a cause of retinitis pigmentosa accompanied by pseudocoloboma[J]. Ophthalmology, 2017, 124(7): 992-1003. DOI:10.1016/j.ophtha.2017.03.010 (  0) 0) |
| [28] |
王镜岩, 朱圣庚, 徐长法, 等. 生物化学[M]. 北京: 高等教育出版社, 2002. WANG Jingyan, ZHU Shenggeng, XU Changfa, et al. Biochemistry[M]. Beijing: Higher Education Press, 2002. (  0) 0) |
| [29] |
MEINKEN J, MIN J. Computational prediction of protein subcellular locations in eukaryotes:An experience report[J]. Computational Molecular Biology, 2012, 2(1): 1-7. DOI:10.5376/cmb.2012.02.0001 (  0) 0) |
| [30] |
ZENG L, MORINIBU A, KOBAYASHI M, et al. Aberrant IDH3α expression promotes malignant tumor growth by inducing HIF-1-mediated metabolic reprogramming and angiogenesis[J]. Oncogene, 2014, 34(36): 4758-4766. (  0) 0) |
 2019, Vol. 17
2019, Vol. 17



