1958年,Crick提出了分子生物学的中心法则:DNA自我复制,DNA转录生成RNA,RNA翻译生成蛋白质。转录是基因表达的第一环节,它把遗传信息和生理功能的主要执行者蛋白质联系到了一起。转录调控,决定着基因的表达量,从而决定了它对于生物体的发育、形态结构、以及各类生理功能的基础性地位。近年来,随着生化检测技术水平的不断提高,学术界发现了一个新现象——转录爆发(Transcriptional bursting)。基因会从沉默的状态突然切换为激活态,启动转录、并在短时间内(通常不超过2~3 min)急剧生成产生大量信使RNA;然后再次进入沉默的状态[1-4]。转录爆发的发现,意味着已有的转录调控知识需要重新审视;也因为如此,转录爆发迅速成为当前生命科学的热点问题,成为后基因组时代的研究重点[5-6]。转录爆发,从简单的原核生物到高等哺乳动物细胞,普遍存在[1-4]。本文综述真核生物的转录爆发及其调控。
新现象的发现,使得科学家们开始不断寻找合适的数学模型对此现象进行定量解释。传统上认为,基因转录是一个泊松过程[7]。但是,在很多情况下,泊松模型并不能与数据吻合。为了对这些数据进行解释,学术界提出了两态模型。两态模型结构简明,应用广泛,与数据拟合较好[8-9]。本文首先介绍转录爆发的现象和模型。
基因转录表现为爆发式,带来一个富有挑战性而又有趣的难题。先前认为,转录水平与基因接受的调控信号的强度正相关——通常用希尔函数表示[10-11]。具体来讲,激活子(也全称为基因特异性转录因子)的浓度越高,基因转录水平越高。转录爆发现象的发现,意味着传统观点不再成立。过去的几年来,多种信号调控机制如雨后春笋般不断涌现,比如转录爆发频率调制[8, 12]、转录爆发尺度调制[9, 13], 以及它们的组合调制等[14]。但是,学术界尚未达成共识,并且都没有拿出压倒性的证据。本文将介绍转录爆发现象转导细胞调控信号的可能机制及其重要的生物学意义,并指出转录爆发领域待解决的问题。
1 转录爆发现象转录爆发现象的最初发现可追溯到1979年。Mcknight和Miller使用电子显微镜在对果蝇胚胎的检测中发现一个现象:转录时,染色体片段因被无转录发生的区域隔开而形成许多的新生RNA集群(RNA cluster)。这个新生RNA的集群现象就是最早的转录爆发。他们认为这种现象是由于转录起始的涨落引起的[15]。但是,这一发现并没有引起足够的重视。转录过程被认为是一个平稳过程,即转录产物的生成速率(单位时间内生成的RNA数量)不随时间变化。考虑到分子间相互作用的随机性,基因的转录速率在其均值的上下轻微波动[10]。因此,基因被认为以常数概率进行转录,信使RNA的生成是一个泊松过程[7]。
因为传统生化技术是从细胞群体水平上进行检测的,所以最终得到的是平均信息,丢失了单细胞的动态信息。1990年,Ko等首先使用一种报告基因(Reporter gene)的方法[16]。报告基因被定义为表达产物既易于检测又易于与内源性蛋白的背景区分的外源性基因。报告基因技术是指将报告基因剪接到目标基因启动子之后,再通过质粒转染到细胞内,在特定条件下培养,最终表达出用于产生信号的报告蛋白(如β-半乳糖苷酶、GFP等)。该技术使得科研人员不再受传统生化技术的阻碍,可以在单细胞水平下观察基因的表达。20世纪90年代,许多研究都陆续运用了reporter gene的方法,这些研究使用的实验技术就是最早的单细胞水平的生化技术[17-19]。
直到21世纪初,随着生物化学技术的不断进步,单细胞水平甚至单分子水平的检测技术成为目前研究转录爆发现象的主要手段[20-21]。单分子荧光原位杂交技术(Single-molecule Fluorescence in situ hybridization),其基本原理是将特定的信使RNA序列与荧光探针结合,从而可在高分辨率显微镜下直接观测到单个RNA分子。MS2标记技术是此类技术的代表,利用了MS2结合位点(MBS)与名为MS2外壳蛋白(MCP)之间的高度亲和性。实验前,MS2序列被插入到了目标基因的起始位点之后;随着转录的进行,MS2被转录出来以后形成茎环结构,该茎环与结合有绿色荧光蛋白的MCP特异性结合。因此,新生成的信使RNA序列可以被跟踪观测[2, 22]。结果表明,基因转录是一个断断续续的爆发式过程[2, 4](见图 1)。在0秒时,基因处于非激活态,不转录;在0到60 s之间,基因突然被激活,转录产生大量信使RNA;在120 s时,基因停止转录,回到非激活态。生成的信使RNA由于降解而逐渐减少。在接下来的时间内,基因不再进行转录,一直处于非激活态;之前转录产生的信使RNA逐渐降解,在视野中渐渐消失。
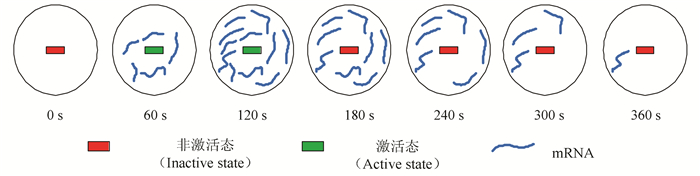
|
图 1 显微镜下转录爆发现象的示意图 Figure 1 Schematic diagram of transcriptional bursting under the microscope |
随着单细胞检测技术的发展,越来越多的学者在研究中观察到了转录爆发现象。无论是酵母菌、盘基网柄菌、果蝇还是哺乳动物,转录爆发普遍存在[1-4]。这说明转录爆发并非偶然现象,而是基因表达的一种基本性质[2]。这些单细胞和单基因水平的测量结果显示,转录并非传统上认为的稳恒泊松过程[10-11],而是间歇性的爆发式的。转录过程中,基因会从一个无转录活性的沉默态突然转换到一个激活态。在激活态,大量信使RNA被急速生成;激活态的寿命一般不超过2~3 min,基因继而再次进入沉默态。图 2示意了细胞内信使RNA的数量随时间的变化。图 2中展示了6次爆发,并示意了第五次爆发的持续时间(两条蓝色直线之间的时间间隔)。
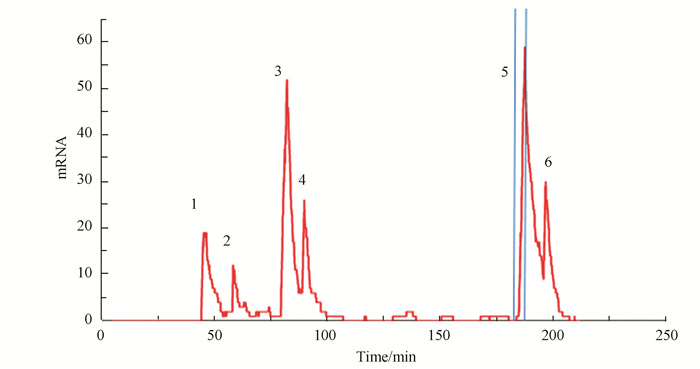
|
图 2 细胞内信使RNA数量随时间变化的关系图 Figure 2 Variation of the amount of messenger RNA with time |
传统的泊松模型,本质上基因只维持在一个状态;基因在以常数速率一直进行着转录。该模型对早期的实验数据有不错的解释[23]。但随着单细胞实验技术的不断进步,这种单态模型无法解释单细胞数据,两态模型应运而生。两态模型是目前学术界研究转录爆发使用最广泛的唯象模型[24]。两态模型,在研究信使RNA和蛋白质分子的数量变化、单个基因的状态切换、激活态基因的稳恒转录、以及信使RNA的指数降解等方面有很好的效果[4, 8-9, 25]。
在两态模型中,基因启动子只有两个状态:转录激活态(ON)和非转录态(OFF)。两态模型由四个参数描述,分别是kON, kOFF, km, γ(见图 3)。kON表示从OFF态到ON态的转换速率;koff表示从ON态到OFF态的转换速率。只有启动子处于ON态时,基因才进行转录,转录速率用km表示,km>0;启动子处于OFF态时,不转录,转录速率km=0。信使RNA的降解速率为γ。其中,kON, kOFF, km, γ都为常数[26]。所以,启动子处于ON态的持续时间服从指数分布[26]。当考虑基因只有ON态而没有OFF态时,转录出的信使RNA数量分布服从泊松分布[25]。
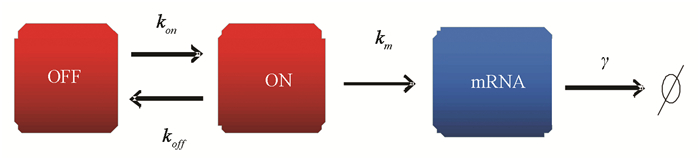
|
图 3 两态模型 Figure 3 Two-state model |
单态模型和两态模型通常用Gillespie算法和主方程方法进行计算机模拟[25, 27-28]。两态模型的动力学参数与爆发频率、爆发尺度直接相关。为了方便对爆发调控机制的理解,利用图 2进行名词进行解释。图 2展示了6次转录爆发,一次爆发的持续时间对应于信使RNA数量上升对应的时间间隔。第5次转录爆发上两条蓝色直线之间的时间间隔即为第5次爆发的持续时间,其它爆发依次类推。爆发幅度(Burst magnitude)被定义为一次转录爆发产生的信使RNA分子数量。爆发的持续时间和爆发幅度合称爆发尺度(Burst size)。爆发频率(Burst frequency)即单位时间内转录爆发发生的次数。
3 转录爆发信号转导机制目前,基因转录爆发现象的普遍性已得到公认。随之而来的问题是,转录爆发现象背后的分子调控机制和信号转导机制是什么?2006年,在诺贝尔化学奖的获奖演说中,Kornberg归纳了真核生物转录机器的基本架构[29]。转录机器的基本架构包括通用转录因子(General transcription factor)、RNA聚合酶Ⅱ、媒介子(Mediator)和激活子(Activator)等。其中,通用转录因子包括TFIIA、TFIIB、TFIID、TFIIE、TFIIF和TFIIH [29-30]。激活子携带来自细胞信号转导通路的信号,激活子与增强子相互结合并以某种方式编码了上游信号。媒介子与结合在增强子上的激活子相互结合,并将激活子带来的上游信号向后传递;最后,媒介子与RNA聚合酶Ⅱ相互作用,转录机器开始起始转录(见图 4)。而其中的编码方式则是学术界研究的转录爆发的信号转导机制[22]。目前学术界出现了多种信号转导机制的猜想与假设,如转录的频率调制[8, 12]、转录爆发的尺度调制[9, 13]、以及这些调制的组合调制[14]。这些调控机制会在下文进行详细介绍。
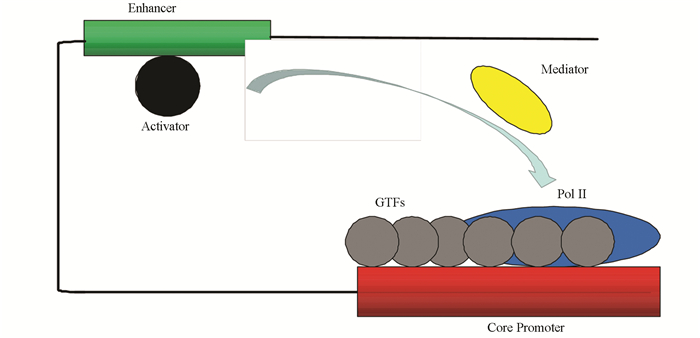
|
图 4 RNA聚合酶Ⅱ的转录机制 Figure 4 Transcription mechanism of RNA polymerase Ⅱ |
为便于理解,本文以两态模型为基础对爆发调控机制进行解释(见图 5)。图中,基因启动子处于爆发的ON态和OFF态的时间分别标注为τon和τoff,虚线表示在某类调制模式下增加的部分。转录爆发的频率调制(Burst frequency)是指在不改变τon和转录速率km的前提下,基因启动子通过缩短时间τoff来增加爆发的次数,从而增加基因表达。转录爆发的幅度调制(Burst magnitude)是指在不改变τon和τoff的前提下,基因启动子通过增加转录速率km来增加基因表达。转录爆发的持续时间调制(Burst duration)是指在不改变转录速率km和τoff的前提下,通过延长时间τon来增加基因表达。转录爆发的幅度(Burst magnitude)调制和时间(Burst duration)调制统一称为转录爆发的尺度(Burst size)调制。
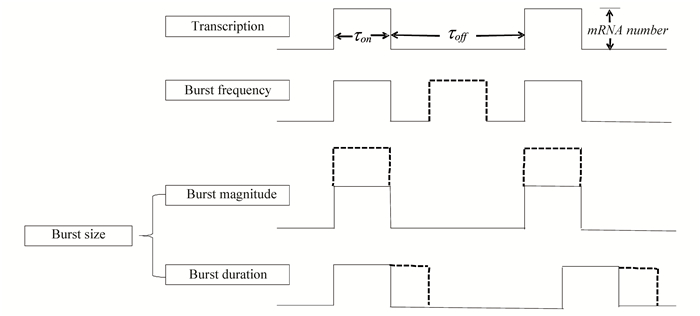
|
图 5 转录爆发调制模式 Figure 5 Modulation mode of transcriptional bursting |
Senecal的研究发现转录爆发中主要的调控机制是频率调制[8]。该文章使用两种不同的诱导手段激活MAPK途径研究了早期响应基因c-Fos的转录响应,并调查了不同激活子(血清诱导主要是ERK,锌诱导主要是p38)对c-Fos基因的转录爆发的影响。该研究表明在MAPK诱导期间,转录因子浓度调节c-Fos的爆发频率,而其他爆发参数基本保持不变。具体表现为,在转录响应时,随着诱导物浓度的变化,细胞的激活子浓度和转录爆发频率发生相似的变化。所以该文认为细胞通过调整激活子浓度来控制爆发频率,进一步控制整体信使RNA的水平。当转录因子浓度较高时,细胞更有可能进行激活和进行信使RNA的高水平表达;当转录因子浓度低时,情况则相反。这种频率调制虽然简单,但对c-Fos基因的转录响应是一种通用的机制。
Molina的研究表明转录爆发中主要的调控机制是爆发尺度调制模式[9]。该文针对哺乳动物单细胞单个等位基因ctgf进行血清诱导和TGF-β1诱导,研究二者引起的不同的转录响应。二者的诱导都引起了基因的转录响应,形成了转录爆发现象。血清诱导引起的是瞬时的转录响应,之后会产生一个约为3小时的不应期。而TGF-β1诱导引起的则是持续的转录响应。该研究表明血清诱导引起的转录爆发的原因是转录爆发的持续时间τon的延长、转录速率km的增加,由于是瞬时的转录响应,所以这是一个相应的动力学参数的增长是一个短期的效应。TGF-β1诱导引起的转录爆发原因是转录爆发的持续时间τon的延长、转录速率km的增加,而转录速率km长时间保持在较高速率,暗示了某些诱导物会通过持续地增加转录速率km来诱导一系列的转录爆发。时间τoff的几乎不变也说明TGF-β1诱导引起的转录爆发几乎不受频率调制的影响。该研究表明这二种诱导引起的精确的转录响应的主要原因是转录速率km的增加和爆发持续时间τon的增加,即这两种诱导引起的转录响应是转录爆发尺度调制。
Dar的研究显示,因基因表达水平的不同,爆发频率与爆发尺度都可以是调制的对象[14]。该文利用noise space[31]作为提取基因表达波动的研究框架,研究了两个问题,分别是人类基因组中占主导地位的表达模式是什么类型的表达、影响占主导地位的表达模式的主要因素是什么。实验发现,转录爆发模式是转录过程的主导形式,爆发频率与爆发尺度在人类基因组都受到了调制。当基因表达水平较低的时候,转录以爆发频率调制为主,即基因启动子会通过增加爆发频率来增加基因的表达。但当爆发频率达到一个阈值之后,转录以爆发尺度调制为主,此时只能通过增加爆发期的转录速率或持续时间来继续增加基因的表达。在人类基因组中,爆发频率和爆发尺度随染色体位置的不同而不同。虽然这种二者的变化与启动子序列无关,但极强地取决于基因座的表达水平。
4 转录爆发的生物学意义在转录爆发现象发现的早期,学术界普遍认为这是一种噪音(Noise)。噪音,即围绕平均值的随机涨落。随着研究的深入,越来越多的学者惊叹,基因转录的噪音水平远远超出了预期。学术界开始思考噪音对细胞信号转导的影响,探索噪音水平是如何被控制削弱的[10]。
随着单细胞单分子测量技术的不断进步,越来越多的证据表明,转录爆发是一种普遍现象。转录爆发存在于从简单的原核细胞到高等真核生物细胞内。学术界不得不承认,转录爆发并非是简单的噪音,而是传递细胞调控信号的方式[1-4]。
转录爆发是如何携带调控信号的?如前所述,目前尚无定论。转录爆发传递信号的可能方式包括频率调制和尺度调制,也可能二者兼有。如果是前者,就意味着转录调控过程中,信号转换是数字式的。另一方面,不同基因的爆发动力学也不相同,这种不同背后又蕴含了什么样的生物学意义?基因转录是爆发式的,意味着基于高通量测量方式的数据需要重新审视。爆发式的转录,客观上要求单分子标记技术和计算机模拟技术的进步。
5 总结本文详细阐述了转录爆发现象和转录爆发的两态模型,并介绍了转录调控领域中的信号转导机制以及转录爆发的生物学意义。下面总结该领域内的难点和疑问。首先,学术界认为最有可能的两种信号调制机制是爆发频率调制机制和爆发尺度调制机制。转录爆发的尺度,是由爆发的持续时间和爆发产生的信使RNA分子的数量决定的。转录爆发的频率是由单位时间内爆发的发生次数决定的。承载信号的究竟是尺度还是频率,抑或二者兼有?关于转录爆发的信息编码机制,学术界仍在积极地探索中。其次,由于实验技术的限制,两次相邻发生的转录爆发的边界难以区分。另外,转录涉及到的许多物质与形态结构也会对转录过程产生影响,比如增强子[3]、局部启动子结构[4]、转录因子[8]、核小体构型[32]等。最后,越来越多的研究发现两态模型并不能对所有爆发现象的实验数据给出满意的解释[33];目前,学术界中已出现了一些更为新颖的转录模型,如连续性模型[28]和多尺度模型[34]等。在研究转录爆发的动力学和分子机制方面,还有许多工作需要做。
| [1] |
LARSON D R, ZENKLUSEN D, WU B, et al. Real-time observation of transcription initiation and elongation on an endogenous yeast gene[J]. Science, 2011, 332(6028): 475-478. DOI:10.1126/science.1202142 (  0) 0) |
| [2] |
CHUBB J R, TRCEK T, SHENOY S M, et al. Transcriptional pulsing of a developmental gene[J]. Current Biology, 2006, 16(10): 1018-1025. DOI:10.1016/j.cub.2006.03.092 (  0) 0) |
| [3] |
FUKAYA T, LIM B, LEVINE M. Enhancer control of transcriptional bursting[J]. Cell, 2016, 166(2): 358-368. DOI:10.1016/j.cell.2016.05.025 (  0) 0) |
| [4] |
RAJ A, PESKIN C S, TRANCHINA D, et al. Stochastic mRNA synthesis in mammalian cells[J]. PLOS Biology, 2006, 4(10): e309. DOI:10.1371/journal.pbio.0040309 (  0) 0) |
| [5] |
唐旭清, 朱平. 后基因组时代生物信息学的发展趋势[J]. 生物信息学, 2008, 6(3): 142-144. TANG Xuqing, ZHU Ping. Developing trend of bioinformatics in post genome era[J]. China Journal of Bioinformatics, 2008, 6(3): 142-144. DOI:10.3969/j.issn.1672-5565.2008.03.015 (  0) 0) |
| [6] |
陈铭. 后基因组时代的生物信息学[J]. 生物信息学, 2004(2): 29-34. CHEN Ming. Bioinformatics in the postgenomic era[J]. China Journal of Bioinformatics, 2004(2): 29-34. DOI:10.3969/j.issn.1673-2588.2001.04.034 (  0) 0) |
| [7] |
SANCHEZ A, CHOUBEY S, KONDEV J. Stochastic models of transcription: From single molecules to single cells[J]. Methods, 2013, 62(1): 13-25. DOI:10.1016/j.ymeth.2013.03.026 (  0) 0) |
| [8] |
SENECAL A, MUNSKY B, PROUX F, et al. Transcription factors modulate c-Fos transcriptional bursts[J]. Cell Reports, 2014, 8(1): 75-83. DOI:10.1016/j.celrep.2014.05.053 (  0) 0) |
| [9] |
MOLINA N, SUTER D M, CANNAVO R, et al. Stimulus-induced modulation of transcriptional bursting in a single mammalian gene[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(51): 20563-20568. DOI:10.1073/pnas.1312310110 (  0) 0) |
| [10] |
RAJ A, VAN OUDENAARDEN A. Nature, nurture, or chance: Stochastic gene expression and its consequences[J]. Cell, 2009, 96(3): 15a-15a. DOI:10.1016/j.cell.2008.09.050 (  0) 0) |
| [11] |
LENSTRA T L, RODRIGUEZ J, CHEN H, et al. Transcription dynamics in living cells[J]. Annual Review of Biophysics, 2016, 45(1): 25. DOI:10.1146/annurev-biophys-062215-010838 (  0) 0) |
| [12] |
BARTMAN C R, HSU S C, HSIUNG C C, et al. Enhancer regulation of transcriptional bursting parameters revealed by forced chromatin looping[J]. Molecular Cell, 2016, 62(2): 237-247. DOI:10.1016/j.molcel.2016.03.007 (  0) 0) |
| [13] |
SKUPSKY R, BURNETT J C, FOLEY J E, et al. HIV promoter integration site primarily modulates transcriptional burst size rather than frequency[J]. PLOS Computational Biology, 2010, 6(9): e1000952. DOI:10.1371/journal.pcbi.1000952 (  0) 0) |
| [14] |
DAR R D, RAZOOKY B S, SINGH A, et al. Transcriptional burst frequency and burst size are equally modulated across the human genome[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(43): 17454-17459. DOI:10.1073/pnas.1213530109 (  0) 0) |
| [15] |
MCKNIGHT S L, MILLER O L. Post-replicative nonribosomal transcription units in D. melanogaster embryos[J]. Cell, 1979, 17(3): 551-563. DOI:10.1016/0092-8674(79)90263-0 (  0) 0) |
| [16] |
KO M S, NAKAUCHI H, TAKAHASHI N. The dose dependence of glucocorticoid-inducible gene expression results from changes in the number of transcriptionally active templates[J]. The EMBO Journal, 1990, 9(9): 2835-2842. DOI:10.1002/j.1460-2075.1990.tb07472.x (  0) 0) |
| [17] |
ROSS I L, BROWNE C M, HUME D A. Transcription of individual genes in eukaryotic cells occurs randomly and infrequently[J]. Immunology and Cell Biology, 1994, 72(2): 177-185. DOI:10.1038/icb.1994.26 (  0) 0) |
| [18] |
NEWLANDS S, LEVITT L K, ROBINSON C S, et al. Transcription occurs in pulses in muscle fibers[J]. Genes & Development, 1998, 12(17): 2748-2758. DOI:10.1101/gad.12.17.2748 (  0) 0) |
| [19] |
TAKASUKA N, WHITE M R H, WOOD C D, et al. Dynamic changes in prolactin promoter activation in individual living lactotrophic cells[J]. Endocrinology, 1998, 139(3): 1361-1368. DOI:10.1210/endo.139.3.5826 (  0) 0) |
| [20] |
FEMINO A M. Visualization of single RNA transcripts in situ[J]. Science, 1998, 280(5363): 585-590. DOI:10.1126/science.280.5363.585 (  0) 0) |
| [21] |
BERTRAND E, CHARTRAND P, SCHAEFER M, et al. Localization of ASH1 mRNA particles in living yeast[J]. Molecular Cell, 1998, 2(4): 437-445. DOI:10.1016/S1097-2765(00)80143-4 (  0) 0) |
| [22] |
TIMOTHÉE L, SINGER R H. Transcription goes digital[J]. EMBO Reports, 2012, 13(4): 313-321. DOI:10.1038/embor.2012.31 (  0) 0) |
| [23] |
ZENKLUSEN D, LARSON D R, SINGER R H. Single-RNA counting reveals alternative modes of gene expression in yeast[J]. Nature Structural & Molecular Biology, 2008, 15(12): 1263-1271. DOI:10.1038/nsmb.1514 (  0) 0) |
| [24] |
KO M S H. Problems and paradigms: Induction mechanism of a single gene molecule: Stochastic or deterministic?[J]. BioEssays, 2010, 14(5): 341-346. DOI:10.1002/bies.950140510 (  0) 0) |
| [25] |
PAULSSON J. Models of stochastic gene expression[J]. Physics of Life Reviews, 2005, 2(2): 157-175. DOI:10.1016/j.plrev.2005.03.003 (  0) 0) |
| [26] |
SUTER D M, MOLINA N, GATFIELD D, et al. Mammalian genes are transcribed with widely different bursting kinetics[J]. Science, 2011, 332(6028): 472-4. DOI:10.1126/science.1198817 (  0) 0) |
| [27] |
GILLESPIE D T. Exact stochastic simulation of coupled chemical-reactions[J]. The Journal of Chemical Physics, 1997, 81(25): 2340-2361. DOI:10.1021/j100540a008 (  0) 0) |
| [28] |
CORRIGAN A M, TUNNACLIFFE E, CANNON D, et al. A continuum model of transcriptional bursting[J]. eLIFE, 2016, 5: e13051. DOI:10.7554/eLife.13051 (  0) 0) |
| [29] |
KORNBERG R. The molecular basis of eukaryotic transcription (nobel lecture)[J]. Angewandte Chemie International Edition, 2007, 46(37): 6956-6965. DOI:10.1073/pnas.0704138104 (  0) 0) |
| [30] |
ORPHANIDES G, LAGRANGE T, REINBERG D. The general transcription factors of RNA polymerase Ⅱ[J]. Genes & Development, 1996, 10(21): 2657-2683. DOI:10.1101/gad.10.21.2657 (  0) 0) |
| [31] |
COX C D, MCCOLLUM J M, ALLEN M S, et al. Using noise to probe and characterize gene circuits[J]. Proceedings of the National Academy of Sciences, 2008, 105(31): 10809-10814. DOI:10.1073/pnas.0804829105 (  0) 0) |
| [32] |
BOEGER H, SHELANSKY R, PATEL H, et al. From structural variation of gene molecules to chromatin dynamics and transcriptional bursting[J]. Genes, 2015, 6(3): 469-483. DOI:10.3390/genes6030469 (  0) 0) |
| [33] |
YAOLAI W, TENGFEI N, WEI W, et al. Gene transcription in bursting: A unified mode for realizing accuracy and stochasticity[J]. Biological Reviews, 2019, 94(1): 248-258. DOI:10.1111/brv.12452 (  0) 0) |
| [34] |
TANTALE K, MUELLER F, KOZULIC-PIRHER A, et al. A single-molecule view of transcription reveals convoys of RNA polymerases and multi-scale bursting[J]. Nature Communications, 2016, 7: 12248. DOI:10.1038/ncomms12248 (  0) 0) |
 2019, Vol. 17
2019, Vol. 17


