miRNA(microRNA)是一类广泛存在于动植物和某些病毒内,长度为19-25 nt的内源性单链非编码小RNA,序列具有高度保守性[1]。miRNA主要通过与靶基因mRNA的3’端非翻译区(3’UTR)结合,导致翻译抑制或者降解,进而调控靶基因的表达[2]。
has-miR-10a-5p是近年来新发现的miRNA,属于miR-10基因家族,其聚集在染色体17长度为22 bp的位置中,具体基因位点在chr17:48 579 838-48 579 947处。近年国内外研究表明,通过与靶基因相互作用,has-miR-10a-5p作为癌基因在多种肿瘤的发生发展中发挥作用[3-7]。目前研究miR-10a-5p与肿瘤抑制和发展的相关问题越来越多,所以对miR-10a-5p进行靶基因相关生物信息学分析对今后探索miR-10a-5p功能和作用十分必要。相较于实验验证,生物信息学能对靶基因及相关通路进行大量多样高速的预测。本研究运用生物信息学方法预测miR-10a-5p的靶基因,并对其靶基因集合进行功能注释(Gene Ontology, GO)和信号通路富集分析(Kyoto Encyclopedia of Genes and Genome Pathway, KEGG Pathway),为后续对miR-10a-5p的靶基因鉴定及生物学功能研究提供理论基础和思路。
1 资料与方法 1.1 miR-10a-5p的基本生物学信息使用在线工具miRbase(http://www.mirbase.org/)查找miR-10a-5p的染色体定位、碱基序列及不同动物的成熟序列。并使用clustalx 2.1软件分析物种保守性。
1.2 miR-10a-5p靶基因的预测分析使用Pubmed(http://www.ncbi.nlm.nih.gov/pubmed/)查找已有文献报道miR-10a-5p靶基因的相关信息。采用Targetscan7.1(http://www.targetscan.org/vert_71/)、miRDB(http://www. http://mirdb.org/)、mirDIP(http://ophid.utoronto.ca/mirDIP/index_confirm.jsp)和DIANA TOOLS(http://diana.imis.athena-innovation.gr/DianaTools/index.php)4个在线靶基因预测工具预测miR-10a-5p的靶基因,并用Venny 2.1 (http://bioinfogp.cnb.csic.es/tools/venny/index.html)绘制4个数据库预测靶基因的韦恩图,取交集用于后续分析。
1.3 miR-10a-5p靶基因的GO功能注释及KEGG富集分析使用在线工具DAVID(http://david.abcc.ncifcrf.gov/)对交集靶基因进行Gene Ontology功能富集分析(GO分析),细胞组分(cellular component, CC)、分子功能(molecular function, MF)、生物学过程(biological process, BP),用超几何分布检验,以P < 0.05为显著性阈值,得到具有统计意义前10个GO注释分析。
用DAVID数据库中KEGG Pathway分析对交集靶基因进行信号通路富集分析,具有统计意义前10个信号通路。
2 结果与分析 2.1 miR-10a-5p的序列保守性分析使用miRBase数据库检索miR-10a-5p的成熟序列,miR-10a-5p在已知的31个物种中存在成熟序列,并且使用数据库下载了人(hsa)、小鼠(mmu)、大鼠(rno)、斑马鱼(dre)等15个物种的成熟序列(见图 1)。利用clustalx 2.1软件,对其序列保守性进行分析,结果表明miR-10a-5p在各物种之间具有高度的保守性的成熟序列为“uacccuguagauccgaauuugug”(见表 1)。

|
图 1 不同物种miR-10a-5p的成熟序列 Figure 1 Mature sequence of miR-10a-5p in different species |
| 表 1 不同物种miR-10a-5p的成熟序列 Table 1 Mature sequence of miR-10a-5p in different species |
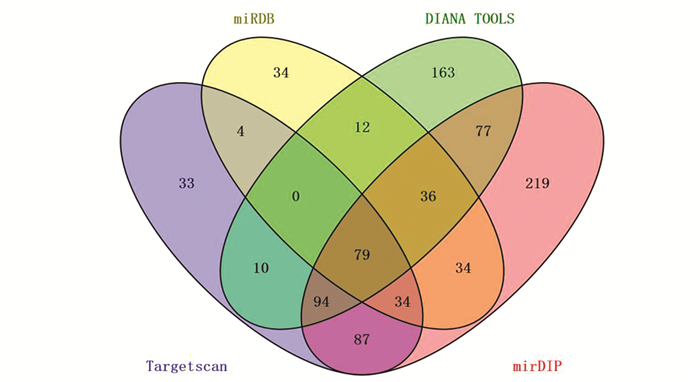
Pubmed检索文献表明,miR-10a-5p通过在转录后水平调控其靶基因参与了多种疾病的发生发展及预后(见表 2)。选用TargetScan、miRDB、mirDIP和DIANA TOOLS 4个在线数据库预测的靶基因个数分别为341、233、660和472,用Venny 2.1对上述结果绘制韦恩图取交集,得到79个预测靶基因的数据集(见表 3)。作为后续分析的基因总集合(见图 2)。
| 表 2 miR-10a-5p调控的靶基因参与人类疾病 Table 2 miR-10a-5p target genes involved in human disease |
| 表 3 miR-10a-5p调控的靶基因数据集 Table 3 Target gene data sets regulated by miR-10a-5p |
|
图 2 miR-10a-5p的预测靶基因个数 Figure 2 Predicted number of target genes of miR-10a-5p |
将4个数据库均能预测到的79个靶基因进行GO分析,发现miR-10a-5p的靶基因主要富集于核质、突触、细胞间粘附链接、核基质、组蛋白甲基转移酶复合物等10个细胞组分(P < 0.05,见图 3);参与肝脏发育、脂肪组织发育、神经细胞凋亡负调控、RNA聚合酶Ⅱ启动子转录的起始、RNA聚合酶启动子转录的正调控、DNA模板转录等10个生物学过程(P < 0.05,见图 4);显著富集于染色质结合、转录活性激活、RNA聚合酶Ⅱ远端增强子序列特异性DNA结合、RNA聚合酶Ⅱ转录因子活性配体激活的序列特异性DNA结合等9个分子功能(P < 0.05,见图 5)。

|
图 3 miR-10a-5p靶基因细胞组分的GO功能分析 Figure 3 GO function analysis of cell components of miR-10a-5p target gene |

|
图 4 miR-10a-5p靶基因生物学过程的GO功能分析 Figure 4 GO function analysis of the biological process of miR-10a-5p target gene |

|
图 5 miR-10a-5p靶基因分子功能的GO功能分析 Figure 5 GO function analysis of molecular function of miR-10a-5p target gene |
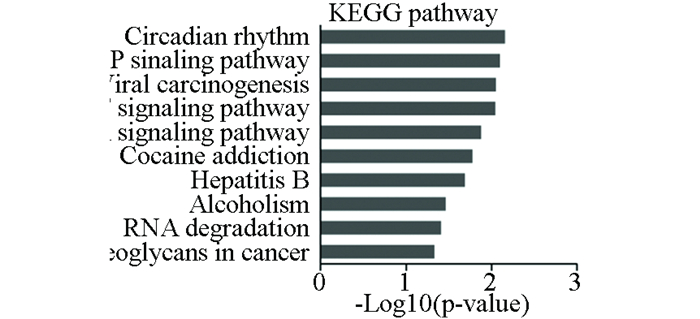
利用DAVID数据库中的工具Functional Annotation对靶基因集合进行生物通路富集分析,其中79个靶基因具有相关的生物通路。以人类全基因组为背景,发现miR-10a-5p的靶基因在cAMP信号通路、TNF信号通路及AMPK信号通路中显著富集(见图 6)。
|
图 6 miR-10a-5p靶基因KEGG通路富集分析 Figure 6 KEGG pathway enrichment analysis of predicted target of miR-10a-5p |
近年来,与miRNA相关的内容已成为研究的热点之一。越来越多的证据表明,miRNA能够通过调控下游靶基因的表达从而发挥其功能,包括重要的生命活动,如细胞分化、增殖、细胞凋亡以及新陈代谢[9],并在肿瘤的发生发展、侵袭与转移[10],以及治疗、预后[11]等方面都有重要作用。而生物信息学在miRNA靶基因的预测等研究中发挥了重要作用,本研究预测靶基因使用了四种计算方法不同的miRNA靶基因预测软件进行,考虑到miRNA与靶基因结合位点的序列保守性、序列匹配、miRNA与mRNA双链结合的热稳定性等因素来预测靶基因,具有高特异性和低假阳性率,将预测的靶基因取交集后作为靶基因集合,因此它作为分析的靶基因集合具有很好的代表性。
本研究中预测得到总共79个miR-10a-5p的靶基因, 靶基因存在细胞的各个组分中,具有染色质结合、转录活性激活、RNA聚合酶Ⅱ远端增强子序列特异性DNA结合和其它分子功能,并显著富集于肝脏发育、脂肪组织发育、神经细胞凋亡负调控等生物学过程;显著富集于cAMP信号通路、TNF信号通路及AMPK信号通路。大量研究发现,miR-10a-5p在细胞增殖和分化[11-12]过程中起着重要的调控作用,有研究发现,阿兹海默症(AD)中淀粉样蛋白-β或TAU病理诱导的神经元miR-10a-5p的升高[13],推测miR-10a-5p能调控神经元及突触的转录[14]。研究还表明滑膜细胞中miR-10a-5p的下调有助于TBX5控制的关节炎症[15],并起到抑制各种炎性细胞因子的产生的作用。
大量研究表明,miR-10a-5p在多种恶性肿瘤中异常表达。例如,miR-10a-5p在宫颈癌和人胰腺导管腺癌中呈现高表达[3, 5]。然而,一些研究发现miR-10a-5p在多种癌细胞中低表达,如胆管癌、肾细胞癌[4, 7]。近年来,一些研究证实miR-10a-5p通过调节其靶基因在抑制肿瘤细胞的增殖和转移中起重要作用,例如miR-10a-5p可通过抑制MAPK8IP1的表达,进而促进胃癌转移[6]。在细胞凋亡和其他肿瘤过程中, 已经鉴定了miRNA表达的双相模式(上调和下调),因此miR-10a-5p未来或许可以考虑作为肿瘤的早期诊断[16]和预后[17]的分子生物标志物及治疗肿瘤的潜在靶标[18-19]。
4 结论本研究采用生物信息学方法对miR-10a-5p的GO功能富集分析和Pathway通路富集分析等数据进行系统的挖掘,结果显示miR-10a-5p在生长发育及肿瘤发生发展过程中的作用不容忽视,且本研究结果与现有对miR-10a-5p调控作用的研究报道基本一致,说明生物信息学分析具有可靠性。这些分析为有关miR-10a-5p在各生物学过程中的作用机制后续研究提供了一定的数据支持和探索思路。但由于预测靶基因过程中仍存在不可避免的假阳性率,因此,由本次实验结果筛选出的靶基因还需进行实验验证,其功能和相关信号通路也有待进一步研究。
| [1] |
LI Defang, LIU Jin, GUO Baosheng, et al. Osteoclast-derived exosomal miR-214-3p inhibits osteoblastic bone formation[J]. Nature Communications, 2016, 7: 10872. DOI:10.1038/ncomms10872 (  0) 0) |
| [2] |
KONG fanyang, LI Lei, WANG Guokun, et al. VDR signaling inhibits cancer-associated-fibroblasts' release of exosomal miR-10a-5p and limits their supportive effects on pancreatic cancer cells[J]. Gut, 2019, 68(5): 950-951. DOI:10.1136/gutjnl-2018-316627 (  0) 0) |
| [3] |
ZHAI Lianchen, LI Yanli, LAN Xinzhi, et al. MicroRNA-10a-5p suppresses cancer proliferation and division in human cervical cancer by targeting BDNF[J]. Experimental & Therapeutic Medicine, 2017, 14(6): 6147-6151. (  0) 0) |
| [4] |
GAO Lili, YANG Xiaoping, ZHANG Hao, et al. Inhibition of miR-10a-5p suppresses cholangiocarcinoma cell growth through downregulation of Akt pathway[J]. Onco Targets Ther, 2018, 11: 6981-6994. DOI:10.2147/OTT (  0) 0) |
| [5] |
XIONG Guangbing, HUANG Hua, FENG Mengyu, et al. miR-10a-5p targets TFAP2C to promote gemcitabine resistance in pancreatic ductal adenocarcinoma[J]. Journal of Experimental & Clinical Cancer Research, 2018, 37(1): 76. DOI:10.1186/s13046-018-0739-x (  0) 0) |
| [6] |
ARAI T, OKATO A, KOJIMA S, et al. Regulation of spindle and kinetochore-associated protein 1 by antitumor miR-10a-5p in renal cell carcinoma[J]. Cancer Science, 2017, 108(10): 2088-2101. DOI:10.1111/cas.13331 (  0) 0) |
| [7] |
LU Yaoyong, WEI Ganbao, LIU Liangbo, et al. Direct targeting of MAPK8IP1 by miR-10a-5p is a major mechanism for gastric cancer metastasis[J]. Oncology Letters, 2017, 13(3): 1131-1136. DOI:10.3892/ol.2016.5544 (  0) 0) |
| [8] |
HUSSAIN N, ZHU W, JIANG C, et al. Down-regulation of miR-10a-5p in synoviocytes contributes to TBX5-controlled joint inflammation[J]. Journal of Cellular and Molecular Medicine, 2018, 22(1): 241-250. DOI:10.1111/jcmm.13312 (  0) 0) |
| [9] |
孙丹, 于萌, 郑明星, 等. hsa-miR-138的生物信息学分析及分子调控通路预测[J]. 中国医科大学学报, 2016, 45(6): 481-486. SUN Dan, YU Meng, ZHENG Mingxing, et al. Bioinformatics analysis and molecular regulation pathway prediction of hsa-miR-138[J]. Journal of China Medical University, 2016, 45(6): 481-486. DOI:10.12007/j.issn.0258-4646.2016.06.001 (  0) 0) |
| [10] |
郭萍, 曹文君, 任晨霞, 等. has-miR-105的靶基因预测及生物信息学分析[J]. 长治医学院学报, 2017, 39(5): 61-64. GUO ping, CAO Wenjun, REN Chenxia, et al. Target gene prediction and bioinformatics analysis of has-miR-105[J]. Journal of Changzhi Medical College, 2017, 39(5): 61-64. DOI:10.3969/j.issn.1006-0588.2018.01.001 (  0) 0) |
| [11] |
EUN K J, WON H J, LEE H S, et al. Hsa-miR-10a-5p downregulation in mutant UQCRB-expressing cells promotes the cholesterol biosynthesis pathway[J]. Scientific Reports, 2018, 8(1): 12407. DOI:10.1038/s41598-018-30530-6 (  0) 0) |
| [12] |
LI Zheng, JIANG Chao, YE Canhua, et al. miR-10a-5p, miR-99a-5p and miR-21-5p are steroid-responsive circulating microRNAs[J]. American Journal of Translational Research, 2018, 10(5): 1490-1497. (  0) 0) |
| [13] |
GUI Yaxing, LIU Hai, ZHANG Lishan, et al. Altered microRNA profiles in cerebrospinal fluid exosome in Parkinson disease and Alzheimer disease[J]. Oncotarget, 2015, 6(35): 37043-53. DOI:10.18632/oncotarget.6158 (  0) 0) |
| [14] |
HAMADA N, FUJITA Y, KOJIMA T, et al. MicrornaRNA expression profiling of NGF-treated PC12 cells revealed a critical role for miR-221 in neuronal differentiation[J]. Neurochemistry International, 2012, 60(8): 743-750. DOI:10.1016/j.neuint.2012.03.010 (  0) 0) |
| [15] |
HUSSAIN N, ZHU Wenhua, JIANG Congshan, et al. Down-regulation of miR-10a-5p promotes proliferation and restricts apoptosis via targeting T-box transcription factor 5 in inflamed synoviocytes[J]. Bioscience Reports, 2018, 38(2): BSR20180003. DOI:10.1042/BSR20180003 (  0) 0) |
| [16] |
WANG R H, HE L Y, ZHOU S H. The role of gene sculptor microRNAs in human precancerous lesions[J]. Onco Targets and Therapy, 2018, 11: 5667-5675. DOI:10.2147/OTT (  0) 0) |
| [17] |
GONZALEZ D A L, ALMEIDA B C D, ALMEIDA T G D, et al. Could miRNA signatures be useful for predicting uterine sarcoma and carcinosarcoma prognosis and treatment?[J]. Cancers (Basel), 2018, 10(9): 315. DOI:10.3390/cancers10090315 (  0) 0) |
| [18] |
YANEZ-MO M, SILJANDER P R, ANDREU Z, et al. Biological properties of extracellular vesicles and their physiological functions[J]. Journal of Extracellular Vesicles, 2015, 4(1): 27066. DOI:10.3402/jev.v4.27066 (  0) 0) |
| [19] |
BROWNE B M, STENSLAND K D, PATEL C K, et al. microRNA expression profiles in upper tract urothelial carcinoma differentiate tumor grade, stage, and survival: Implications for clinical decision-making[J]. Urology, 2019, 123(1): 93-100. DOI:10.1016/j.urology.2018.10.004 (  0) 0) |
 2019, Vol. 17
2019, Vol. 17


