过去的20年里,斑马鱼逐渐成为研究人类基因功能的重要模式动物。因为它们的寿命相对较短,但与人类一样,它们仍然会逐渐老化。并且,由于它的生命周期短,适合做生物学实验。此外,它的保守分子的广泛表征细胞生理学使斑马鱼成为揭示老化,疾病和修复的潜在机制的极好模型。了解斑马鱼衰老基因的分子机制,例如氧化蛋白质在肌肉中的积累都至关重要[1-2]。尽管没有现成的斑马鱼衰老基因组,但大多数的性状都需要涉及到一个复杂的分子之间相互作用,因此,衰老也不例外。
最近,有了更多关于斑马鱼神经系统老化的消息,使得对于它的衰老分子机制的研究更加明朗。斑马鱼尤其有可能在老年学研究中具有很大的价值,因为发展-精神和基因研究的丰富性已经推动了基本斑马鱼生物学和相关实验技术的广泛特征化。这种小型脊椎动物具有几个独特的特征,为该领域提供许多优势。此外,大多数斑马鱼因组已被测序,并被证明与人类具有高度相似性,具有许多人类基因的直系同源物。一个复杂的哺乳动物样发育基因程序生成了一个整合的神经系统,为此所有的主要脑区都被映射,并赋予这些斑马鱼高级认知功能和社会行为[3]。此外,斑马鱼专用实验程序(获得和丧失功能方法,体内成像技术)和资源(克隆,抗体)的广泛可用性将进一步支持老化。在之前的斑马鱼研究中,衰老一直被认为是微不足道的,因为它们经历了不确定的生长并具有非常强大的再生能力。过去二十年的不断努力,已经开始研究出斑马鱼内潜伏老龄化途径,说明了与其他脊椎动物物种的许多相似之处。它们表现出相同的典型标记和降低的β-半乳糖苷酶表达,并显示出通常类似于人类中已知的年龄相关病理学的衰老表型和骨关节炎[13]。
研究表明,由于斑马鱼复杂和多效的性质,其衰老状态难以用少数特征来表征。越来越明显的是,许多老龄驱动因素相互关联并产生相互影响。不幸的是,到目前为止,对于斑马鱼衰老分子机制的分子相互作用很少,所以高度需要斑马鱼衰老蛋白质分子相互作用交互图。在这种情况下,使用不同的动物模型系统进行精确的遗传或药理学操作确实可以在不同的生理系统和/或功能中鉴别衰老剂。因此,似乎只有将不同模型中的数据组合起来才有助于破译造成老龄化的主要因素。
1 数据来源和分析数据来源National Center of Biotechnology Information(NCBI)基因表达综合数据库(GEO)数据库(http://www.ncbi.nlm.nih.gov/geo/),GPL1319平台([Zebrafish] Affymetrix Zebrafish Genome Array)应用于表达式阵列。本平台的斑马鱼数据集包含12个样本,下载这些数据(CEL表单)和注释文件以进行进一步分析。由于智人与斑马鱼具有很高的同源性,最后从47 036个斑马鱼基因中,选择了67个与智人具有同源性、具有代表性的斑马鱼衰老相关基因[4-7]。使用SPSS对数据进行标准化处理,然后将获得的67个基因放入NCBI的GEO数据集中。已经发现85.38%的基因被用于功能基因组学研究,因此我们有理由相信这些数据可以作为斑马鱼衰老机制的基因。获得原始数据后,对数据进行归一化,使得GEO数据集中斑马鱼基因与人类基因之间的同源性系数在0到1之间,然后进行单样本T检验方法鉴定Hgs(同源性基因),得出GEO中与人类同源性高的斑马鱼基因。选择置信区间为99%作为截止标准(见表 1)。
| 表 1 来自GEO数据库的同源化基因的分析结果 Table 1 Analysis of homologous genes from the GEO database |
KEGG(京都基因与基因组百科全书)是基因组破译方面的数据库。它是一个公认的综合性数据库,同时它是一个系统的分析基因功能、基因组信息数据库,它有助于研究者把基因及表达信息作为一个整体网络进行研究, 包括各种生物化学途径[5-8]。因此,在这项工作中,我们对Hgs应用了KEGG数据库研究Hgs的富集分析,寻找可能影响斑马鱼衰老发生的生物化学途径。DAVID用于进行KEGG通路富集分析,p值< 0.05,基因计数>2。为了进一步了解Hgs的功能,DAVID应用于识别显着失调的KEGG途径。来自所有衰老相关基因的p值< 0.05和基因计数> 2获得的途径分别显示在表 2中。根据富集结果,显著富集FoxO singaling pathway、ErbB singaling pathway、p53 singaling pathway、Focal adhesion等。其中也不乏一些之前预测像细胞新陈代谢之类的通路,我们有理由相信通过这些蛋白质相互作用,可以得出或者预测之前不曾考虑到的斑马鱼致衰老因素[6]。
| 表 2 同源性基因的KEGG通路分析 Table 2 Analysis of the KEGG pathway of homologous genes |
由于蛋白质很少独立发挥其功能,因此通过研究蛋白质的较大功能群来了解这些蛋白质的相互作用是重要的[9-10]。所以在这项研究中,STRING在线工具被用来分析Hgs的PPIs,其综合得分> 0.15的截止标准。从以前的研究中,大多数获得的PPI网络服从无标度归因[16-18]。同时,网络的节点度被分析并用于获得PPI网络中的中枢蛋白质, 选择连接度>35作为中枢节点,并预测可能在斑马鱼的衰老研究进展中占重要比重。STRING工具被用来获得Hgs的PPI关系[11-12]。综合得分> 0.4时,共获得767个PPI关系。在滤除了度数≤5的节点后,我们最终构建了包含78个节点和478个边的网络(见图 1)。计算PPI网络各节点的连通度,输出见图 2。高连接度>35的这些基因(kita、mapk3、mapk1、ptneb、kras、mtor、jak2b、hrasb、sirt1、myca、pickc3、arf1)被选为影响斑马鱼衰老的重要因素。
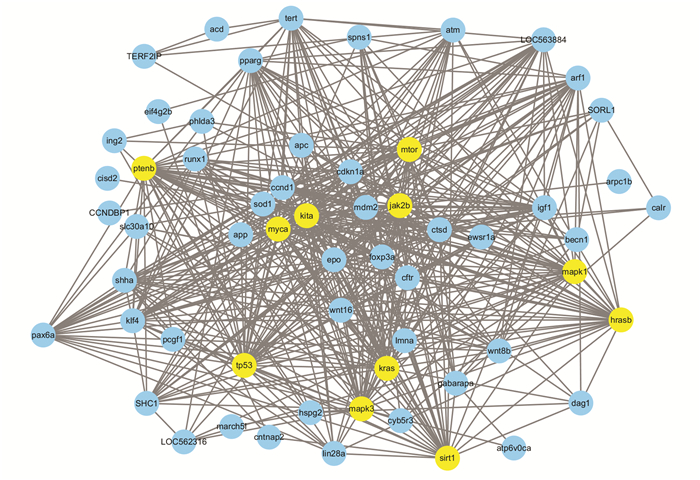
|
图 1 通过STRING在线工具获取网络图,并对选定的一些中心基因进行标记 Figure 1 Network map obtained through the STRING online tool with some selected central genes marked |
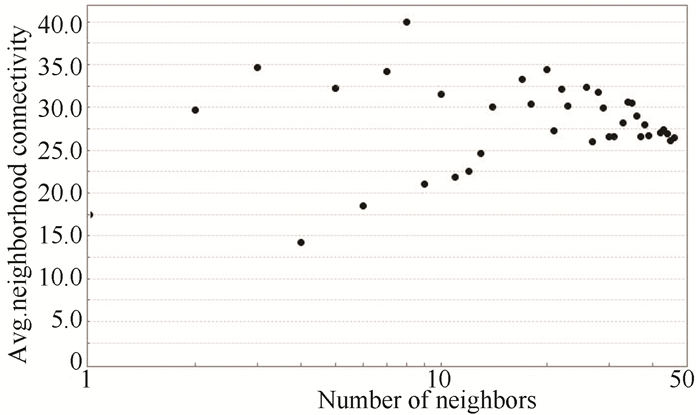
|
图 2 PPI网络中各节点的连通度 Figure 2 Connectivity of each node in PPI network |
由于PPI网络的节点和边缘非常复杂,我们需要使用ClusterONE Cytoscape插件进行富集分析。在运行ClusterONE Cytoscape以披露PPI网络的丰富功能模块之前,设置参数最小尺寸> 5且最小密度 < 0.05。我们还对模块基因进行了GO(基因本体论)功能富集分析,以分析分子水平上的基因功能。此外,我们还使用DAVID进行最佳富集模块KEGG通路富集分析,同时进行了Kmeans聚类。PPI网络富集是研究和鉴定功能蛋白质的主要方法之一[23]。在这项研究中,有8个重要模块(p值< 1×10-3)通过ClusterONE Cytoscape插件进行富集,参数最小> =5,最小密度 < 0.05。最重要的富集模块A(p=1.0×10-3),模块B(p=9.083×10-5)和模块C(p=1.2×10-3)如图 3所示。根据图 3,显然模块A可能是最好的模块,因为它有42个节点和487条边,而模块B有10个节点和44条边,模块C有7个节点和21条边[15]。为了进一步研究肿瘤进展过程中的功能变化,我们在模块A中进行了GO功能注释[19-20]。模块A的GO富集分数分别为18.28。因此,模块A可能是进一步功能分析最合适的模块。模块A中有42个基因(见图 3),它们和之前我们所假设的中枢基因不谋而合,所假设的中枢基因也在图一中标注出来。因此模块A可能是进一步功能分析最合适的模块。模块A有42个基因,它们在细胞老化、细胞周期、代谢过程的调节等生物过程功能富集,然后通过KEGG富集分析研究结果。
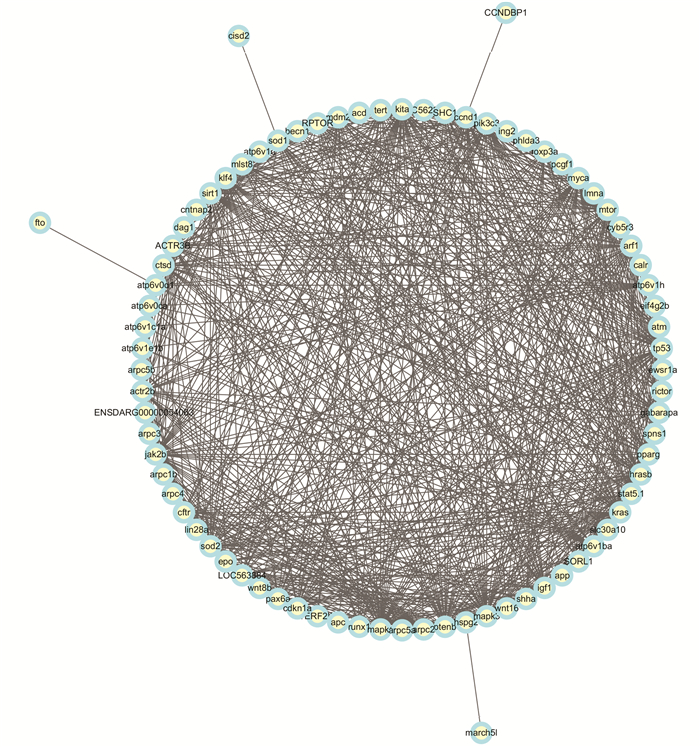
|
图 3 Hgs的PPI模块化分析中的模块A Figure 3 Module A in PPI modular analysis of Hgs |
本项目介绍了斑马鱼这种模式生物衰老机制,涵盖了衰老相关的87个蛋白质之间的767个相互作用。这不仅包含准确预测的PPI,还包含从文献收集以及实验所得的那些分子相互作用。然后进行网络化,找到中枢基因(mapk1、mtor、tp35、kita、pik3cs、myca、sirt1、kras、mapk3、arf1、jak2b),与后面的模块富集得出的重要基因不谋而合。在对斑马鱼衰老过程进行模块A分析的过程中,得出生物调节、生物过程、分子代谢过程等功能富集,说明端粒磨损、细胞老化对斑马鱼衰老过程中起到重要的作用[21-22]。同时,参考López-Otín等人(2013年)描述衰老与生物体的基本功能相关的九个主要参数,并且通常与衰老过程相关,进一步佐证我们的预测。
6 讨论与展望斑马鱼已成为脊椎动物基因功能研究的热门生物。该物种几乎透明的胚胎以及通过基因敲低或过表达加速基因研究的能力已经导致斑马鱼在脊椎动物基因功能的详细研究中的广泛使用,并且越来越多地研究人类遗传疾病[27-29]。但是,为了有效模拟人类遗传疾病,了解斑马鱼基因和基因结构与直系同源人类基因的相关程度很重要。在这项研究中,根据斑马鱼与人类的同源性,从NCBI(https://www.ncbi.nlm.nih.gov/)47 036个斑马鱼基因中,选择了67个具有代表性的斑马鱼衰老相关基因。这些基因大多富含生物过程和生物调节,部分基因富含细胞代谢过程等。
通过分析网络中的功能富集,发现细胞有很多重要的生物过程[24-26]。P53是许多的肿瘤类型起着肿瘤抑制剂的作用,根据生理情况和细胞类型诱导生长停滞或细胞凋亡。作为反式激活因子参与细胞周期调控,通过控制该过程所需的一组基因来负向调节细胞分裂。其中一个激活的基因是细胞周期蛋白依赖性激酶抑制剂。细胞凋亡诱导似乎是通过刺激bax和fas抗原表达或通过抑制Bcl-2表达来介导的。
此外,使用STRING工具获得Hgs的PPI关系,并获得了具有87个节点和767个边缘的网络。在网络中,选择kita、mapk3、mapk1、ptneb、kras、mtor、jak2b、hrasb、sirt1、myca、pickc3、arf1作为集线器节点,因为它们的连通度> 35。最后,我们进行了PPI网络的模块分析。含有42个基因的模块A被证明与斑马鱼衰老密切相关[14-15]。通过GO分析,模块A中的基因在生物调节、生物过程, 分子代谢过程等功能富集,说明端粒磨损、细胞老化对斑马鱼衰老过程中起到重要的作用。
7 结论作为研究的结果,我们证实斑马鱼衰老机制与FoxO singaling pathway、ErbB singaling pathway、p53 singaling pathway、Focal adhesion、分子代谢过程有关。此外,我们还发现mapk1、mtor、tp35、kita、pik3cs、myca、sirt1、kras、mapk3、arf1,jak2b等基因可能在衰老机制中发挥重要作用,并且它们是预测的源基因。
| [1] |
刘成龙, 武乔乔, 于滨, 等. 蛋白质对玉米淀粉理化特性的影响[J]. 齐鲁工业大学学报, 2018(6): 29-33. LIU Chenglong, WU Qiaoqiao, YU bin, et al. Effect of protein on physicochemical properties of corn starch[J]. Journal of Qilu University of Technology, 2018(6): 29-33. DOI:10.16442/j.cnki.qlgydxxb.2018.06.006 (  0) 0) |
| [2] |
刘桂霞, 王沫沅, 苏令涛, 等. 基于深度神经网络的蛋白质相互作用预测框架[J]. 吉林大学学报(工学版), 2019(2): 1-8. LIU Guixia, WANG Moyuan, SU Lingtao, et al. Prediction framework of protein interaction based on deep neural network[J]. Journal of Jilin University (Engineering), 2019(2): 1-8. DOI:10.13229/j.cnki.jdxbgxb20171101 (  0) 0) |
| [3] |
滕文静, 李洪霖, 李雁. 基于Cytoscape等生物信息学分析软件预测荜茇明碱抗肿瘤靶基因[J]. 广州中医药大学学报, 2017, 34(5): 785-789. TENG Wenjing, LI Honglin, LI Yan. Based on bioinformatics analysis software such as Cytoscape predict piperlongumine antitumor target genes[J]. Journal of Guangzhou University of Chinese Medicine, 2017, 34(5): 785-789. DOI:10.13359/j.cnki.gzxbtcm.2017.05.035 (  0) 0) |
| [4] |
吴卓. 基于Cytoscape软件对人类疾病网络图的构建与关联性分析[J]. 周口师范学院学报, 2017, 34(5): 14-16. WU Zhuo. Construction and correlation analysis of Cytoscape software on human disease network graph[J]. Journal of Zhoukou Normal University, 2017, 34(5): 14-16. DOI:10.13450/j.cnki.jzknu.2017.05.004 (  0) 0) |
| [5] |
邢伟, 谢伟东, 何杰, 等. 人(Homo sapiens)海马神经元microRNA调控网络的构建及其基本性质研究[J]. 现代生物医学进展, 2013, 13(2): 201-204. XING Wei, XIE Weidong, HE Jie, et al. Construction and studies of microRNA regulatory networks in Homo sapiens hippocampal neuron[J]. Progress in Modern Biomedicine, 2013, 13(02): 201-204. DOI:10.13241/j.cnki.pmb.2013.02.001 (  0) 0) |
| [6] |
卢纯惠. 细胞突变与人类的疾病及衰老[J]. 环境保护, 1982(3): 23-24. LU Chunhui. Cell mutation and human disease and aging[J]. Environmental Protection, 1982(3): 23-24. DOI:10.14026/j.cnki.0253-9705.1982.03.009 (  0) 0) |
| [7] |
胡赛, 熊慧军, 李学勇, 等. 多关系蛋白质网络构建及其应用研究[J]. 自动化学报, 2015, 41(12): 2155-2163. HU Sai, XIONG Huijun, LI Xueyong, et al. Construction and application of multi-relationship protein network[J]. Acta Automatic Sinica, 2015, 41(12): 2155-2163. DOI:10.16383/j.aas.2015.c150187 (  0) 0) |
| [8] |
胡赛, 熊慧军, 陈治平, 等. 基于不确定网络的关键蛋白质识别[J]. 四川大学学报(工程科学版), 2014, 46(05): 116-120. HU Sai, XIONG Huijun, CHEN Zhiping, et al. Identification of key proteins based on uncertain networks[J]. Journal of Sichuan University (Engineering Science Edition), 2014, 46(05): 116-120. DOI:10.15961/j.jsuese.2014.05.047 (  0) 0) |
| [9] |
赵振华, 陈新雨, 周鹏方, 等. 酵母重复基因在蛋白质互作网络中的分歧进化[J]. 西北农林科技大学学报(自然科学版), 2010, 38(10): 165-170. ZHAO Zhenhua, CHEN Xinyu, ZHOU Pengfang, et al. Bifurcation evolution of yeast repeat genes in protein interaction networks[J]. Journal of Northwest Agricultural and Forestry University (Natural Science Edition), 2010, 38(10): 165-170, 177. DOI:10.13207/j.cnki.jnwafu.2010.10.007 (  0) 0) |
| [10] |
孙鹏飞, 张健沛. 基于量子遗传算法的蛋白质折叠结构预测[J]. 哈尔滨工程大学学报, 2010, 1(1): 92-97. SUN Pengfei, ZHANG Jianpei. A method for predicting protein folding based on a quantum genetic algorithm[J]. Journal of Harbin Engineering University, 2010, 1(1): 92-97. DOI:10.3969/j.issn.1006-7043.2010.01.016 (  0) 0) |
| [11] |
吕志鹏, 黄文奇. 求解蛋白质结构预测问题的局部搜索算法[J]. 计算机科学, 2005, 2(11): 148-151. LÜ Zhipeng, HUANG Wenqi. Local search algorithm for solving protein structure prediction problem[J]. Computer Science, 2005, 2(11): 148-151. DOI:10.3969/j.issn.1002-137X.2005.11.040 (  0) 0) |
| [12] |
王庆喜, 朱丽华. 基于布谷鸟搜索算法的蛋白质能量优化[J]. 浙江农业学报, 2017, 9(7): 1216-1220. WANG Qingxi, ZHU Lihua. Protein energy optimization based on cuckoo search algorithm[J]. Acta Agriculturae Zhejiangensis, 2017, 9(7): 1216-1220. DOI:10.3969/j.issn.1004-1524.2017.07.22 (  0) 0) |
| [13] |
张菁, 郭丹. 拟蛇算法在蛋白质折叠模拟中的应用可行性研究[J]. 软件, 2017, 8(2): 75-79. ZHANG Jing, GUO Dan. Application feasibility study on snake algorithm of protein folding simulation[J]. Computer Engineering & Software, 2017, 8(2): 75-79. DOI:10.3969/j.issn.1003-6970.2017.02.017 (  0) 0) |
| [14] |
CUI Z, ZHANG Y. Swarm intelligence in bioinformatics: Methods and implementations for discovering patterns of multiple sequences[J]. Journal of Nanoscience & Nanotechnology, 2014, 4(2): 1746-1757. DOI:10.1166/jnn.2014.9196 (  0) 0) |
| [15] |
SHIN J M, LEE B, CHO K H. A new efficient conformational search method for ab initio protein folding study:Window growth evolutionary algorithm[J]. Bulletin of the Korean Chemical Society, 2016, 37(12): 1971-1976. DOI:10.1002/bkcs.11006 (  0) 0) |
| [16] |
胥晓莎, 潘理, 叶苏杨, 等. 蛋白质相互作用时序网络模型及动态性质分析[J]. 湖南理工学院学报(自然科学版), 2008, 31(1): 16-19, 75. XU Xiaosha, PAN Li, YE Suyang, et al. Protein-protein interaction temporal network modal and its dynamics[J]. Journal of Hunan Institute of Science and Technology (Natural Sciences), 2008, 31(1): 16-19, 75. DOI:10.16740/j.cnki.cn43-1421/n.2018.01.004 (  0) 0) |
| [17] |
赵振华, 陈新雨, 周鹏方, 等. 酵母重复基因在蛋白质互作网络中的分歧进化[J]. 西北农林科技大学学报(自然科学版), 2010, 38(10): 165-170, 177. ZHAO Zhenhua, CHEN Xinyu, ZHOU Pengfang, et al. Bifurcation evolution of yeast repeat genes in protein interaction networks[J]. Journal of Northwest Agricultural and Forestry University (Natural Science Edition), 2010, 38(10): 165-170, 177. DOI:10.13207/j.cnki.jnwafu.2010.10.007 (  0) 0) |
| [18] |
WORSLEY S D, PONDER B A J, DAVIES B R. Overexpression of cyclin D1 in epithelial ovarian cancers[J]. Gynecologic Oncology, 1997, 64(2): 189-195. DOI:10.1006/gyno.1996.4569 (  0) 0) |
| [19] |
SUI L, TOKUDA M, OHNO M, et al. The concurrent expression of p27kip1 and cyclin D1 in epithelial ovarian tumors[J]. Gynecologic Oncology, 1999, 66(3): 202-209. DOI:10.1006/gyno.1999.5373 (  0) 0) |
| [20] |
GILKS C B. Subclassification of ovarian surface epithelial tumors based on correlation of histologic and molecular pathologic data[J]. International Journal of Gynecologic Pathology, 2004, 23(3): 200-205. DOI:10.1097/01.pgp.0000130446.84670.93 (  0) 0) |
| [21] |
BOWEN N J, WALKER L D, MATYUNINA L V, et al. Gene expression profiling supports the hypothesis that human ovarian surface epithelia are multipotent and capable of serving as ovarian cancer initiating cells[J]. BMC Med Genomics, 2009(2): 71. DOI:10.1186/1755-8794-2-71 (  0) 0) |
| [22] |
RITCHIE M E, SILVER J, OSHLACK A, et al. A comparison of background correction methods for two-colour microarrays[J]. Bioinformatics, 2007, 23(20): 2700-2707. DOI:10.1093/bioinformatics/btm412 (  0) 0) |
| [23] |
SCHULDT C, LAPTEV I, CAPUTO B. Recognizing human actions: A local SVM approach[A]. Proceedings of the 17th International Conference on Pattern Recognition. Piscataway: IEEE, 2004: 32-36. DOI: 10.1109/ICPR.2004.133 2004.ICPR 2004.
(  0) 0) |
| [24] |
VON MERING C, HUYNEN M, JAEGGI D, et al. STRING: A database of predicted functional associations between proteins[J]. Nucleic Acids Research, 2003, 31(1): 258-261. DOI:10.1093/nar/gkg034 (  0) 0) |
| [25] |
KANEHISA M, ARAKI M, GOTO S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Research, 2007, 36(suppl_1): D480-D484. DOI:10.1093/nar/gkm882 (  0) 0) |
| [26] |
KANEHISA M, GOTO S, KAWASHIMA S, et al. The KEGG resource for deciphering the genome[J]. Nucleic Acids Research, 2004, 32(suppl_1): D277-D280. DOI:10.1093/nar/gkh063 (  0) 0) |
| [27] |
O'CONNOR B P. SPSS and SAS programs for determining the number of components using parallel analysis and Velicer's MAP test[J]. Behavior Research Methods, Instruments, & Computers: A Journal of the Psychonomic Society, Inc, 2000, 32(3): 396-402. DOI:10.3758/BF03200807 (  0) 0) |
| [28] |
KANEHISA M, GOTO S, FURUMICHI M, et al. KEGG for representation and analysis of molecular networks involving diseases and drugs[J]. Nucleic Acids Research, 2009, 38(suppl_1): D355-D360. DOI:10.1093/nar/gkp896 (  0) 0) |
| [29] |
SCHULDT C, LAPTEV I, CAPUTO B. Recognizing human actions: A local SVM approach[A]. Proceedings of the 17th International Conference on Pattern Recognition. Piscataway: IEEE, 2004: 32-36. DOI: 10.1109/ICPR.2004.133446.
(  0) 0) |
 2019, Vol. 17
2019, Vol. 17


