2. 贵州医科大学 基础医学院,贵阳 550025;
3. 同济大学 医学院附属上海市肺科医院 上海市结核病重点实验室,上海 200433;
4. 贵州医科大学 公共卫生学院,贵阳 550025
2. School of Basic Medical Sciences, Guizhou Medical University, Guiyang 550025, China;
3. Shanghai Key Laboratory of Tuberculosis, Shanghai Pulmonary Hospital, Tongji University School of Medicine, Shanghai 200433, China;
4. School of Public Health, Guizhou Medical University, Guiyang 550025, China
结核病是由结核分枝杆菌(Mycobacterium tuberculosis, Mtb)引起的一种与机体细胞免疫应答密切相关的慢性消耗性传染病,宿主免疫细胞可通过其表面受体识别结核分枝杆菌抗原蛋白中特殊的化学基团,即表位(Epitope)而刺激机体产生免疫应答,但其机制和宿主免疫保护机制仍不清楚[1]。因此,探索结核分枝杆菌蛋白抗原表位的免疫学特性,对结核分枝杆菌致病机制的研究和表位疫苗的研发具有重要意义[2]。越来越多的研究显示,结核分枝杆菌在体内生长必需的基因及其产物可作为有效的药物研发靶点和候选疫苗[3]。
核苷酸是结核分枝杆菌生长和分化的必需物质,主要由从头合成途径产生[4]。其中,乳清酸核苷-5’-磷酸脱羧酶(Orotidine 5’-monophosphate decarboxylase, OMPdecase; Rv1385)是嘧啶核苷酸从头合成途径最后一步反应的酶,催化乳清酸核苷-5’-磷酸脱羧生成尿嘧啶核苷酸[5]。尿嘧啶核苷酸是合成其他嘧啶核苷酸的前体,对促进结核分枝杆菌的分化和增殖具有重要作用[6]。
Rv1385蛋白由结核分枝杆菌pyrF基因编码,包含274个氨基酸残基,富含丙氨酸、甘氨酸和缬氨酸等疏水性氨基酸,其相对分子质量为27.4 KD。生物信息学分析显示,Rv1385蛋白不含信号肽,亚细胞定位可能为细胞膜或细胞浆[7]。迄今,对Rv1385的抗原性研究尚无报道,本研究应用生物信息学的方法对Rv1385的抗原性进行预测分析,以期为后续深入研究结核分枝杆菌的致病机制和新的药物靶点奠定基础。
1 材料与方法 1.1 氨基酸序列从NCBI数据库检索获得结核分枝杆菌(Mycobacterium tuberculosis, Mtb)H37Rv及15株M. tuberculosis菌株的Rv1385蛋白氨基酸序列,序列号分别为:AJF02736、WP_031708180.1、WP_031734789.1、WP_017487696.1、WP_118962198.1、WP_031658710.1、WP_016720430.1、WP_070892765.1、WP_055366474.1、WP_052635252.1、WP_057361138.1、WP_055375288.1、WP_116258713.1、WP_057370066.1, WP_015631297.1和WP_019283631.1。
1.2 Rv1385的二级结构预测应用在线软件PSIPRED Server (http://bioinf.cs.ucl.ac.uk/psipred/)预测该蛋白的二级结构。
1.3 Rv1385的B细胞抗原表位预测应用在线软件BepiPred 1.0 Server(http://www.cbs.dtu.dk/services/BepiPred-1.0/)和ABCpred(http://crdd.osdd.net/raghava/abcpred/ABC_submission.html)预测该蛋白的B细胞抗原表位。
1.4 Rv1385的CTL抗原表位预测应用BIMAS(https://www-bimas.cit.nih.gov/molbio/hla_bind/)、SYFPEITHI(http://www.syfpeithi.de/bin/MHCServer.dll/EpitopePrediction.htm)及NetCTLpan 1.1 Server(http://www.cbs.dtu.dk/services/NetCTLpan/)预测该蛋白的CTL表位。
1.5 Rv1385的Th抗原表位预测应用SYFPEITHI(http://www.syfpeithi.de/bin/MHCServer.dll/EpitopePrediction.htm)和NetMHCIIpan 3.2 Server (http://www.cbs.dtu.dk/services/NetMHCIIpan/)预测该蛋白的Th细胞表位。
1.6 Rv1385氨基酸序列的同源性预测应用Clustal Omega(http://www.ebi.ac.uk/Tools/msa/clustalo)对该蛋白的氨基酸序列进行同源性分析。
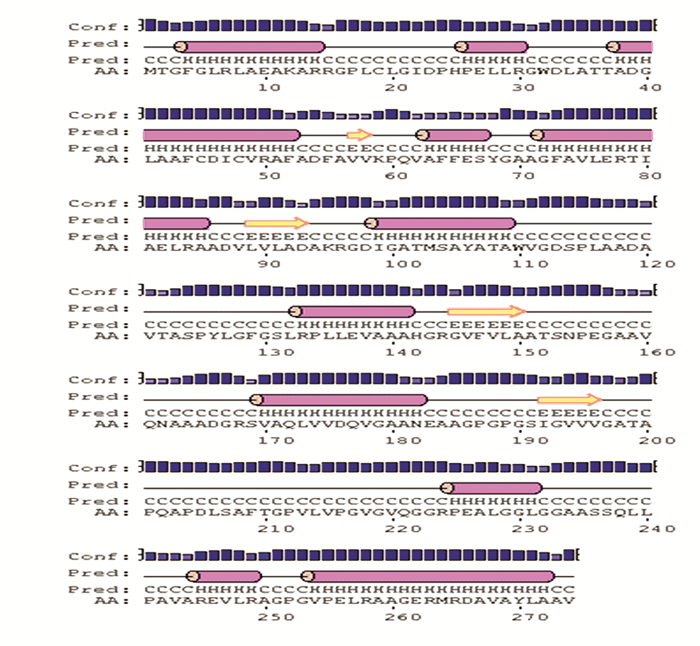
2 结果与分析 2.1 Rv1385的二级结构PSIPRED Server结果见图 1,其中α螺旋(H)、β折叠(E)、无规卷曲(C)分别占51.8%、41.6%和6.6%。
|
图 1 Rv1385二级结构预测结果 Figure 1 Predicted secondary structure of Rv1385 |
BepiPred 1.0 Server预测结果显示95~101、109~124、152~169、178~192、198~209、218~236, 249~260等氨基酸区段可能具有抗原表位;ABCpred预测具有抗原表位可能性最大的位点依次为109~124、94~109、120~135、189~204、195~210、147~162、65~80, 168~183位点。综合两种软件预测结果的分值(≥0.65),同时结合Rv1385蛋白的二级结构预测结果无规卷曲存在的区段进行分析,推测B细胞抗原表位较有可能位于109~124、152~169、178~192, 198~209等氨基酸区段。
2.3 Rv1385的CTL表位应用3种生物信息学软件预测,分值较高的CTL表位结果见表 1,综合分析,推测可能性较高的CTL表位存在于OMPdecase蛋白的下述氨基酸区段:263~271、87~95, 240~248。
| 表 1 Rv1385CTL表位预测结果 Table 1 Predicted CTL epitopes of Rv1385 |
SYFPEITHI和NetCTLpan 3.2 Server在线软件预测,分值较高的Th表位结果见表 2,综合2种软件结果推测Th表位可能位于77~91、87~101、234~247、70~84, 46~60。
| 表 2 Rv1385Th表位预测结果 Table 2 Predicted Th epitopes of Rv1385 |
运用NCBI的Blast对M. tuberculosis complex数据库的101株菌株的Rv1385进行氨基酸序列比对,结果显示一致性为81.32%~100%。然后从中选择一致性较低的16株M. tuberculosis菌株进行同源性分析,结果见图 2。

|
图 2 不同菌株Rv1385氨基酸序列的同源性分析 Figure 2 Multiple sequence alignment of amino acid sequences of Rv1385 from different strains |
结核病仍然是全球性健康性疾病,据2018年世界卫生组织的结核病报告报道,目前全世界有近1/3人口感染结核分枝杆菌,每年大约有180万人死于结核病[8]。尽管自1990年以来,结核病的发病率和新增病例有所下降,但随着多重耐药和广泛耐药结核菌株的增多,以及结核和人类免疫缺陷病毒(Human Immunodeficiency Virus, HIV)共同感染患者的出现,给结核病的防治带来巨大挑战[9]。
根除结核病的一个重要问题是需要长期的药物治疗[10-11]。其原因之一是结核病的病原菌(Mycobacterium tuberculosis,Mtb)能够进入一种非复制的持留状态,导致对传统抗结核药物的敏感性降低[12]。因此,需要新的药物来缩短疗程,同时,急切需要开发新的疫苗控制结核病的发展。为了实现这个目的,对结核分枝杆菌的生理功能研究迫在眉睫,找到一些对结核分枝杆菌在体内生长必需的基因及其产物以开发新的药物靶点和设计疫苗成为当务之急[3]。核苷酸是结核分枝杆菌生长和分化的必需物质,同时核苷酸还可参与到结核分枝杆菌的其他代谢过程,如糖代谢、脂代谢和氨基酸代谢,在结核分枝杆菌的增殖、DNA损伤修复和细胞周期等生理生化过程中发挥重要作用[4]。嘧啶核苷酸主要通过从头合成途径生成,包含6个连续的酶促反应过程[13],简述如下:氨基甲酰磷酸合成酶Ⅱ催化谷氨酰胺、HCO3-和ATP生成氨基甲酰磷酸;天冬氨酸氨基甲酰转移酶催化天冬氨酸与氨基甲酰磷酸生成氨基甲酰天冬氨酸;二氢乳清酸酶催化氨基甲酰天冬氨酸脱水环化形成二氢乳清酸;二氢乳清酸脱氢酶催化二氢乳清酸生成乳清酸;乳清酸磷酸核糖转移酶催化乳清酸与磷酸核糖焦磷酸反应生成乳清酸核苷-5’-磷酸;乳清酸核苷-5’-磷酸脱羧酶(Orotidine 5’-monophosphate decarboxylase, OMPdecase; Rv1385)催化乳清酸核苷-5’-磷酸脱羧生成尿嘧啶核苷酸。分枝杆菌中OMPdecase由274个氨基酸残基组成,相对分子质量27.4 KD[14]。在真核细胞中,编码最后两步反应的OMPdecase与乳清酸磷酸核糖转移酶的基因发生融合,使得上述两种酶位于同一条多肽链上,即尿嘧啶核苷酸合成酶[15-16],包含480个氨基酸残基。由于一级结构的差异,结核分枝杆菌和人OMPdecase的氨基酸序列同源性较低,产生交叉反应的概率较低,该酶可作为新的特异性结核诊断及抗结核疫苗的候选靶标。随着生物信息学的快速发展,生物信息学分析软件已成为预测抗原表位的常用工具,受到越来越多的科研工作者的青睐[17]。本研究运用生物信息学方法,预测分析Rv1385的B细胞和T细胞抗原表位,分析结果将为下一步寻找新的抗原靶点,为结核早期快速诊断和候选疫苗的筛选奠定基础。
4 结论Rv1385含有潜在的B细胞和T细胞抗原表位,本文结果为结核特异诊断和多表位结核疫苗的研究奠定了基础。
| [1] |
谢龙祥, 党艺方, 谢建平. 翻译后修饰与结核分枝杆菌耐药调控网络[J]. 生物工程学报, 2018, 34(8): 1279-1287. XIE Longxiang, DANG Yifang, XIE Jianping. Post-translational modification and regulatory network of Mycobacterium tuberculosis antibiotic resistance[J]. Chinese Journal of Biotechnology, 2018, 34(8): 1279-1287. DOI:10.13345/j.cjb.170530 (  0) 0) |
| [2] |
BROWN E D, WRIGHT G D. Antibacterial drug discovery in the resistance era[J]. Nature, 2016, 529(7286): 336-343. DOI:10.1038/nature17042 (  0) 0) |
| [3] |
DONINI S, FERRARIS D M, MIGGIANO R, et al. Structural investigations on orotate phosphoribosyltransferase from Mycobacterium tuberculosis, a key enzyme of the de novo pyrimidine biosynthesis[J]. Scientific Reports, 2017, 7(1): 1-11. DOI:10.1038/s41598-017-01057-z (  0) 0) |
| [4] |
WARNER D F, EVANS J C, MIZRAHI V. Nucleotide metabolism and DNA replication[J]. Microbiology Spectrum, 2014, 2(5): 1-20. DOI:10.1128/microbiolspec.MGM2-0001-2013 (  0) 0) |
| [5] |
YUAN J, CARDENAS A M, GIBERT H F, et al. Determination of the amino acid sequence requirements for catalysis by the highly proficient orotidine monophosphate decarboxylase[J]. Protein Science, 2011, 20(11): 1891-1906. DOI:10.1002/pro.728 (  0) 0) |
| [6] |
FUJIHASHI M, MNPOTRA J S, MISHRA R K, et al. Orotidine monophosphate decarboxylase - a fascinating workhorse enzyme with therapeutic potential[J]. Journal of Genetics and Genomics, 2015, 42(5): 221-234. DOI:10.1016/j.jgg.2015.04.005 (  0) 0) |
| [7] |
黄劲, 王洁, 牛雪可, 等. 结核分枝杆菌pyrF基因的生物信息学分析[J]. 中国病原生物学杂志, 2019, 14(2): 125-130, 136. HUANG Jin, WANG Jie, NIU Xueke, et al. Bioinformatics analysis of pyrF gene encoding protein orotidine 5'-monophosphate decarboxylase in Mycobacterium tuberculosis[J]. Journal of Pathogen Biology, 2019, 14(2): 125-130, 136. DOI:10.13350/j.cjpb.190201 (  0) 0) |
| [8] |
World Health Organization. Global tuberculosis report 2018[M]. Geneva: World Health Organization, 2018.
(  0) 0) |
| [9] |
袁秋露, 付玉荣, 伊正君. 结核分枝杆菌BfrB蛋白的生物信息学分析[J]. 中国病原生物学杂志, 2018, 13(2): 131-134. YUAN Qiulu, FU Yurong, YI Zhengjun. Bioinformatic analysis of Rv3841 gene from Mycobacterium tuberculosis[J]. Journal of Pathogen Biology, 2018, 13(2): 131-134. DOI:10.13350/j.cjpb.180206 (  0) 0) |
| [10] |
罗微. 结核分枝杆菌RD区在结核病诊断及疫苗中的研究进展[J]. 中华传染病杂志, 2017, 35(11): 701-704. LUO Wei. Progress of Mycobacterium tuberculosis RD region in tuberculosis diagnosis and vaccine[J]. Chinese Journal of Infectious Diseases, 2017, 35(11): 701-704. DOI:10.3760/cma.j.issn.1000-6680.2017.11.013 (  0) 0) |
| [11] |
GUPTA A, KAUL A, TSOLAKI A G, et al. Mycobacterium tuberculosis: Immune evasion, latency and reactivation[J]. Immunobiology, 2012, 217(3): 363-374. DOI:10.1016/j.imbio.2011.07.008 (  0) 0) |
| [12] |
OKOMBO J, CHIBALE K. Insights into integrated lead generation and target identification in malaria and tuberculosis drug discovery[J]. Accounts of Chemical Research, 2017, 50(7): 1606-1616. DOI:10.1021/acs.accounts.6b00631 (  0) 0) |
| [13] |
TANAKA Y, TERAMOTO H, INUI M. Regulation of the expression of de novo pyrimidine biosynthesis genes in Corynebacterium glutamicum[J]. Journal of Bacteriology, 2015, 197(20): 3307-3316. DOI:10.1128/JB.00395-15 (  0) 0) |
| [14] |
COLE S T, BROSCH R, PARKHILL J, et al. Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence[J]. Nature, 1998, 393(6685): 537-544. DOI:10.1038/31159 (  0) 0) |
| [15] |
BEN-SAHRA I, HOWELL J J, ASARA J M, et al. Stimulation of de novo pyrimidine synthesis by growth signaling through mTOR and S6K1[J]. Science, 2013, 339(6125): 1323-1328. DOI:10.1126/science.1228792 (  0) 0) |
| [16] |
LANE A N, FAN T W. Regulation of mammalian nucleotide metabolism and biosynthesis[J]. Nucleic Acids Research, 2015, 43(4): 2466-2485. DOI:10.1093/nar/gkv047 (  0) 0) |
| [17] |
CARLOS P, ROUPIE V, HOLBERT, et al. In silico epitope analysis of unique and membrane associated proteins from Mycobacterium avium subsp. paratuberculosis for immunogenicity and vaccine evaluation[J]. Journal of Theoretical Biology, 2015, 384: 1-9. DOI:10.1016/j.jtbi.2015.08.003 (  0) 0) |
 2019, Vol. 17
2019, Vol. 17


