蛋白质序列数据库的创建可以追溯到半个多世纪以前。二十世纪六十年代中期,美国国家生物医学基金会(National Biomedical Foundation, NBRF) Margaret Dayhoff领导的研究小组着手收集蛋白质序列数据,以《蛋白质序列和结构图集》(Atlas of Protein Sequence and Structure)为书名编著出版,以后又多次更新,一共出了5卷;最后一卷共470页,于1978年出版。1983年,Dayhoff不幸病逝(1925-1983),她的同事Winona Barker继续从事蛋白质序列收集和蛋白质家族分类研究。1984年,这一项目获美国国立卫生研究院(National Institutes of Health, NIH)资助,Barker和NBRF主任Robert Ledley一起,成立了蛋白质信息资源部(Protein Information Resource, PIR),开发了蛋白质资源鉴定系统(Protein Identification Resource)。该系统包括数据库和分析软件两部分,其中数据库则以蛋白质序列为主,也包括核酸序列[1]。1988年,NBRF联合德国慕尼黑蛋白质序列信息中心(Munich Information Center for Protein Sequence, MIPS)和日本国际蛋白质信息数据库(Japan International Protein Information Database, JIPID),在原有PIR的基础上成立了国际蛋白质序列数据库(PIR-International Protein Sequence Database, PIR-PSD)。PIR-PSD是当时数据量最大的蛋白质序列数据库,根据序列注释信息的不同,将所收集的蛋白质序列分为PIR1-PIR4四个不同级别[2]。
1.2 人工审阅和注释的瑞士蛋白质序列数据库Swiss-Prot1986年,瑞士日内瓦大学医学院Amos Bairoch创建了蛋白质序列数据库Swiss-Prot,并作为他博士学位论文的一部分[3]。Swiss-Prot数据库的格式借鉴1981年创建的欧洲核酸序列数据库(EMBL),其数据来源除上述PIR-PSD数据库外,还包括核酸序列数据库EMBL中编码区序列翻译得到的蛋白质序列,以及文献中收集的蛋白质序列。该数据库的特色是对序列条目进行人工审阅和注释,包括物种分类学来源、功能、定位、表达等,同时也包括与其它数据库的链接。1987年起,Swiss-Prot由日内瓦大学和位于德国海德堡的欧洲分子生物学实验室(European Molecular Biology Laboratory, EMBL)共同管理和发布。1994年,EMBL下属欧洲生物信息学研究所(European Bioinformatics Institute, EBI)在英国剑桥南郊基因组园区建立,成为仅次于美国NCBI的国际生物信息中心,欧洲分子生物学实验室负责维护的数据库移交EBI。
1.3 核酸序列翻译所得的蛋白质序列数据库TrEMBL20世纪90年代,随着核酸序列测定技术的发展,核酸序列数据激增,由核酸序列通过计算机程序翻译得到的蛋白质序列也急剧增长。1996年,欧洲生物信息学研究所Rolf Apweiler和瑞士日内瓦大学Bairoch共同创建了蛋白质序列数据库TrEMBL,作为Swiss-Prot数据库的补充和后备,专门存放核酸序列数据库EMBL中蛋白质编码序列翻译所得氨基酸序列。1998年,瑞士生物信息研究所(Swiss Institute of Bioinformatics,SIB)成立,主要负责管理、维护、发布和进一步开发Swiss-Prot数据库,而EBI主要负责管理、维护和发布TrEMBL数据库。
1.4 蛋白质数据库UniProt2002年,获美国国立卫生研究院(National Institutes of Health)和美国科学基金会(National Science Foundation)、欧盟(European Union),以及瑞士联邦政府教育和科研联合办公室等机构资助,Swiss-Prot、TrEMBL和PIR三个国际上主要蛋白质序列数据库合并,建立了通用蛋白质资源(Universal Protein Resource, UniProt),统一收集、管理、注释、发布蛋白质序列数据及注释信息[4]。UniProt的核心数据是蛋白质序列,因此也常被称为蛋白质序列数据库,或简称蛋白质数据库。目前,UniProt已经成为欧洲生命科学大数据联盟(European Life Science Infrastructure for Biological Information, ELIXIR)主要核心数据资源之一,研究开发团队共100多人,欧洲生物信息学研究所Alex Bateman、瑞士生物信息研究所Alan Bridge和美国特拉华大学(University of Delaware)生物信息学和计算生物学中心Cathy Wu共同担任项目负责人,基金资助来源包括美国和欧洲多个政府部门。UniProt从创建至今,一直遵循人类基因组计划实施时国际科学界达成的共识,即基因组、蛋白组等生物信息数据资源应该为全人类共享,为世界各国公众提供无偿服务。不言而喻,UniProt已经成为生命科学研究和生物技术开发不可或缺的蛋白质序列信息资源。
2 UniProt数据库主要内容UniProt包括三个主要部分,即蛋白质知识库(UniProt Knowledgebase, UniProtKB)、蛋白质序列归档库(UniProt Sequence Archive, UniParc)和蛋白质序列参考集(UniProt Reference Clusters, UniRef)。为适应蛋白组学研究的需要,UniProt数据库还新增了蛋白组(Proteome)和参考蛋白组数据。此外,UniProt数据库还包括文献引用(Literature Citations)、物种分类学来源(Taxonomy)、亚细胞定位(Subcellular Locations)、数据库交叉链接(Cross-reference Databases)、相关疾病(Diseases)和关键词(Keywords)等辅助数据。
2.1 蛋白质知识库UniProtKB蛋白质知识库UniProtKB是UniProt的精华,除核心数据蛋白质序列外,还包含大量注释信息[5]。这些信息是从学术文献和其它数据库中通过人工阅读和计算机提取得到的,内容包括蛋白质功能基因本体(Gene Ontology, GO)注释、物种名及分类、亚细胞定位、蛋白质加工修饰、表达等信息。此外,UniProtKB还提供与基因组、核酸序列、蛋白质结构、蛋白质家族、蛋白质功能位点、蛋白质相互作用等其它数据库的交叉链接。
UniProtKB分为Swiss-Prot和TrEMBL两个子库。两个子库序列条目分类相似,主要差别在于Swiss-Prot子库中的序列条目以及相关信息都经过手工注释(Manual Annotation)和人工审阅(Reviewed),由瑞士生物信息研究所团队负责。该团队由经验丰富的分子生物学家和生物化学家组成,专门从事蛋白质序列数据的搜集、整理、分析、注释,力图为用户提供高质量的蛋白质序列和丰富的注释信息。
TrEMBL子库由欧洲生物信息学研究所团队负责,所有序列条目由计算机程序根据一定规则进行自动注释,内容包括蛋白质名、基因名、物种名、分类学地位等基本信息,功能、表达、定位、家族和结构域等注释信息,以及与其它数据库的交叉链接。需要说明的是,TrEMBL子库中的序列未经手工注释,也未经人工审阅(Unreviewed),可靠性远不及Swiss-Prot子库中的序列,使用时需谨慎。TrEMBL和Swiss-Prot采用统一的数据库格式和登录号系统,TrEMBL中的序列经手工注释和人工审阅后,归并到Swiss-Prot子库中,不再在TrEMBL子库中保留。这两个子库的数据量差别很大,如2019年1月发布的统计报表,Swiss-Prot子库约含56万条序列,而TrEMBL子库的数据量达到1.4亿条。
2.2 蛋白质序列归档库UniParc蛋白质序列归档库UniParc是目前数据最为齐全的非冗余蛋白质序列数据库。由于数据来源、测定方法、递交时间、审阅方式和更新周期等多种原因,同一蛋白质可能存放于多个数据库,而某个数据库中收录的若干条目其序列也可能相同。为避免上述冗余问题,UniParc归档库对相同序列归并到同一个记录中,并赋予特定标识符(Unique Identifier,UPI)。特定标识符一旦赋予,就不再改变,也永不删除。UniParc定期更新,若源数据库中某个序列有了更新,可以在UniParc中查到更新记录。通过上述策略,UniParc以序列唯一性为标准,将国际上不同蛋白质序列数据库整合在一起,搜索UniParc,就相当于同时搜索这些数据库。UniParc的数据来源除UniProtKB知识库外,还包括国际核酸序列数据库(EMBL/DDBJ/GenBank)、NCBI参考序列数据库(Reference Sequence, RefSeq)、基因组数据库(Ensembl)、脊椎动物基因组注释(Vertebrate Genome Annotation, VEGA)、拟南芥等模式生物数据库、蛋白质三维结构数据库(Protein Data Bank, PDB),以及欧美、日本、韩国等蛋白质序列专利数据库,共计二十多个。2019年1月发布的UniParc归档库中,约含2.5亿个记录。
每个UniParc记录除包含特定标识符UPI、序列、循环冗余校验码(Cyclic Redundancy Check Number)等基本信息外,同时列出源数据库,包括源数据库名称、源数据库中该序列的登录号、版本号、最早收录时间和最近更新时间。不论这些序列条目源自何处,具有同一标识符的所有条目序列完全相同。若源数据库已经不复存在或源数据库中该序列条目已经不复存在,则标注为无效(Inactive)条目。以血红蛋白alpha亚基为例,其UniParc标识符为UPI0000000239,共包括214个有效条目,1 178个无效条目(2019年1月发布的版本)。通过有效条目中的登录号,可以查看源数据库中该序列条目。通过无效条目,可以追踪该序列历史,搞清该序列曾经出现在哪些数据库中。
2.3 蛋白质序列参考集UniRef蛋白质序列参考集UniRef分为三个数据集(Sequence Cluster),分别为UniRef100、UniRef90和UniRef50,其数据主要来自UniProtKB知识库,同时也包括UniParc归档库中部分条目[6]。2019年1月发布的数据,UniRef100数据集约含1.7亿个记录,UniRef90数据集记录数约为UniRef100的一半,约9千万个,而UniRef50数据集含3千多万个记录。UniRef三个数据集的构建采用一定算法,分三步进行。第一步是把不同物种中长度不小于11个氨基酸的相同序列和序列片段合并在一起,得到UniRef100数据集。第二步是按相同位点所占序列全长比例90%为阈值,将UniRef100数据集中高度相似序列合并在一起,产生UniRef90数据集。第三步则是按相同位点所占序列全长比例50%为阈值,将UniRef90数据集中具有一定相似性的序列合并在一起,所得数据集即UniRef50。
UniRef三个数据集中每个记录都有一个标识符,便于查询和比较。例如,标识符为UniRef100_P01308的记录中含4条胰岛素序列(确切地说,应该是胰岛素原前体Pre-proinsulin),3条为人胰岛素(Insulin),1条为大猩猩(Gorilla)胰岛素。若对这4条序列进行多序列比对,所有位点完全相同。需要说明的是,3条人胰岛素序列中有2条为TrEMBL中未经审阅的序列,其中1条为序列片段,长度为94 aa,而全长胰岛素原前体长度为110 aa。UniRef三个数据集的每个记录中,选取序列长度最长的一条序列作为种子序列(Seed),选择注释最为详尽的序列为代表序列(Representative)。例如,上述UniRef100_P01308记录4条序列中,种子序列为大猩猩胰岛素(登录号Q6YK33),长度为110 aa;代表序列为人胰岛素(登录号P01308)。
UniRef90_P01308记录中包含11条序列,其相同位点比例不低于90%。除上述UniRef100_P01308中的4条序列外,其它7条序列来自6种灵长类动物:倭黑猩猩(Pygmy Chimpanzee)、婆罗洲猩猩(Bornean Orangutan)、苏门答腊猩猩(Sumatran Orangutan)、白颊长臂猿(Northern White-cheeked Gibbon)、白颈白眉猴(Sooty Mangabey)、金丝猴(Golden Snub-nosed Monkey),以及1种非灵长类动物树鼩(Tree Shrew)。这11条序列中,种子序列为来自UniParc的白颊长臂猿胰岛素剪接变体(Isoform1 X1),序列长度为134 aa;代表序列仍为人胰岛素(P01308)。
UniRef50_P01308记录共含79条序列,其相同位点比例不低于50%,物种分布范围则包括灵长类、啮齿类、兽类、两栖类、鱼类等多个谱系。这79条序列中,种子序列为来自UniParc的岩鸽(Rock Dove)胰岛素剪接变体,长度为139 aa;代表序列仍为人胰岛素(P01308)。
2.4 蛋白组Proteome除UniProtKB知识库、UniRef参考序列集和UniParc归档库外,自2011年9月起,UniProt又增加了蛋白组数据。英文“蛋白组”(Proteome)这一术语,由澳大利亚学者Marc Wilkins于1994年在一次学术讨论会上提出。我们知道,一个物种的基因组DNA序列只有一套,而转录组mRNA序列则不止一套,不同组织、不同发育阶段、不同环境条件的转录组均不相同。即使是同一套转录组,经过翻译后处理、修饰等,其最终产物蛋白组也很不相同。尽管目前已经可以通过质谱等手段直接测定肽段序列,并经过拼接后获得蛋白质序列,与基因组和转录组测序相比,无论是测序成本还是测序通量,仍有较大距离。UniProt数据库中的蛋白组数据,主要是指已经完成全基因组测序物种的核酸序列翻译所得的蛋白质序列。
截至2019年1月,UniProt数据库中收录的蛋白组数据共18.8万多组,每组数据都赋予蛋白组特定标识符(Unique Proteome Identifier),如大肠杆菌K12菌株的标识符为UP000000625。上述蛋白组数据绝大部分来自细菌和病毒,如大肠杆菌就有6 000多套蛋白组数据,而艾滋病毒也有近6 000套蛋白组数据。由于研究背景、测序质量、注释程度等多方面原因,同一物种不同蛋白组的数据质量也参差不齐。为此,UniProt数据库挑选测序质量较好、数据比较完整、注释比较详尽的蛋白组为参考蛋白组(Reference Proteome),目前总计有1.3万多套参考蛋白组数据。参考蛋白组通常为具有代表性的蛋白组,有的通过人工选择确定,有的则通过一定算法由计算机选择得到。大肠杆菌共三套参考蛋白组数据,分别来自K12、O157:H7和ISC11三个不同菌株,而艾滋病毒则有6套蛋白组数据,包括Ⅰ型和Ⅱ型两种不同亚型的数据。真核生物蛋白组数据远比细菌和病毒少,2019年1月发布的版本中不到1 500套。
需要说明的是,参考蛋白组中的序列条目并非都经过人工审阅,如上述大肠杆菌三个参考蛋白组的数据,K12菌株共有4 446个条目,其中4 345个条目已经人工审阅;O157:H7(蛋白组标识符UP000000558)共有5 062个条目,其中2 028个已经人工审阅;而ISC11(蛋白组标识符UP000019194)全部6 130个条目均未经人工审阅。人类基因组计划指定的模式生物酿酒酵母(Saccharomyces cerevisiae, strain ATCC 204508 / S288c, 蛋白组标识符UP000002311)所有6 049个序列条目,均已经过人工审阅。人类参考蛋白组(蛋白组标识符UP000005640)共计约7.4万个序列条目,其中约2万个已经人工审阅。
3 UniProt网站功能模块与NCBI和EBI等国际著名生物信息中心一样,UniProt网站(https://www.uniprot.org/)的用户界面简洁明了,使用十分方便[7]。主页面中用一句话说明UniProt的宗旨:为广大用户无偿提供完整的、高质量的蛋白质序列和功能信息。主页面上方列出了UniProt主要组成部分名称和简单说明,即蛋白质知识库UniProtKB及其两个子库Swiss-Prot和TrEMBL的数据量,蛋白质序列参考集UniRef,蛋白质序列归档库UniParc和蛋白组Proteome,以及文献引用、交叉数据库、物种分类学来源、疾病、亚细胞定位、关键词等主要辅助信息。主页面右侧新闻(News)专栏可供用户了解数据库更新等情况。此外,UniProt还提供了常用工具、数据下载、统计报表、数据递交、应用程序接口等多个功能模块。而该网站高级检索功能、帮助文档、以及蛋白质分子精选(Protein Spotlight),则是UniProt数据库的特色板块。限于篇幅,下面对这些功能模块只作简单介绍。
3.1 高级检索方便实用的数据库检索功能是UniProt网站最大特色之一,特别是高级检索功能。利用强大的数据库索引技术,基于数据库条目中不同字段,对数据库中大量注释信息作了索引,为快速准确查找特定信息提供了方便。值得一提的是,UniProt网站为该数据库中的不同数据集提供了统一的检索界面,点击检索框左侧下拉式菜单,即可列出所有可检索的数据集,包括UniProtKB知识库、UniRef参考序列集、UniParc归档库、Proteome蛋白组,以及文献、物种等辅助数据集,就连帮助文档也可按关键词进行检索。此外,UniProt数据库也支持基于逻辑运算的高级检索,便于用户依据序列条目注释信息进行精确检索。
3.2 帮助文档丰富的帮助文档,是UniProt数据库另外一大特色。无论是用户指南中给出的文本检索实例(Text Search),还是有关UniProt数据库的基本介绍(About UniProt),或者是常见问题解答(FAQ),以及UniProtKB用户手册(User Manual),都提供了大量数据库使用的帮助信息。而所有这些帮助信息,均在帮助页面(https://www.uniprot.org/help/)中分门别类地列出,供用户浏览;也可在主页面上方的检索框中指定检索对象为帮助文档(Help)后输入关键词进行全文搜索。
3.3 在线工具UniProt数据库中提供的在线工具包括数据库相似性搜索工具(Blast)、序列比对工具(Sequence Alignment)、数据批量提取和登录号映射(Retrieve/ID Mapping)工具和多肽搜索工具(Peptide Search)。在线获得多序列比对结果后,用户可根据注释信息和氨基酸特性用不同颜色标注不同位点的序列特征信息。
3.4 数据下载UniProt所有数据均可免费下载,其数据发布基于国际知识共享(Creative Commons Attribution CCBY 4.0)许可(https://creativecommons.org/licenses/by/4.0/)。该网站数据下载页面详细列出UniProtKB、UniRef和UniParc等不同数据集专用文件下载服务器(FTP)的链接,同时包括常见问题回答(FAQ)和必读文档(README)等,为用户特别是初学者提供了有用的信息。值得一提的是,UniProt还提供基因组注释数轨(Genome Annotation Tracks),用户可用基因组浏览器(Genome Browser)查看UniProt序列条目的注释信息,为基因组研究提供了很大帮助。需要说明的是,UniProt数据库某些数据集的数据量极大,需要很大的存储空间和网络带宽,下载时须谨慎。
3.5 统计报表UniProt网站统计报表提供了大量信息,内容十分丰富,本文第4部分专门介绍。
3.6 数据递交尽管UniProt数据库的绝大部分数据均由数据库开发团队收集,用户也可向UniProt递交序列数据,包括用质谱等方法测得的蛋白质序列和用DNA测序所得的核苷酸序列,后者在存放到数据库前由计算机自动翻译成蛋白质序列。此外,UniProt鼓励用户对数据库中的条目提交校正和更新信息,以提高数据库注释质量。
3.7 应用程序接口UniProt网站提供的应用程序接口(Application Programming Interface, API),为通过计算机程序查询和获取UniProt数据库中的序列或各种注释信息提供了方便。通过基于表征性状态转移规范(Representational State Transfer, REST)的网页访问应用程序接口,既可访问单个序列条目,也可批量访问多个序列条目;既可通过网页地址直接访问某个序列条目,也可通过查询语句访问指定的序列条目。UniProt帮助文档中给出了Perl, Python, RUBY, Java等计算机语言程序实例,可供用户参考。此外,UniProt还提供了利用REST应用程序接口访问序列条目在基因组上位置信息以及基因组注释信息。而利用数据库查询应用程序接口(SPARQL API),则可批量获取UniProt后台数据库中数据,构建本地数据库。此外,UniProt还提供了用于Java应用程序接口的Java程序库。
3.8 蛋白质分子精选UniProt网站另一个特色板块是科普短文蛋白质分子精选(Protein Spotlight),由Vivienne Gerritsen撰写和维护。自2000年9月起,Gerritsen从UniProtKB知识库中每月挑选一个特色蛋白质,用生动幽默的语言,讲述该蛋白质的故事,或介绍某个蛋白质的特殊功能,如绿色荧光蛋白;或描述某个蛋白质的发现过程,如胰岛素;或关注某个蛋白质背后的科学家,如血红蛋白。通过文末文献和登录号,可进一步了解该蛋白质研究背景和最新进展,在UniProtKB知识库中查看其详细注释信息。截至2019年1月,一共撰写了210篇科普短文。
4 UniProtKB统计报表 4.1 统计报表概况UniProtKB知识库通常每四周更新发布一次。每次发布新版时,同时发布Swiss-Prot和TrEMBL两个子库的统计报表(Release Statistics),除数据总量、更新情况、数据类别、物种分布等基本信息外,还列出所有注释信息更新情况,包括常规注释信息(General Annotation)、序列特征注释信息(Sequence Feature)和数据库交叉链接(Database Cross-reference)等。熟悉这些注释信息,不仅有助于了解UniProtKB知识库主要内容,而且有助于通过高级检索从数据库中快速高效地获取所需信息,有助于利用数据库条目中丰富的注释信息和数据库交叉链接,深入了解研究课题相关或感兴趣的蛋白质。
根据分工,Swiss-Prot子库原始统计报表由瑞士生物信息学研究所发布,而TrEMBL子库原始统计报表由欧洲生物信息学研究所发布,而在UniProt网站上的统计报表则是则两个原始报表的简化版(见表 1)。
| 表 1 UniProtKB知识库统计报表网址 Table 1 URLs of the UniProtKB statistics |
UniProtKB知识库每次发布新版,统计报表中均给出新增条目数和更新条目数。更新条目绝大部分为注释信息更新,仅有极少量条目的序列有所更新,如2019年1月发布的版本中,更新的36万多条序列中,仅有22条的序列信息与上一版不同。
最近几年,随着DNA测序技术的不断改进,测序成本快速下降,全基因组测序已经成为基因组学和基础医学等研究的常规手段。因此,TrEMBL数据库中数据量按指数级别快速增长。由于同一细菌的不同菌株由不同国家、不同研究机构的不同测序结果翻译成蛋白质序列后,都存放到TrEMBL数据库中,而绝大部分细菌不同菌株的同一个基因编码的蛋白质序列相同,这就带来了数据库冗余的问题。例如,2015年2月发布的TrEMBL子库约含1 700个结核分枝杆菌(Mycobacterium tuberculosis)的近600万条序列。数据库的高度冗余,不仅增加了维护管理成本,也不便于用户查询、搜索。为此,自2015年4月起,TrEMBL对上述冗余数据进行适当处理,去除了约4 700万条冗余数据,整个数据库容量减少约一半。
4.2 数据类别需要特别注意的是,UniProtKB知识库中并非所有条目都具有蛋白质存在证据(Protein Existence)。所谓蛋白质存在证据,是指已经通过实验手段分离纯化获得该蛋白质。即使是人工审阅的Swiss-Prot子库,其中大部分条目也是由计算机推断所得,人工审阅过程仅为审阅计算机推断信息是否可靠,并不意味经实验手段进行验证。计算机推断则包括根据转录本推断、根据同源序列推断和从头预测三类(见表 2)。具有编码某蛋白质的mRNA序列的条目称为具有转录水平证据,而直接从DNA序列推断得到的序列又根据是否具有已知同源序列分为同源推断和从头预测两类。这几类不同蛋白质存在证据的条目所占比例在Swiss-Prot和TrEMBL两个子库中很不相同。
| 表 2 UniProtKB知识库数据类别 Table 2 Dataset type of the UniProtKB |
此外,Swiss-Prot子库中尚有部分存疑序列(Uncertain)。2009年1月发布的版本共包括1 834条存疑序列,列在前四位的699条来自酿酒酵母、576条来自人、117条来自拟南芥、108条来自大肠杆菌K12株。这些条目通常带有“假想蛋白”(Putative Protein)或“未鉴定蛋白”(Uncharacterized Protein)等注释信息,在功能注释栏目下还有“警示”(Caution)信息,说明该蛋白质序列有可能由假基因翻译得到,或来自不太可靠的预测结果。为慎重起见,对这些已经收录的存疑序列,在没有确定的证据前,一般仍加以保留。
4.3 物种分类学来源物种分类学来源是蛋白质序列最基本的注释信息之一。UniProtKB知识库中绝大部分序列条目都包含物种分类学来源信息,Swiss-Prot和TrEMBL两个子库统计报表中分别以饼状图方式给出物种分布大体情况。值得注意的是,无论是人工审阅序列还是未经审阅序列,均以细菌序列居多,在两个子库中均占一半以上。真核生物序列在Swiss-Prot子库中约占三分之一。除细菌和真核生物外,其余为古菌(Archaea)和病毒序列,比例较小,各占5%左右。真核生物序列又分为动物、植物、真菌三大类,而动物来源的序列又细分为哺乳动物、昆虫和其它后生动物(Metazoa)。
通过Swiss-Prot子库原始统计报表,还可以进一步查询某些物种具体序列条目数。例如,人的序列最多,共2万多条,约占Swiss-Prot子库总量的3.5%,其次是小鼠和拟南芥,均超过1.5万条,而大鼠、斑马鱼、果蝇、线虫、酵母以及水稻等其它模式生物的序列条目数也均超过3 000(见表 3)。值得一提的是,大肠杆菌(Escherichia coli)、枯草杆菌(Bacillus subtilis)和结核分枝杆菌(Mycobacterium tuberculosis)的基因组均远小于真核生物,编码的蛋白质总数也仅几千,但由于这三种细菌在分子生物学、工业生产和人类疾病研究中的重要性,Swiss-Prot注释团队对它们“情有独钟”,收录的序列条目数均名列前茅,大肠杆菌和结核分枝杆菌各有两个菌株的序列条目数排在前20位。
| 表 3 UniProtKB知识库Swiss-Prot子库中数据条目数前20位的物种 Table 3 The first 20 species in the UniProtKB/Swiss-Prot based on entry numbers |
UniProtKB知识库对收录的蛋白质序列长度进行了分布统计,并以直方图形式展示(见图 1)。我们知道,生物多样性是生命与非生命的重要区别之一,而生物多样性的基础很大程度上取决于种类繁多、大小不一的蛋白质。UniProtKB知识库中,长度为100-500 aa的序列数目最多,长度为500-2 000 aa的序列数目随长度增加而逐渐减少,而长度为2 000-4 000 aa的序列则更少了。Swiss-Prot子库中长度超过4 000个氨基酸的序列有300多个,其中分子量最大的是肌联蛋白(Titin)。小鼠的肌联蛋白(登录号A2ASS6)共35 213个氨基酸,由300多个结构域组成,包括144个免疫球蛋白类结构域(Ig-like)、132个Ⅲ型纤维连接蛋白(Fibronectin type-Ⅲ),以及多个重复序列片段。把肌联蛋白比作蛋白质分子中的“巨无霸”一点也不过分,它是脊椎动物横纹肌的重要组成部分,与肌肉收缩有关。人的肌联蛋白(登录号Q8WZ42)仅次于小鼠肌联蛋白,共34 350个氨基酸。
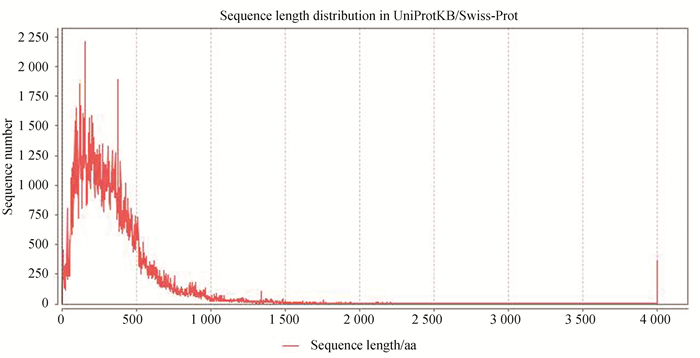
|
图 1 UniProtKB知识库Swiss-Prot子库序列长度分布(2019年1月) Figure 1 The sequence length distribution of UniProt/Swiss-Prot (released in Jan 2019) 注:X-轴为序列长度,Y-轴为序列条目数,长度大于4 000 aa的序列条目合并显示。 |
需要说明的是,长度小于50个氨基酸(50 aa)的序列习惯上称为肽(Peptide)或多肽(Polypepetide)。Swiss-Prot子库中将近1万条序列的长度小于50 aa,其中包括一大类多肽毒素,如蛇毒(Snake Toxin)、蝎毒(Scorpion Toxin)、蜘蛛毒素(Spider Toxin)、芋螺毒素(Conotoxin)等。上世纪九十年代以来,湖南师范大学梁宋平教授课题组一直从事蜘蛛毒素序列、结构和功能研究[8]。他们从我国广西、海南等丘陵地带特有的捕鸟蛛(Chinese Bird Spider)毒液中分离纯化得到一系列多肽类毒素,测定了它们的氨基酸序列和核磁共振溶液构象。UniProtKB知识库中收录的海南捕鸟蛛(Cyriopagopus hainanus)和虎纹捕鸟蛛(Cyriopagopus schmidti)多肽类毒素就达300多个。以九十年代初最早测定序列和溶液构象的虎纹捕鸟蛛毒素-I(Huwentoxin-I)为例(UniProt登录号P56676),编码该多肽毒素的mRNA序列全长458 bp(GenBank登录号AY263711),编码区序列243 bp(71-313 bp),共编码长度为81个氨基酸的虎纹捕鸟蛛毒素-I前体原(Pre-prohuwentoxin-I),在N-端1-24 aa信号肽的引导下,分泌到细胞外间质中,信号肽切除后折叠成具有一定构象但没有活性的虎纹捕鸟蛛毒素-I前体(Prohuwentoxin-I);而在捕猎时切除第22-48 aa共27个氨基酸,剩下的C-端33个氨基酸(49-81 aa)即为最终产物虎纹捕鸟蛛毒素-I。这种蜘蛛体型足有手掌大小,能捕食鸟类、青蛙等小动物,通过鳌爪将毒素注入猎物体内,阻断其细胞膜上的钙离子通道,抑制神经肌肉信号传递。
4.5 氨基酸含量尽管地球上的物种多种多样,无论是动物还是植物,无论是细菌还是病毒,组成其蛋白质的基本单元为氨基酸。氨基酸种类繁多,常见的有20种,而20种氨基酸的含量在蛋白质中所占比例相去甚远(见表 4),有些氨基酸的含量接近平均水平5%,如脯氨酸、苏氨酸,有的则远高于平均水平,接近10%,如亮氨酸、丙氨酸,而有的则含量偏低,如色氨酸仅1%,半胱氨酸仅1.3%。
| 表 4 UniProtKB知识库中20种氨基酸含量百分比 Table 4 Distribution percentage of 20 amino acids in UniProtKB |
如前文所述,除基本信息外,UniProtKB知识库统计报表中还提供了大量注释信息统计数据。注释信息主要包括两大类,一类是基于整条序列的常规注释信息,如功能、表达、亚细胞定位等。表 5列出2019年1月发布的Swiss-Prot子库中所有常规注释信息的中英文名称和数量,及其在数据库中条目数和占数据库条目总量比例。例如,2019年1月发布的Swiss-Prot子库条目总数为559 077,其中445 565条序列具有功能注释,占数据库条目总数的83%,注释信息共466 212条(有些条目的注释信息不止一条)。
| 表 5 UniProtKB知识库Swiss-Prot子库中常规注释信息统计表 Table 5 Statistics of general annotation in UniProt/Swiss-Prot |
UniProtKB知识库中另一类注释信息不是基于整条序列或整个蛋白质,而是基于序列特定区域或特定位点,因此也称序列特征注释信息。序列特征注释信息共分以下七大类。
(1) 分子加工(Molecular Processing)
包括信号肽(Signal Peptide)、转移肽(Transition Peptide)、前体肽(Propeptide)、N-末端甲硫氨酸等。
(2) 序列区域(Region)
包括结构域(Domain)、序列模体(Motif)、重复序列(Repeat)、无规卷曲(Coiled Coil)、跨膜螺旋(Transmembrane)、锌指结构(Zinc Finger)、DNA结合区(DNA Binding)、核苷酸结合区(Nucleotide Binding)、钙结合区(Calcium Binding)等。
(3) 序列位点(Site)
包括活性位点(Active Site)、金属结合位点(Metal Binding)等。
(4) 氨基酸修饰(Amino Acid Modification)
包括二硫键(Disulfide Bond)、糖基化(Glycosylation)、脂质化(Lipidation)、交联键(Cross-link)、非标准氨基酸(Non-standard Residue)等。
(5) 天然变异(Natural Variations)
包括天然突变位点和选择性剪接产物(Alternative Splicing)。
(6) 实验信息(Experimental Information)
包括突变(Mutagenesis)、非连续氨基酸(Non-adjacent Residues)、非末端氨基酸(Non-terminal Residues)、存疑序列(Sequence Uncertainty)、矛盾序列(Sequence Conflict)等。
(7) 二级结构(Secondary Structure)
包括alpha螺旋(Alpha Helix)、beta折叠(Beta Sheet)、beta回折(Beta Turn)。
4.8 数据库交叉链接除上述常规注释信息和序列特征注释信息外,UniProtKB知识库中序列条目与其它数据库的交叉链接则是另外一类重要注释信息。
生物信息数据库种类繁多,内容千差万别,数据量大小、数据质量也参差不齐。据中国科学院基因组研究所大数据中心构建的“生物数据库目录”(Database Commons)网站(http://bigd.big.ac.cn/databasecommons/)不完全统计,国际上已经发表的生物数据库共计4 500多个。1996年起,英国牛津大学出版社出版的《核酸研究》半月刊(Nucleic Acids Research, NAR)于每年第一期出版专集,专门刊登有关生物信息数据库论文。2009年,牛津大学出版社创刊的网络杂志《生物数据库和审编》(The Journal of Biological Databases and Curation, JBDC)上线,专门发表生物信息数据库相关论文。除上述两个杂志外,牛津大学出版社出版的《生物信息学》(Bioinformatics)和《生物信息学简报》(Briefings in Bioinformatics)、英国生物医学核心期刊出版集团(Biomed Central, BMC)出版的《BMC生物信息学》(BMC Bioinformatics)等杂志也不定期刊登生物信息数据库相关论文。
为便于用户快速查看某个蛋白质在其它数据库中的信息,UniProtKB知识库中收录了100多个重要生物信息数据库,并通过序列条目中的数据库交叉链接,直接查看该数据库中有关该蛋白质的信息,如蛋白质编码基因序列、基因组定位、蛋白质代谢通路、蛋白质相互作用、蛋白质三维结构、蛋白质表达、亚细胞定位、蛋白质家族和结构域、演化和系统发生等。UniProtKB知识库统计报表中将上述100多个数据库分为以下几类,每类包括几个或十几个各具特色的数据库。
(1) 序列数据库(Sequence Databases)
包括NCBI人和小鼠共有编码序列数据库(Consensus Coding Sequences, CCDS)、NCBI参考序列数据库(RefSeq)、EBI核酸序列数据库(EMBL)等。
(2) 蛋白质三维结构数据库(3D Structure Databases)
包括国际蛋白质结构数据库(Protein Data Bank, PDB)、EBI蛋白质结构概览(PDBSUM)、国际蛋白质结构模型数据库(Protein Model Portal)、瑞士生物信息研究所蛋白质结构同源模型数据库(Swiss Model Repository, SMR)等。
(3) 蛋白质相互作用数据库(Protein-protein Interaction Databases)
包括国际模式生物基因和蛋白质相互作用数据库(The Biological General Repository for Interaction Datasets, BioGRID)、EBI生物大分子相互作用数据库(Molecular Interaction Database, IntAct)和手工注释的生物大分子复合物数据库(Complex Portal)、欧洲分子生物学实验室(European Molecular Biology Laboratory, EMBL)与瑞士生物信息学研究所等合作维护的蛋白质相互作用数据库(Search Tool for Recurring Instances of Neighbouring Genes, STRING)、德国哺乳动物蛋白质复合物综合资源库(Comprehensive Resource for Mammalian Protein Complex, CORUM)、美国加州大学洛杉矶分校具有实验证据的蛋白质相互作用数据库(Database of Interacting Proteins)等。
(4) 化学小分子数据库(Chemistry Databases)
包括EBI药物类生物活性分子数据库(ChEMBL)、加拿大阿尔贝塔大学(University of Alberta)药物和药物靶标数据库(DrugBank)、国际基础和临床药理学会(International Union of Basic and Clinical Pharmacology, IUPHAR)和英国药理学会(British Pharmacology Society)合作构建的药理信息网站(Guide to Pharmacology)、加州大学圣地亚哥分校(UCSD)蛋白质和化学小分子结合数据库(Binding Database)等。
(5) 特殊类别蛋白质数据库(Family/Group Databases)
包括国际食品过敏特异免疫治疗联盟(Food Allergy Specific ImmunoTherapy)过敏分子数据库(Allergome)、EBI蛋白酶数据库(MEROPS)、法国艾克斯-马赛大学(Aix Marseille University)糖代谢酶(Carbohydrate-Active Enzyme Database, CAZy)和多功能蛋白质数据库(MoonDB)、法国图卢兹大学(University of Toulouse)过氧化物酶数据库(PeroxiBase)、新英格兰生物实验室(New England BioLabs)限制性内切酶数据库(REBASE)、美国加州大学戴维斯分校(UC Davis)转运蛋白分类数据库(Transporter Classification Database, TCDB)、瑞士生物信息研究所凝集素数据库(UniLectin)、加拿大康考迪亚大学(Concordia University)真菌源木质纤维素蛋白质数据库(mycoCLAP)等。
(6) 翻译后修饰数据库(Post-translational Modification (PTM) Databases)
包括蛋白质翻译后修饰数据库(iPTMNet)、蛋白质羰基化位点数据库(CarbonylDB)、蛋白质糖基化数据库(Glyconnect)、糖生物学数据库(UniCarbKB)人类去磷酸化数据库(DEPOD)等。
(7) 多态性和突变体数据库(Polymorphism Databases)
包括NCBI单核苷酸多态性数据库(dbSNP)、美国乔治-华盛顿大学癌症相关单核苷酸多态性数据库(BioMuta)等。
(8) 双向凝胶电泳数据库(2D Gel Databases)
包括瑞士双向聚丙烯酰胺凝胶电泳数据库(Swiss-2DPage)、南京大学医学院生殖相关双向聚丙烯酰胺凝胶电泳数据库(Reproduction-2DPage)、爱尔兰都柏林大学双向聚丙烯酰胺凝胶电泳数据库(UCD 2D-Page)等。
(9) 蛋白组数据库(Proteome Databases)
包括EBI蛋白质组鉴定数据库(Pride)、国际蛋白组联盟蛋白组数据库(CTDB)、苏格兰蛋白组动态百科全书(EPD)、瑞士生物信息研究所蛋白质丰度数据库(PaxDB)、德国马普所蛋白组数据库(MaxDB)、西雅图蛋白组中心肽段数据库(PeptideAtlas)、日本蛋白组数据库(jPOST)、奥地利维也纳大学蛋白组数据库(ProMex)等。
(10) 基因组注释数据库(Genome Annotation Databases)
包括EBI基因组注释平台和数据库(Ensembl)、美国加州大学圣克鲁兹分校的基因组浏览器(UCSC)、NCBI基因数据库(GeneID)、日本京都大学基因和基因组百科全书(KEGG)、国际植物基因组注释数据库(Gramene)、美国过敏和传染病研究所病原菌信息资源中心(Patric)和无脊椎动物病原菌数据库(VectorBase)等。
(11) 特殊物种数据库(Organism-specific Databases)
UniProt数据库中与特殊物种数据库的交叉链接共三十多个,其中大部分是模式生物基因组数据库,包括小鼠(MGI)、大鼠(RGD)、非洲爪蟾(Xenbase)、斑马鱼(ZFIN)、果蝇(Flybase)、线虫(WormBase);拟南芥(TAIR和Araport)、玉米(MaizeDB);酿酒酵母(SGD)、裂殖酵母(PomBase);大肠杆菌(EcoBase)、结核分枝杆菌(TubercuList)、嗜肺性军团病杆菌(LegioList)、念珠菌(CGD)、盘基网柄菌(dictyBase);丙肝病毒(euHCVdb)等。另一类是与人类健康关系密切的基因和蛋白质数据库,如人类孟德尔单基因疾病数据库(MIM)、人类基因、蛋白、疾病数据库(GeneCards)、人类蛋白质组织特异性表达(HPA)、药理遗传学和基因组数据库(PharmGKB)、比较环境毒理学数据库(CTD)、人类基因及变异与疾病相关数据库(DisGeNet)、真核生物病原菌数据库(EuPathDB)。第三类是某些特殊物种的特殊蛋白质数据库,如蜘蛛毒素(ArachnoSever)和芋螺毒素(ConoSever)。此外还包括人类和脊椎动物基因命名数据库(HGNC和VGNC)。
(12) 系统发生数据库(Phylogenomic Databases)
包括Ensembl基因树数据库(GeneTree)、EBI动物基因树(TreeFam数据库)、欧洲分子生物学实验室直系同源簇和功能注释数据库(eggNOG)、瑞士生物信息研究所直系同源基因数据库(OrthoDB)、瑞士苏黎世大学直系同源数据库(OMA)、瑞典直系同源簇数据库(inParanoid)等。
(13) 酶和代谢通路数据库(Enzyme and Pathway Databases)
包括国际生物反应和过程知识库(REACTOME)、德国酶学数据库(BRENDA)、意大利信号网络开放资源(SIGNOR)、德国海德堡生物化学反应和动力学数据库(SABIO-RK)、日本京都大学代谢通路数据库(KEGG)等。
(14) 基因表达数据库(Gene Expression Databases)
包括EBI基因表达数据库(ExpressionAtlas)、瑞士生物信息学研究所正常组织基因表达数据库(Bgee)等。
(15) 蛋白质家族和结构域数据库(Family/Domain Databases)
包括EBI综合蛋白质序列分类数据库和分析平台(InterPro)和蛋白质家族和结构域数据库(Pfam)、欧洲分子生物学实验室蛋白质结构域分类数据库和分析平台(SMART)、NCBI保守结构域数据库(CDD)、美国南加州大学蛋白组功能和演化数据库(PANTHER)、美国乔治敦大学基于全长序列的蛋白组分类数据库(PIRSF)、伦敦大学蛋白质分类数据库CATH中结构域数据库(Gene3D)、英国剑桥大学蛋白质结构和功能注释数据库(SuperFamily)、英国曼切斯特大学蛋白组指纹图谱数据库(PRINTS)、瑞士生物信息研究所蛋白质功能位点数据库(Prosite)、法国蛋白质结构域数据库(ProDom)等。
5 讨论 5.1 本文统计数据说明本文列出了许多统计数据,以便读者对UniProt数据库中不同数据集的数据量有一个大体了解,如上文提到的“Swiss-Prot子库约含56万条序列,而TrEMBL子库的数据量将近1.4亿条”。读者不必拘泥于具体数字,UniProt数据库每4周更新一次,这些统计数字随着UniProt数据库的更新而不断变化。本文初稿于2019年2月初完成,文中数据均来自UniProt数据库网站2019年1月16日发布的版本,而文章于4月底定稿时文中统计数据和最新版统计报表中有所不同。读者若需要了解不同版本的统计数据,可以查阅每个版本的统计报表。
5.2 现有蛋白质序列是个很小的子集随着基因组测序不断进展,UniProt数据库数据量快速增长。然而,目前我们所测得的序列,只是蛋白质序列空间(即所有可能序列)的一小部分。我们知道,蛋白质序列是由20种不同氨基酸组成的线性分子,以序列长度为6个氨基酸为例,理论上可有6 400万(206)种排列方式,即6 400万种不同序列;当序列长度增加到8时,则有256亿种排列方式;当序列长度增加到10时,则有1 024万亿种排列方式。而通常蛋白质序列长度远远不止10个氨基酸,理论上蛋白质序列空间是个天文数字。根据UniParc统计数据,迄今为止已收录到数据库中的蛋白组序列总数约2.5亿条,而UniRef100参考集中各不相同的序列约1.7亿条。这就是说,今天我们从地球上现存所有生物体中鉴定到的所有蛋白质序列,是蛋白质序列空间的一个很小子集。已故理论物理学和生物信息学家郝柏林先生在“基因组测序永无止境的根本原因”一文中指出:“从自然界中抽提出来的生物学符号序列,不是随机序列,而属于同等长度或更长的序列集合中的非典型序列子集合,对它们几乎要一条一条地具体研究”。言下之意,就是说自然界中实际存在的蛋白质序列,是亿万年演化的结果,而不是随机产生的[10]。
5.3 UniProt给我们的启示从统计报表可以看出,最近几年,UniProt数据库总体数据量增长很快,而Swiss-Prot子库数据量增长很慢(见表 6)。这是因为,Swiss-Prot子库所有条目都需人工审阅。
| 表 6 UniProt数据库主要统计数据 Table 6 Main statistics of different datasets in UniProt |
尽管目前瑞士生物信息研究所有一支将近四十人的数据库注释团队,仍远远不能满足需求。二十年前,已故北京大学生命科学学院教授顾孝诚曾向瑞士科学基金会建议,从我国生命科学领域选派若干博士生或博士后,加入Swiss-Prot数据库注释团队,为国际生物信息数据库资源建设作出应有贡献,也向国际同行学习数据库开发的有益经验。这一建议虽得到当时瑞士科学基金会和Swiss-Prot负责人支持,由于各种各样的原因,却始终未能实现。最近三十多年来,基因组、转录组、蛋白组和蛋白质三维结构等数据飞速增长,生命科学研究大数据时代已经到来,这些数据中的信息有待于我们挖掘,而数据库注释则是数据挖掘的基础,需要大量人力物力。十分遗憾的是,无论是国际核酸序列数据库联盟(International Nucleotide Sequence Database Collaboration, INSDC),还是国际蛋白质结构数据库(Worldwide Protein Data Bank, wwPDB),或者是UniProt国际蛋白质序列数据库,均没有我国参与,这与世界第二经济体的大国地位很不相称。为改变上述情况,中国科学院北京基因组研究所(Beijing Institute of Genomics, BIG)于2015年底成立了大数据中心(BIG Data Center, BIGD, http://bigd.big.ac.cn/)。几年来,BIGD在基因组数据汇交、整合、发布,专业数据库构建、注释等方面取得了卓有成效的进展[9]。UniProt蛋白质数据库及其前身PIR-PSQ, Swiss-Prot和TrEMBL等创建三十多年来,一直秉承为广大用户提供公益性无偿服务的宗旨,已经成为生命科学和生物工程研究开发不可或缺的宝贵资源,其成功经验值得借鉴。
5.4 后记UniProt是以蛋白质序列为核心的蛋白质知识宝库,内容十分丰富。自2001年起,笔者在北京大学生命科学学院和中国农业科学院研究生院开设《实用生物信息技术》(Applied Bioinformatics Course, ABC)研究生课程[11]。ABC是一门上机操作课(http://abc.cbi.pku.edu.cn/),UniProt数据库是本课程主要内容之一[12]。选修本课程的同学,通过高级检索从UniProtKB知识库中查找自己研究课题相关蛋白质,浏览该蛋白质注释信息,并通过数据库交叉链接,进一步查看该蛋白质及其编码基因信息,为课题实验研究提供参考。教学实践中深刻体会到,若要快速高效找到感兴趣的蛋白质,并充分利用UniProtKB中该蛋白质的注释信息,有必要了解UniProt数据库的基本内容,搞清常规注释信息、序列特征信息,数据库交叉链接等基本概念。希望本文能为生命科学和生物技术研究开发人员对UniProt数据库的使用有所裨益。限于笔者水平,对该数据库也只是有个粗浅了解,本文许多地方也只是浅尝辄止。文中谬误和遗漏之处,恳请读者发送邮件(uniprot@pku.edu.cn)指正,笔者将在正在编写的教科书中予以更正。在了解该数据库基本情况后,读者可结合课题研究实际需要,参阅UniProt网站帮助文档,阅读相关文献,边学边用、边用边学,在使用过程中逐步熟悉和用好UniProt,也欢迎读者通过邮件交流使用过程中的心得体会。
致谢 本文撰写过程中,得到了北京蛋白组研究中心朱伟民、Henning Hermjacob的帮助;感谢两名审稿人以及江志强、文可佳、周群飞、杨冬英对本文初稿所提宝贵的修改意见。
| [1] |
GEORGE D G, BARKER W C, HUNT L T. The protein identification resource (PIR)[J]. Nucleic Acids Research, 1986, 14(1): 11-15. DOI:10.1093/nar/16.5.1869 (  0) 0) |
| [2] |
BARKER W C, GEORGE D G, MEWES H W, et al. The PIR-International Protein Sequence Database[J]. Nucleic Acids Research, 1992, 20(Suppl): 2023-2026. DOI:10.1093/nar/20.suppl.2023 (  0) 0) |
| [3] |
BAIROCH A, BOECKMANN B. The Swiss-Prot protein sequence data bank[J]. Nucleic Acids Research, 1991, 19(Suppl): 2247-2249. DOI:10.1093/nar/20.suppl.2019 (  0) 0) |
| [4] |
APWEILER R, BAIROCH A, WU C H, et al. UniProt: the Universal Protein knowledgebase[J]. Nucleic Acids Research, 2004, 32(D1): D115-119. DOI:10.1093/nar/gkh131 (  0) 0) |
| [5] |
UNIPROT CONSORTIUM. UniProt: A worldwide hub of protein knowledge[J]. Nucleic Acids Research, 2019, 47(D1): D506-D515. DOI:10.1093/nar/gky1049 (  0) 0) |
| [6] |
SUZEK B E, HUANG H, MCGARVEY P, et al. UniRef: Comprehensive and non-redundant UniProt reference clusters[J]. Bioinformatics, 2007, 23(10): 1282-1288. DOI:10.1093/bioinformatics/btm098 (  0) 0) |
| [7] |
BOUTET E, LIEBERHERR D, TOGNOLLI M, et al. UniProtKB/Swiss-Prot, the manually annotated section of the UniProt KnowledgeBase: How to use the entry view[J]. Plant Bioinformatics (Part of the Methods in Molecular Biology book series), 2016, 1374: 23-54. DOI:10.1007/978-1-4939-3167-5_2 (  0) 0) |
| [8] |
梁宋平, 张云. 中国动物多肽毒素[M]. 北京: 科学出版社, 2016: 1-498. LIANG Songping, ZHANG Yun. Peptide toxins of Chinese animals[M]. Beijing: Science Press, 2016: 1-498. (  0) 0) |
| [9] |
Big Data Center Members. Database resources of the BIG Data Center in 2019[J]. Nucleic Acids Research, 2019, 47(D1): D8-D14. DOI:10.1093/nar/gkx897 (  0) 0) |
| [10] |
郝柏林. 基因组测序永无止境的根本原因[J]. 科学, 2011, 63(5): 5-10. HAO Bailin. Fundamental reason for never ending genome sequencing[J]. Science, 2011, 63(5): 5-10. DOI:10.3969/j.issn.0368-6396.2011.05.005 (  0) 0) |
| [11] |
LUO J. Teaching the ABCs of bioinformatics: A brief introduction to the applied bioinformatics course[J]. Briefings in Bioinformatics, 2013, 15(6): 1004-1013. DOI:10.1093/bib/bbt065 (  0) 0) |
| [12] |
罗静初. 实用生物信息技术课程教学实例[J]. 生物技术通报, 2015, 31(11): 102-111. LUO Jingchu. Teaching examples of applied bioinformatics course[J]. Biotechnology Bulletin, 2015, 31(11): 102-111. DOI:10.13560/j.cnki.biotech.bull.1985.2015.07.001 (  0) 0) |
 2019, Vol. 17
2019, Vol. 17

