2. 武汉大学人民医院 消化内科,武汉 430060
2. Department of Gastroenterology, Renmin Hospital of Wuhan University, Wuhan 430060, China
乳腺癌是女性最常见的恶性肿瘤,发病率逐年升高,严重威胁女性生命健康[1]。NOP56核糖核蛋白(NOP56 ribonucleoprotein, NOP56)是一种核仁蛋白,是C/D盒小核仁核糖核酸复合物(snoRNPs)的重要组成部分,在60S核糖体亚基组装中发挥关键作用,涉及rRNA前体的早中期加工[2](MIM#614154)。研究报道,NOP56表达受原癌基因c-Myc调控[3],同时,NOP56也可调节c-Myc从而促进Burkitt淋巴瘤生长[4]。NOP56可以与hPygo2蛋白结合,促进人卵巢癌细胞生长[5]。在人乳腺癌细胞中,NOP56基因敲除后细胞集落形成能力和生长能力明显降低[6]。然而,NOP56在人乳腺癌组织中的表达情况及临床意义的大数据分析尚未见报道。
通过对美国生物技术信息中心(National Center for Biotechnology Information,NCBI)的基因表达综合数据库(Gene Expression Omnibus,GEO)[7]和美国国立健康中心(The National Institude of Health, NIH)的癌症基因图集数据库(The Cancer Genome Atlas, TCGA)[8]公共数据集进行分析, 探索NOP56在人乳腺癌组织中的表达及临床病理关系,同时在K-M plotter在线数据库中研究NOP56的预后价值。进一步利用String、GSEA数据库,挖掘乳腺癌中与NOP56存在互相作用的蛋白网络,筛选与NOP56存在相互作用的基因并建立基因调控网络,WebGestalt数据库寻找靶向药物[9],为乳腺癌的分子机制研究提供新的切入点和同时进一步探讨临床靶向药物治疗的分子机制。
1 材料与方法 1.1 数据资料收集从NCBI的GEO数据库下载原始数据,通过MAS5进行标准化,乳腺癌组织登录号为GSE42568、GSE15852、GSE10780和GSE45255。从TCGA数据库下载原始数据,通过RSEM标准化。纳入的数据基本信息见表 1。
| 表 1 纳入的4套乳腺癌基因芯片基本信息 Table 1 Basic information of four GEO datasets of breast cancer |
在数据集GSE15852中,通过配对样本T检验比较乳腺癌和配对癌旁组织NOP56表达差异。在数据集GSE42568和GSE10780中,通过非配对样本T检验比较乳腺癌和正常组织NOP56表达差异;在TCGA数据库中,通过非配对样本T检验比较不同临床病理分期的乳腺癌和配对癌旁组织NOP56表达差异;在TCGA数据库中,通过非配对样本T检验比较不同临床病理分期乳腺癌组织和配对癌旁组织NOP56表达差异。
1.3 数据集筛选与临床病理学参数相关性研究TCGA数据集和GSE45255基因样本量大且附带临床信息,利用此数据集进行进一步的相关性研究。其中临床资料缺失的样本被排除,根据表达谱数据,对样本的NOP56表达进行由高到低排序, 高于信号log2中位数的样本作为高表达组,低于信号log2中位数的样本作为低表达组。
1.4 Kaplan-Meier plotter在线数据库分析利用在线数据库Kaplan-Meier plotter (http://www.kmplot.com/breast)获得的3 951例乳腺癌患者信息,以NOP56表达中位值(Median)将样本分为低表达和高表达组,利用缺省设置,在线分析NOP56对乳腺癌患者总生存期和无病生存期的预后价值。
1.5 NOP56互相作用基因筛选和功能富集分析利用String(https://string-db.org)在线数据库筛选人乳腺癌样本中与NOP56存在互作关系的蛋白质并且获得互作蛋白网络图。通过R3.5软件计算互作基因在分子功能、生物过程、细胞组分以及三者平均的语义相似性并绘制热图。利用GSEA3.0软件对上述互作基因进行富集分析。
1.6 靶向药物富集分析利用WebGestalt(http://www.webgestalt.org)在线数据库富集分析互作基因的靶向药物。
1.7 采用SPSS 20.0软件进行统计学分析生存分析采用Kaplan-Meier和log-rank检验法。临床病理参数相关性分析,组间比较采用χ2检验及Fisher确切概率法。以P<0. 05为差异有统计学意义。筛选共表达基因采用spearman和Pearson检验,spearman分数和Pearson分数均大于0.3的基因被认为有共表达关系。功能分析采用Fisher精确检验,P<0.05的功能集合被认为差异有统计学意义。
2 结果分析 2.1 NOP56在人乳腺癌组织中高表达在GEO数据库的3个独立数据集GSE42568、GSE15852和GSE10780中,与正常组织相比,NOP56在人乳腺癌组织中显著高表达。在TCGA数据库的数据集中,1 093例乳腺癌组织与112例正常组织相比,NOP56在人乳腺癌组织中显著高表达(见图 1);不同临床病理分期的乳腺癌组织与正常组织相比,NOP56均高表达(P<0.001)(见表 2)。

|
图 1 NOP56在人乳腺癌组织中高表达 Figure 1 Overexpressed NOP56 in human breast cancer |
| 表 2 分期与p值 Table 2 Stage and P |
(PRG) 状态、人表皮生长因子受体2(HER2)状态、组织学分级和肿瘤大小等临床病理参数与NOP56表达高低的相关性。结果显示,NOP56高表达在组织学分级为G3的肿瘤样本中更常见,而在分化程度较高的G1、G2肿瘤样本中,NOP56低表达更多见(P<0.001),提示NOP56高表达与乳腺癌的进展呈正相关。同时,NOP56与乳腺癌HER2状态相关,在HER2状态阳性的患者中高表达(P=0.019)。NOP56的表达与年龄、淋巴结转移、PRG状态、ER状态和肿瘤大小无显著相关性(见表 3)。
| 表 3 不同NOP56表达与GEO数据库139例乳腺癌患者临床病理参数的关系 Table 3 Correlation between NOP56 expression and clinicopathological parameters in 139 patients with breast cancer |
(PRG) 状态、人表皮生长因子受体2(HER2)状态和病理分期等临床病理参数与NOP56表达高低的相关性。结果显示,NOP56与乳腺癌HER2状态、年龄、PRG状态相关,在HER2状态阳性的患者中高表达(P=0.019),在年龄 < 55岁的患者中高表达(P < 0.001),在PRG阴性的患者中高表达(P < 0.000 1)。NOP56的表达与年龄、淋巴结转移和ER状态无显著相关性(见表 4)。GEO数据库与TCGA数据库结果一致。
| 表 4 不同NOP56表达与数据TCGA数据库库1 093例乳腺癌患者临床病理参数的关系 Table 4 Correlation between NOP56 expression and clinicopathological parameters in 1 093 patients with breast cancer in TCGA |
K-M plotter生存分析显示,NOP56高表达患者的无进展存活期(Disease free survival,DFS)和总存活期(Overall survival,OS)均明显较低表达患者短(P<0.001,见图 2)。这表明NOP56低表达与患者生存期缩短显著相关,NOP56可能成为预测乳腺癌的预后指标(见图 2)。
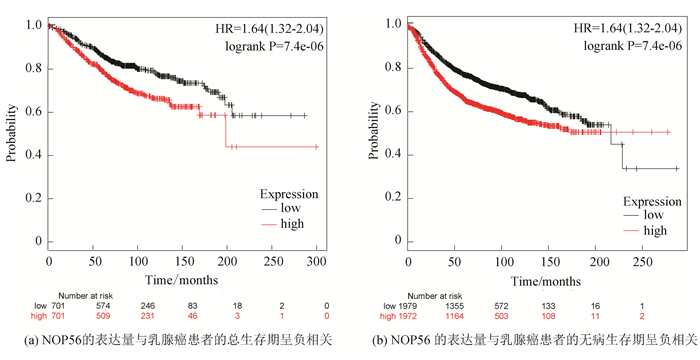
|
图 2 NOP56表达水平与乳腺癌患者生存时间关系 Figure 2 Correlation between NOP56 expression and the prognosis of breast cancer patients |
String在线数据库筛选人乳腺癌样本中与NOP56存在互作关系较强的基因10个。(见图 3(a)) R3.5进行语义相似性分析(见图 3(b)、(c)、(d)、(e))
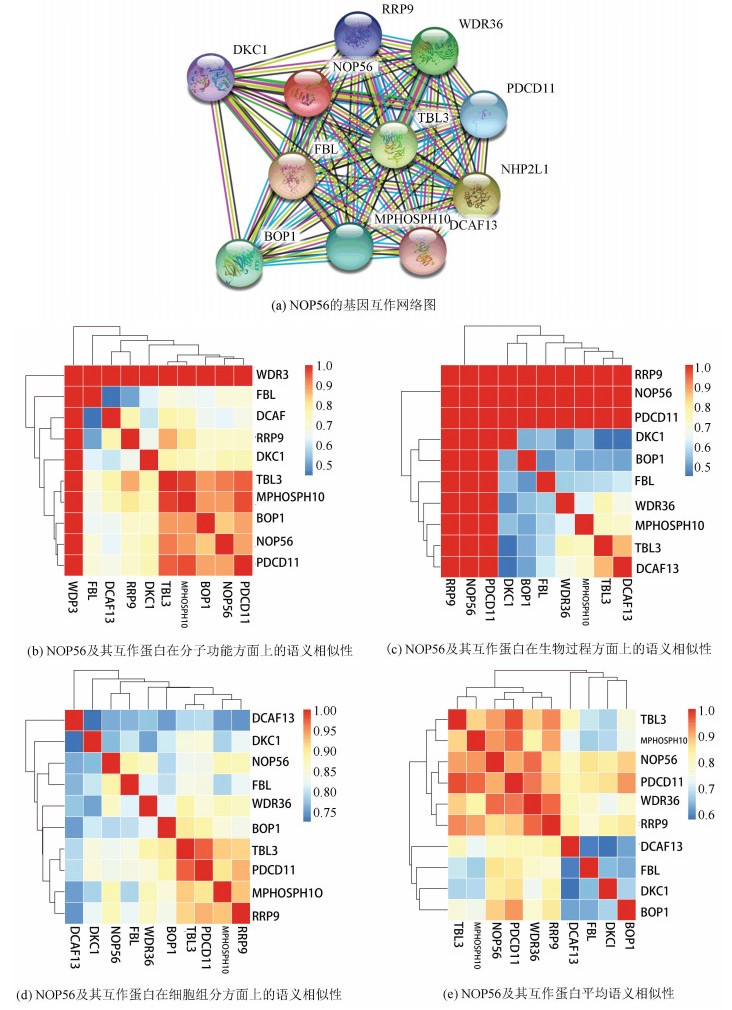
|
图 3 NOP56蛋白互作网络以及共表达基因与NOP56分子功能、生物过程、细胞组分以及平均语义相似性 Figure 3 Gene interaction network diagram of NOP56 and the molecular function, biological process, cellular component, and the mean semantic similarity between NOP56 and its co-expressed genes |
利用GSEA数据库对互作基因进行富集分析,结果显示这些基因主要富集在RNA加工,细胞分裂和DNA复制等与肿瘤发生发展关系密切的功能上(见图 4)。

|
图 4 NOP56共表达基因主要富集在RNA加工和细胞分裂等与肿瘤发生发展关系密切的功能上 Figure 4 NOP56 co-expression genes are mainly enriched in the functions closely related to the tumor development such as RNA processing and cell division |
WebGestalt网站分析显示,以上互作基因靶向药物有5种,其中互作基因作为靶基因最多的为放线菌素D(见表 5)
| 表 5 靶向基因药物 Table 5 Targeted gene drugs |
乳腺癌的发生发展离不开异常的蛋白质生物合成[6],而细胞中蛋白质的合成需求与核糖体生物合成活性相协调,当某些关键的核糖体蛋白水平上调后,可通过调节其他蛋白的表达影响细胞生长代谢[10]。研究表明,核糖体生物合成速率增加促进原癌基因Myc表达,增加癌细胞增殖能力[8],同时可通过释放MDM2蛋白降解抑癌基因p53[12]和pRb[13],增加癌症患病风险。核糖体生物合成活跃可促进乳腺癌细胞侵袭能力,增加乳腺癌恶性程度[14]。NOP56是小分子核仁核糖核蛋白(snoRNP)复合体的核心蛋白质成员,直接对rRNA前体核糖部分进行甲基化修饰,在核糖体生物合成中发挥重要作用[15]。NOP56突变可导致60S核糖体亚基装配缺陷,rRNA加工受抑[15]。研究表明,NOP56可调节原癌基因c-Myc促进Burkitt淋巴瘤细胞生长[4],同时可以与hPygo2蛋白结合,促进卵巢癌细胞生长[5]。本研究通过生物信息学分析,首次在人乳腺癌组织中发现NOP56显著高表达,且NOP56高表达与乳腺癌不良临床病理参数和预后相关。为进一步揭示其作用机制,我们探索了乳腺癌中NOP56共表达基因、互作蛋白及其功能,并构建蛋白互作网络图。
通过GEO和TCGA数据库,我们首次发现NOP56在人乳腺癌中显著高表达。在GSE45255中,数据显示NOP56高表达患者乳腺癌分化程度低。这与已有的研究一致,Su H等人发现NOP56促进人乳腺癌细胞集落形成能力和生长能力[6]。此外,我们发现NOP56在HER2阳性患者中高表达。HER2过表达与乳腺癌的发生、发展、侵袭和转移密切相关[16]。HER2基因编码酪氨酸激酶活性跨膜糖蛋白,与相应配体结合导致胞内酪氨酸激酶磷酸化,引起细胞生长、增殖相关信号通路如NF-κB等的连锁反应,促进细胞增殖分化[17]。HER2阳性乳腺癌细胞c-Myc表达水平高,敲低HER2基因后c-Myc表达降低[18],而c-Myc可直接促进NOP56表达[3]。这说明乳腺癌中HER2可能通过激活细胞生长增殖相关信号通路上调c-Myc等原癌基因从而促进NOP56表达。我们进一步利用在线数据库K-M plotter,对3 951例乳腺癌患者进行预后分析,发现NOP56高表达乳腺癌患者生存期显著降低。乳腺癌组织中NOP56表达水平,或许可以作为乳腺癌患者新的检测指标和远期预后评价指标,但还需大样本的临床数据进一步验证。
为了进一步理解乳腺癌中NOP56异常表达的机制,筛选出NOP56最显著的互作基因10个。单一的基因异常往往伴随下游基因的连锁异常反应,这是多种癌症相关基因异常促进癌症发展的重要原因之一。功能分析显示,NOP56共表达基因主要参与RNA加工,细胞分裂和DNA复制等与肿瘤发生发展关系密切的功能。这些结果与现有的实验结果一致,Victoria H.等[5]发现NOP56基因过表达后,Burkitt淋巴瘤细胞的增殖明显加快;Yuichi J.等[19]发现在乳腺癌上皮细胞中Nop56促进细胞周期G1期缩短,是细胞周期进程的重要介质。研究证明NOP56共表达网络的准确性,提示NOP56可能通过影响这些共表达基因来发挥其促癌作用。
最后,我们筛选出了NOP56互作基因富集的靶向药物,选取了富集基因最多的放线菌素类中的放线菌素D。放线菌素D(更生霉素)是第一种显示出具有抗肿瘤活性的抗生素,是临床上广泛使用的抗肿瘤药物,现在常用于各种小儿肿瘤,如肾母细胞瘤,横纹肌肉瘤和尤因氏肉瘤的治疗[20-22]。放线菌素D是一种DNA嵌入剂,它更偏向于富含GC的DNA序列[23]。Perry RP等人发现低浓度的放线菌素D可以破坏核糖体正常的生物过程,最终导致细胞周期停滞和细胞凋亡[23-25]。Das T等人研究发现放线菌素D可以下调SOX2表达,从而诱导乳腺癌干细胞死亡[26]。通过这些研究我们可以假设,放线菌素D通过靶向作用于NOP56以及其互作基因达到对乳腺癌的治疗作用,放线菌素D的治疗作用可能与NOP56的表达量相关,而这些假设还需要进一步的实验以及临床验证。
综上所述,利用公共数据库证明乳腺癌组织中NOP56高表达,NOP56促进肿瘤分化,在HER阳性患者中表达量高,且与乳腺癌患者的不良预后密切相关。机制上,我们研究了乳腺癌中NOP56共表达基因,其功能主要为RNA加工,细胞分裂和DNA复制等与肿瘤发生发展关系密切的功能,并构建基因互作网络图,发现互作基因靶向药物明显富集在放线菌素D上。系统地研究了NOP56在乳腺癌中的表达与临床预后、临床病理状态以及组织学分级的关系,探讨了分子机制以及与临床靶向药物的关系,为乳腺癌防治提供潜在的新的预测指标,完善了临床靶向药物使用的分子机制。
| [1] |
TORRE L A, BRAY F, SIEGEL R L, et al. Global cancer statistics, 2012[J]. CA: A Cancer Journal for Clinicians, 2015, 65(2): 69-90. DOI:10.3322/caac.21262 (  0) 0) |
| [2] |
LYKKE-ANDERSEN S, ARDAL B K, HOLLENSEN A K, et al. Box C/D snoRNP autoregulation by a cis-Acting snoRNA in the NOP56 pre-mRNA[J]. Molecular Cell, 2018, 72(1): 99-111. DOI:10.1016/j.molcel.2018.08.017 (  0) 0) |
| [3] |
RUGGERO D. The role of Myc-induced protein synthesis in cancer[J]. Cancer Research, 2009, 69(23): 8839-8843. DOI:10.1158/0008-5472.Can-09-1970 (  0) 0) |
| [4] |
COWLING V H, TURNER S A, COLE M D. Burkitt's lymphoma-associated c-Myc mutations converge on a dramatically altered target gene response and implicate Nol5a/Nop56 in oncogenesis[J]. Oncogene, 2014, 33(27): 3519-3527. DOI:10.1038/onc.2013.338 (  0) 0) |
| [5] |
HE Zhijian. Identification of pygopus 2 as a component of the ribosomal RNA transcription complex in cancer[D]. Newfoundland: Memorial University of Newfoundland, 2008.
(  0) 0) |
| [6] |
SU H, XU T, GANAPATHY S, et al. Elevated snoRNA biogenesis is essential in breast cancer[J]. Oncogene, 2014, 33(11): 1348-1358. DOI:10.1038/onc.2013.89 (  0) 0) |
| [7] |
BARRETT T, TROUPD B, WILHITE S E, et al. NCBI GEO: Archive for functional genomics data sets--10 year on[J]. Nucleic Acids Research, 2011, 39(Database issue): D1005-D1010. 41: D991-995. DOI: 10.1093/nar/gks1184.
(  0) 0) |
| [8] |
TOMCZAK K, CZERWINSKA P, WIZNEROWICZ M. The Cancer Genome Atlas (TCGA): An immeasurable source of knowledge[J]. Contemporary Oncology, 2015, 19(1a): A68-77. DOI:10.5114/wo.2014.47136 (  0) 0) |
| [9] |
WANG J, VASAIKAR S, SHI Z, et al. WebGestalt 2017: A more comprehensive, powerful, flexible and interactive gene set enrichment analysis toolkit[J]. Nucleic Acids Research, 2017, 45(W1): W130-w137. DOI:10.1093/nar/gkx356 (  0) 0) |
| [10] |
GENTILELLA A, KOZMA S C, THOMAS G. A liaison between mTOR signaling, ribosome biogenesis and cancer[J]. BBA. Gene Regulatory Mechanisms, 2015, 1849(7): 812-820. DOI:10.1016/j.bbagrm.2015.02.005 (  0) 0) |
| [11] |
GOMEZ-ROMAN N, FELTON-EDKINS Z A, KENNETH N S, et al. Activation by c-Myc of transcription by RNA polymerases Ⅰ, Ⅱ and Ⅲ[J]. Biochemical Society Symposium, 2006(73): 141-154. DOI:10.1042/bss0730141 (  0) 0) |
| [12] |
MONTANARO L, TRERÉ D, DERENZINI M. Changes in ribosome biogenesis may induce cancer by down-regulating the cell tumor suppressor potential[J]. Biochimica et Biophysica Acta-Reviews on Cancer, 2012, 1825(1): 101-110. DOI:10.1016/j.bbcan.2011.10.006 (  0) 0) |
| [13] |
RIDDLE V G H, PARDEE A B, ROSSOW P W. Growth control of normal and transformed cells[J]. Journal of Cellular Biochemistry, 2010, 11(4): 529-538. DOI:10.1002/jss.400110411 (  0) 0) |
| [14] |
BELIN S, BEGHIN A, SOLANOGONZÀLEZ E, et al. Dysregulation of ribosome biogenesis and translational capacity is associated with tumor progression of human breast cancer cells[J]. Plos One, 2009, 4(9): e7147. DOI:10.1371/journal.pone.0007147 (  0) 0) |
| [15] |
HAYANO T, YANAGIDA M, YAMAUCHI Y, et al. Proteomic analysis of human Nop56p-associated pre-ribosomal ribonucleoprotein complexes: Possible link between Nop56p and the nucleolar protein treacle responsible for Treacher Collins syndrome[J]. Journal of Biological Chemistry, 2003, 278(36): 34309-34319. DOI:10.1074/jbc.M304304200 (  0) 0) |
| [16] |
IRVIN W J R, MUSS H B, MAYER D K J O. Symptom management in metastatic breast cancer[J]. Oncologist, 2011, 16(9): 1203-1214. DOI:10.1634/theoncologist.2011-0159 (  0) 0) |
| [17] |
GUPTA P, SRIVASTAVA S J M O. HER2 mediated de novo production of TGFβ leads to SNAIL driven epithelial-to-mesenchymal transition and metastasis of breast cancer[J]. Molecular Oncology, 2014, 8(8): 1532-1547. DOI:10.1016/j.molonc.2014.06.006 (  0) 0) |
| [18] |
PAPANIKOLAOU V, ILIOPOULOS D, DIMOU I, et al. Survivin regulation by HER2 through NF-kappaB and c-myc in irradiated breast cancer cells[J]. Journal of Cellular and Molecular Medicine, 2011, 15(7): 1542-1550. DOI:10.1111/j.1582-4934.2010.01149.x (  0) 0) |
| [19] |
MACHIDA Y J, CHEN Y, MACHIDA Y, et al. Targeted comparative RNA interference analysis reveals differential requirement of genes essential for cell proliferation[J]. Molecular Biology of the Cell, 2006, 17(11): 4837-4845. DOI:10.1091/mbc.e06-04-0340 (  0) 0) |
| [20] |
HOLLSTEIN U J C L. Actinomycin, chemistry and mechanism of action[J]. Chemistry Letters, 1974, 74(6): 625-652. DOI:10.1021/cr60292a002 (  0) 0) |
| [21] |
MALOGOLOWKIN M, COTTON C A, GREEN D M. Treatment of Wilms tumor relapsing after initial treatment with vincristine and actinomycin D: A report from the National Wilms Tumor Study Group[J]. Pediatric Blood & Cancer, 2008, 50(2): 236-241. DOI:10.1002/pbc.21267 (  0) 0) |
| [22] |
JAFFE N, PAED D, TRAGGIS D, et al. Improved outlook for Ewing's sarcoma with combination chemotherapy (vincristine, actinomycin D and cyclophosphamide) and radiation therapy[J]. Cancer, 1976, 38(5): 1925-1930. DOI:10.1002/1097-0142(197611)38:5<1925::aid-cncr2820380510>3.0.co;2- (  0) 0) |
| [23] |
PERRY R P, KELLEY D E. Inhibition of RNA synthesis by actinomycin D: Characteristic dose-response of different RNA species[J]. Journal of Cellular Physiology, 1970, 76(2): 127. DOI:10.1002/jcp.1040760202 (  0) 0) |
| [24] |
DONATI G, PEDDIGARI S, MERCER C A, et al. 5S ribosomal RNA is an essential component of a nascent ribosomal precursor complex that regulates the Hdm2-p53 checkpoint[J]. Cell Reports, 2013, 4(1): 87-98. DOI:10.1016/j.celrep.2013.05.045 (  0) 0) |
| [25] |
HILL C R, COLE M, ERRINGTON J, et al. Characterisation of the clinical pharmacokinetics of actinomycin D and the influence of ABCB1 pharmacogenetic variation on actinomycin D disposition in children with cancer[J]. Clinical Pharmacokinetics, 2014, 53(8): 741-751. DOI:10.1007/s40262-014-0153-2 (  0) 0) |
| [26] |
DAS T, NAIR R R, GREEN R, et al. Actinomycin D down-regulates SOX2 expression and induces death in breast cancer stem cells[J]. Anticancer Research, 2017, 37(4): 1655-1663. DOI:10.21873/anticanres.11496 (  0) 0) |
 2019, Vol. 17
2019, Vol. 17


