目前幽门螺杆菌感染(Helicobacter pylori,HP)已经非常常见,平均大约50%的人群有感染[1],特别是在贫困地区,因为自然环境、饮食卫生条件、及民俗习惯等感染率会更高。HP感染者可能会导致慢性活动性胃炎,糜烂性胃炎,以及消化道溃疡等消化道疾病,甚至造成食管腺癌,胃萎缩,胃癌和结直肠癌的潜在治病因素[2]。目前对于HP感染已经有多种根治治疗方案,但由于耐药菌的增多,部分患者效果较差[3]。同时一部分感染者并不是都会出现炎性改变或者癌变,为了认识HP感染对于胃粘膜组织损伤的分子机制,设计了对于HP相关芯片的生物信息学分析。
1 材料和方法 1.1 芯片数据来源从GEO网站下载相关的基因芯片数据(GSE5081)[4]。该芯片使用微阵列比较了HP阳性和HP阴性胃糜烂和正常相邻粘膜的全基因组基因表达谱,对HP感染后mRNA表达模式进行分析。从8例HP阴性糜烂性胃窦炎患者和8例HP阳性糜烂性胃窦炎患者的冷冻活检标本中提取总RNA。同时,从所有16名患者的邻近(距离侵蚀至少3 cm)肉眼可见的正常窦腔样品取样。其中本研究涉及HP阴性患者包括三名男性患者和五名女性患者,年龄为50~88岁(中位数67.5岁)。组织学炎症程度轻度6例,中度2例。其中两人患有肠化生。本研究涉HP阳性感染者年龄为32~75岁(中位数58.5岁),5名男性和3名女性患者。组织学炎症程度轻度3例,中度1例,严重4例。其中两人有中度萎缩,一人有局灶性肠化生。在内镜检查时,没有患者接受质子泵抑制剂或抗生素治疗。所有患者属于高加索人种族。他们都没有吸烟,还有其他已知的疾病。
1.2 芯片数据生物信息分析首先芯片的质量评估[5],通过分组差异基因的筛选[6],并功能分析、注释。把组织样本分为4组,A) ER(+)HP(+), B) ER(+)HP(-), C) ER(-)HP(+), D) ER(-)HP(-), 其中HP为幽门螺杆菌,而ER为损伤粘膜组织。分别分析受损组织,健康组织组内的HP感染与否的差异基因变化,最后合并分析。具体是A、B组进行了差异分析得到分析结果差异分析1。C、D组进行了差异分析得到分析结果差异分析2(Q值< 0.05,Fold change≥1.2,阈值设定为分析软件推荐值)。将两组获得的差异基因取并集,随后进行GO分析、通路分析,以及基因共表达网络分析,基因相互作用网络分析。
1.3 实时定量PCR验证收集2016年1月至2016年6月,在解放军联勤保障部队第925医院C14尿素氮呼气试验阳性者(CPM>100)为感染者,检测阴性者为健康对照各10例,考虑诊断为慢性糜烂性胃炎,并且在内镜检查时,没有接受质子泵抑制剂或抗生素治疗,排除其他疾病。感染者与对照组一般临床特征无统计学差异,均取受损黏膜组织,在获取胃黏膜组织后,使用Trizol试剂(Invitrogen,Carlsbad,CA)提取总RNA,然后进行反转录。cDNA被适当稀释并用于PCR。使用SYBR Premix EX Taq(TAKARA)和ABI PRISM 7300实时PCR系统(Applied Biosystems,Life Technologies,Carlsbad,CA)以β-actin作为参考对照,一式三份进行实时PCR。引物由上海生工公司合成,CXCR4引物上游:5'-CCTATGCAAGGCAGTCCATGT-3',下游:5'CCTATGCAAGGCAGTCCATGT 3',内参基因β-actin引物:上游:5'-TGGCACCCAGCACAATGAA -3',下游:5'-CTAAGTCATAGTCCGCCTAGAAGCA-3'。CXCL2上游引物:CGCAGCAGGAGCGCC下游引物: TGGATGTTCTTGAGGTGAATTCC CXCL5上游引物:GGAAGGAAATTTGTCTTGATCC下游引物: TTTCCTTGTTTCCACCGTC。参与本研究的所有HP感染患者和正常人均已签署知情同意书,并通过医院伦理委员会批准。
1.4 统计方法差异基因筛选主要利用文献的常用方法(Significance Analysis of Microarray,SAM)。两组间计量资料采用使用T检验(平均数±SD),计数采用卡方检验,p < 0.05,有统计学差异。统计及分析软件使用spss23,以及GCBI在线实验室分析软件。
2 结果分析 2.1 芯片质量分析总样本数量共31个。对获得的样本,首先进行质量分析,每张芯片的RLE(Relative Log Expression)计算了芯片信号值估计在整体芯片中的相对变化率,进而反映了所检测基因的变化特征。本芯片RLE一致性较高,芯片本身质量无问题(见图 1)。质量较高的芯片,才有必要进行下一步的研究分析,否则需要舍去不合格的样本。
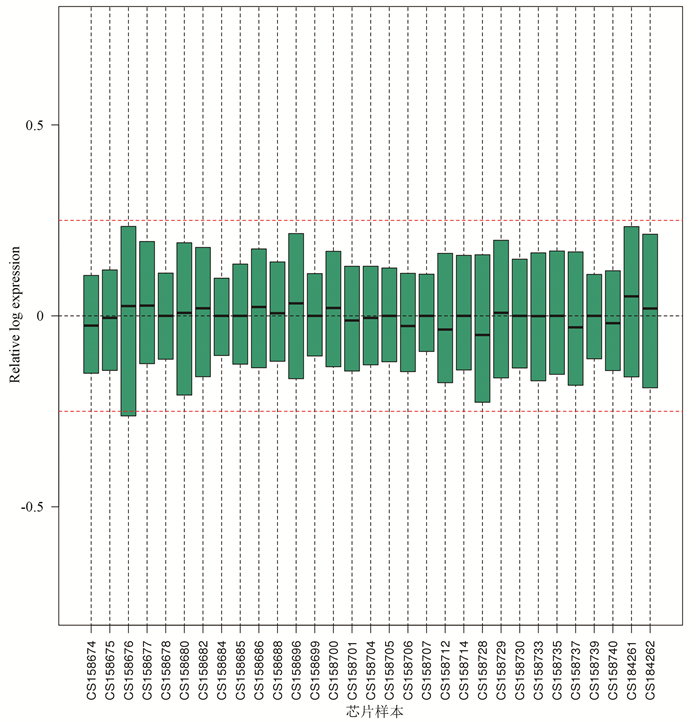
|
图 1 基因芯片质量分析箱形图 Figure 1 Quality analysis box chart |
应用SAM法[7]分析在预先设定的分组下筛选具有显著性差异的基因。通过分析A,B组,得到在受损的胃粘膜组织,HP(+)vs HP(-)的差异基因1,共28个(见图 2)。
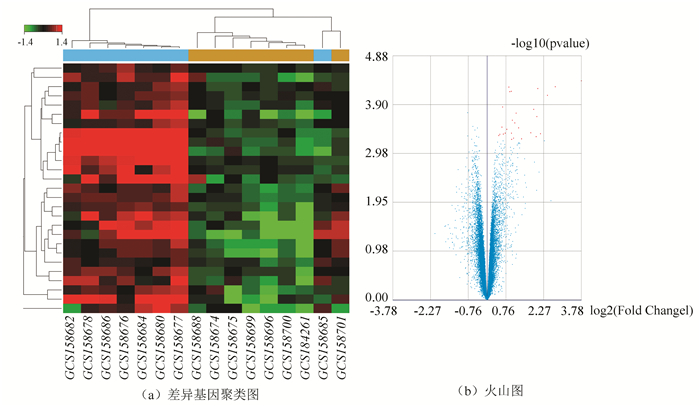
|
图 2 A、B组差异分析1聚类分析和火山图 Figure 2 Cluster analysis chart of Group A and B, volcano map in DEG1 注:(a)中红色代表上调基因,绿色代表下调基因(颜色详见电子版http://swxxx.alljournals.cn/ch/login.aspx.(2019年第1期));(b)中横坐标代表差异倍数,纵坐标代表P值均取log |
通过分析C,D组,我们得到在健康的胃粘膜组织,HP(+)vs HP(-)的差异基因(见图 3),共27个。同时两个差异基因集取并集获得上调差异基因44个。提示在HP感染后,这些基因的高表达与疾病的发生发展有关。
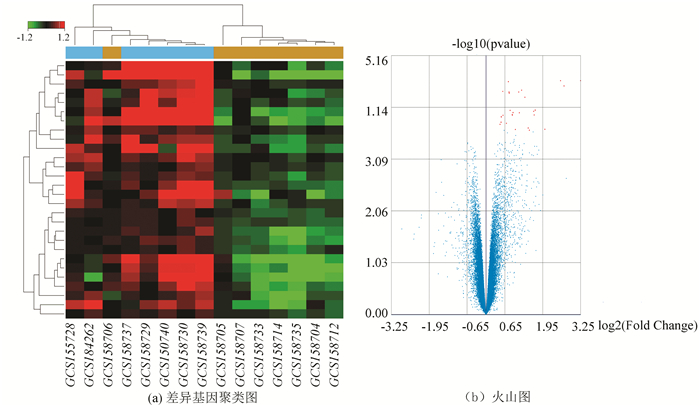
|
图 3 C、D组差异分析2聚类分析和火山图 Figure 3 Cluster analysis chart of Group C and D volcano map in DEG2 |
基因功能注释的目的是发现这些差异基因在生物学功能,代谢途径中的地位和意义,研究基因表达调控网络,及其功能机制。Go分析[8]发现这些差异基因主要涉及免疫反应,炎症反应,抗原提呈,细胞间信号通路等,而KEGG信号通路分析[9],主要包括:细胞因子信号通路,因子受体相互作用,细胞粘附等通路。
2.4 核心节点基因网络分析基因相互作用(Gene signal network)分析解构了KEGG数据库,突破限制获得单一信号通路中基因之间的相互作用。因此,可以获得一些基因的上下游分子关系。通过分析发现差异基因的相互作用:CXCR4, CCL20, CXCL13, CXCL3, CXCL5, CXCL2, HLA-DMA, HLA-DQA1, HLA-DPA1, HLA-DOA, HLA-DQB1存在有网络作用,其中核心分子为CXCR4(见图 4)。
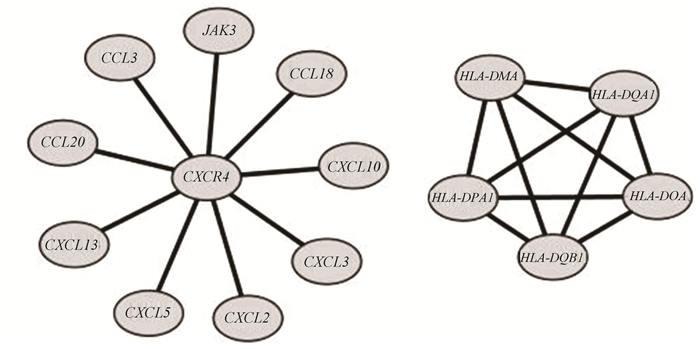
|
图 4 基因相互作用网络图 Figure 4 Gene interaction network diagram |
共表达网络清楚的揭示了两基因之间的关系,并找到调控网络的关键基因。这是通过基因间相关系数来拟合基因无标度网络关系实现的。分析基因共表达得到CCL20,PLEK,HLA-DMA,JAK3,BCL2A1,TNFAIP2,CXCL13,PTPRC,CXCL3,FGR,FAM65B,C3,CXCL5,MMP9,VNN2,ADAMDEC1,GZMK, TLR10之间共表达关系,其中重要的节点分子为CCL20,JAK3,TNFAIP2,PLEK,HLA-DMA,PTPRC,CXCL13,BCL2A1(见图 5)。
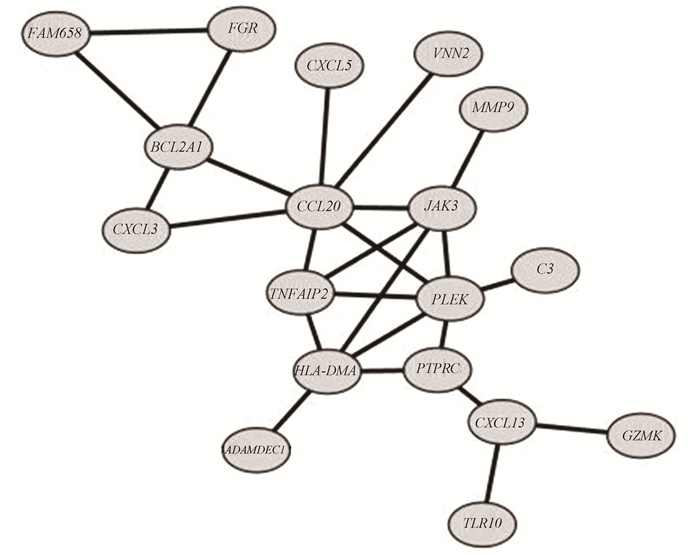
|
图 5 基因共表达分析网络图 Figure 5 Gene co-expression network diagram |
通过实施定量PCR方法,验证在感染后的胃黏膜组织部分相关基因的表达情况。研究发现CXCR4, CXCL5, CXCL2在HP阳性的胃黏膜组织表达高于HP阴性对照组,且具有统计学意义(P < 0.05)(见图 6)。
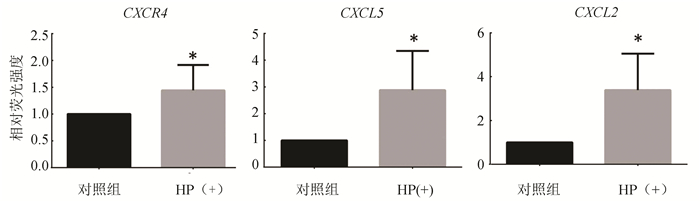
|
图 6 实时定量PCR检测CXCR4, CXCL5, CXCL2在胃黏膜组织中的表达(*提示P < 0.05) Figure 6 Real-time quantitative PCR detection of CXCR4, CXCL5, and CXCL2 expression in gastric mucosa |
| 表 1 主要涉及GO基因分类(前10) Table 1 Top 10 GO mainly involved gene classification |
| 表 2 主要涉及的信号通路(前10) Table 2 Top 10 mainly involved signaling pathway |
幽门螺旋杆菌是一种革兰氏阴性细菌,最初是由Warren和Marshall于1983在胃上皮的管腔表面发现,并分离出来,从第一次发现幽门螺杆菌,研究其与临床消化道疾病关系以来,已经经历三十多年[10]。绝大多数HP感染患者不会有任何临床意义上的表现,仅仅在部分患者当作引起消化道疾病治病因素,其中毒力因子空泡毒素(VacA)[11],细胞毒素相关基因A (CagA)[12]被认为是重要的治病因素。为了认识该细菌,以及了解其致病机制,做了许多研究,包括体内以及体外研究。
目前发现幽门螺杆菌与消化道疾病关系紧密,但同时与心血管疾病,贫血,脑梗,糖尿病等疾病均有关联[13-16]。为了进一步认识幽门螺杆菌对胃粘膜组织的分子层面影响,分析幽门螺杆菌相关芯片。Go分析:主要涉及免疫反应,炎症反应,抗原提呈,细胞信号通路等。Pathways通路分析:提示主要涉及的信号通路包括:细胞因子通路,因子受体关联,细胞粘附分子等。
基因相互作用分析中发现重要的CXCR4基因,与趋化因子的互相作用,在人类发育、免疫应答、癌转移均起到了重要的作用。已经有临床研究证明CXCR4在HP患者当中高表达[17],部分的HP感染患者可能会引起消化道肿瘤,CXCR4与消化道肿瘤的高表达有关系[18],这是否和CXCR4的过度激活有关?同时在已有研究中发现胃淋巴瘤观察到CXCR4和Ki-67表达之间的相关性,并提示CXCR4可作为诊断和治疗MALT型胃淋巴瘤潜在靶点[19]。不仅如此,近年来研究表明在慢性幽门螺杆菌感染过程中,骨髓间充质干细胞迁移到胃组织,也可能是胃腺癌的起源[20]。
基因共表达分析中重要的节点分子为趋化因子CCL20[21],CXCL13[22],CXCL5[23],CXCL2[24]以及其他包括增殖,炎症,免疫,凋亡相关重要分子JAK3, TNFAIP2, PLEK, HLA-DMA, PTPRC, BCL2A1的激活,而这些基因与胃癌的发生发展有密切关系[25-28]。有研究证实幽门螺杆菌诱导的STAT3激活直接上调JAK3,可能有助于胃癌的发生和发展[25]。为了进一步验证分析相关分子的表达情况,进行了实时定量PCR实验验证,CXCR4,CXCL5,CXCL2在幽门螺杆菌感染后的胃黏膜组织均高于对照组,更多的机制探讨需要进一步实验验证。
4 结论幽门螺杆菌感染后,存在多个信号通路的激活,包括炎症,免疫反应,细胞凋亡信号,而后出现组织细胞修复、增殖活化等。而这些在组织细胞间异常表达的基因,提示我们是否可能通过相关信号通路药物,从而避免幽门螺杆菌感染所造成的严重后果,同时也给我们提供了进一步研究的方向。
| [1] |
MOCANU V, DANG J T, SWITZER N, et al. The effect of helicobacter pylori on postoperative outcomes in patients undergoing bariatric surgery:a systematic review and meta-analysis[J]. Obesity Surgery, 2017, 28(5): 1-7. DOI:10.1007/s11695-017-3024-8 (  0) 0) |
| [2] |
KOUNTOURAS J, POLYZOS S A, DOULBERIS M, et al. Potential impact of helicobacter pylori-related metabolic syndrome on upper and lower gastrointestinal tract oncogenesis[J]. Metabolism Clinical & Experimental, 2018, 87(10): 18-24. DOI:10.1016/j.metabol.2018.06.008 (  0) 0) |
| [3] |
ZAGARI R M, RABITTI S, EUSEBI L H, et al. Treatment of helicobacter pylori infection:a clinical practice update[J]. European Journal of Clinical Investigation, 2017, 48(1): e12857. DOI:10.1111/eci.12857 (  0) 0) |
| [4] |
BARRETT T, WILHITE S E, LEDOUX P, et al. NCBI GEO:archive for functional genomics data sets-update[J]. Nucleic Acids Research, 2013, 39(Database issue): 1005-1010. DOI:10.1093/nar/gks1193 (  0) 0) |
| [5] |
KAPUR K, XING Y, OUYANG Z, et al. Exon arrays provide accurate assessments of gene expression[J]. Genome Biology, 2007, 8(5): R82. DOI:10.1186/gb-2007-8-5-r82 (  0) 0) |
| [6] |
GRACE C, NACHEVA E P. Significance analysis of microarrays (sam) offers clues to differences between the genomes of adult philadelphia positive all and the lymphoid blast transformation of cml[J]. Cancer Informatics, 2012, 11(11): 173-183. DOI:10.4137/CIN.S9258 (  0) 0) |
| [7] |
LARSSON O, WAHLESTEDT C, TIMMONS J A. Considerations when using the significance analysis of microarrays (SAM) algorithm[J]. BMC Bioinformatics, 2005, 6(1): 129. DOI:10.1186/1471-2105-6-129 (  0) 0) |
| [8] |
The Gene Ontology consortium. Expansion of the Gene Ontology knowledgebase and resources[J]. Nucleic Acids Research, 2016, 45(D1): D331-D338. DOI:10.1093/nar/gkw1108 (  0) 0) |
| [9] |
DU J, LI M, YUAN Z, et al. A decision analysis model for KEGG pathway analysis[J]. BMC Bioinformatics, 2016, 17(1): 407. DOI:10.1186/s12859-016-1285-1 (  0) 0) |
| [10] |
SALAMA N R, HARTUNG M L, MULLER A. Life in the human stomach:persistence strategies of the bacterial pathogen Helicobacter pylori[J]. Nature Reviews Microbiology, 2013, 11(6): 385-399. DOI:10.1038/nrmicro3016 (  0) 0) |
| [11] |
NEJATI S, KARKHAH A, DARVISH H, et al. Influence of helicobacter pylori virulence factors CagA and VacA on pathogenesis of gastrointestinal disorders[J]. Microbial Pathogenesis, 2018, 117: 43-48. DOI:10.1016/j.micpath.2018.02.016 (  0) 0) |
| [12] |
PARK J Y, FORMAN D, WASKITO L A, et al. Epidemiology of helicobacter pylori and CagA-Positive infections and global variations in gastric cancer[J]. Toxins (basel), 2018, 10(4): 163. DOI:10.3390/toxins10040163 (  0) 0) |
| [13] |
FERRARA M, CAPOZZI L, RUSSO R. Influence of helicobacter pylori infection associated with iron deficiency anaemia on growth in pre-adolescent children[J]. Hematology, 2009, 14(3): 173-176. DOI:10.1179/102453309X402287 (  0) 0) |
| [14] |
HUGHES W S. An hypothesis:the dramatic decline in heart attacks in the United States is temporally related to the decline in duodenal ulcer disease and Helicobacter pylori infection[J]. Helicobacter, 2014, 19(3): 239-241. DOI:10.1111/hel.12123 (  0) 0) |
| [15] |
OSMAN S M, MUBARAK S M, OMER I M, et al. Helicobacter pylori infection and the onset of type 1 diabetes mellitus in Sudanese children[J]. Sudanese Journal of Paediatrics, 2016, 16(2): 59. (  0) 0) |
| [16] |
LI J Z, LI J Y, WU T F, et al. Helicobacter pylori infection is associated with type 2 diabetes, not type 1 diabetes:an updated meta-analysis[J]. Gastroenterology Research and Practice, 2017, 2017(5): 5715403. DOI:10.1155/2017/5715403 (  0) 0) |
| [17] |
ZHAO C, LU X, BU X, et al. Involvement of tumor necrosis factor-alpha in the upregulation of CXCR4 expression in gastric cancer induced by Helicobacter pylori[J]. BMC Cancer, 2010, 10(1): 419. DOI:10.1186/1471-2407-10-419 (  0) 0) |
| [18] |
YU S, WU T, CHENG C, et al. Combined evaluation of expression of cxcr4 and nrf2 as prognostic factor for patients with gastric carcinoma[J]. Anticancer Agents In Medicinal Chemistry, 2018, 18(3): 388-393. DOI:10.2174/1871520617666171103112019 (  0) 0) |
| [19] |
STOLLBERG S, KÄMMERER D, NEUBAUER E, et al. Erratum to:differential somatostatin and CXCR4 chemokine receptor expression in MALT-type lymphoma of gastric and extragastric origin[J]. Journal of Cancer Research & Clinical Oncology, 2016, 142(11): 2239-2247. DOI:10.1007/s00432-016-2312-3 (  0) 0) |
| [20] |
FAKHARI S, KALANTAR E, NIKZABAN M, et al. Effect of helicobacter pylori infection on stromal-derived factor-1/CXCR4 axis in bone marrow-derived mesenchymal stem cells[J]. Advanced Biomedical Research, 2014(3): 19. DOI:10.4103/2277-9175.124650 (  0) 0) |
| [21] |
CHEN J P, WU M S, KUO S H, et al. IL-22 negatively regulates Helicobacter pylori-induced CCL20 expression in gastric epithelial cells[J]. PLoS One, 2014, 9(5): e97350. DOI:10.1371/journal.pone.0097350 (  0) 0) |
| [22] |
NAKASHIMA Y, ISOMOTO H, MATSUSHIMA K, et al. Enhanced expression of CXCL13 in human Helicobacter pylori-associated gastritis[J]. Digestive Diseases & Sciences, 2011, 56(10): 2887-2894. DOI:10.1007/s10620-011-1717-8 (  0) 0) |
| [23] |
RAJA U M, GOPAL G, SHIRLEY S, et al. Immunohistochemical expression and localization of cytokines/chemokines/growth factors in gastric cancer[J]. Cytokine, 2017, 89: 82-90. DOI:10.1016/j.cyto.2016.08.032 (  0) 0) |
| [24] |
WU D, CAO M, PENG J, et al. The effect of trimethylamine N-oxide on Helicobacter pylori-induced changes of immunoinflammatory genes expression in gastric epithelial cells[J]. International Immunopharmacology, 2017, 43: 172-178. DOI:10.1016/j.intimp.2016.11.032 (  0) 0) |
| [25] |
ZHAO J, DONG Y, KANG W, et al. Helicobacter pylori-induced STAT3 activation and signalling network in gastric cancer[J]. Oncoscience, 2014, 1(6): 468-475. (  0) 0) |
| [26] |
WEST A C, TANG K, TYE H, et al. Identification of a TLR2-regulated gene signature associated with tumor cell growth in gastric cancer[J]. Oncogene, 2017, 36(36): 5134-5144. DOI:10.1038/onc.2017.121 (  0) 0) |
| [27] |
ZHANG L, LIU Y, WANG X, et al. The extent of inflammatory infiltration in primary cancer tissues is associated with lymphomagenesis in immunodeficient mice[J]. Scientific Reports, 2015, 5: 9447. DOI:10.1038/srep09447 (  0) 0) |
| [28] |
XU Y, MA H, YU H, et al. The miR-184 binding-site rs8126 T>C polymorphism in TNFAIP2 is associated with risk of gastric cancer[J]. PLoS One, 2013, 8(5): e64973. DOI:10.1371/journal.pone.0064973 (  0) 0) |
 2019, Vol. 17
2019, Vol. 17

