miRNAs(microRNAs)是一类长度约为19~25个核苷酸的非编码小分子RNA,由长度约为60~70 nt具有发夹结构的pre-miRna(Precursor microRna)经Dicer酶剪切而来,普遍存在于各种生物中,人类细胞中约有1/3的蛋白编码基因受miRNAs的调控,主要通过与信使RNA(mRNA)的3’非编码(3’-UTR)区结合,抑制mRNA翻译或促进其降解,从而在转录后水平负性调控靶基因的表达[1]。不同时间、不同疾病、不同组织和细胞中miRNA的表达水平有显著差异。miRNA与多种疾病的发生发展都有密切联系,在肿瘤、心血管疾病、糖尿病等等疾病中都有举足轻重的作用,因而深入研究miRNA具有重要意义。研究表明,miRNAs参与了许多生命活动过程的调节,包括机体生长发育、激素分泌、信号转导、细胞增殖、凋亡、分化以及对外界环境变化的应答等过程[2]。
hsa-miR-192-3p位于染色体11q13.1,近年研究结果显示肾病、胃癌、结肠癌等疾病都与miR-192-3p的失调有关[3-5]。有研究提示miR-192与肾癌有关[6],Lokeshwar SD和Talukder A等人用正常和肿瘤标本通过qPCR分析7个异常表达的miRNA,其中miR-192下调3~60倍。然而,对miRNAs具体作用机制的认识仍然存在局限,如许多miRNA的靶基因没有确定,并且对其进行靶基因分析和鉴定的难度较大。因此,准确的预测miRNA的靶基因并对预测出来的靶基因进行生物信息学分析是研究miRNA作用机制的关键。
目前已有很多工作者使用生物信息学工具进行数据分析,常凯、江忠勇等人发表在重庆医科大学学报上的文章就曾用过TargetScan、miRanda和DAVID等数据库进行预测分析[7]。本研究也将综合多种生物信息学工具预测hsa-miR-192-3p的靶基因并对其进行表达分析、功能富集分析和信号转导通路富集分析,进一步挖掘数据中的生物学知识为后续的功能研究提供理论依据和数据支持。
1 材料与方法 1.1 hsa-miR-192-3p的保守性分析通过miRbase(http://www.mirbase.org/)和UCSC(http://genome.ucsc.edu/)在线数据库分析hsa-miR-192-3p在各物种之间的保守性。
1.2 hsa-miR-192-3p在各组织中的表达情况通过miGator v3.0(http://mirgator.kobic.re.kr/)数据库查看hsa-miR-192-3p在各个组织器官中的表达情况,在miGator v3.0数据库中输入hsa-miR-192-3p后,查询结果。miGator v3.0汇编了GEO、TCGA等机构发布的70多个人类miRNA深度测序数据集,使用者可以通过miGator v3.0数据库在线查询所研究的miRNA的序列和表达情况。
1.3 hsa-miR-192-3p的靶基因预测运用TargetScan和miRanda两种靶基因预测软件。TargetScan是目前应用最为广泛的,这款软件的算法提出了一个新概念即种子匹配(Seed match),其定义是靶基因mRNA的3'UTR非编码区上的一段7nt(Nucleotide)与miRNA的5'端的2~8位碱基完全互补的序列,miRNA上的这7个核苷酸叫做miRNA的种子区,软件还同时考虑了序列在各个物种之间的保守性来进行分析预测。miRanda是开发的第一个预测靶基因的软件,不会受到物种的限制,因而适用范围较为广泛。Betel等人在传统的miRNA与靶基因碱基的要求之外更新了算法mirSVR,作为一种新方法,mirSVR不需要种子区亚类(Seed subclasses),将靶点位置序列与结构特征(Contextual features)结合分析,并进行回归分析。通过TargetScan 7.2(http://www.targetscan.org/vert_72/)和miRanda(http://www.microrna.org/)两种在线数据库预测hsa-miR-192-3p的靶基因,取两个数据库预测结果的交集,以便进行下一步研究。
1.4 hsa-miR-192-3p靶基因的功能富集分析DAVID 6.8(https://david.ncifcrf.gov/)是一个生物信息数据库,整合了生物学数据和分析工具,为大规模的基因或蛋白列表提供系统综合的生物功能注释信息,帮助用户从中提取生物学信息。DAVID工具目前可以实现以下功能:识别富集的生物条目;发现功能相关的基因集合;聚类注释条目;寻找其他功能相关的但是不在列表中的基因;列出相互作用的蛋白质,此外,DAVID还可以进行基因ID的转换。
将靶基因的交集进行功能富集分析,通过DAVID在线分析工具对靶基因交集进行功能富集分析,选择本物种所有的基因作为背景基因,以P < 0.05为显著性阈值,得到的功能注释相对于背景具有统计意义。
1.5 hsa-miR-192-3p靶基因的信号转导通路富集分析将2个在线数据库预测到的hsa-miR-192-3p的靶基因交集,采用DAVID 6.8在线数据库进行信号转导通路富集分析,以P < 0.05为显著性阈值,得到基因集合的信号转导通路相对于背景具有统计学意义。
2 结果分析 2.1 hsa-miR-192-3p的保守性分析通过miRbase和UCSC在线数据库对hsa-miR-192-3p进行保守性分析,发现miRbase及UCSC均显示此miRNA在人、鸭嘴兽、家鼠、褐家鼠、等多个物种之间具有高度保守性(P < 0.05),如表 1所示,提示hsa-miR-192-3p具有潜在的重要生物学功能。
| 表 1 不同物种hsa-miR-192-3p成熟序列 Table 1 Mature sequence of hsa-miR-192-3p in different species |
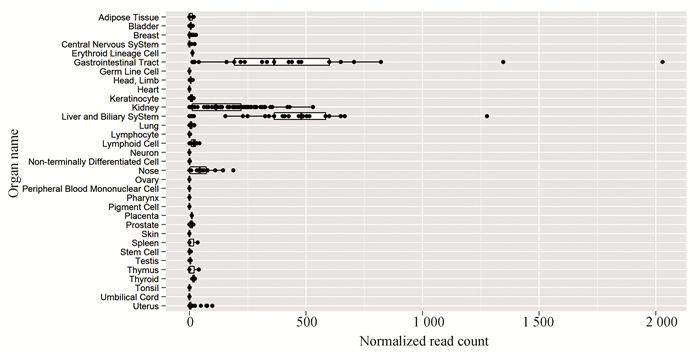
hsa-miR-192-3p在各个器官的表达情况,下载结果图片见图 1,其中在胃肠道、肾脏、肝胆系统、干细胞、鼻、脾、胸腺中表达丰富度较高。
|
图 1 hsa-miR-192-3p在各个组织器官中的表达情况 Figure 1 Expression abundance of hsa-miR-192-3p in organs |
TargetScan预测的hsa-miR-192-3p靶基因数量为3 477个,miRanda预测的靶基因个数为886个,两个数据库预测的靶基因取交集共筛选出190个靶基因,其中包括与癌症信号通路和催乳素信号通路相关的基因,与癌症信号通路相关的靶基因为BCL2L11、CCND2、ZEB2等,与催乳素信号通路相关的靶基因为ELF5、ESR1、SOCS5等。
2.4 hsa-miR-192-3p靶基因的功能富集分析将2个数据库都预测到基因交集的190个hsa-miR-192-3p的靶基因用DAVID数据库作功能富集分析发现hsa-miR-192-3p的靶基因富集在细胞质、细胞核、质膜、高尔基体等15个细胞组件(p < 0.05),参与蛋白结合、GTP酶活性、锌离子跨膜转运蛋白活性、转录激活因子等7个分子功能(p < 0.05),涉及金属离子运输、RNA聚合酶Ⅱ启动子的转录阳性调控、基因表达调节、钙离子跨膜运输、胚胎发育等18个生物过程(p < 0.05),结果见表 2。
| 表 2 hsa-miR-192-3p靶基因功能富集分析部分结果 Table 2 Functional enrichment analysis of target genes of hsa-miR-192-3p |
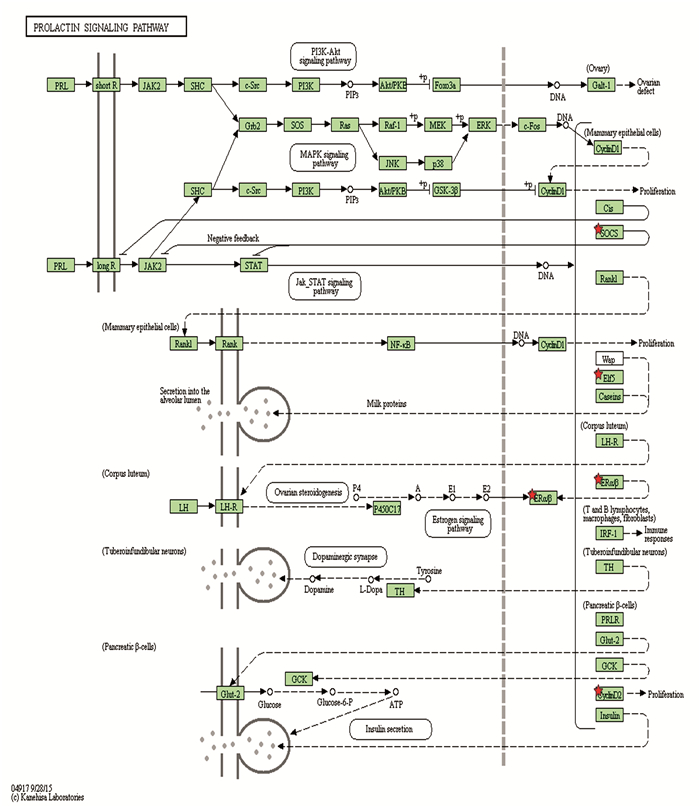
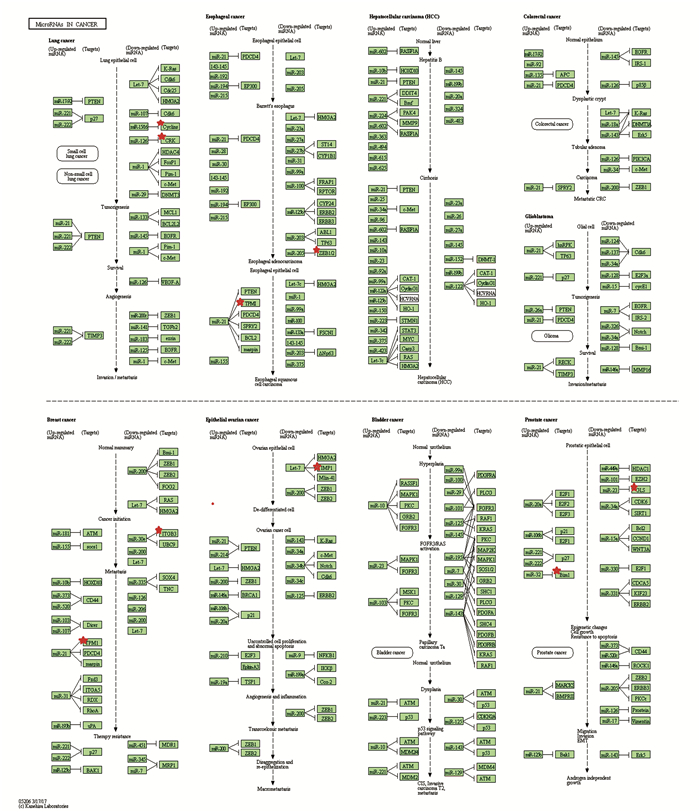
利用DAVID数据库对基因集合的190个候选靶基因进行生物通路富集分析。结果显示,预测靶基因集合显著富集于催乳素信号通路与癌症通路(P < 0.05),结果如表 3所示;并给出两个信号通路图,截获结果如图 2、图 3所示。
| 表 3 hsa-miR-192-3p靶基因的信号转导通路富集分析结果 Table 3 Signal pathway enrichment analysis of target genes of hsa-miR-192-3p |
|
图 2 hsa-miR-192-3p的靶基因参与催乳素信号通路信 Figure 2 Target genes of hsa-miR-192-3p articipated in prolactin signal pathway 注:红色五角星:hsa-miR-192-3p靶基因;绿色方块:催乳素信号通路基因(彩图详见电子版:http://swxxx.alljournals.cn/ch/index.aspx2018年第4期)。 |
|
图 3 hsa-miR-192-3p的预测靶基因参与癌症信号通路 Figure 3 Predicted target genes of hsa-miR-192-3p articipated in cancer signal pathway 注:红色五角星:hsa-miR-192-3p靶基因;绿色方块:癌症信号通路基因(彩图详见电子版:http://swxxx.alljournals.cn/ch/index.aspx)。 |
近年来,许多研究表明,miRNA与多种癌症有关,相关miRNA可能起到原癌基因或者抑癌基因的作用。因此,通过利用在线数据库预测miRNA调控的靶基因,进而研究miRNA的生物学功能,对临床疾病尤其是癌症的诊断与治疗具有重要的价值。
本研究中,两个数据库预测的hsa-miR-192-3p靶基因取交集后共190个,其中癌症信号通路的靶基因有BCL2L11、CRK、CCND2、IGF2BP1、ITGB3、ZEB2。一些研究已经表明hsa-miR-192与癌症具有相关性,包括乳腺癌[8]、肺癌[9]、膀胱癌[10]、肝癌[11]、结肠直肠癌[12]和急性淋巴细胞白血病[9]等。本研究预测出来的两条通路,其中一条就是癌症通路,hsa-miR-192-3p的部分靶基因聚集在肺癌和乳腺癌的通路中,且另一条通路是催乳素信号通路,两个通路存在共同的靶基因CCND2,说明CCND2在癌症疾病中起着重要的作用,尤其是在乳腺癌中,已有相关报道证明CCND2和乳腺癌有关,报道在肿瘤和正常组织之间差异表达的基因共有9个,其中一个基因就是CCND2[13]。已有相关报道证明BCL2L11与乳腺癌和肺癌具有相关性;ZEB2与食管癌,骨肉瘤,直肠癌等癌症有关。Matsumoto R和Tsuda M等人的研究发现CRK可引起浸润性膀胱癌的发生,CRK可能是膀胱癌的一个有效分子靶点,特别是对于预防癌细胞转移[14]。另有研究表明miR-126/CRK轴的失调可能促进人胃癌的恶性进展,miR-126和CRK联合表达可作为晚期胃癌患者总生存期的独立预测因子[15]。Elmansuri A和Tanino MA等人在人肺癌组织中观察到CRK的过度表达,并且与不良预后显著相关[16]。Baudry D癌症和Faussillon M等人检测到CCND2在肾母细胞瘤中特异性上调[17]。
上述这些已经证明与癌症有关的本基因都是本研究对has-miR-192-3p进行生物信息预测出来的靶基因,说明has-miR-192-3p很可能与癌症有关,并通过这些基因在癌症的发生发展中起到关键作用,另外那些没有得到验证与癌症有关的靶基因很可能也在疾病中起到关键作用,本研究预测的两条通路中富集的靶基因就具有很好的提示作用,这些基因都可能与癌症的发生发展有关,虽然癌症机制还尚未完全清楚,但本研究为以后研究癌症机制提供了线索和方向。
4 结论(1) hsa-miR-192-3p在肿瘤中的作用不可忽视。
(2) 通过生物信息学预测软件进行靶基因预测,得到靶基因集合,并对靶基因集合进行分子功能、生物过程和信号转导通路富集分析,使我们对hsa-miR-192-3p靶基因所参与的分子功能、生物过程等有一个较为全面的认识,已有研究报道,miR-192-3p与其靶基因在肿瘤中的作用与本文中的分析结果一致,生物信息学分析方法具有一定的可靠性,但生物预测所得到的靶基因会有假阳性的情况存在,所以还需进一步的RT-PCR、荧光素酶报告基因,MTS、Western blotting等实验对预测所得的靶基因和其所参与的生物学功能等进行验证。
(3) 生物预测分析为后期深入研究miRNA在疾病中的作用机制提供了一定的方向和思路。
| [1] |
CHUA J H, ARMUGAM A, JEYASEELAN K, et al. MicroRNAs: Biogenesis, function and applications[J]. Current Opinion in Molecular Therapeutics, 2009, 11(2): 189-199. (  0) 0) |
| [2] |
WANG J, FANG M, LIU X Y, et al. A novel GATA4 mutation responsible for congenital ventricular septal defects[J]. International Journal of Molecular Medicine, 2011, 28(4): 557-564. DOI:10.3892/ijmm.2011.715.Epub2011Jun1 (  0) 0) |
| [3] |
JASWANI P, PRAKASH S, PRASAD N, et al. Predicting miRNA association with corresponding target genes and single nucleotide polymorphisms in altered renal pathophysiology[J]. Microrna, 2017, 6(3): 213-221. DOI:10.2174/2211536606666171016151846 (  0) 0) |
| [4] |
SMID D, KULDA V, SRBECKA K, et al. Tissue microRNAs as predictive markers for gastric cancer patients undergoing palliative chemotherapy[J]. International Journal of Oncology, 2016, 48(6): 2693-2703. DOI:10.3892/ijo.2016.3484.Epub2016Apr12 (  0) 0) |
| [5] |
KRATTINGER R, BOSTRÖM A, SCHIÖTH H B, et al. MicroRNA-192 suppresses the expression of the farnesoid X receptor[J]. American Journal of Physiology Gastrointestinal & Liver Physiology, 2016, 310(11): 1044-1051. DOI:10.1152/ajpgi.00297.2015 (  0) 0) |
| [6] |
LOKESHWAR S D, TALUKDER A, YATES T J, et al. Molecular characterization of renal cell carcinoma: A potential three-microRNA prognostic signature[J]. Cancer Epidemiology Biomarkers Prevention, 2018, 27(4): 464-472. DOI:10.1158/1055-9965.EPI-17-0700 (  0) 0) |
| [7] |
常凯, 江忠勇, 任俊龙, 等. 糖尿病肾病相关miR-130b-3p靶点的生物信息学预测[J]. 重庆医科大学学报, 2018(6): 788-792. CHANG Kai, JIANG Zhongyong, REN Junlong, et al. Bioinformatics prediction of miR-130b-3p target genes associated with diabetic nephropathy[J]. Journal of Chongqing Medical University, 2018(6): 788-792. (  0) 0) |
| [8] |
XIANG M, ZENG Y, YANG R, et al. U6 is not a suitable endogenous control for the quantification of circulating microRNAs[J]. Biochemical Biophysical Research Communication, 2014, 454(1): 210-214. DOI:10.1016/j.bbrc.2014.10.064 (  0) 0) |
| [9] |
CAO J, HE Y, LIU H Q, et al. MicroRNA 192 regulates chemo-resistance of lung adenocarcinoma for gemcitabine and cisplatin combined therapy by targeting Bcl-2[J]. International Journal of Clinical and Experimental Medicine, 2015, 8(8): 12397-12403. (  0) 0) |
| [10] |
HU Yingbo, CHENG Cheng, HONG Zhengdong, et al. Independent prognostic miRNAs for bladder urothelial carcinoma[J]. Oncology Letters, 2017, 14(3): 3001-3005. DOI:10.3892/ol.2017.6471 (  0) 0) |
| [11] |
ZHU H T, LIU R B, LIANG Y Y, et al. Serum microRNA profiles as diagnostic biomarkers for HBV-positive hepatocellular carcinoma[J]. Liver International Official Journal of the International Association for the Study of the Liver, 2017, 37(6): 888-896. (  0) 0) |
| [12] |
CARTER J V, ROBERTS H L, PAN J, et al. A highly predictive model for diagnosis of colorectal neoplasms using plasma microRNA: Improving specificity and sensitivity[J]. Annals of Surgery, 2016, 264(4): 575-584. DOI:10.1097/SLA.0000000000001873 (  0) 0) |
| [13] |
LI Z, HENG J, YAN J, et al. Integrated analysis of gene expression and methylation profiles of 48 candidate genes in breast cancer patients[J]. Breast Cancer Research and Treatment, 2016, 160(2): 371-383. DOI:10.1007/s10549-016-4004-8 (  0) 0) |
| [14] |
MATSUMPTO R, TSUDA M, WANG L, et al. Adaptor protein CRK induces epithelial-mesenchymal transition and metastasis of bladder cancer cells through HGF/c-Met feedback loop[J]. Cancer Science, 2015, 106(6): 709-717. DOI:10.1111/cas.2015.106.issue-6 (  0) 0) |
| [15] |
FENG R, SAH B K, BEEHARRY M K, et al. Dysregulation of miR-126/Crk protein axis predicts poor prognosis in gastric cancer patients[J]. Cancer Biomarkers, 2017, 21(2): 335-343. DOI:10.3233/CBM-170472 (  0) 0) |
| [16] |
ELMANSURI A Z, TANINO M A, MAHABIR R, et al. Novel signaling collaboration between TGF-β and adaptor protein Crk facilitates EMT in human lung cancer[J]. Oncotarget, 2016, 7(19): 27094-27107. DOI:10.18632/oncotarget.8314 (  0) 0) |
| [17] |
BAUDR Y D, FAUSSILLON M, CABANIS M O, et al. Changes in WT1 splicing are associated with a specific gene expression profile in Wilms' tumour[J]. Oncogene, 2002, 21(36): 5566-5573. DOI:10.1038/sj.onc.1205752. (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


