2. 淮海工学院 药学院,江苏 连云港 222005
2. Pharmacy School, Huaihai Institute of Technology, Lianyungang 222005, Jiangsu, China
蛋白质折叠预测问题[1]是在已知蛋白质氨基酸序列的情况下,直接通过序列顺序求解出蛋白质的三维空间结构,即求解蛋白质的最小能量。结构生物学研究已经验证,蛋白质的空间结构决定了蛋白质的特定功能[2]。蛋白质的氨基酸序列直接决定了其在三维空间中的结构,而蛋白质的结构又决定了其特殊的生物功能,因此蛋白质折叠问题的研究在生物学中具有及其重要意的义[3]。蛋白质折叠问题已成为生物信息学领域中的核心研究问题之一,对其求解是后基因时代蛋白质研究的一项重要任务[4]。虽然通过特殊的技术手段方法可以得到一部分蛋白质的三维空间中的结构,但却极其耗时,并且还需要收到较多限制。目前为止,还有大部分的蛋白质没有得到有效的三维构想,因此通过理论方法,从蛋白质氨基酸序列入手预测其三维空间结构已成为分子生物学研究中的一个迫切需求。
蛋白质折叠预测是一个典型的非确定性NP困难问题,随着蛋白质中氨基酸序列的增多,其计算量呈指数级增加。在蛋白质折叠预测主要即为对蛋白质最小能量进行求解也称为蛋白质能量优化,所以蛋白质折叠预测问题的核心是使用全局优化算法进行蛋白质能量优化。基于HP(Hydrophobie-Polar)网格的蛋白质折叠预测模型[5],通过逐步减少固定蛋白质内部氨基酸位置从而可以使连续的序列进行离散化,可以有效地对蛋白质折叠方式进行探索和解读。尽管基于HP模型蛋白质折叠已经是一个去掉较多约束的简化预测模型,但仍然是NP困难问题,难以建立精确的数学模型对其进行高效而准确的求解。近些年来,研究人员使用了多种优化算法对其进行分析求解,如模拟退火算法、遗传算法、人工神经网络和蚁群算法等[6-10],这些不同的方法与算法推动了蛋白质折叠预测研究,并取得了一定的进展。
Cannabinoid receptor Type 2(简称CB2)是大麻素受体的一种亚型,由360个氨基酸组成。其受体分布表达造成了其独特的生理药理作用,如免疫抑制作用、抑制肿瘤细胞生长、促进骨生成[11]、镇痛、抗肝纤维化、抗神经损伤[12-13]、促神经生长[14]等作用。除此之外,大麻素Ⅱ型受体(CB2)的激活可促进干细胞(如造血干/祖细胞和神经祖细胞)的增殖.心脏祖细胞激活和增殖在心肌梗死(心梗)后心肌内源性再生和修复中发挥重要作用[15],并且,CB2选择性激动剂AM1241可激活原位CPCs, 促进心肌梗死后心肌组织内源性再生, 轻心梗后心肌组织纤维, 改善心脏功能[16]。CB2受体的生理药理作用表明其是十分重要的药物靶标,特别在止疼、抗炎和抑制咳嗽方面,因为其无中枢神经副作用,不会产生成瘾性及耐受性,显示出了非常好的开发前景和潜在的应用价值。更不要说其作为免疫调节剂、神经保护剂和抗癌药的巨大市场价值。因此开发适合于人体用药的选择性CB2受体激动剂、反向激动剂及拮抗剂,是一件非常有意义的探索。目前,CB2蛋白的空间结构还未被测定出来,对于CB2的折叠问题研究也开展的较少,针对此问题本文进行了一定的探索工作。
1 理论与方法 1.1 蛋白质折叠蛋白质折叠研究的主要目标是根据蛋白质的氨基酸序列(也称一级序列),推测出蛋白质在空间中的折叠形式,从而最终确定在三维空间中的具有特定功能的蛋白质构象。Anfinsen等在对牛胰核糖核酸酶的复性实验研究中发现,蛋白质的氨基酸序列唯一确定了其在三维空间中构象,并依此提出了蛋白质的自由能最小构象即应为其自然构象的这一著名热力学假说。目前的各种蛋白质折叠结构预测的理论与方法都是基于此假说。蛋白质折叠问题自20世纪中期就被广泛研究,但到目前尚无满意的解决方法。如果通过计算机枚举方法进行搜索蛋白质的自由能最小构象,则对其求解的计算时间耗费将变得不可承受。因此,蛋白质折叠问题的研究无论是在计算机模拟还是实验研究上,仍然是非常困难的事情[17]。
1.2 HP折叠模型蛋白质内部的疏水作用力是促使其折叠的主要因素,球状蛋白质折叠结构通常是由一个疏水核心紧密堆积而形成特定的空间结构[18]。基于HP的折叠模型,由于只在在二维空间中进行蛋白质的折叠进行研究,保证了相对的折叠精度下又可有效的提高求解速度,在计算机模拟方法研究中获得了研究人员的广泛关注。
在HP模型中,蛋白质序列中的氨基酸被分为疏水性(H)和亲水性(P)两类氨基酸。对于蛋白质序列中的多种氨基酸,可分别使用字母H和P进行代表,从而把蛋白质序列形成简化的表示形式。基于HP折叠模型,蛋白质折叠问题可简化为:将二维空间划分等距的网格空间,将每个氨基酸简化成一个节点放入网格之中。一个合法的蛋白质序列的空间构象需要同时满足以下三个条件[19]:
1) 任一氨基酸(H或P)必须放在排放在二维空间整数点坐标上;
2) 链序列中相邻的结点在摆放后仍相邻(距离为1);
3) 二维坐标任一格点上最多只能放一个氨基酸节点。
基于HP的蛋白质折叠模型中,其氨基酸序列所对应的能量函数可表示为:
| $ E = \sum\limits_{i < j} {{E_{\sigma i\sigma j}}\mathit{\Delta }\left( {{r_i} - {r_j}} \right)} $ |
当氨基酸序列上第j个结点rj与其之前的第i个节点ri同为H型氨基酸,他们在一级序列中并不相连而在二维格点空间中ri与rj相邻,则计EσiσjΔ(ri-rj)=-1;否则记EσiσjΔ(ri-rj)=0。通过将蛋白质序列进行HP模型表示,可以较为简单的计算得到某二维构想所对应的能量值。
1.3 大麻素受体CB2Cannabinoid receptor Type 2(简称CB2)是大麻素受体的一种亚型,共有360个氨基酸组成。氨基酸序列分析显示CB2受体的结构中包括7次亲酯跨膜α螺旋结构[20],是典型的G蛋白耦联受体。1993年Munro等用PCR技术首次克隆出CB2的基因,并发现其对Δ9-四氢大麻酚具有高亲和力。CB2受体有两个同种型,分别命名为CB2A和CB2B[21-22]。
CB2受体则主要分布于外周免疫系统[23]。研究表明,健康的脑细胞中没有CB2受体的表达,只有具有活性炎症和变性的脑区才有CB2受体的迁移和渗透[24]。大麻素受体除了在上述组织中存在外,在其他病变的组织如肿瘤中也有表达[25-26]。
1.4 蒙特卡罗(Monte Carlo,MC)方法蒙特卡罗方法是一种随机模拟方法,也称统计模拟或随机抽样方法。它是以概率论和数理统计等理论为基础,通过随机产生的随机数来解决具体计算问题的一种方法。蒙特卡罗方法兴起的背景是由于科学技术的发展特别是电子计算机的发明,是一类非常重要的数值计算方法。通常需要使用某一概率模型来描述具体需要解决的问题,在得到与所求问题同解的概率模型以后,通过随机实验方式对此概率模型进行模拟和计算,实验最后所得的统计值即所求问题的近似可行解[27]。
近些年来,随着计算机技术的飞速发展,在计算机的帮助下很多实验过程可以进行模拟,变成了简单快速的计算机计算,这为蒙特卡罗方法的发展提供了基础。目前,蒙特卡罗方法在多个研究领域得到了广泛的应用,取得了较好的研究成果,包括计算生物学、生物医学、计算物理学等研究领域。特别是此方法能很好地用来对于问题的维数并不敏感,该方法的计算复杂性也不再依赖于维数,所以一些无法计算的问题现在也可以计量。在具体应用时,对于一些复杂繁琐问题,如果直接求解变得几乎不可行,那么可采用蒙特卡罗方法从其他角度进行有效求解。在本文中,使用了蒙特卡罗方法对于优化问题进行求解,取得了很好的效果。
2 实验过程 2.1 生成CB2氨基酸序列的格点位置创建CB2氨基酸序列空间格点位置时,先将空间中原有信息清空,固定第一个氨基酸的位置(格点空间的中央);其次,将后续氨基酸的位置分为:上摆放、右摆放、下摆放、左摆放四种情况并编号。在摆放氨基酸位置时,除第一个氨基酸,所有的氨基酸的摆放位置都是随机产生的。通过不断循环,将所需排列的氨基酸依次排列,并将其相对于上一个氨基酸摆放的位置记录在一个特定的数组中(当向上移动一个位置时,数组存数值1;当向右移动一个位置时,数组存数值2;当向下移动一个位置时,数组存数值3;当向左移动一个位置时,数组存数值4)。当所有氨基酸都已经摆放完毕后,即创建出一个合法的CB2空间排放序列。
2.2 回溯机制在顺序排列氨基酸序列时,会出现序列后方的氨基酸重叠在之前已经拜访了氨基酸的情况,从而导致序列排放非法。随着氨基酸序列的增长,发生前述冲突的几率也会变大。为了防止这种情况发生,本文采取回溯机制[28]。当出现氨基酸重叠的情况时,将退回到此氨基酸的上一个氨基酸的摆放位置,由存放氨基酸摆放位置的数组可知此氨基酸的摆放位置,通过此摆放位置返回到上一个氨基酸的位置,并重新选择位置,同时记录不能摆放的位置和能够摆放氨基酸的位置。若所有位置都已验证是不可以摆放,那么将会再向上退回前一个氨基酸的位置。若可以摆放的位置剩下两个,则在剩下的两个位置中随机选一个;若可以摆放的位置剩下三个,则在剩下的三个位置中随机选一个。
回溯可以较好的解决了冲突,但是如果不限制回溯次数,极端情况下将会退化到初始氨基酸,将会极大的限制算法的执行效率。对于10 000个随机序列进行回溯次数的测定,实验结果如表 1所示。
| 表 1 回溯次数比较 Table 1 Comparison of backtracking times |
评估(计算)氨基酸能量的方法:从第一个氨基酸开始判断,当氨基酸不为H型氨基酸(疏水性氨基酸)时,则继续判断下一个氨基酸是否为H型氨基酸,当氨基酸为H型氨基酸(疏水性氨基酸)时,让此H型氨基酸的位置固定,通过存放氨基酸摆放位置的数组对后面的氨基酸进行摆放,同时对其后面的所有氨基酸(保证两个氨基酸在序列中不相连)判断是否同为H,当有后续氨基酸为H型氨基酸时,再判断此后续氨基酸与位置固定的第一个氨基酸的距离是否为1(即两个氨基酸在X轴上的坐标相同,在Y轴上的坐标相差1或者在Y轴上的坐标相同,在X轴上的坐标相差1),这是即可得到一个单位的能量值。通过遍历序列中的所有氨基酸,最后即可得到某个排放方式下CB2所对应的能量。
2.4 算法流程图本文中使用了蒙特卡罗算法,具体流程图见图 1。
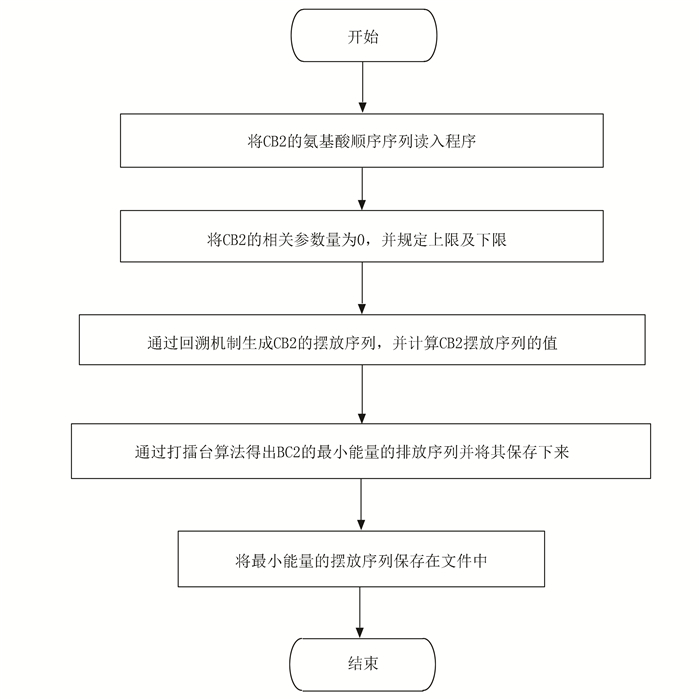
|
图 1 蒙特卡罗流程图 Figure 1 Monte Carlo flowchart |
实验计算机配置为:处理器:Intel 64位CPU,2.53 GHz;内存,4 GB;硬盘空间,128 GB。Windows 7(64位)操作系统,软件开发平台为Microsoft Visual C++ 6.0。项目代码为独立开发,源代码下载网址为:https://github.com/niceliangfan/CB2-Folding-Based-on-HP-Model-and-Monte-Carlo-Method/blob/master/Source%20Code。
3 实验结果通过对于大麻素受体CB2的HP折叠模型表达,使用带有回溯机制的蒙特卡罗方法进行求解。对本方法取得的三种最优值(E=-67)和其他较好结果的空间折叠构象排列表达如图 2所示。在图 2中,使用实心点表示H型氨基酸,空心表示P型氨基酸,加粗实心表示此处有能量累计。由图直观可知,节点排列越紧密,氨基酸的能量值就越小;反之,能量值越大。

|
图 2 CB2的空间折叠结果 Figure 2 Spatial folding results for CB2 |
由图 2可知,对于CB2序列而言,即使能量最低的三种折叠排列形式(见图 2中的(a)、(b)、(c),在空间表达上相互差别较大,且这三种折叠方式对于其他随机选取的折叠方式(见图 2中的(d)、(e)而言同样差比较大。这说明使用蒙特卡罗方法可以完全跳出多数智能优化算法的局部最优困扰,可以在全局范围内进行最优求解。
4 总结与展望目前,以HP模型为基础的大麻素受体CB2的二维空间结构测定的研究较少,对于CB2的二维折叠问题研究也开展的较少。本文工作对于CB2的空间构象研究具有一定的指导意义,对后续的分子对接和虚拟筛选等工作提供了很好的支撑。
在本文中使用了HP折叠预测模型对于大麻素受体的亚型CB2蛋白的折叠情况进行了研究,并使用了蒙特卡罗方法进行全局优化求解,取得了较好的折叠排列结果。
本文中使用的蒙特卡罗方法虽然具有全局寻优能力,但也存在收敛速度慢等问题,且在对大量的数据进行筛选时没有一定的针对性,既浪费了大量的计算时间又存在较多的偶然性因素,导致实验结果还有进一步的提升空间。在接下来的工作中,将引入遗传算法等对序列排放方法进行进一步优化,以期得到CB2更好的折叠构象能量值。
蛋白质在体内是在三维空间中进行折叠的,因此在二维空间研究蛋白质折叠具有局限性。在接下来的工作中,将对序列排放方法进行进一步优化并尝试将把CB2放在三维空间中进行折叠研究,以期得到CB2的更为准确的符合实际生活的三维空间折叠构象能量值。
| [1] |
MUÑOZ V, CAMPOS L A, SADQI M. Limited cooperativity in protein folding[J]. Current Opinion in Structuaral Biology, 2016, 36: 58-66. DOI:10.1016/j.sbi.2015.12.001 (  0) 0) |
| [2] |
MIRCEVA G, DAVCEV D. Incorporating several features in the protein ray descriptor for more accurate protein 3D structure retrieval[C]. ACM(Firenze, Italy): Workshop on 3d Object Retrieval, 2010.DOI: 10.1145/1877808.1877820. https://dl.acm.org/citation.cfm?id=1877808.1877820
(  0) 0) |
| [3] |
ENGLANDER S W, MAYNE L, KAN Z Y, et al. Protein folding-how and why: By hydrogen exchange, fragment separation, and mass spectrometry[J]. Annual Review of Biophysics, 2016, 45: 135-152. DOI:10.1146/annurev-biophys-062215-011121 (  0) 0) |
| [4] |
孙鹏飞, 张健沛. 基于量子遗传算法的蛋白质折叠结构预测[J]. 哈尔滨工程大学学报, 2010, 31(1): 92-97. SUN Pengfei, ZHANG Jianpei. A method for predicting protein folding based on a quantum genetic algorithm[J]. Journal of Harbin Engineering University, 2010, 31(1): 92-97. DOI:10.3969/j.issn.1006-7043.2010.01.016 (  0) 0) |
| [5] |
吕志鹏, 黄文奇. 求解蛋白质结构预测问题的局部搜索算法[J]. 计算机科学, 2005, 32(11): 148-151. LÜ Zhipeng, HUANG Wenqi. Local search algorithm for solving protein structure prediction problem[J]. Computer Science, 2005, 32(11): 148-151. DOI:10.3969/j.issn.1002-137X.2005.11.040 (  0) 0) |
| [6] |
王庆喜, 朱丽华. 基于布谷鸟搜索算法的蛋白质能量优化[J]. 浙江农业学报, 2017, 29(7): 1216-1220. WANG Qingxi, ZHU Lihua. Protein energy optimization based on cuckoo search algorithm[J]. Acta Agriculturae Zhejiangensis, 2017, 29(7): 1216-1220. DOI:10.3969/j.issn.1004-1524.2017.07.22 (  0) 0) |
| [7] |
张菁, 郭丹. 拟蛇算法在蛋白质折叠模拟中的应用可行性研究[J]. 软件, 2017, 38(2): 75-79. ZHANG Jing, GUO Dan. Application feasibility study on snake algorithm of protein folding simulation[J]. Computer Engineering & Software, 2017, 38(2): 75-79. DOI:10.3969/j.issn.1003-6970.2017.02.017 (  0) 0) |
| [8] |
CUI Z, ZHANG Y. Swarm intelligence in bioinformatics: Methods and implementations for discovering patterns of multiple sequences[J]. Journal of Nanoscience & Nanotechnology, 2014, 14(2): 1746-1757. DOI:10.1166/jnn.2014.9196 (  0) 0) |
| [9] |
SHIN J M, LEE B, CHO K H. A new efficient conformational search method for ab initio protein folding study: Window growth evolutionary algorithm[J]. Bulletin of the Korean Chemical Society, 2016, 37(12): 1971-1976. DOI:10.1002/bkcs.11006 (  0) 0) |
| [10] |
THANGSUNAN P, KITTIWACHANA S, MEEPOWPAN P, et al. Rapid activity prediction of HIV-1 integrase inhibitors: Harnessing docking energetic components for empirical scoring by chemometric and artificial neural network approaches[J]. Journal of Computer-Aided Molecular Design, 2016, 30(6): 471-488. DOI:10.1007/s10822-016-9917-0 (  0) 0) |
| [11] |
SCUTT A, WILLIAMSON E M. Cannabinoids stimulate fibroblastic colony formation by bones marrow cells indirectly via CB2 receptors[J]. Calcified Tissue International, 2007, 80(1): 50-59. DOI:10.1007/s00223-006-0171-7 (  0) 0) |
| [12] |
ARÉVALO-MARTÍN A, VELA J M, MOLINA-HOLGADO E, et al. Therapeutic action of cannabinoids in a murine model of multiple sclerosis[J]. The Journal of Neuroscience: the Official Journal of the Society for Neuroscience, 2003, 23(7): 2511-2516. DOI:10.1523/JNEUROSCI.23-07-02511.2003·Source:PubMed (  0) 0) |
| [13] |
BAKER D, PRYCE G, CROXFORD J L, et al. Cannabinoids control spasticity and tremor in a multiple sclerosis model[J]. Nature, 2000, 404(6773): 84-87. DOI:10.1038/35003583 (  0) 0) |
| [14] |
BROMBERG K D, MA'AYAN A, NEVES S R. Design logic of a cannabinoid receptor signaling network that triggers neurite outgrowth[J]. Science, 2008, 320(5878): 903-909. DOI:10.1126/science.1152662 (  0) 0) |
| [15] |
JIANG S, ALBERICH-JORDA M, ZAGOZDZON R, et al. Cannabinoid receptor 2 and its agonists mediate hematopoiesis and hematopoietic stem and progenitor cell mobilization[J]. Blood, 2011, 117(3): 827-838. DOI:10.1182/blood-2010-01-265082 (  0) 0) |
| [16] |
马赛, 王亚斌, 王强, 等. 大麻素Ⅱ型受体通过激活心脏祖细胞促进小鼠心肌梗死后内源性的心肌再生[J]. 中国科学:生命科学, 2014, 44(7): 685-693. MA Sai, WANG Yabin, WANG Qiang, et al. Effects of cannabinoid receptor type 2 on endogenous myocardial regeneration by activating cardiac progenitor cells in mouse infarcted heart[J]. Science China: Life Science, 2014, 44(7): 685-693. DOI:10.1007/s11427-013-4604-z (  0) 0) |
| [17] |
史小红. 基于HP模型的蛋白质折叠问题的研究[J]. 生物信息学, 2016, 14(2): 112-116. SHI Xiaohong. Research on protein folding based on HP model[J]. Chinese Journal of Bioinformatics, 2016, 14(2): 112-116. DOI:10.3969/j.issn.1672-5565.2016.02.08 (  0) 0) |
| [18] |
刘赟, 王存新, 王宝翰, 等. 基于格子模型的蛋白质设计方法[J]. 生物化学与生物物理进展, 2004, 31(2): 172-176. LIU Yun, WANG Cunxin, WANG Baohan, et al. A Protein design procedure based on the lattice model[J]. Progress in Biochemistry and Biophysics, 2004, 31(2): 172-176. DOI:10.3321/j.issn:1000-3282.2004.02.014 (  0) 0) |
| [19] |
黄文奇, 李宗旻. 基于模拟退火算法的蛋白质折叠问题求解[J]. 计算机工程与应用, 2005, 41(7): 40-41. HUANG Wenqi, LI Zongmin. Solutions for protein folding problem based on simulated annealing algorithm[J]. Computer Engineering and Applications, 2005, 41(7): 40-41. DOI:10.3969/j.issn.1002-8331-(2005)07-0040-02 (  0) 0) |
| [20] |
LAGNEUX C, LAMONTAGNE D. Involvement of cannabinoids in the cardioprotection induced by lipopolysaccharide[J]. British Journal of Pharmacology, 2010, 132(4): 793-796. DOI:10.1038/sj.bjp.0703902 (  0) 0) |
| [21] |
LIU Q R, PAN C H, HISHIMOTO A, et al. Species differences in cannabinoid receptor 2(cnr2gene): Identification of novel human and rodent cb2 isoforms, differential tissue expression and regulation by cannabinoid receptor ligands[J]. Genes Brain & Behavior, 2010, 8(5): 519-530. DOI:10.1111/j.1601-183X.2009.00498.x (  0) 0) |
| [22] |
THOMPSON M D, COLE D E C, CAPRA V, et al. Pharmacogenetics of the g protein-coupled receptors[J]. Methods in Molecular Biology, 2014, 1175(4): 189. DOI:10.1007/978-1-4939-0956-8_9 (  0) 0) |
| [23] |
RAJESH M, MUKHOPADHYAY P, HASKÓ G, et al. CB2 cannabinoid receptor agonists attenuate TNF-alpha-induced human vascular smooth muscle cell pro1iferation and migration[J]. British Journal of Pharmacology, 2010, 153(2): 347-357. DOI:10.1038/sj.bjp.0707569 (  0) 0) |
| [24] |
PICONE R P, KENDALL D A. Minireview:From the bench, toward the clinic: Therapeutic opportunities for cannabinoid receptor modulation[J]. Molecular Endocrinology, 2015, 29(6): 801-813. DOI:10.1210/me.2015-1062 (  0) 0) |
| [25] |
SARFARAZ S, ADHAMI V M, SYED D N, et al. Cannabinoids for cancer treatment: Progress and promise[J]. Cancer Research, 2008, 68(2): 339-342. DOI:10.1158/0008-5472.CAN-07-2785 (  0) 0) |
| [26] |
MORALES P, BLASCO-BENITO S, ANDRADAS C, et al. Selective, Nontoxic CB2 Cannabinoid o-Quinone with in vivo activity against triple-negative breast cancer[J]. Journal of Medicinal Chemistry, 2015, 58(5): 2256-2264. DOI:10.1021/acs.jmedchem.5b00078 (  0) 0) |
| [27] |
李正夫.药物分子对接优化算法及在云平台中的应用[D].大连: 大连理工大学, 2014.DOI: 10.7666/d.y2728903. LI Zhengfu.Optimization algorithms for drug moleclue docking and application on cloud platform[D].Dalian: Dalian University of Technology, 2014. DOI: 10.7666/d.y2728903. http://cdmd.cnki.com.cn/Article/CDMD-10141-1015573918.htm (  0) 0) |
| [28] |
JOHNSON C M, KATIKIREDDY A. A genetic algorithm with backtracking for protein structure prediction[C]. ACM: Conference on Genetic and Evolutionary Computation, 2006.DOI: 10.1145/1143997.1144049. http://citeseerx.ist.psu.edu/viewdoc/download?doi=10.1.1.420.5804&rep=rep1&type=pdf
(  0) 0) |
 2018, Vol. 16
2018, Vol. 16


