乙肝病毒(HBV)感染是一个在全球范围危害公众健康的问题[1]。全球约有20亿人感染过乙肝病毒,超过3.5亿慢性乙肝携带者[2]。其中大约有15%~40%的乙肝患者会发展成肝硬化、肝功能衰竭或肝细胞癌[3]。每年大约有50~120万人因乙肝死亡[4-5],感染乙肝病毒已成为全球第十大死因[6]。感染乙肝病毒使肝细胞癌的发病率增加[7],每年约有30~50万人因感染乙肝病毒罹患肝细胞癌,肝细胞癌已成为常见的癌症[8],是全球癌症死亡的第二大原因[9]。全球人口大约有45%居住在HBV高发地区[10-11]。
随着高通量平台大量应用于医学,生物芯片成为临床样本病理分析的有效工具,可以快速、高效、精确、低成本地进行信号通路探索、疾病预测分子诊断、新药靶点发现、肿瘤反应预测等工作[12-13],本研究便采取了芯片分析的方法来探索乙肝感染造成的基因表达和信号通路的改变,从GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)下载了基因表达谱数据集GSE83148后对样本进行质量检测、数据标准化后筛选出fold change≥2,p-value < 0.05的差异表达基因,接着对差异表达基因进行了GO和KEGG富集分析和GeneMANIA(http://genemania.org/)基因网络互作分析,发现了一些HBV感染细胞后的关键基因和信号通路,这对我们研究HBV感染发病以及癌变的具体机制有一定意义。
1 材料和方法 1.1 材料芯片数据集GSE83148从GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)下载,pHBV1.3质粒,pcDNA3.1质粒,hepG2细胞为实验室自有,RNA提取试剂盒购自天根生化科技(北京)有限公司,PCR生化反应试剂购自宝生物工程(大连)有限公司,引物由华津生物科技有限公司合成,veriti96-well thermal cycler PCR仪购置于Applied Biosystems公司,5424型高速离心机购置于eppendorf公司。
1.2 实验方法 1.2.1 获得差异表达基因GSE83148基因表达谱数据集由美国Affymetrix公司制作,基于GPL570平台。GSE83148包含122个HBV感染者的肝细胞样本和6个健康对照肝细胞样本。下载CEL格式的原始数据,使用R语言做质量检测后将质量合格的芯片做数据标准化处理,然后用经典t检验筛选出fold change≥2,p-value < 0.05的差异表达基因。
1.2.2 差异表达基因的GO分析和KEGG分析将筛选获得的差异表达基因上传到DAVID生物信息学数据库(https://david.ncifcrf.gov/),进行GO分析和KEGG分析,找出差异表达基因的分子功能、细胞组成、生物过程和富集的KEGG信号通路。
1.2.3 差异表达基因互作网络分析将获得的差异表达基因上传到GeneMANIA(http://genemania.org/)数据库进行基因网络互作分析,选择合适的”network”和”function”,获得差异表达基因间的网络互作关系和枢纽基因图。
1.2.4 Real-time PCR验证上述预测结果(1) 转染细胞。复苏HepG2细胞,传代3次后,布6孔板,待细胞有60%融合度时,实验组每孔转染4 μg乙肝病毒1.3倍体质粒pHBV1.3,对照组转染4 μg空载pcDNA3.1质粒,48 h后收集样品。
(2) 总RNA提取。收取样品后,进行RNA的提取。收集样品时,弃去细胞孔中的培养液,用PBS清洗3次,各1~2 min。然后向每个细胞孔中添加400 μL Trizol试剂,室温放置2~3 min后,充分吹打以保证细胞从孔壁上完全脱落,然后将液体收集于RNase free的EP管中。
提取RNA的步骤如下:①将上述EP管放在混匀器上剧烈振荡20 s,然后室温静置15 min;②向每个EP管内加入80 μL氯仿,置于混匀器剧烈振荡2 s,室温放置5 min后,12 000 r/min离心15 min。离心后取上层的水相到另一个RNase free的EP管内,然后加等量异丙醇,混合均匀,-20 ℃放置30 min以上;③在4 ℃,12 000 r/min离心15 min,弃去上清,向EP管内加入300 μL 75%的无水乙醇,洗涤沉淀,然后4 ℃,12 000 r/min离心5 min。弃去上清,将EP管盖子打开,置于超净台中,直到沉淀被吹至透明后,向每个EP管内加入20 μL RNase free水。
(3) 反转录。按照反转录试剂盒(TaKaRa公司)提供的说明书获得cDNA。步骤简述如下:在10 μL体系中,5X PrimScript RT Master Mix 2 μL,total RNA 4 μL,RNase free水4 μL。反应体系中,RNA的用量最大为500 ng。将反应体系设置为37 ℃ 15 min;85 ℃ 5 s。
(4) 引物的设计和合成。根据GenBank公布的全基因序列作为设计引物的参考序列,用Primer Premier 5设计,引物合成由上海华津生物科技有限公司完成。目的和内参引物序列见表 1。
| 表 1 基因引物列表 Table 1 List of gene primers |
Real-time PCR反应程序为 95 ℃ 30 s, 95 ℃ 5 s,62 ℃ 31 s,一共40个循环;95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s。结果用β-actin做为内参,用2-△△ct法进行相对定量分析。
2 结果分析 2.1 获得差异表达基因下载GSE83148数据集(CEL格式),芯片信息如表 2。
| 表 2 芯片GSE83148的基本信息 Table 2 Basic information of chip GSE83148 |
对样品芯片进行灰度扫描、RNA降解分析、以及用simpleaffy包进行分析后,表明芯片制作的技术误差在允许范围内,因此将122张HBV样本和6张对照样本全部纳入分析,图 1为122张HBV芯片和6张对照组芯片灰度扫描箱线图。
|
图 1 芯片灰度扫描箱线图 Figure 1 Chip grayscale scanning box plot |
从芯片中提取基因数据37 707个,用RMA法进行背景校正,中位数标准化后,筛选出HBV组相较于对照组fold change≥2,p-value < 0.05的上调表达基因44个(见表 3)。
| 表 3 fold change≥2,p-value < 0.05的上调基因 Table 3 Up-regulated genes with fold change≥2, p-value < 0.05 |
对获得的差异表达基因进行GO和KEGG分析(见表 4)。GO分析结果表明,HBV感染的肝细胞上调的差异表达基因主要富集的细胞过程有:对伽玛辐射的反应,蛋白质四聚化,细胞对紫外线反应,对药物反应;主要富集的细胞代谢区域为线粒体。BAK1基因和TP63基因被富集入多条信号通路。BAK1基因编码的蛋白属于BCL2蛋白家族, BCL2家族成员形成寡聚体或异二聚体并且充当涉及各种细胞活性的抗凋亡或促凋亡调节剂, 这种蛋白定位于线粒体,并起到诱导细胞凋亡的作用, 它与线粒体电压依赖性阴离子通道的开放相互作用并加速其开放,这导致膜电位的丧失和细胞色素c的释放, 暴露于细胞应激后,该蛋白还与肿瘤抑制因子P53相互作用[14]。TP63基因编码的蛋白是p53家族转录因子的成员[15],与NF-KB信号通路相互作用[16]。
| 表 4 差异表达基因的GO分析 Table 4 GO analysis of differentially expressed genes |
将上述获得的差异表达基因做KEGG通路富集(见表 5),未获得富集基因的信号通路。将筛选条件放宽至fold change≥1.5, p-value < 0.05后获得差异表达基因富集KEGG信号通路,获得的信号通路有:乙型肝炎信号通路、病毒癌变信号通路、FoxO信号通路、PI3K-Akt信号通路。FoxO信号通路在细胞凋亡,细胞周期控制,葡萄糖代谢,氧化应激抗性等方面有着重要作用[17],FoxO信号通路的调节机制是响应于胰岛素以及几种生长因子,通过磷脂酰肌醇3-激酶(PI3K)下游的丝氨酸-苏氨酸激酶Akt /蛋白激酶B(Akt / PKB)磷酸化,三个保守残基磷酸化导致FoxO蛋白从细胞核输出到细胞质,从而降低FoxO靶基因的表达。而应激激活的c-Jun N端激酶(JNK)和能量感应AMP激活蛋白激酶(AMPK)在氧化和应激刺激下磷酸化并激活FoxO。除了PKB,JNK和AMPK之外,FoxO还受到多个参与者的调控,包括磷酸化,乙酰化,甲基化和泛素化等。磷脂酰肌醇3’-激酶(PI3K)-Akt信号通路可被多种类型的细胞刺激或毒性损伤激活,调节转录,翻译,增殖,生长和存活等基本的细胞功能。生长因子与其受体酪氨酸激酶(RTK)或G蛋白偶联受体(GPCR)的结合分别刺激PI3K的Ia和Ib亚型。PI3K催化细胞膜上磷脂酰肌醇-3, 4, 5-三磷酸(PIP3)的产生。第二信使PIP3激活Akt,激活后Akt通过磷酸化参与凋亡,蛋白质合成,代谢和细胞周期调节等过程。
| 表 5 差异表达基因的KEGG分析 Table 5 KEGG analysis of differentially expressed genes |
差异表达基因生成的互作网络图如图 2所示:BAK1、GNG5、ATP5B、NDUFS1、NDUFS2、IMMT、MLX、OPA1等9个基因被富集,这些基因均与线粒体氧化呼吸链有关,基因NDUFS1、NDUFS2、ATP5B、COX7B、OPA1处于互作网络中的核心位置。

|
图 2 GeneMANIA网络图 Figure 2 GeneMANIA network diagram 注:彩图见电子版(http://swxxx.alljournals.cn/ch/index.aspx.2018年第4期)。 |
NDUFS1、NDUFS2是线粒体呼吸链复合体1的核心基因,复合体1是线粒体内膜上最大的蛋白质复合物,催化NADH氧化,既是电子传递体又是质子传递体。呼吸作用会产生活性氧,活性氧化学性质活泼,既是电子供体又是电子受体,是生命活动中不可缺失的活性物质,可以提高某些酶的活性,如肝脏中一些合成酶和解毒酶,分析结果显示与NDUFS1基因共表达的基因有NDUFS2, ATP5B, IMMT, MLX, PRELID3B, CAST, TFRC, GNG5,COX7B, GRSF1,METAP1,NCKAP1,PGK1, 共定位的基因有COX7B 和NDUFS2; 与NDUFS2共表达的基因有NDUFS1,OPA1,GSS, ATP5B, BAK1,METAP1,MTRF1L, COX7B, G3BP2,ALAS1,MLX, CTNNA1,SPTLC2,TFRC, PRELID3B, RBM5,CLCN4,LDHA, IMMT, MRTTL2A, GRSF1,共定位的基因有NDUFS1和COX7B。COX7B编码细胞色素c氧化酶的一个的结构亚基,线粒体呼吸链复合物IV的重要组成部分,是线粒体呼吸不可缺失的部分[18],本研究筛选出的差异表达基因中与COX7B共表达的基因有IMMT, NDUFS1,NDUFS2,ATP5B, PGK1,ALAS1,GNG5,KRT10,CAST, 与COX7B共定位的基因有ATP5B,PGK1,GRSF1,AK1。ATP5B编码ATP合酶的Fl部分的β亚基,β亚基是ATP酶氧化磷酸化合成ATP的限速组分,它是ATP酶复合体中唯一的一个催化亚单位[19],筛选的差异表达基因中与ATP5B共表达的基因有NDUFS1,NDUFS2,MGST1,IMMT, GRSF1,VAV2,MTRF1L, METAP1,ALAS1,MLX, CTNNA1,SPTLC2,PRELID3B, TFRC, 共定位的基因有COX7B, SPTLC2,KRT10,GRSF1,AK1。OPA1位于线粒体内膜,参与线粒体嵴重塑,阻止细胞色素C释放,是调节线粒体分裂与融合的关键基因[20],与OPA1共表达的基因有CAST, CDYL, G3BP2,ANKRD17,NDUFS2,ZFC3H1,NCKAP1。这些表达上调说明HBV感染后肝细胞氧化还原反应活跃。
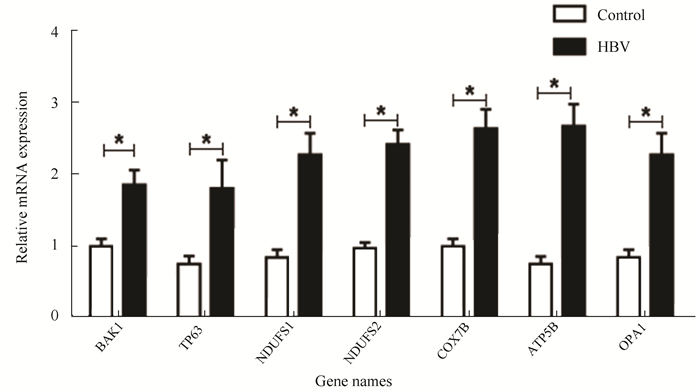
2.5 Real-time PCR验证芯片分析结果如图 3所示,X轴表示基因名称,Y轴表示基因表达量之比,HepG2细胞在转染乙肝病毒1.3倍体质粒48h后,BAK1、TP63、NDUFS1、NDUFS2、COX7B、ATP5B、OPA1的mRNA表达水平均显著升高(P < 0.05),与预测结果一致。
|
图 3 Real-time PCR验证芯片分析结果 Figure 3 Real-time PCR verification chip analysis results 注:图中结果以β-actin为内参,用2-△△ct法进行相对定量分析,转乙肝病毒1.3倍体质粒组中BAK1、TP63、NDUFS1、NDUFS2、COX7B、ATP5B、OPA1mRNA表达水平,与正常对照相比, *P < 0.05 (n=3). |
本研究从GSE83148基因表达谱中获得37 707个基因,从这37 707个基因中获得HBV组fold change≥2,px-value < 0.05的差异表达基因44个,对这44个上调差异表达基因做了一系列分析后获得7个关键基因,BAK1,TP63,NDUFS1,NDUFS2,COX7B,ATP5B,OPA1,这些基因均与线粒体以及氧化呼吸链相关,提示患者感染乙肝病毒后线粒体氧化还原反应活跃。氧化还原活跃后会产生更多的活性氧, 已有研究表明感染乙肝病毒后会,细胞会产生大量活性氧,活性氧增多导致机体氧化应激反应(Oxidative stress)造成细胞损伤,这一损伤参与多种病理机制[21]。线粒体靶向抗氧化剂可以显著改善HBV感染后的线粒体和抗病毒CD8T功能[22]。但HBV感染后如何造成这些基因表达量增高的具体原因尚不明了,仍需进一步探索研究。本研究发现HBV感染者差异表达基因富集的信号通路有:乙型肝炎信号通路、病毒癌变信号通路、FoxO信号通路、PI3K-Akt信号通路。FoxO信号通路响应于胰岛素以及几种生长因子,通过磷脂酰肌醇3-激酶(PI3K)下游的丝氨酸-苏氨酸激酶Akt /蛋白激酶B(Akt / PKB)磷酸化,在细胞凋亡,细胞周期控制,葡萄糖代谢,氧化应激抗性等方面有着重要作用,且与PI3K-Akt信号通路联系紧密。筛选出的关键基因和富集的信号通路均与细胞氧化应激相关,表明HBV感染后的氧化损伤可能是造成感染者肝细胞损伤及细胞癌变的关键因素,但具体机制尚不明确,仍需进一步探究。
| [1] |
范竹萍, 邱德凯. 乙型肝炎的流行病学[J]. 胃肠病学, 2002, 7(5): 289-292. FAN Zhuping, QIU Dekai. Epidemiology of hepatitis B[J]. Gastroenterology, 2002, 7(5): 289-292. DOI:10.3969/j.issn.1008-7125.2002.05.010 (  0) 0) |
| [2] |
PREVISANI N, LAVANCHY D. World health organization. Fact sheet[J]. Indian Journal of Chest Diseases & Allied Sciences, 2000, 42(2): 126. (  0) 0) |
| [3] |
LOK A S. Chronic hepatitis B[J]. New England Journal of Medicine, 2002, 346(22): 1682-1683. DOI:10.1002/hep.21513 (  0) 0) |
| [4] |
MAHONEY F J. Update on diagnosis, management, and prevention of hepatitis B virus infection[J]. Clinical Microbiology Reviews, 1999, 12(2): 351-366. DOI:10.1128/CMR.12.2.351 (  0) 0) |
| [5] |
LIAW Y F, CHU C M. Hepatitis B virus infection[J]. New England Journal of Medicine, 2007, 12(3): 160-167. DOI:10.1016/j.siny.2007.01.013 (  0) 0) |
| [6] |
徐舒.白背叶根抗鸭乙肝病毒的实验研究[D].广州: 南方医科大学(原中国人民解放军第一军医大学), 2006. XU Shu, Experimental study on anti-duck hepatitis B virus in white back leaf root[D].Guangzhou: Southern Medical University, 2006. http://cdmd.cnki.com.cn/article/cdmd-90023-2006177254.htm (  0) 0) |
| [7] |
赵作银, 佟月婷. 原发性肝癌与乙型肝炎病毒感染关系再探讨[J]. 中西医结合肝病杂志, 2006, 16(3): 180-181. ZHAO Zuoyin, TONG Yueting. Further discussion on the relationship between primary liver cancer and hepatitis B virus infection[J]. Chinese Journal of Integrated Traditional and Western Medicine, 2006, 16: 180-181. DOI:10.3969/j.issn.1005-0264.2006.03.026 (  0) 0) |
| [8] |
PARKIN D M, BRAY F, FERLAY J, et al. Estimating the world cancer burden: Globocan 2000[J]. International Journal of Cancer, 2001, 94(2): 153. DOI:10.1002/ijc.1440 (  0) 0) |
| [9] |
LEVRERO M, ZUCMAN-ROSSI J. Mechanisms of HBV-induced hepatocellular carcinoma[J]. Journal of Hepatology, 2016, 64(1 Suppl): S84. DOI:10.1016/j.jhep.2016.02.021 (  0) 0) |
| [10] |
LAFARO K J, DEMIRJIAN A N. Epidemiology of hepatocellular carcinoma[J]. Surgical Oncology Clinics of North America, 2015, 24(1): 1-17. DOI:10.1016/j.soc.2014.09.001 (  0) 0) |
| [11] |
BANATVALA J E, VAN D P. Hepatitis B vaccine-do we need boosters?[J]. Journal of Viral Hepatitis, 2003, 10(1): 1. DOI:10.1046/j.1365-2893.2003.00400.x (  0) 0) |
| [12] |
FENG L, DU J, LIU J, et al. Identification of key target genes and pathways in laryngeal carcinoma[J]. Oncology Letters, 2016, 12(2): 1279-1286. DOI:10.3892/ol.2016.4750 (  0) 0) |
| [13] |
GUO Y, BAO Y, MA M, et al. Identification of key candidate genes and pathways in colorectal cancer by integrated bioinformatical analysis[J]. International Journal of Molecular Sciences, 2017, 18(4): 722. DOI:10.3390/ijms18040722 (  0) 0) |
| [14] |
NAM K H, LI J. BRI1/BAK1, a receptor kinase pair mediating brassinosteroid signaling[J]. Cell, 2002, 110(2): 203-212. DOI:10.1016/S0092-8674(02)00814-0 (  0) 0) |
| [15] |
THOMASON H A, DIXON M J, DIXON J. Facial clefting in Tp63 deficient mice results from altered Bmp4, Fgf8 and Shh signaling[J]. Developmental Biology, 2008, 321(1): 273-282. DOI:10.1016/j.ydbio.2008.06.030 (  0) 0) |
| [16] |
陈中, 张嘉玲, 杨歆萍, 等. 抑癌基因TP53及新家族成员TP63和TP73的研究新进展[J]. 内蒙古医科大学学报, 2013, 35(1): 57-62. CHEN Zhong, ZHANG Jialing, YANG Xinping, et al. New progress in the study of tumor suppressor gene TP53 and new family members TP63 and TP73[J]. Journal of Inner Mongolia Medical University, 2013, 35(1): 57-62. DOI:10.3969/j.issn.1004-2113.2013.01.012 (  0) 0) |
| [17] |
ESSERS M A, WEIJZEN S, DE VRIES MITS A M, et al. FOXO transcription factor activation by oxidative stress mediated by the small GTPase Ral and JNK[J]. The EMBO Journal, 2014, 23(24): 4802-4812. DOI:10.1038/sj.emboj.7600476 (  0) 0) |
| [18] |
INDRIERI A, VANRAHDEN V A, TIRANTI V, et al. Mutations in COX7B cause microphthalmia with linear skin lesions, an unconventional mitochondrial disease[J]. American Journal of Human Genetics, 2012, 91(5): 942-949. DOI:10.1016/j.ajhg.2012.09.016 (  0) 0) |
| [19] |
王春枝.低氧胁迫对鲢线粒体ATP酶活性及β、γ、δ亚基表达的影响[D].武汉: 华中农业大学, 2014. DOI: 10.7666/d.Y2565765. WANG Chunzhi. Effects of mitochondria ATPase activity and the expressions of β, γ, δ subunits during hypoxia stress in hypophthalmichthys molitrix[D].Wuhan: Huazhong Agricultural University, 2014.DOI: 10.7666/d.Y2565765. http://cdmd.cnki.com.cn/Article/CDMD-10504-1014231428.htm (  0) 0) |
| [20] |
孙丽娟.前脑特异性Presenilins双敲除小鼠模型中线粒体结构与功能的探讨[D].上海: 华东师范大学, 2010. DOI: 10.7666/d.y1742228. SUN Lijuan. Discussion on mitochondrial structure and function in forebrain-specific Presenilins double knockout mouse model[D]. Shanghai: East China Normal University, 2010.DOI: 10.7666/d.y1742228. http://cdmd.cnki.com.cn/Article/CDMD-10269-2010199095.htm (  0) 0) |
| [21] |
YUAN K, LEI Y, CHEN H N, et al. HBV-induced ROS accumulation promotes hepatocarcinogenesis through Snail-mediated epigenetic silencing of SOCS3[J]. Cell Death & Differentiation, 2016, 23(4): 616-627. DOI:10.1038/cdd.2015.129 (  0) 0) |
| [22] |
FISICARO P, BARILI V, MONTANINI B, et al. Targeting mitochondrial dysfunction can restore antiviral activity of exhausted HBV-specific CD8 T cells in chronic hepatitis B[J]. Nature Medicine, 2017, 23(3): 327. DOI:10.1038/nm.4275. (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


