2. 山西医科大学 基础医学院生物化学与分子生物学教研室,太原 030001;
3. 长治医学院 附属和济医院骨科,山西 长治 046000
2. Department of Biochemistry and Molecular Biology, Shanxi Medical University, Taiyuan 030001, China;
3. Department of Orthopaedics, Heji Hospital Affiliated to Changzhi Medical College, Changzhi 046000, Shanxi, China
FOXD3最早在胚胎干细胞中被发现,在神经嵴发育和干细胞生物学过程中发挥重要作用[1]。在小鼠早期胚胎中,FOXD3基因敲除鼠在发育过程中会过早死亡,推测FOXD3对于建立体外小鼠胚胎干细胞系至关重要,能够维持内细胞团、神经嵴和滋养层祖细胞中多能细胞的特性[2]。此外,FOXD3还能够调控细胞周期、细胞分化、增殖、衰老、凋亡和代谢等诸多生物学过程[3]。近期研究表明,FOXD3在细胞癌变过程中扮演重要角色,与癌症的发生发展和化疗耐药有关[4]。
研究人员发现在黑色素瘤、神经母细胞瘤和乳腺癌等很多癌组织内FOXD3基因表达水平下调,推测FOXD3在多种肿瘤细胞中起到抑癌作用,但下调机制及其下游靶基因尚不清楚[5-6]。胃癌、结直肠癌和慢性淋巴细胞白血病细胞中,启动子高甲基化可能引起FOXD3表观遗传失活,但启动子甲基化是否为造成FOXD3下调的主要原因尚待研究[7-9]。
另外,FOXD3在癌细胞中的功能和作用机制尚未完全阐明。胶质瘤细胞中FOXD3过量表达抑制细胞增殖,促进血清饥饿诱导的细胞凋亡[1]。甲状腺未分化癌细胞中,下调FOXD3表达能够促进癌细胞侵袭和上皮间质转化,抑制细胞凋亡; 沉默FOXD3促使p-ERK表达水平升高,推测FOXD3负调控MAPK/ERK信号途径[10]。结肠癌细胞中下调FOXD3表达促进癌细胞增殖和侵袭,抑制细胞凋亡; 沉默FOXD3可激活EGFR/Ras/Raf/MEK/ERK通路[11]。由此推测,FOXD3在不同类型的恶性肿瘤中,可能通过不同机制发挥抑癌作用。
越来越多的证据表明,FOXD3是一个有价值的肿瘤预后标志物。Chu等发现FOXD3表达水平下调与乳腺癌患者预后差有关[5]。Cheng等发现FOXD3启动子甲基化的胃癌患者,其生存时间更短[7]。Li等研究表明,FOXD3高表达与神经母细胞瘤患者预后良好有关[6]。因此,FOXD3作为一个潜在的肿瘤标志物,其分子功能和作用机制值得深入研究。本文使用生物信息学分析方法,研究FOXD3蛋白的理化性质和结构功能,筛选与其相互作用的蛋白,旨在为深入研究FOXD3的作用机制提供思路。
1 材料与方法 1.1 FOXD3蛋白序列的获得使用美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)的蛋白质数据库(https://www.ncbi.nlm.nih.gov/),以“FOXD3+物种名”为关键词,搜索得到人FOXD3蛋白的氨基酸序列信息。
1.2 系统进化树构建在NCBI中,使用BLASTp搜索得到不同物种中FOXD3的同源序列。运用Clustal2.1软件对同源蛋白间的多重序列进行比对。运用MEGA6软件,Neighbor-joining方法,设置Boot-strap分析重复数为1 000,构建系统进化树[12]。
1.3 FOXD3的理化性质分析运用ProtParam(http://web.expasy.org/protparam/)在线工具,对FOXD3的分子式、等电点、酸碱性和稳定性等理化性质进行分析。运用Protscale在线工具(http://web.expasy.org/protscale/)预测FOXD3的亲疏水性。运用SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)预测FOXD3有无信号肽和切割位点,运用TMHMM 2.0在线工具(http://www.cbs.dtu.dk/services/TMHMM/)预测FOXD3有无跨膜区域。
1.4 FOXD3的组织表达特异性和亚细胞定位预测运用NCBI中UniGene数据库的EST结果,预测FOXD3在正常组织和癌变组织中的表达情况。运用PSORTII(https://psort.hgc.jp/)对FOXD3的亚细胞定位进行预测。
1.5 FOXD3的二级结构和高级结构分析运用SOPMA工具(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)对FOXD3的二级结构及各成分所占比例进行预测。运用NCBI的Conserved Domain数据库对FOXD3的结构域进行分析。运用SWISS-MODEL建模服务器(https://swissmodel.expasy.org/)预测FOXD3的三维结构。
1.6 FOXD3的相互作用蛋白预测运用STRING数据库(http://string-db.org/),设置为高置信度0.7,不限制数量,构建与FOXD3相互作用的蛋白网络。
2 结果与分析 2.1 FOXD3蛋白质多序列比对和进化关系分析在NCBI数据库中搜索到人的FOXD3蛋白在哺乳动物、鱼类和两栖类中的同源序列。人FOXD3蛋白的氨基酸序列与黑猩猩、野猪、小鼠、鸡、大鼠、黄鳝、非洲爪蟾和斑马鱼的相似性分别为99.58%、96.05%、89.81%、64.66%、61.71%、60%、59.46%和58.42%,与物种进化程度相一致,表明FOXD3蛋白在进化过程中高度保守(见图 1)。
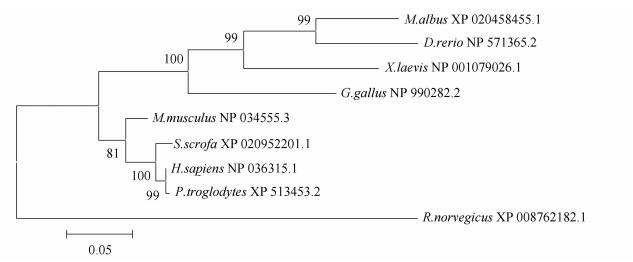
|
图 1 FOXD3蛋白的分子进化树 Figure 1 Phylogenetic tree of FOXD3 protein |
人FOXD3基因(NM_012183.2)位于1号染色体上(1p31.3),含有1个外显子,编码产物NP_036315.1为该基因的共识编码序列。FOXD3蛋白分子式为C2091H3279N597O656S11,含有478个氨基酸,分子量为47 630.30 Da。该蛋白理论等电点为6.01,整个序列中带负电荷的酸性氨基酸(Asp和Glu)共有37个,带正电荷的碱性氨基酸(Arg和Lys)共有32个,因此属于酸性蛋白质。FOXD3在哺乳动物网织红细胞内半衰期为30 h,不稳定系数为53.49,属于不稳定蛋白质。脂肪系数为71.55,总的平均亲水性为-0.181。
ProtScale工具预测可知,FOXD3亲水性最强的位点是第236位的精氨酸,分值为-3.411;疏水性最强的位点是第151位的苏氨酸,分值为2.878。由图 2可知,FOXD3蛋白的亲水区域多于疏水区域,属于亲水蛋白质。
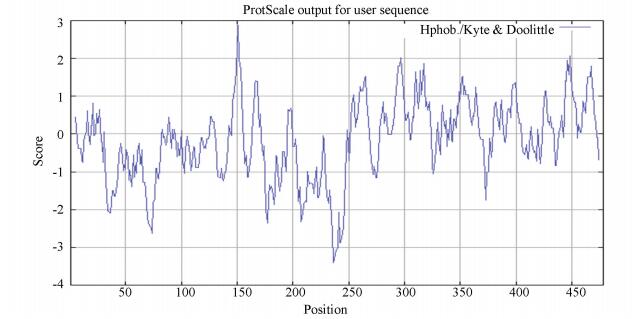
|
图 2 FOXD3蛋白的亲疏水性分析 Figure 2 Hydrophilic-hydrophobic analysis of FOXD3 protein |
TMHMM 2.0预测FOXD3无跨膜结构域(见图 3)。SignalP 4.0预测FOXD3蛋白无信号肽序列(见图 4)。原始剪切位点C值最高点位于第58位氨基酸,分值为0.111;被结合的剪切点Y值最高点位于第11位氨基酸,分值为0.112;信号肽S值最高点在第1位氨基酸,分值为0.150;1-10位氨基酸序列的平均信号肽分值为0.103,不足以形成经典的信号肽区域。
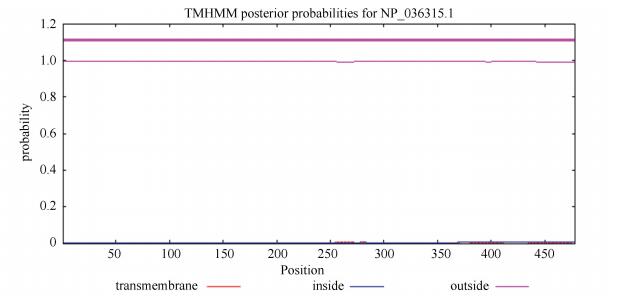
|
图 3 FOXD3蛋白跨膜结构分析 Figure 3 Trans-membrane domain analysis of FOXD3 |
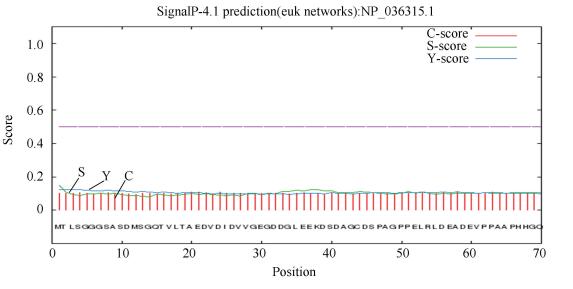
|
图 4 FOXD3蛋白信号肽分析 Figure 4 Signal peptides analysis of FOXD3 |
由NCBI的UniGene数据库中EST结果可知,FOXD3在以下正常组织中均有表达,拷贝数分别是:耳朵62,眼睛4,心脏33,肠4,肾脏4,睾丸2。在生殖细胞肿瘤组织中也有表达,拷贝数为26。生殖细胞肿瘤很罕见,主要是15至35岁男性易患的一种恶性肿瘤。FOXD3能够调控正常和恶性胚胎干细胞的增殖与分化。因此,George等对原始生殖细胞组织中FOXD3的表达情况进行分析,结果表明,21个样本中有5个病患肿瘤组织中FOXD3高表达[13]。
PSORTII预测,FOXD3定位于细胞核的可能性最大(78.3%),其次可能定位于细胞质(17.4%)和细胞骨架(4.3%)。由此推测,FOXD3主要在细胞核中发挥生理功能,也动态存在于其他亚细胞结构中。
2.4 FOXD3蛋白质结构分析在FOXD3蛋白质的二级结构中,无规卷曲(图中紫色)占51.26%,α-螺旋(图中蓝色)占26.36%,延伸链(图中红色)占12.34%,β-转角(图中绿色)占10.04%(见图 5)。NCBI的Conserved Domain数据库预测FOXD3蛋白属于FH超家族,含有一个FH (forkhead)结构域,该结构域的C末端含有两个柔性环或“翼”,因此FH结构域又被称为“翼螺旋”(winged helix)(见图 6)。此外,FH结构域能够以单体形式结合B型DNA,结合的DNA序列通常为5’-A [AT]T[AG]TTTGTTT-3’。FH结构域也存在于肝细胞核因子(Hepatocyte nuclear factor,HNF)蛋白中,进行基因调节时具有组织特异性[14]。使用SWISS-MODEL建模服务器预测FOXD3蛋白三维结构(见图 7),GMQE为0.12,QMEAN为-3.24,序列相似度为98.9%,该模型结构合理。
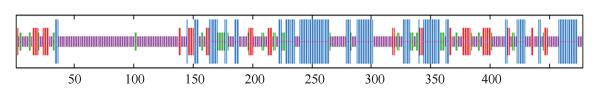
|
图 5 FOXD3的二级结构分析 Figure 5 FOXD3 secondary structure analysis |
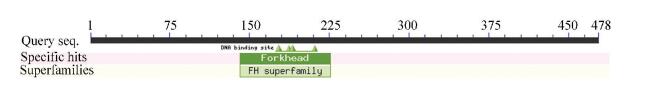
|
图 6 FOXD3功能结构域分析 Figure 6 Functional domain analysis of FOXD3 |
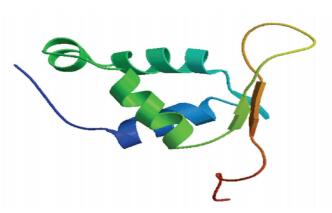
|
图 7 FOXD3三维结构预测 Figure 7 Three dimensional structure prediction of FOXD3 |
使用STRING数据库搜索与FOXD3相互作用的蛋白质信息,设置为高置信度0.7,不限制数量,构建FOXD3蛋白相互作用网络(见图 8)。与FOXD3相互作用的蛋白质主要包括SOX2(SRY (sex determining region Y)-box 2)、SOX10(SRY (sex determining region Y)-box 10)、POU5F1(POU class 5 homeobox 1)、NANOG(Nanog homeobox)和谷胱甘肽过氧化物酶7(Glutathione peroxidase 7, GPX7)、TFAP2A(Transcription factor AP-2 alpha (activating enhancer binding protein 2 alpha))、PAX6(Paired box 6)、PAX3(Paired box 3)、SNAI2(Snail homolog 2 (Drosophila))、MITF(Microphthalmia-associated transcription factor)。
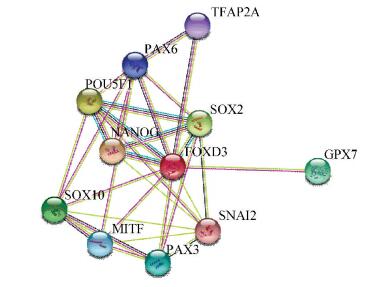
|
图 8 FOXD3蛋白相互作用网络 Figure 8 Protein-protein interaction network for FOXD3 |
对以上互作蛋白的基因进行GO分析和KEGG通路分析。GO分析结果表明,互作基因主要参与序列特异性DNA结合和染色质结合等分子功能(见表 1),涉及核酸模板转录的负调控、RNA聚合酶Ⅱ结合启动子转录过程的调控、干细胞的发生和分化等生物过程(见表 2)。KEGG通路分析结果表明,互作基因显著富集于癌症信号通路、MAPK信号通路和谷胱甘肽代谢等通路中(见表 3)。由此推测,FOXD3与互作蛋白中的转录因子,调控细胞干性的蛋白和调控肿瘤细胞特性的蛋白之间的关系值得进一步探究。
| 表 1 GO分子功能分类结果 Table 1 GO molecular function results |
| 表 2 GO生物学过程分类结果 Table 2 GO biological process results |
| 表 3 KEGG pathway路径列表 Table 3 KEGG pathway list |
本研究通过生物信息学方法,分析得到FOXD3是酸性不稳定的亲水蛋白,无信号肽和跨膜区域,定位于细胞核的可能性最大。FOXD3的主要二级结构是无规卷曲,属于FH超家族。根据GO分析、KEGG通路分析结果,结合亚细胞定位情况和结构分析结果,推测FOXD3在细胞内作为转录因子起作用。
根据FOXD3互作蛋白的基因GO分析和KEGG通路分析结果可知,互作基因主要参与序列特异性DNA结合等分子功能,涉及核酸模板转录调控等生物过程,显著富集于MAPK信号通路中。因此,FOXD3与互作蛋白中的转录因子SOX2、SOX10和MITF之间的关系引起关注。SOX2是一个重要的转录因子,能够在转录水平调控细胞干性和肿瘤细胞的抗凋亡特性[15-16]。SOX10主要参与维持神经嵴干细胞的全能性,在黑色素细胞和肠神经系统的发育和分化过程中扮演重要角色[17]。SOX10-MITF通路参与维持黑色素瘤细胞的增殖和致瘤特性,与黑色素瘤细胞周期调控、转移和其它生化过程有关[18]。从FOXD3与这三个转录因子的关系和参与的MAPK信号通路入手,探讨FOXD3在调控发育过程中的作用机制,有助于深化对FOXD3分子功能的理解。
根据GO分析和KEGG通路分析结果可知,互作基因主要涉及干细胞的发生和分化等生物过程,显著富集于癌症信号通路中。因此,FOXD3与调控细胞干性和肿瘤细胞特性的蛋白NANOG和SNAI2之间的关系值得继续探究。NANOG是一个转录因子,能够调控胚胎发育和细胞重编程过程。NANOG诱导体细胞重编程后,体细胞发生无限制的自我更新和分裂,并与其他干细胞因子相互作用继而引发癌变[19]。此外,NANOG在多种恶性肿瘤中高表达,其表达水平与肿瘤患者预后差和生存时间短呈正相关,预示其亦可作为肿瘤预后标志物[20-22]。另外,SNAI2在基质成纤维细胞中过量表达,能够使正常的成纤维细胞癌变,且与肿瘤基质激活和卵巢癌预后差有关,推测其作为致癌因子起作用[23]。FOXD3作为抑癌因子,在肿瘤细胞中与NANOG和SNAI2作用相反,目前也尚无关于FOXD3与NANOG和SNAI2之间关系的研究报道,深入探究它们之间的相互作用,有助于进一步认识FOXD3的作用机制。
此外,根据FOXD3互作蛋白的KEGG通路分析结果,互作基因显著富集于谷胱甘肽代谢通路中。因此,在FOXD3互作蛋白中,GPX7引起关注。正常细胞具有完整的抗氧化系统,保护其免受活性氧(Reactive oxygen species, ROS)诱导的DNA损伤和细胞损伤。GPXs(谷胱甘肽过氧化物酶家族)是细胞中主要的抗氧化酶家族,能够以还原型谷胱甘肽的形式催化过氧化物的还原[24]。GPX7是谷胱甘肽过氧化物酶家族的一个新成员,研究表明,GPX7能够保护食道细胞不受胆汁酸诱导产生的ROS的影响,使细胞免于氧化应激损伤和氧化性DNA损伤[25]。此外,Chen等发现下调GPX7易使胃癌细胞抗氧化系统紊乱,氧化性DNA损伤和突变积累,进而引发癌变[26]。由目前的报道可知,FOXD3与GPX7在肿瘤细胞中都起抑癌作用,但它们之间的关系尚未见报道,本文分析得出它们之间存在相互作用,有望成为阐明FOXD3作用机制的突破口。
4 结论1) FOXD3的理论分子量为47 630.30 Da,理论等电点为6.01,预测为酸性蛋白质。不稳定系数为53.49,预测为不稳定蛋白质。脂肪系数为71.55,总平均亲水性为-0.181,预测为亲水蛋白质。无信号肽和跨膜结构域。
2) FOXD3的二级结构主要为无规卷曲,属于FH超家族,含有一个FH结构域。
3) FOXD3表达的组织特异性不强,定位于细胞核的可能性较大。
4) 系统进化树显示,人FOXD3蛋白在进化过程中高度保守,该蛋白的同源性与物种间亲缘关系呈正相关。
5) 根据GO分析和KEGG通路分析结果,FOXD3与互作蛋白中的转录因子,调控细胞干性的蛋白和调控肿瘤细胞特性的蛋白之间的关系值得进一步探究。
| [1] |
DU W, PANG C, WANG D, et al. Decreased FOXD3 expression is associated with poor prognosis in patients with high-grade gliomas[J]. PLoS One, 2015, 10(5): e0127976. DOI:10.1371/journal.pone.0127976 (  0) 0) |
| [2] |
LABOSKY P A, LIU Y. Regulation of embryonic stem cell self-renewal and pluripotency by Foxd3[J]. Stem Cells, 2008, 319(2): 2475-2484. DOI:10.1634/stemcells.2008-0269 (  0) 0) |
| [3] |
YONG J S, INTRIAGOBALDEóN D P, LAM E W. FOXD3 controls pluripotency through modulating enhancer activity[J]. Stem Cell Investigation, 2016, 3(17): 17. DOI:10.21037/sci.2016.05.03 (  0) 0) |
| [4] |
LAM E W, BROSENS J J, GOMES A R, et al. Forkhead box proteins: tuning forks for transcriptional harmony[J]. Nature Reviews Cancer, 2013, 13(7): 482-495. DOI:10.1038/nrc3539 (  0) 0) |
| [5] |
CHU T L, ZHAO H M, LI Y, et al. FoxD3 deficiency promotes breast cancer progression by induction of epithelial-mesenchymal transition[J]. Biochemical and Biophysical Research Communications, 2014, 446(2): 580-584. DOI:10.1016/j.bbrc.2014.03.019 (  0) 0) |
| [6] |
LI D, MEI H, QI M, et al. FOXD3 is a novel tumor suppressor that affects growth, invasion, metastasis and angiogenesis of neuroblastoma[J]. Oncotarget, 2013, 4(11): 2021-2044. DOI:10.18632/oncotarget.1579 (  0) 0) |
| [7] |
CHENG A S, LI M S, KANG W, et al. Helicobacter pylori causes epigenetic dysregulation of FOXD3 to promote gastric carcinogenesis[J]. Gastroenterology, 2013, 144(1): 122-133. DOI:10.1053/j.gastro.2012.10.002 (  0) 0) |
| [8] |
VAN ROON E H, BOOT A, DIHAL A A, et al. BRAF mutation-specific promoter methylation of FOX genes in colorectal cancer[J]. Clinical Epigenetics, 2013, 5(1): 1-10. DOI:10.1186/1868-7083-5-2 (  0) 0) |
| [9] |
CHEN S S, RAVAL A, JOHNSON A J, et al. Epigenetic changes during disease progression in a murine model of human chronic lymphocytic leukemia[J]. Proceedings of the National Academy of Science, 2009, 106(32): 13433-13438. DOI:10.1073/pnas.0906455106 (  0) 0) |
| [10] |
YIN H, MENG T, ZHOU L, et al. FOXD3 regulates anaplastic thyroid cancer progression[J]. Oncotarget, 2017, 8(20): 33644-33651. (  0) 0) |
| [11] |
LI K, GUO Q, YANG J, et al. FOXD3 is a tumor suppressor of colon cancer by inhibiting EGFR-Ras-Raf-MEK-ERK signal pathway[J]. Oncotarget, 2017, 8(3): 5048-5056. (  0) 0) |
| [12] |
TAMURA K, DUDLEY J, NEI M, et al. MEGA4: molecular evolutionary genetics analysis(MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24(8): 1596-1599. DOI:10.1093/molbev/msm092 (  0) 0) |
| [13] |
GEORGE D W, FOSTER R S, HROMAS R A, et al. Update on late relapse of germ cell tumor: a clinical and molecular analysis[J]. Journal of Clinical Oncology, 2003, 21(1): 113-122. DOI:10.1200/JCO.2003.03.019 (  0) 0) |
| [14] |
SUTTON J, COSTA R, KLUG M, et al. Genesis, a winged helix transcriptional repressor with expression restricted to embryonic stem cells[J]. Journal of Biological Chemistry, 1996, 271(38): 23126-23133. DOI:10.1074/jbc.271.38.23126 (  0) 0) |
| [15] |
MASUI S, NAKATAKE Y, TOYOOKAY, et al. Pluripotency governed by Sox2 via regulation of Oct3/4 expression in mouse embryonic stem cells[J]. Nature Cell Biology, 2007, 9(6): 625-635. DOI:10.1038/ncb1589 (  0) 0) |
| [16] |
JIA X, LI X, XU Y, et al. SOX2 promotes tumorigenesis and increases the anti-apoptotic property of human prostate cancer cell[J]. Journal of Molecular Cell Biology, 2011, 3(4): 230-238. DOI:10.1093/jmcb/mjr002 (  0) 0) |
| [17] |
STOLT C C, WEGNER M. SoxE function in vertebrate nervous system development[J]. The International Journal of Biochemistry & Cell Biology, 2010, 42(3): 437-440. DOI:10.1016/j.biocel.2009.07.014 (  0) 0) |
| [18] |
TUDREJ K B, CZEPIELEWSKA E, KOZLOWSKA-WOJCIECHOWSKA M. SOX10-MITF pathway activity in melanoma cells[J]. Archives of Medical Science, 2017, 13(6): 1493-1503. DOI:10.5114/aoms.2016.60655 (  0) 0) |
| [19] |
JETER C R, YANG T, WANG J, et al. Concise review: NANOG in cancer stem cells and tumor development: an update and outstanding questions[J]. Stem Cells, 2015, 33(8): 2381-2390. DOI:10.1002/stem.2007 (  0) 0) |
| [20] |
LI X Q, YANG XL, ZHANG G, et al. Nuclear beta-catenin accumulation is associated with increased expression of Nanog protein and predicts poor prognosis of non-small cell lung cancer[J]. Journal of Translational Medicine, 2013, 11(1): 114. DOI:10.1186/1479-5876-11-114 (  0) 0) |
| [21] |
PARK E, PARK S Y, SUN P L, et al. Prognostic significance of stem cell-related marker expression and its correlation with histologic subtypes in lung adenocarcinoma[J]. Oncotarget, 2016, 7(27): 42502-42512. (  0) 0) |
| [22] |
DU Y, MA C, WANG Z, et al. Nanog, a novel prognostic marker for lung cancer[J]. Surgical Oncology, 2013, 22(4): 224-229. DOI:10.1016/j.suronc.2013.08.001 (  0) 0) |
| [23] |
YANG Z, YANG X, XU S, et al. Reprogramming of stromal fibroblasts by SNAI2 contributes to tumor desmoplasia and ovarian cancer progression[J]. Molecular Cancer, 2017, 16(1): 163. DOI:10.1186/s12943-017-0732-6 (  0) 0) |
| [24] |
BRIGELIUS-FLOHE R, KIPP A. Glutathione peroxidases in different stages of carcinogenesis[J]. Biochimica et Biophysica Acta (BBA) -General Subjects, 2009, 1790(11): 1555-1568. DOI:10.1016/j.bbagen.2009.03.006 (  0) 0) |
| [25] |
PENG D, BELKHIRI A, HU T, et al. Glutathione peroxidase 7 protects against oxidative DNA damage in oesophageal cells[J]. Gut, 2012, 61(9): 1250-1260. DOI:10.1136/gutjnl-2011-301078 (  0) 0) |
| [26] |
CHEN Z, HU T, ZHU S, et al. Glutathione peroxidase 7 suppresses cancer cell growth and is hypermethylated in gastric cancer[J]. Oncotarget, 2017, 8(33): 54345-54356. (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


