蛋白激酶(Protein Kinase,PK)是生物体内重要的调控蛋白,对很多重要的生理功能(如生长、代谢、衰老等)起关键调控作用。Sch9是芽殖酵母中重要的蛋白激酶,现已被证实Sch9参与调控多种信号途径和细胞过程,如细胞凋亡[1]、细胞自噬[2]、染色体重组与基因组稳定性[3]及寿命调控[4-5]等,近年来Sch9在寿命调控中的作用正被广泛研究。蛋白激酶S6K1是哺乳动物雷帕霉素靶标蛋白(mTOR)和磷脂酰肌醇-3-激酶(PI3K)的底物,且与很多人类疾病相关,包括肥胖症、糖尿病和癌症[6]。研究显示,S6K1也参与寿命调控,且发现Sch9与S6K1为同源蛋白[7]。
在芽殖酵母中Pkh1/2调节下游的AGC蛋白激酶家族(包括Ypk1、Pkc1、Sch9)[8]。Pkh1/2可以磷酸化Sch9中活性环位点的苏氨酸(PDK1位点)来激活Sch9[9]。另外研究表明Sch9蛋白激酶C末端737位苏氨酸为另一个保守的磷酸化位点(PDK2位点[10-11]。最近系列研究表明Sch9与酵母的寿命调控密切相关[12-13],主要机制是Sch9是寿命调控重要因子TORC1(Target of Rapamycin Complex 1)的下游靶标,TORC1可通过磷酸化Sch9 C末端6个残基而调控包括寿命调控在内的多种细胞功能[5, 7]。同时发现Sch9参与细胞热胁迫应答[14]。在雷帕霉素(Rapamycin,一种TORC1的特异抑制剂)的作用下Sch9可发生明显去磷酸化,调节核蛋白的产生[7]。
mTOR是哺乳动物中重要的寿命调控蛋白,属丝氨酸/苏氨酸蛋白激酶。研究表明在mTOR复合物1(mTORC1)中mTOR与调节蛋白“Raptor”结合,而在mTOR复合物2(mTORC2)中,mTOR与雷帕霉素不敏感组分“Rictor”结合,雷帕霉素主要作用于mTORC1而非mTORC2 [15]。mTORC1激活磷脂酰肌醇3-激酶(PI3K)/蛋白激酶B(PKB / Akt),从而磷酸化下游靶标核糖体蛋白S6激酶1(S6K1,Sch9的同源蛋白)和真核起始因子4E结合蛋白1(4E-BP1)而促进蛋白质合成[16]。研究显示mTORC1特异磷酸化S6K1 Thr389,从而激活S6K1,进而磷酸化核糖体蛋白S6而促进蛋白合成[17-18]。
鉴于Sch9及S6K1蛋白在生物体内的重要的调控功能,且目前尚缺乏系统的对Sch9及S6K1进行性质、结构及功能的分析研究,本文利用生物信息学手段较全面分析了Sch9与S6K1蛋白的理化性质、结构特点及其相互作用蛋白,为进一步深入研究Sch9与S6K1蛋白分子功能与作用机制提供了思路。
1 材料与方法 1.1 材料Sch9及S6K1蛋白的序列信息来自于NCBI数据库,本文选择酿酒酵母(Saccharomyces cerevisiae)中Sch9(检索号:CAA40853)与人的S6K1(检索号:P23443.2)进行研究。
1.2 方法 1.2.1 Sch9与S6K1理化性质分析使用ExPASy数据分析系统中的ProtParam(http://web.expasy.org/protparam/)工具,对Sch9的分子量、分子式、酸碱性和稳定性等理化性质进行分析。使用Protscale工具(http://web.expasy.org/protscale/)对Sch9与S6K1的亲疏水性进行分析。使用SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)分析Sch9与S6K1有无切割位点和信号肽,TMHMM 2.0工具(http://www.cbs.dtu.dk/services/TMHM-M/)分析Sch9与S6K1的跨膜区域。
1.2.2 Sch9与S6K1亚细胞定位预测使用PSORTII(https://psort.hgc.jp/)进行Sch9与S6K1的亚细胞定位预测。
1.2.3 Sch9与S6K1二级结构和高级结构分析使用SOPMA工具(https://npsa-prabi.ibcp.fr-/cgi-bin/secpred_sopma.pl)预测Sch9与S6K1的二级结构及各成分所占比例。使用NCBI的Conserved Domain数据库分析Sch9与S6K1的结构域。
1.2.4 相互作用蛋白分析使用STRING数据库(http://string-db.org/),设置高置信度为0.7,构建Sch9与S6K1相互作用的蛋白网络。
1.2.5 Sch9与S6K1系统进化树构建通过BLASTp搜索得到Sch9在不同种属酵母中的同源蛋白、S6K1在不同物种中的同源蛋白质序列。使用Clustal2.1软件进行Sch9与S6K1同源蛋白间的多重序列比对。使用MEGA6软件,Neighbor-joining方法,设置Boot-strap分析重复数为1 000,构建分子进化树。
2 结果与分析 2.1 基因的编码产物分析S6K1蛋白有a、b、c、d、e五种亚型,其对应的转录产物有:NM_003161.3、NM_001272042.1、NM_001272043.1、NM_001272044.1、NM_001272060.1,Sch9与S6K1基因具体基因编码产物分析如表 1。
| 表 1 Sch9与S6K1基因编码产物分析 Table 1 Gene encoding product analysis of Sch9 and S6K1 |
通过ExPA数据库中的ProtParam在线工具预测,可得到Sch9与S6K1蛋白相应理化性质,如表 2。
| 表 2 Sch9与S6K1蛋白理化性质 Table 2 Chemical properties of Sch9 and S6K1 protein |
由表 2可以看出,Sch9蛋白与S6K1蛋白均为酸性蛋白质,Sch9蛋白与S6K1蛋白不同主要表现在:Sch9蛋白为稳定蛋白质,S6K1为不稳定蛋白质,二者含量最高的氨基酸不相同。
利用ExPASy数据库中的ProtScale在线工具预测,确定Sch9与S6K1亲疏水性(见表 3及图 1)。由图 1可知,Sch9与S6K1亲水区域多于疏水区域,均属于亲水蛋白质蛋白。
| 表 3 Sch9与S6K1蛋白亲疏水性分析 Table 3 Hydrophobicity analysis of Sch9 and S6K1 protein |
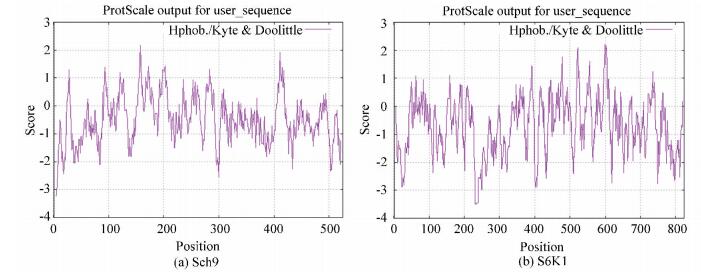
|
图 1 Sch9与S6K1蛋白的亲疏水性分析 Figure 1 Hydrophilic-hydrophobic analysis of Sch9 and S6K1 protein |
利用PSORTII分析Sch9与S6K1蛋白的亚细胞定位,结果见表 4,可看出Sch9与S6K1蛋白均最有可能定位在细胞核。
| 表 4 Sch9与S6K1蛋白的亚细胞定位分析 Table 4 Subcellular localization of Sch9 and S6K1 protein |
SignalP 4.0预测Sch9蛋白与S6K1蛋白均无信号肽序列(见图 2)。不足以形成经典的信号肽区域。信号肽预测结果见表 5。
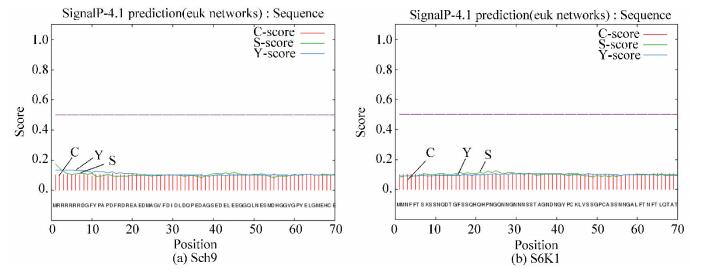
|
图 2 Sch9与S6K1蛋白信号肽分析 Figure 2 Signal peptides analysis of Sch9 and S6K1 protein |
| 表 5 Sch9与S6K1蛋白信号肽预测分析 Table 5 Signal peptide analysis of Sch9 and S6K1 protein |
经TMHMM 2.0预测Sch9与S6K1蛋白均无跨膜结构域(见图 3)。二者位于膜外(粉色细线)的概率几乎为100 %,位于膜内(蓝色细线)和跨膜区域(红色细线)的概率几乎为0。粉色粗线代表多肽链中跨膜区域所在位置,因二者没有跨膜区域,所以在粗线上不显示相应标记。
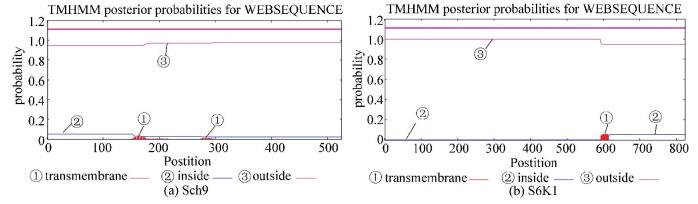
|
图 3 Sch9与S6K1蛋白跨膜结构分析 Figure 3 Trans-membrane domain analysis of Sch9 and S6K1 protein |
利用SOPMA分析Sch9与S6K1蛋白的二级结构(见图 4),与相应二级结构占比(见表 6)由此可见,Sch9与S6K1蛋白的二级结构非常相似,各种构象所占百分率很接近。
|
图 4 Sch9和S6K1的二级结构分析 Figure 4 Secondary structure analysis of Sch9 and S6K1 protein |
| 表 6 Sch9与S6K1蛋白的二级结构分析 Table 6 Secondary structure analysis of Sch9 and S6K1 protein |
通过NCBI的Conserved Domain数据库预测Sch9蛋白属于C2超家族和PKc_like超家族(见图 5)。PKc_like超家族中的STKc结构域能够催化磷酸基团从ATP转移到蛋白底物的丝氨酸/苏氨酸残基上,从而使蛋白底物磷酸化。海藻糖作为碳水化合物储备和应激代谢物,在酵母对环境变化的响应中起着重要的作用,Sck1在由葡萄糖和氮源触发的海藻糖酶活化中起作用。二糖海藻糖在海藻糖酶催化作用下裂解为葡萄糖。C2结构域首先在PKC中被鉴定。许多C2结构域是Ca2 +依赖性膜靶向模块,其结合多种物质,包括结合磷脂,肌醇多磷酸酯和细胞内蛋白质等。大多数C2结构域蛋白是含有单个C2结构域的信号转导酶,例如蛋白激酶C,或含有至少两个C2结构域的膜运输蛋白,如突触结合蛋白1。然而,具有钙结合区的C2结构域具有带负电荷的残基,主要是天冬氨酸,其充当钙离子的配体。Ypk1是细胞生长所必需的,在酵母的鞘脂介导的信号传导途径中起着下游激酶的作用,在高效的胞吞作用和维持细胞壁完整性方面也发挥作用。
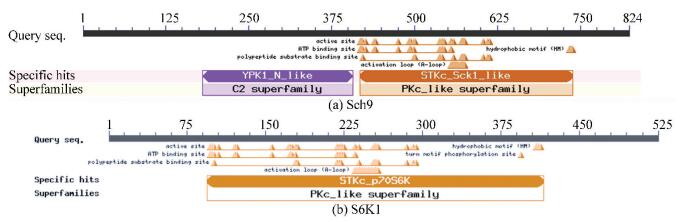
|
图 5 Sch9和S6K1的功能结构域分析 Figure 5 Functional domains analysis of Sch9 and S6K1 protein |
S6K1蛋白也属于PKc_like超家族(见图 5),其位点为丝氨酸/苏氨酸激酶的催化结构域,其属于70kDa核糖体蛋白S6激酶。STKs催化γ-磷酰基从ATP转移到蛋白质底物上的丝氨酸/苏氨酸残基。它作为STK mTOR(哺乳动物雷帕霉素靶标)的下游效应物,在蛋白质合成期间在翻译机制的调节中起作用。其靶标有S6,翻译起始因子eIF3和胰岛素受体底物IRS-1等。
2.6 Sch9与S6K1蛋白质相互作用分析使用STRING数据库预测Sch9及S6K1的相互作用的蛋白质,设置为高置信度0.7,构建Sch9及S6K1蛋白相互作用网络(见图 6)。与Sch9相互作用的蛋白质主要包括Cyr1、TOR1(Target of Repamycin complex 1)、TOR2(Target of Repamycin complex 2)、Pkh1/2等,该网络涉及到的生物过程有:MAPK级联反应(GO:0000165MAPK cascade)、细胞内信号转导(GO:0035556intracellular signal transduction)、参与细胞壁组织或生物合成的MAPK级联反应(GO:0000196MAPK cascade involved in cell wall organization or biogenesis),涉及到的分子功能有:ATP结合(GO:0005524ATP binding)、丝氨酸/苏氨酸蛋白激酶活性(GO:0004674protein serine/threonine kinase activity),涉及到的细胞组成有:TORC1复合体(GO:0031931TORC1 complex)。

|
图 6 Sch9及S6K1蛋白相互作用网络 Figure 6 Protein-protein interaction network for Sch9 and S6K1 protein |
与S6K1相互作用的蛋白质主要包括PIK3CA、RHEB、Rps6、RPTOR、mTOR等。该网络涉及到的生物过程有:胰岛素受体信号传导途径(GO:0008286insulin receptor signaling pathway)、细胞对胰岛素刺激反应(GO:0032869cellular response to insulin stimulus)、跨膜受体蛋白酪氨酸激酶信号通路(GO:0007169transmembrane receptor protein tyrosine kinase signaling pathway),涉及到的分子功能有:RNA聚合酶Ⅲ 1型启动子DNA结合(GO:0001030RNA polymerase Ⅲ type 1 promoter DNA binding)、RNA聚合酶Ⅲ 2型启动子DNA结合(GO:0001031RNA polymerase Ⅲ type 2 promoter DNA binding)、RNA聚合酶Ⅲ 3型启动子DNA结合(GO:0001031RNA polymerase Ⅲ type 3 promoter DNA binding),涉及到的细胞组成有:TOR复合体(GO:0038201TOR complex)、细胞质(GO:0005829cytosol),涉及到的信号通路有:mTOR信号通路(04150mTOR signaling pathway)、胰岛素信号通路(04910Insulin signaling pathway)、PI3K-Akt信号通路(04151PI3K-Akt signaling pathway)、AMPK信号通路(04152AMPK signaling pathway)。
2.7 Sch9与S6K1蛋白多序列比对和进化关系分析对不同物种Sch9蛋白进行多重序列比对,并构建分子进化树(见图 7)。酿酒酵母(Saccharomyces cerevisiae)Sch9蛋白的氨基酸序列与Saccharomyces arboricola、Naumovozyma castellii、Vanderwaltozyma polyspora、Naumovozyma dairenensis、Kazachstania naganishii、Zygosaccharomyces rouxii、Zygosaccharomyces bailii、Zygosaccharomyces parabailii、Candida的相似性分别为91%、67%、70%、68%、66%、67%,68%、68%、68%。与物种进化程度相一致,表明Sch9蛋白在进化过程中具有保守的分子功能,可以在科属级水平上进行归类。

|
图 7 Sch9蛋白的分子进化树 Figure 7 Phylogenetic tree of Sch9 protein |
对不同物种S6K1蛋白进行多重序列比对,并构建分子进化树(见图 8)。人S6K1蛋白的氨基酸序列与家犬、黑猩猩、河狸、牛、抹香鲸、绵羊、大鼠、家猪、鸡和小鼠的相似性分别为99%、99%、99%、99%、97%、99%、99%、95%、97%、94%,与物种进化程度相一致,表明S6K1蛋白在进化过程中具有非常保守的分子功能。

|
图 8 S6K1蛋白的分子进化树 Figure 8 Phylogenetic tree of S6K1 protein |
本研究通过生物信息学方法,分析得到Sch9是酸性稳定的亲水蛋白,无信号肽和跨膜区域,定位于细胞核的可能性最大。Sch9的主要二级结构是无规卷曲,属于C2超家族和PKc_like超家族,具有利于蛋白间相互作用的结构信息。S6K1是酸性不稳定的亲水蛋白,无信号肽和跨膜区域,定位于细胞核的可能性最大。S6K1的主要二级结构是无规卷曲,属于PKc_like超家族,具有利于蛋白间相互作用的结构信息。可见Sch9与S6K1在理化性质与结构功能有诸多相同,且两个蛋白在进化上相当保守。
通过STRING分析,Sch9除与已被功能性实验鉴定的Tor1、Tor2、Pkh1/2相互作用外,还与Cyr1蛋白有相互作用关系。Cyr1是腺苷酸环化酶,是调控cAMP产生和cAMP依赖性蛋白激酶信号传导的关键酶,活性受Ras蛋白和Gpr1的控制。部分研究指出Sch9可调节磷酸化酶[19],推测Sch9可通过Cyr1实现对Ras信号通路的反馈抑制。
在哺乳动物细胞中,mTOR1在上游受到PI3K/Akt信号通路调节,并作用于下游靶点S6K1与4EBP1,而4EBP1与eIF4B可相互作用并且影响翻译过程,营养因子、能量供给等因素会显著影响到此信号通路,然而具体机制尚待深入研究。
由于Sch9与S6K1理化性质、结构功能相似,Sch9蛋白已经被证实其PDK1位点磷酸化调控与热量限制延长芽殖酵母时序寿命密切相关[13],Selman等研究发现,敲除S6K1基因延长小鼠寿命,其基因表达情况与热量限制的小鼠及其相似,证实S6K1对衰老的调控与热量限制有关[20-21],和Sch9蛋白在芽殖酵母中相似,进一步研究可从激活其酶活的C端自主调控区的多个丝氨酸/苏氨酸磷酸化位点入手,进一步分析热量限制如何影响S6K1的表达和功能。
通过对Sch9和S6K1理化性质、结构功能及相互作用蛋白网络的全面比较分析,为进一步深入研究Sch9与S6K1的功能及调控提供理论参考。
| [1] |
BURTNER C R, MURAKAMI C J, KENNEDY B K, et al. A molecular mechanism of chronological aging in yeast[J]. Cell Cycle, 2009, 8(8): 1256-1270. DOI:10.4161/cc.8.8.8287 (  0) 0) |
| [2] |
YoRIMITSU T, ZAMAN S, BROACH J R, et al. Protein kinase A and Sch9 cooperatively regulate induction of autophagy in Saccharomyces cerevisiae[J]. Molecular Biology of the Cell, 2007, 18(10): 4180. DOI:10.1091/mbc.E07-05-0485 (  0) 0) |
| [3] |
MADIA F, WEI M, YUAN V, et al. Oncogene homologue Sch9 promotes age-dependent mutations by a superoxide and Rev1/Polzeta-dependent mechanism[J]. Journal of Cell Biology, 2009, 186(4): 509-523. DOI:10.1083/jcb.200906011 (  0) 0) |
| [4] |
FABRIZIO P, POZZA F, PLETCHER S D, et al. Regulation of Longevity and Stress Resistance by Sch9 in Yeast[J]. Science, 2001, 292(5515): 288. DOI:10.1126/science.1059497 (  0) 0) |
| [5] |
KAEBERLEIN M, KENNEDY B K. Regulation of yeast replicative life span by TOR and Sch9 in response to nutrients[J]. Science, 2005, 310(5751): 1193-1196. DOI:10.1126/science.1115535 (  0) 0) |
| [6] |
张会, 梁俊玉, 张继. 激酶S6K1研究现状及展望[J]. 生物医学工程学杂志, 2014(04): 935-938. ZHANG Hui, LIANG Junyu, ZHANG Ji. Research on progress and prospect of kinase S6K1[J]. Journal of Biomedical Engineering, 2014(04): 935-938. DOI:10.7507/1001-5515.20140176 (  0) 0) |
| [7] |
URBAN J, SOULARD A, HUBER A, et al. Sch9 is a major target of TORC1 in Saccharomyces cerevisiae[J]. Current Biology, 2007, 26(5): 663-674. DOI:10.1016/j.molcel.2007.04.020 (  0) 0) |
| [8] |
LIU K, ZHANG X, SUMANASEKERA C, et al. Signalling functions for sphingolipid long-chain bases in Saccharomyces cerevisiae[J]. Biochemical Society Transactions, 2005, 33(5): 1170-1173. DOI:10.1042/BST20051170 (  0) 0) |
| [9] |
BIONDI R M. Phosphoinositide-dependent protein kinase 1, a sensor of protein conformation[J]. Trends in Biochemical Sciences, 2004, 29(3): 136-142. DOI:10.1016/j.exphem.2017.12.006 (  0) 0) |
| [10] |
KOBAYASHI T, COHEN P. Activation of serum- and glucocorticoid-regulated protein kinase by agonists that activate phosphatidylinositide 3-kinase is mediated by 3-phosphoinositide-dependent protein kinase-1 (PDK1) and PDK2[J]. Biochemical Journal, 1999, 339 (Pt 2)(3): 319. DOI:10.1042/bj3390319 (  0) 0) |
| [11] |
ROELANTS F M, TORRANCE P D, THORNER J. Differential roles of PDK1- and PDK2-phosphorylation sites in the yeast AGC kinases Ypk1, Pkc1 and Sch9[J]. Microbiology, 2004, 150(10): 3289-3304. DOI:10.1099/mic.0.27286-0 (  0) 0) |
| [12] |
TROTT A, SHANER L, MORANO K A. The molecular chaperone Sse1 and the growth control protein kinase Sch9 collaborate to regulate protein kinase A activity in Saccharomyces cerevisiae[J]. Genetics, 2005, 170(3): 1009-1021. DOI:10.1534/genetics.105.043109 (  0) 0) |
| [13] |
刘军, 闾磊, 郄蓓蓓, 等. 环境变化和时序衰老过程中酵母蛋白激酶Sch9磷酸化的调控[J]. 生物化学与生物物理进展, 2014(02): 192-201. LIU Jun, LÜ Lei, QIE Beibei, et al. Regulation of the phosphorylation of yeast protein kinase Sch9 under environmental changes and during chronological aging[J]. Progress in Biochemistry and Biophysics, 2014(02): 192-201. DOI:10.3724/SP.J.1206.2013.00025 (  0) 0) |
| [14] |
闾磊, 郄蓓蓓, 黄鹏, 等. 酵母蛋白激酶Sch9激活环磷酸化状态参与热胁迫应答调控[J]. 应用与环境生物学报, 2012(03): 369-372. LÜ Lei, QIE Beibei, HUANG Peng, et al. Phosphorylation of yeast protein kinase Sch9 regulates heat stress response[J]. Chinese Journal of Applied and Environmental Biolohy, 2012(03): 369-372. DOI:10.3969/j.issn.0490-6756.2009.04.049 (  0) 0) |
| [15] |
ZONCU R, SABATINI D M, EFEYAN A. mTOR: from growth signal integration to cancer, diabetes and ageing[J]. Nature Reviews Molecular Cell Biology, 2011, 12(1): 21. DOI:10.1038/nrm3025 (  0) 0) |
| [16] |
MA M X J, BLENIS J. Molecular mechanisms of mTOR-mediated translational control[J]. Nature reviews. Molecular cell biology, 2009, 10(5): 307. DOI:10.1038/nrm2672 (  0) 0) |
| [17] |
BURNETT P E, BARROW R K, COHEN N A, et al. RAFT1 phosphorylation of the translational regulators p70 S6 kinase and 4E-BP1[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(4): 1432. DOI:10.1073/pnas.95.4.1432 (  0) 0) |
| [18] |
ISOTANI S, HARA K, TOKUNAGA C, et al. Immunopurified mammalian target of rapamycin phosphorylates and activates p70 S6 kinase alpha in vitro[J]. The Journal of Biological Chemistry, 1999, 274(48): 34493-34498. DOI:10.1074/jbc.274.48.34493 (  0) 0) |
| [19] |
ZHANG A, SHEN Y, GAO W, et al. Role of Sch9 in regulating Ras-cAMP signal pathway in Saccharomyces cerevisiae[J]. Febs Letters, 2011, 585(19): 3026-3032. DOI:10.1016/j.febslet.2011.08.023 (  0) 0) |
| [20] |
MAGNUSON B, EKIM B, FINGAR D C. Regulation and function of ribosomal protein S6 kinase (S6K) within mTOR signalling networks[J]. Biochemical Journal, 2012, 441(1): 1. DOI:10.1042/BJ20110892 (  0) 0) |
| [21] |
SELMAN C, TULLET J M A, WIESER D, et al. Ribosomal protein S6 kinase 1 signaling regulates mammalian lifespan[J]. Science, 2009, 326(5949): 140-144. DOI:10.1126/science.1177221 (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


