测序技术的进步、物种测序工作的迅速展开以及基因组数据的快速增长为生物信息学的研究发展提供了良好的数据材料。基因组数据的挖掘工作具有十分重要的意义。目前,人们发现“重组”造成的基因置换远超过以前认识,基因置换与遗传重组不同,它是染色单体间DNA的单向交换。而且,研究发现基因置换经常涉及染色体的一小部分,通常仅由十位或千位碱基对组成。关于基因组非正常重组的研究只在部分基因组中做了一些工作,在绝大多数中尚未展开。现今,国内外基本上都已认识到了基因置换在同源染色体间发生的可能更为频繁,致使激发了国内外的研究兴趣,诸如分析研究哪些因素与基因置换的发生有关,基因置换对基因组结构、基因组进化、物种形成及物种存留有什么样的影响都已成为国内外研究热点。本文以同源基因四联子为模板,对基因置换事件进行了推断,为研究基因置换对物种进化的影响奠定了基础。
1 基因置换的当前发展趋势 1.1 基因置换与遗传重组的区别遗传重组指交换分别来自两个亲本的基因连锁群,形成两个亲本所没有的性状组合,产生具有重组性状的后代(重组体)的现象。其不仅可以修复遗传过程中损伤的DNA序列,而且可以传递同源序列间的信息(同源重组)[1]。基因置换,又称基因替代作用,它指的是遗传信息单向的从一个序列向其同源序列传递的过程[2]。在原核生物及真核生物中已广泛的发现了基因置换的现象。
遗传重组和基因置换都可以进行DNA的修复,但在基础层面上两者是不同的。研究发现,与遗传重组相比,基因置换影响的DNA长度更小,仅由十位或千位碱基对组成,而遗传重组影响的DNA的长度达百万碱基。其次,基因置换因为是一个等位基因去覆盖另一个等位基因导致改变等位基因频率,进而影响基因组进化,而遗传重组不改变等位基因频率。再者,遗传重组是染色体间DNA的互惠交换,而基因置换是遗传信息单向传递的过程。研究还发现,在遗传重组不能够发生的基因组区域内基因置换也可以发生,如基因置换可以在着丝点DNA中发生,而遗传重组在这些遗传区域是禁止的。
1.2 基因置换的影响相对于遗传重组,基因置换也对遗传多样性、基因组进化、物种形成和物种存留产生独特的影响。越来越多的证据表明,基因置换能够通过均质化相关的等位基因直接的影响遗传多样性,除此之外,基因置换也可以通过破坏连锁来影响遗传多样性。研究表明基因置换是使连锁不平衡退化的最有影响力的因素[3]。基因置换因其涉及的染色体片段极小而在倒位杂合子中基因通量的机制中占主导地位,对于基因置换在倒位杂合子中发生的频率虽还没有完全了解,但有充足的证据表明基因置换促进了倒位杂合子中的基因通量,破坏了连锁不平衡,降低了种间适应性。使用群体遗传数据和基于连锁不平衡的方法来估计倒位杂合子体内基因置换的频率的研究表明,基因置换在除倒位断点附近的倒置区域之间有规律的发生[4]。这些数据可以用来理解基因置换在倒位杂合子中会产生多少基因通量,从而了解倒位杂合子对物种持久性的重要性。
在多倍体方面,基因置换对物种形成提供了独特的贡献。来自被子植物的证据表明,基因置换保持了旁系同源基因同源序列的相似性,但加速了与其他物种的直系同源基因的差异[5]。基因置换也会影响协同进化,理论研究表明,基因置换促进的协同进化有效的提高了多基因家族的选择效率,形成了基因家族的分子进化[6];除了理论工作外,实证研究也注意到通过基因置换的协同进化与选择之间的相互作用,通过基因置换的协同进化复杂了选择性清扫的特征[7],而且通过基因置换的协同进化使基因新功能化的过程复杂化[8]。
基因置换的另一个结果是,基因置换可能会使GC含量发生变化。在发生GC偏好的基因置换的生物体中,基因置换所涉及的基因位点的GC的积累会随着时间的推移导致更高的GC含量。有证据表明,GC偏好的基因置换在哺乳动物基因组GC含量的演变中发挥了重要作用[9]。
1.3 有关基因置换的研究随着对基因置换的认识,越来越多的学者开始致力于基因置换的研究。王金朋等[10]研究了谷子、玉米重复基因间可能的基因置换,研究发现谷子中18.9%、玉米中11.9%的重复基因发生了基因置换,研究还发现基因在染色体上的物理位置与发生基因置换的概率显著相关,统计分析表明,重复基因对越靠近染色体末端越易发生基因置换;Laurits Skov等[11]为研究人类Y染色体的两个回文臂中的变异模式分析了2.7 Mb的大反向重复区域,研究最终确认了416个基因置换事件;Ezawa K等[12]研究了大鼠、小鼠中由全基因组加倍产生的重复基因,研究发现在分化之前有18%的重复基因对发生了基因置换;Xu S[13]等应用GENECONV的方法研究了水稻626个多基因组家族,研究发现其中的377个发生了基因置换。以上研究结果为基因置换在同源染色体之间发生的很频繁提供了有力的证据。
Elena Goicoechea de Jorge等[14]研究了CFH基因和CFHR1基因之间的基因置换事件,研究发现由基因置换事件产生的H因子与非典型溶血性尿毒综合征有很大的关联;Wentao Shi等[15]研究了人类Y染色体上的基因置换事件,研究发现与双交叉相比,基因置换更有可能是引起拷贝数变异的原因,而拷贝数变异可以导致呈罕见疾病、遗传的单基因病以及其他复杂疾病;Brenda Maria A.Issangya等[16]通过下一代测序分析检测到了一种由基因置换产生的新HLA等位基因。以上研究表明基因置换可导致遗传物质发生改变,继而可导致某些疾病的发生。
基因置换是形成基因组多样化和物种进化的重要部分,但基因置换的程度如何调节尚不清楚,Yann Duroc等为此做出了贡献。Yann Duroc等[17]研究了MutLβ异质二聚物和Mer3解旋酶对减数分裂重组所产生的基因置换的影响,研究发现MutLβ异质二聚物和Mer3解旋酶协同作用调节全范围的减数分裂基因置换,提供了一个在有机体内限制基因置换的机制;Paulina M Budzynska等[18]为了研究Bach2在AID介导的免疫球蛋白基因多样化过程中的作用,建立了BACH 2缺陷的DT40B细胞系,研究发现Bach2可通过增加AID的表达来促进基因置换,提供了一个促进基因置换的机制。
2 基因置换的推断几个统计实验足以鉴别基因置换事件。Stephens[19]提出了从一组DNA序列识别基因置换事件的方法,虽然适用于非常小的序列,但现在已经引入了更多用途的测试。Sawyer[20]提出的方法现在是检测基因置换软件包的基础,包括GENECONV。Betran等[21]人为了获得基因置换的频率和长度的估计,描述了一个算法纳入了一个统计模型。例如,利用果蝇种群数据,研究了基因置换。这种推断方法有一个优点,不需要对一个家系中的大量个体进行测序。
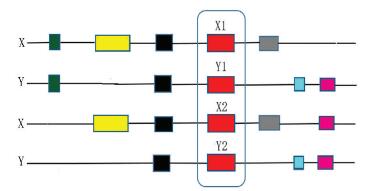
推断可能的基因置换事件的常用模型是同源基因四联子,它是由一个物种的两个旁系同源基因X1和X2,以及X1和X2在另一物种上所对应的直系同源基因Y1和Y2组成,如图 1所示。
|
图 1 同源基因四联子 Figure 1 Homologous genes quadruple |
图 1中,两个不同的物种用X和Y来表示,同源染色体片段上的基因用带颜色的方框来表示, 其中颜色一致的方框代表同源基因。X1和X2分别表示的是X物种两个旁系同源基因,Y1和Y2分别表示的是X1和X2在Y物种上所对应的直系同源基因,X1、X2、Y1、Y2共同构成同源基因四联子。
对亲缘关系上比较接近的物种的全基因组进行Blast比对分析,得到基因同源性;用共线性分析工具MCScanX、Colinearscan分析,推断亲缘关系上比较接近的基因组间的共线性同源基因,从而构建两者间共有的多倍化事件产生的同源基因四联子;应用多重序列比对工具Clustalw对找到的同源基因四联子进行多重序列比对,分析比对结果,删除氨基酸一致性小于40%、总序列长度被序列的空位占50%以上的同源基因四联子;基于最大似然法(ML)、最大简约法(MP)和邻接法(NJ)算法,运用系统发育分析软件MEGA构建同源基因四联子的系统发育树,通过分析研究同源基因四联子构建的系统发育树的拓扑结构的变化,推断全基因置换。
由于物种进化的发生早于直系同源基因对的产生,因此两个物种直系同源基因对的相似性理论上应高于所对应旁系同源基因对,若物种发生全基因置换,直系同源基因对的相似性理论上有可能低于所对应旁系同源基因对。对同源基因四联子构建的系统发育树的拓扑结构的变化进行分析,推断是否发生全基因置换,对发生基因置换的系统发育树进行置换检验(Bootstrap), 可以得到置换基因的置信度。各同源基因间的同义核苷酸的置换率(Ks)可度量序列的相似性,一般来说可以使用进化分析软件(PAML)中的Nei-Gojobori方法来计算同义核苷酸的置换率。若物种的一对旁系同源基因间的同义核苷酸置换率小于所对应的直系同源基因间同义核苷酸置换率,则可以说明在物种分化之后,这对旁系同源基因发生了全基因置换。
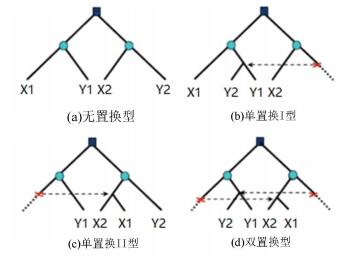
在图 2中,方框表示的是共同祖先基因组中的基因多倍化事件,圆形表示的是物种的分化。图 2(a)展示的是无置换型,即没有发生基因置换时基因四联子间的遗传进化关系;图 2(b)和图 2(c)分别展现的是发生单置换时基因四联子间的遗传进化关系;图(d)展示的是发生双置换时基因四联子间的遗传进化关系。例如,图 2(b)展示的是基因Y2被Y1置换的情况;图 2(c)展示的是基因X1被X2置换的情况;图 2(d)展示的是两种置换都发生的情况。
|
图 2 基因置换推断 Figure 2 Gene conversion to deduce |
然而,王金朋等[10]在谷子和玉米中研究重复基因间的基因置换的研究中发现,在谷子、玉米重复基因间发生基因置换的事件中,发生部分基因置换事件的概率远高于发生全基因置换事件的概率。部分基因置换事件不能通过基因序列的相似性来推断,常用动态规划与系统发育树相结合的方法来推断。
3 总结与展望现有的研究表明基因置换广泛的存在于物种基因组中,可直接影响染色体基因结构和功能,并证明了基因发生置换的概率与其在染色体上的物理位置有很大的联系,即越靠近染色体末端,越易发生基因置换。目前,我们已初步了解了影响基因置换的因素,基因置换对遗传多样性、物种进化和物种存留的影响,但仍然存在一些尚未解决的问题,如哪些因素导致染色体发生基因置换、基因置换对染色体基因结构和功能有哪些影响及基因置换对更深层次的生物进化起什么样的作用等一系列问题。
随着对基因置换的深入认识,对物种间基因置换事件进行推断的方法将更加的精确,为基因置换的进一步研究奠定了基础。
| [1] |
PUCHTA H, DUJON B, HOHN B, et al. Two different but related mechanisms are used in plants for the repair of genomic double-strand breaks by homologous recombination[J]. Proceedings of the National Academy of Science of the United States of Amercia, 1996, 93(10): 5055-5060. DOI:10.1073/pnas.93.10.5055 (  0) 0) |
| [2] |
DATTA A, HENDRIX M, LIPSITCH M, et al. Dual roles for DNA sequence identity and the mismatch repair system in the regulation of mitotic crossing-over in yeast[J]. Proceedings of the National Academy of Science of the United States of Amercia, 1997, 94(18): 9757-9762. DOI:10.1073/pnas.94.18.9757 (  0) 0) |
| [3] |
ANDOLFATTO P, NORDBORG M. The effect of gene conversion on intralocus associations[J]. Genetics, 1998, 148(3): 607-615. (  0) 0) |
| [4] |
ROZAS J, AGUADE M. Gene conversion is involved in the transfer of genetic information between naturally occurring inversions of Drosophila[J]. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(24): 11517-11521. DOI:10.1073/pnas.91.24.11517 (  0) 0) |
| [5] |
WANG X Y, PATERSON A H. Gene conversion in angiosperm genomes with an emphasis on genes duplicated by polyploidization[J]. Genes, 2011, 2(1): 1-20. DOI:10.3390/genes2010001 (  0) 0) |
| [6] |
MANO S, INNAN H. The evolutionary rate of duplicated genes under concerted evolution[J]. Genetics, 2008, 180(1): 493-505. DOI:10.1534/genetics.108.087676 (  0) 0) |
| [7] |
HANIKENNE M, KROYMANN J, TRAMPCZYNSKA A, et al. Hard selective sweep and ectopic gene conversion in a gene cluster affording environmental adaptation[J]. PLoS Genetics, 2013, 9(8): e1003707. DOI:10.1371/journal.pgen.1003707 (  0) 0) |
| [8] |
TESHIMA K M, INNAN H. Neofunctionalization of duplicated genes under the pressure of gene conversion[J]. Genetics, 2008, 178(3): 1385-1398. DOI:10.1534/genetics.107.082933 (  0) 0) |
| [9] |
GALTIER N, PIGANEAU G, MOUCHIROUD D, et al. GC-content evolution in mammalian genomes: the biased gene conversion hypothesis[J]. Genetics, 2001, 159(2): 907-911. (  0) 0) |
| [10] |
王金朋, 聂林曼, 王振怡, 等. 谷子、玉米重复基因间基因置换的系统发育分析[J]. 河南农业科学, 2014, 43(12): 34-39. WANG Jinpeng, NIE Linman, WANG Zhenyi, et al. Phylogenetic analysis of gene conversion between duplicated genes in Setria italica and Zea mays[J]. Henan Agricultural Science, 2014, 43(12): 34-39. DOI:10.15933/j.cnki.1004-3268.2014.12.008 (  0) 0) |
| [11] |
SKOV L, CONSORTIUM T D P G, SCHIERUP M H. Analysis of 62 hybrid assembled human Y chromosomes exposes rapid structural changes and high rates of gene conversion[J]. PLoS Genetics, 2017, 13(8): e1006834. DOI:10.1371/journal.pgen.1006834 (  0) 0) |
| [12] |
EZAWA K, OOTA S, SAITOU N. Genome-wide search of gene conversions in duplicated genes of mouse and rat[J]. Molecular Biology and Evolution, 2006, 23(5): 927-940. DOI:10.1093/molbev/msj093 (  0) 0) |
| [13] |
XU S Q, CLRK T, ZHENG H K, et al. Gene conversion in the rice genome[J]. BMC Genomics, 2008, 9(1): 93. DOI:10.1186/1471-2164-9-93 (  0) 0) |
| [14] |
ELENA G J, AGUSTIN T, SHEILA P G, et al. Factor H competitor generated by gene conversion events associates with atypical hemolytic uremic syndrome[J]. Journal of the American Society of Nephrology, 2017, 29(1): 240-249. DOI:10.1681/ASN.2017050518 (  0) 0) |
| [15] |
WENTAO S, MASSAIA S, LOUZADA R, et al. Copy number variation arising from gene conversion on the human Y chromosome[J]. Human Genetics, 2017, 137(01): 73-83. DOI:10.1007/s00439-017-1857-9 (  0) 0) |
| [16] |
BRENDA M A, ISSANGYA R L, UPCHURCH E A, et al. A case of a novel HLA allele generated by gene conversion detected by a next-generation sequencing assay[J]. Human Immunology, 2017, 78: 164. DOI:10.1016/j.humimm.2017.06.208 (  0) 0) |
| [17] |
YANN D, RAJEEV K, LEPAKSHI R, et al. Concerted action of the MutLβ heterodimer and Mer3 helicase regulates the global extent of meiotic gene conversion[J]. eLife, 2017, 6: e21900. DOI:10.7554/eLife.21900 (  0) 0) |
| [18] |
PAULINA M B, MINNA K, TEEMU K, et al. Bach2 regulates AID-mediated immunoglobulin gene conversion and somatic hypermutation in DT40 B cells[J]. European Journal of Immunology, 2017, 47(06): 993-1001. DOI:10.1002/eji.201646895 (  0) 0) |
| [19] |
STEPHENS J C. Statistical methods of DNA sequence analysis: detection of intragenic recombination or gene conversion[J]. Molecular Biology and Evolution, 1985, 2(6): 839-856. DOI:10.1093/oxfordjournals.molbev.a040371 (  0) 0) |
| [20] |
SAWYER S. Statistical tests for detection gene conversion[J]. Molecular Biology and Evolution, 1989, 6(5): 526-538. DOI:10.1093/oxfordjournals.molbev.a040567 (  0) 0) |
| [21] |
BETRAN E, ROZAS J, NARBADILLA A. The estimation of the number and the length distribution of gene conversion tracts from population DNA sequence data[J]. Genetics, 1997, 146(1): 89-99. (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


